La version française de l’article est disponible à la suite de la version anglaise
1. Introduction
Understanding the molecular mechanisms underlying seed development and metabolism that enable seeds to accumulate storage compounds, pause embryo growth, and ultimately germinate under favourable conditions, constitutes an important topic in plant biology and ecology. Seeds have contributed to the tremendous success of spermatophytes on the earth (today, they represent 95% of the plant species), allowing most of them to cope with unfavourable environmental conditions by interrupting their life cycle and resuming growth when placed under favourable conditions [1]. Moreover, seeds are of the utmost importance for the humankind. They provide more than half of the world’s intake of dietary proteins, oils and starch, minerals or vitamins and thus are the main source of human nutrition and animal feed [2]. Over the coming decades, world population will continue to increase and is expected to reach about 10 billion by 2050 [3]. This growth rate needs to be matched by an increase of global food production of at least 50% [4]. Furthermore, seeds accumulate storage carbon compounds such as oil or starch, which represent an alternative to fossil carbon chains as precursors for the chemical industry, as well as high added-value compounds with ever-expanding applications in health and medicine. Finally, seeds constitute the main vector for the improvement of agronomic practices and the management of genetic resources, two key factors for the development of sustainable agricultures and the preservation of biodiversity and environment. These different needs already position a global commercial seed market to about $60 billion in 2020, and this is expected to reach more than $100 billion by 2031 (https://www.alliedmarketresearch.com/seed-market).
Michel Caboche had long been a precursor and a catalyst for research in plant genetics and physiology, including seed biology [5]. Among the different labs he has contributed to develop, the Seed Biology Laboratory created in 1995, which is now part of the Institute Jean-Pierre Bourgin (IJPB, INRAE-AgroParisTech), has been focusing on the study of the molecular mechanisms and on the genes involved in the control of seed development and filling, as well as in the regulation of seed dormancy and germination [6, 7, 8, 9]. Genetic analysis of seed development was initiated with the isolation of a large number of mutants (Lepiniec et al. unpublished) from the Versailles collection of T-DNA insertion mutants [10, 11]. This genetic approach led to led to the discovery of a diversity of actors involved in the biosynthesis of lipid reserves such as the acetyl-CoA carboxylase ACC1 [12] and the diacylglycerol acyltransferase TAG1 [13], in the post-translational modification of proteins e.g., the glucosidase GCS1 [14], or in the regulation of flavonoid biosynthesis and seed coat differentiation (the “transparent testa” (TT) proteins [15, 16, 17, 18]. These genetic approaches, facilitated over time by the ever-increasing availability of T-DNA insertion lines and associated resources, have paved the way for a systematic study of seed-expressed transcription factors and their regulatory networks that is still underway in the lab [19, 20].
In 2010, Michel Caboche coordinated the preparation of a manuscript entitled “Arabidopsis seed secrets unravelled after a decade of genetic and omics-driven research” [1]. The aim of this review was to illustrate important progress achieved in the field over the past decade and covered different topics including seed development (embryo, endosperm, and integuments), seed reserve accumulation, seed dormancy, and germination. At that time, emerging “omics” tools, including the inventory of genes deduced from the sequencing of the Arabidopsis thaliana genome, were speeding up the discovery of biological functions associated with seeds. The access to “omics” resources enabled molecular genetic approaches and attracted a large part of the seed community to work with the model species Arabidopsis. Following the untimely passing away of M. Caboche last year [5], we have decided to update this review in his memory. We have extended the scope of the paper to encompass emerging research of interest such as the characterization of seed biodiversity, recent progress in the analysis of synthesis and modifications of proteins and specialized metabolites, and studies on the impact of the environment on seed quality. Recent developments in high throughput approaches have facilitated the implementation of omics studies in a large spectrum of species, including crops. The characterisation of seed diversity in wild and crop species now appears essential to understand what makes seeds original in terms of biochemical composition, tolerance to adverse environmental conditions, germination features and competition with other species. Today, efficient research can be carried out directly with cultivated species to advance knowledge and to adapt new traits of interest using targeted genetic approaches such as TILLING or CRISPR-based genomic editing systems.
2. Seed (gen)omics
Research in seed biology has greatly benefited from various Arabidopsis genomic tools and resources, including genome sequencing, reverse genetics using T-DNA insertion libraries, and bioinformatics [1]. Over the last decade, the development of high-throughput technologies has led to the establishment of useful resources for plant (epi)genomic, transcriptomic, or proteomic data. Bio-informatic tools have continuously been improved facilitating the access and use of these huge sets of “omics” data [21]. However, most of these “omics” approaches were initially implemented on whole seeds, that are comprised of several different tissues (seed coat, endosperm and embryo), with these tissues themselves encompass different cell types known to exhibit contrasting properties and fates. Accordingly, they are characterized by specific patterns of gene expression, protein composition and metabolite contents. Until very recently, the detailed characterization of seed tissue- or cell-types has therefore represented a critical bottleneck in seed biology, because of the small size and entanglement of these different tissues and the inevitable cross-contaminations associated with their dissection.
2.1. Tissue- and cell-specific omics
To overcome these limitations, dedicated tools have been developed including fluorescence-activated cell/nuclei sorting (FACS/FANS), translating ribosome affinity purification (TRAP), the isolation of specific cell tagged nuclei (INTACT) or micro-dissection [22, 23, 24, 25]. Laser Assisted Microdissection (LAM) allows precise recovery of specific tissues based on their morphology or prior staining. Coupling LAM to quantitative RT-PCR or DNA chips has been successfully achieved with different seed tissues [26, 27]. Nowadays, Next Generation Sequencing (NGS) technologies are highly sensitive and permit the quantification of low-abundance transcripts even in species for which no reference genome sequences are available. Combining ultra-low RNA sequencing with laser microdissection is therefore feasible and has facilitated precise and comprehensive analyses of the epidermal versus mesophyll cell transcriptomes in the cotyledons of the torpedo stage Arabidopsis embryos for example [24]. More recently, fluorescence-activated nuclei sorting coupled with single-nucleus mRNA sequencing allowed a gene expression atlas to be constructed for Arabidopsis early-stage embryos at single-cell resolution [28]. Nevertheless, these increasingly sensitive techniques are still limited by the need for protoplasting or genetic transformations with cell type-specific markers, and biases related to mRNA amplification [29].
2.2. Integrating multi-omics data
The development of high-throughput “multi-omics” strategies, together with the rapid evolution of data technologies, has improved our understanding of seed biology and the management of traits of agronomical and ecological importance related to seed composition, dormancy, longevity, and vigour. Multi-dimensional -omics data that are systematically investigated in seed lots by computational analysis using statistics and bioinformatics approaches enables the identification of key factors and molecular pathways, and have led to the characterization of large number of potential targets for breeding programs or seed treatments to improve their quality and stress resistance. Combining multi-omics data allowed identifying biological entities that share similar patterns across different physiological or environmental conditions. For instance, extensive analyses of transcriptomic data led to the identification of groups of genes that are co-expressed and involved in similar processes such as seed physiology or responses to adverse environmental conditions. A seed specific network, namely SeedNet (http://vseed.nottingham.ac.uk), was developed to provide gene modules associated with Arabidopsis seed dormancy and germination [30, 31, 32]. Nevertheless, some studies have pinpointed a default of correlation between mRNA abundance and protein synthesis, underlining that selective and temporal mRNA translation occurs during seed germination [33]. This observation was further supported by recent studies on the regulation of translation in Arabidopsis seeds by polysome profiling [34, 35, 36]. Due to both post-transcriptional (e.g. splicing or editing) and post-translational (e.g. proteolysis or post-translational modifications, PTMs) regulations, a single gene can give rise to a diversity of protein isoforms that may differ for their function, stability, localization and interactions (see hereafter). For instance, recent work based on protein–protein interactions, highlighted this protein functional diversification in the signalling system for abscisic acid (ABA) [37], as detailed hereafter.
Besides genomic, transcriptomic, and proteomic approaches, the improvement of targeted and untargeted metabolomic sensitivity and data analysis allowed a better characterization of seed physiological status and quality. Metabolomics, the large-scale study of primary and specialized metabolites (SMs), is considered as the omics technique that is closest to the phenotype [38]. Over the past few years, we have witnessed a rise in the number of untargeted and, to a lesser extent, targeted metabolomic studies focusing on seed specialized usual or unusual fatty acids and lipids, and free amino-acids diversity and regulation using metabolomics alone or in combination with other omics techniques [39, 40, 41, 42].
3. Seed development
Seed development can be divided into two successive phases named embryogenesis and maturation [1, 43, 44]. In angiosperms, the “double” fertilization of the embryo sac triggers the development of three genetically distinct tissues comprising seeds, the seed coat (or “testa”) of maternal origin arising from the two integuments of the ovule, the triploid endosperm and the diploid embryo [1]. Over the last few years, it has been demonstrated that fertilization removes the negative epigenetic control exerted by the PcG (Polycomb Group) proteins over seed development [45]. Seed coat differentiation is initiated by ovule fertilization. The endosperm develops with a syncytial phase followed by cellularization and differentiation events. The zygote divides into an apical and a basal cell that further divides and give rise to an advanced embryo and its suspensor, respectively. At the heart stage, the main structures of the Arabidopsis embryo (i.e. cotyledons, shoot meristem, hypocotyl, and embryonic root) are already established, cell divisions start to subside, and the maturation programme is triggered that lead to the accumulation of storage compounds, seed desiccation and the acquisition of dormancy. Auxin signalling plays a major role during embryogenesis, while ABA regulates multiple aspects of seed maturation, including storage reserve accumulation, and seed dormancy (see below).
3.1. Coordinated growth of the seed coat, the endosperm, and the embryo
Proper seed development requires the coordinated growth of the seed coat, the endosperm and the embryo and thus relies on communication between these different compartments [1, 46]. For example, abnormal seed coat growth affects endosperm development and reduces overall seed size. On the same line, the endosperm provides critical signals for embryo development. some cysteine-rich peptides (EMBRYO SURROUNDING FACTOR, ESF) found in the endosperm are involved in suspensor development [47]. Recently, it has been demonstrated that a peptide-mediated signalling pathway takes place between the embryo and the endosperm that controls the formation of embryo cuticle, a protective layer of surface lipids deposited above its epidermis [48]. The TWISTED SEED1 (TWS1) peptide produced by the embryo [49], is activated by an endosperm subtilase, leading to embryo cuticle reinforcement and protection [48]. Another example of these interactions is the mediation of the antagonist development of the nucellus (maternal tissue) and the endosperm by the MADS transcription factors TRANSPARENT TESTA16 (TT16) and its paralog GORDITA [50].
3.2. The LAFL network and the control of seed development and maturation
The LAFL transcriptional regulators namely LEC1 (LEAFY COTYLEDON1), LEC2 (LEAFY COTYLEDON2), ABI3 (ABSCISIC ACID INSENSITIVE3), and FUS3 (FUSCA3) in Arabidopsis, are required for correct seed formation and seems to be conserved in the angiosperms (Figure 1) [51]. LEC1 is a member of the NF-YB protein family (NF-YB9, HAP3 subunit of the CCAAT-box binding factors). ABI3, FUS3, and LEC2 belong to a plant-specific family of B3 domain transcription factors (TFs) and thus are also named “AFL-B3” regulators. LEC1 and LEC2 are the master regulators of the network, but the four LAFL have partially overlapping and redundant functions and can interact to form different regulatory complexes [52, 53]. These complexes control the expression of both structural and regulatory genes involved in different pathways, triggering and maintaining embryo development and maturation, but also repressing seed germination [51, 54]. Many seed-specific genes that participate in storage compound accumulation, including those encoding oleosins, globulins (e.g. 12S cruciferin) and albumins (e.g. 2S arabins) for instance, are direct targets of the LAFLs [52]. Moreover, several other TFs act downstream or in parallel to the LAFLs during maturation such as AGL15, ABI4, and different bZIPs [51]. Another example is WRINKLED1 (WRI1), a member of the AP2/EREB transcription factor family, that controls the fatty acid biosynthetic pathways downstream of the LAFL [55]. WRI1 activity is also largely regulated at the post-translational level [56]. Recently, it has been shown that WRI1 interacts with BLISTER to regulate chromatin dynamics at target promoters [57]. Finally, the ectopic expression of LEC1 or LEC2 in vegetative tissues can trigger the formation of somatic embryos, implying that these TFs are sufficient to confer embryonic cell identity. Consistent with the key functions of the LAFL genes in the induction and control of seed development, their expression is tightly controlled through numerous transcriptional and chromatin-based mechanisms as described in more details below (Figure 1), while the LAFL proteins are subjected to post-translational regulations [19, 58, 59].
The LAFL network controls seed development and maturation. The master regulators LEC1 and LEC2 control both, embryo development and seed maturation. The B3 regulators, namely LEC2, FUS3, and ABI3 have partially redundant function in the control of seed maturation and the acquisition of desiccation tolerance. They control directly and indirectly (through other regulators such as WRI1 and different bZIPs), the expression of genes involved in these processes. The LAFL regulatory network and thus, seed development program, is strongly repressed during vegetative development through various epigenetic regulations including the PRC1/PRC2 complexes. Finally, LAFLs would indirectly induce their own repression at the end of the maturation phase.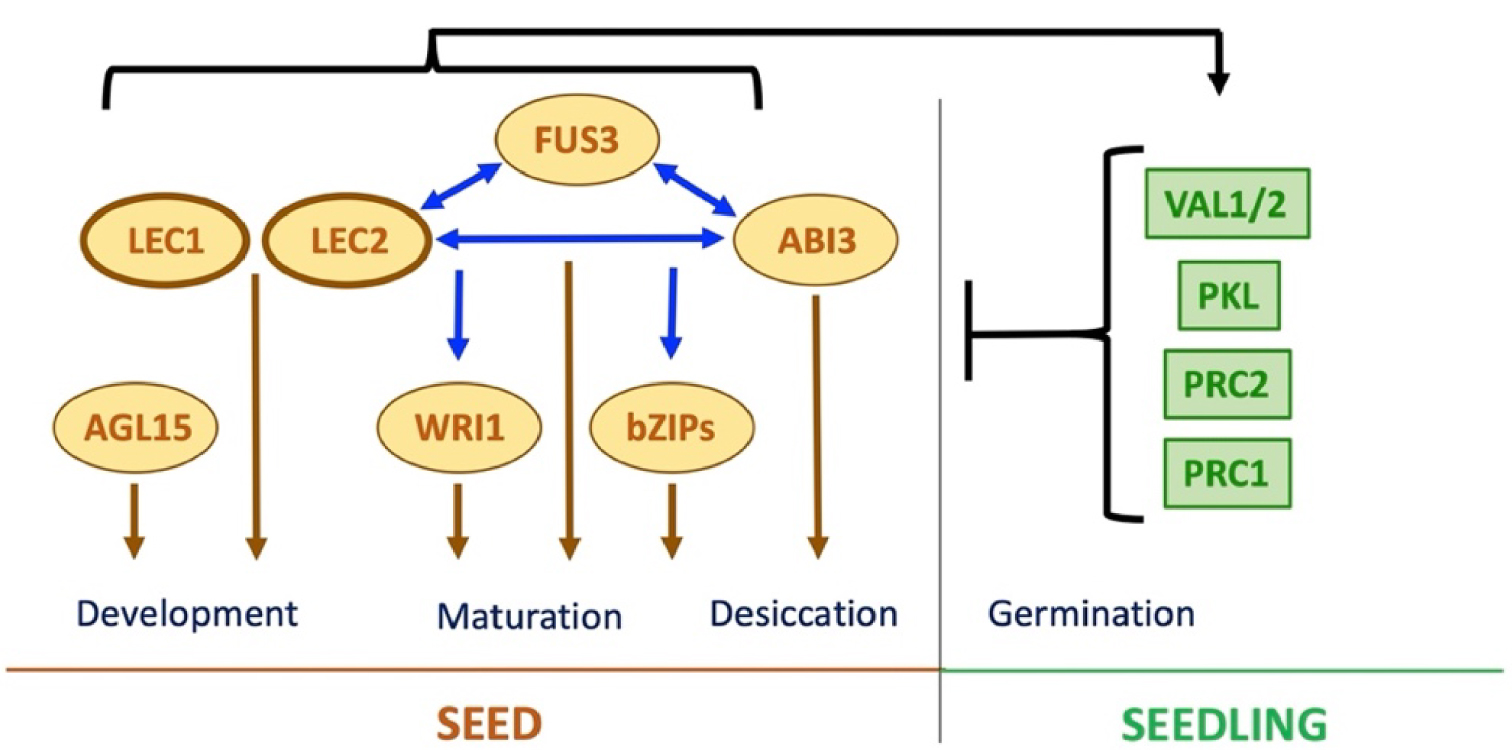
3.3. The LAFL and chromatin regulations
LAFL mRNAs are detected early during Arabidopsis embryo development, LEC1 and LEC2 expression peaking at the heart stage of embryo development, whereas ABI3 and FUS3 peak about a week later, during maturation [51]. Hormonal control of LAFL functioning is predominantly exerted by the auxin and ABA, acting as positive regulators of embryogenesis and maturation, respectively. In addition, the LAFL are involved in self-activating feedback loops, consistent with the occurrence of a minimum threshold level of the AFL “B3” TFs being necessary for their action [1]. The importance of such regulations is exemplified by the functional characterization of several positive and negative cis-regulatory elements in the promoter regions of LEC2, ABI3 and FUS3 [19, 60, 61]. A wealth of results demonstrated that LAFL genes are repressed in vegetative tissues through multiple pathways including chromatin-based mechanisms with POLYCOMB REPRESSIVE COMPLEXES 1 and 2 (PRC1 and PRC2) (Figure 1). PRC2 is an evolutionary conserved histone H3 lysine 27 methyltransferase (H3K27me3), whereas PRC1 carries out H2AK121 mono-ubiquitination (H2AK121ub) [62]. Several results suggested that the LAFL may act not only as direct transcriptional activators, but also as “pioneer factors”, that stably alter the chromatin landscape by recruiting different chromatin regulators [19, 63, 64, 65]. This role has been confirmed for the activation of the FLOWERING LOCUS C (FLC) during early embryo development [66, 67]. The LAFL antagonize VIVIPAROUS1/ABI3-LIKE1 (VAL1) and VAL2, two B3-type TFs involved in recruiting PRC to FLC [68, 69, 70, 71] or the CHD3 chromatin remodelling factor PICKLE to ABI3 and AGL15, to repress seed maturation [72]. Interestingly, FLC has been shown to be involved in the modulation of seed dormancy in response to temperature [73, 74].
Moreover, LEC1 and FUS3 also control the expression of PKL, VAL1, and RING1b (encoding a PRC1 subunit) during late maturation, that repress the expression of the LAFL genes [75]. On this line, LEC1 also induces the expression of SEED DORMANCY 4-LIKE (SDR4L) leading to its own repression [76]. These later results suggest that the LAFL could have an essential role in inducing the negative feedback regulation of their own expression at the end of the maturation phase. Chromatin modifications together with transcriptional regulations exerted by the LAFL are essential, therefore, for controlling the transitions between the different phases of seed development, including seed maturation.
4. Seed coat differentiation and functions
The maternal seed coat or testa plays an essential role in the protection of the endosperm and embryo from adverse environmental conditions during storage of mature dry seeds, and as a physico-chemical barrier regulating seed germination and dispersion. To fulfil these important functions, the Arabidopsis seed coat accumulates a battery of specialized metabolites (SMs) such as flavonoids, polysaccharides (mucilage) and lipid polyesters, through metabolic pathways that are tightly regulated at the spatio-temporal level [18, 77, 78] (Figure 2). As introduced above, seed coat differentiation is initiated by ovule fertilization [18], with auxin produced by the fertilized central cell and exported to the maternal tissues releasing the epigenetic block triggered by Polycomb Group (PcG) regulators [79]. The Arabidopsis seed coat comprises five fully developped and superimposed cell layers that undergo specialisation and differential accumulation of SMs [18, 80]. Seed coat differentiation is completed before then end of embryo maturation, with cells undergoing programmed cell death, so that the mature seed coat is a dead tissue.
Coordinated differentiation of proanthocyanidin- and mucilage polysaccharide-accumulating cell layers in the developing Arabidopsis seed coat. (A) Simplified transcriptional regulatory networks controlling the spatio-temporal biosynthesis of proanthocyanidins (PCs) and mucilage are presented. Trafficking steps toward final subcellular destinations, vacuole and apoplast respectively, are also shown. Proteins discussed in the main text are indicated by their abbreviation in pink upper-case letters with the corresponding mutant names in blue lower-case letters. Dashed arrows indicate multiple steps. (B) Histological localization of proanthocyanins and mucilage in the developing seed coat. Left, schematic representation of a developing seed section detailing integumentary cell layers. Right, section of developing seed with toluidine Blue O staining proanthocyanidins (blue) and mucilage (pink), corresponding to the colours indicated on the innermost and outermost seed coat cell layers of the schematic representation.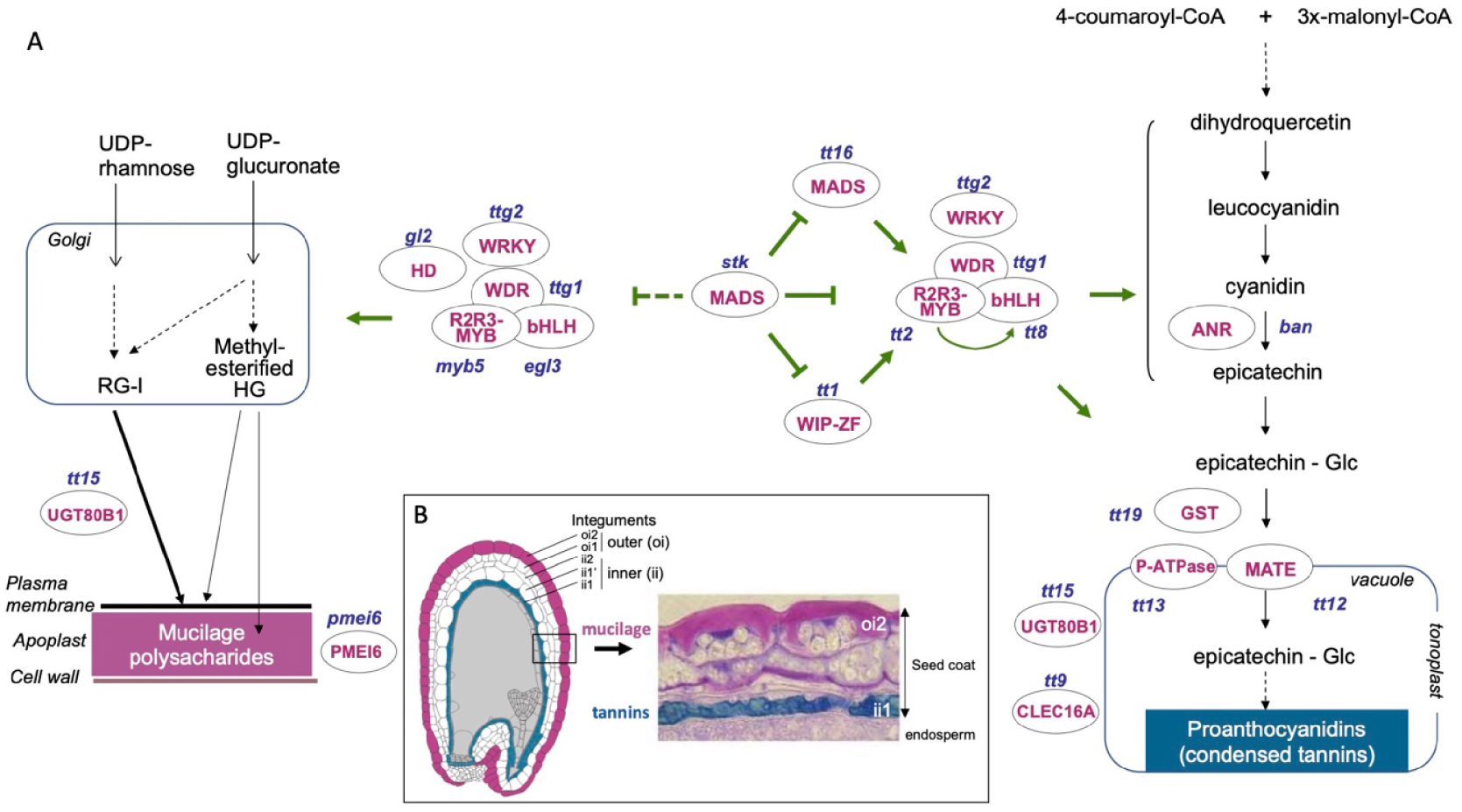
4.1. Flavonoid biosynthesis
In Arabidopsis seed coat, flavonoids consist in proanthocyanidins (condensed tannins) of the procyanidin (PC) type and flavonols including quercetin 3-O-rhamnoside, kaempferol-rhamnoside, and isorhamnetin-rhamnoside [81]. PCs and their epicatechin (EC) flavan-3-ol monomers are accumulated in the innermost cell layer of the inner integument (also termed endothelium), in a few adjacent micropylar cells and in chalazal cells of the pigment strand that joins endothelial edges. Afterwards the testa undertakes programmed cell death [18]. While flavonols can be detected in the whole seed coat, they are preferentially accumulated in the subepidermal cell layer of the outer integument. Notably, PCs are present specifically in the testa, while flavonols are present also in the endosperm and embryo [82].
It is now well established that flavonoid biosynthesis is catalysed by an enzymatic metabolon anchored at the external face of the endoplasmic reticulum. In contrast, the trafficking mechanisms by which the diverse end-products are transported to their final subcellular destinations have not been fully elucidated [83, 84]. Much progress in this field has benefitted from the isolation and functional characterization of Arabidopsis mutants affected in PC metabolism, collectively named transparent testa (tt) mutants because of a modified testa pigmentation [85]. PCs are initially stored in the vacuole as colourless compounds (Figure 2). Upon seed desiccation, their solubility decrease du to oxidation by a polyphenol oxidase, the TT10 laccase, thus colouring the wild-type seed coat brown [18, 82, 86]. A current model for PC vacuolar compartmentation predicts EC monomers to be glucosylated and EC-Glucose translocated into the vacuole lumen by the TT12 Multidrug and Toxic Compound Extrusion (MATE) secondary transporter located at the tonoplast [87, 88]. Energization of TT12 would be ensured by the tonoplastic TT13 P3A-ATPase [89]. The glutathione S-transferase (GST) TT19 would then act as a ligandin to protect EC-Glcucose from degradation in the cytosol during its transit towards TT12 [90, 91]. Moreover the molecular characterization of TT9 as a putative C-type LECtin 16A-like (CLEC16A-like) protein, which belongs to the HOmotypic fusion and Protein Sorting (HOPS) complex involved in vacuolar fusion, strongly suggests that vesicular trafficking is also involved in PC vacuolar compartmentation [92]. How the transporter/ligandin and vesicular trafficking routes cooperate to fulfill this function remains to be established. Interestingly TT9 is also involved in the trafficking of seed storage proteins (SSP) [92]. Thus it is likely that PC and SSP share part of their trafficking route to the vacuole. As hypothesized by Dixon and Sarnala [84], the endomembrane system may also hold the key to the long-standing mystery of flavan-3-ol monomer condensation, which mechanism (enzymatic or not) and subcellular site are still unclear.
4.2. Interplay between seed coat polymer function and production
Upon seed coat cell death, vacuolar PAs cross-link with cell wall components and also diffuse into the endothelial cuticle, thereby increasing the impermeability of the seed coat [93]. As this cuticle is adjacent to the endosperm, it indirectly decreases endosperm permeability and thus reinforces seed dormancy [94]. Suberin deposited in the subepidermal cell layer of the testa and hilum have similar effects on the permeability and physiology of seeds [95]. The characterization of tt15 mutants affected in a sterol 3-β-glucosyltransferase enzyme revealed that sterol glucoside composition is important not only for PC, cutin and suberin accumulation [94, 96, 97], but in the outer layer of seed coat cells also influences mucilage and cell wall polysaccharide accumulation [98]. Excess sterol accumulation has also been shown to impact seed coat differentiation [99]. Furthermore, the TT1/WIP-Zinc Finger and TT16/MADS-domain TFs determine both endothelium identity, and thereby indirectly PA metabolism, as well as endothelial cuticle deposition [100] suggesting that accumulation of the polymers reinforce seed coat physico-chemical properties could be coordinated.
4.3. Transcriptional regulation of seed coat flavonoid and mucilage accumulation
In Arabidopsis, the R2R3-MYB TT2 and MYB5 specifically activates PA and mucilage biosynthesis, respectively, through their interaction with the bHLH TT8 or EGL3 and the WDR protein TTG1, thus forming similar MBW complexes (Figure 2A) [20, 85]. Over the last decades, the key roles of conserved MYB, bHLH and WDR proteins have been characterized and now constitute one of the best-described transcriptional regulatory systems in plants [20, and references inside]. The direct or indirect interactions of TTG1 with TTG2/WRKY add a further level of complexity to this regulatory network [101]. Moreover SEEDSTICK (STK)/MADS negatively regulates not only TT16 and the MBW complex but also directly BANYULS (BAN), the master gene of the PA pathway encoding the anthocyanidin reductase (ANR) that catalyses the formation of epicatechin from cyanidin, by modifying its chromatin state [102]. In seed coat epidermal cells, STK has also been shown to directly induce the expression of a pectin methylesterase inhibitor, PMEI6, that plays a key role in modifying the properties of the outermost cell wall to facilitate the release of mucilage polysaccharides [103]. Apart from developmental cues, environmental signals such as temperature experienced by developing seeds on the mother plant can modulate soluble PC and quercetin (flavonol) deposition [104, 105] and tissular specificity of transcription factors such as TT2 and MYB5 [106].
5. Seed storage compounds
Seeds accumulate substantial amounts of storage compounds during the maturation phase. Depending on the species, these can be starch, β-glucans, storage lipids (triacylglycerols or waxes), or seed storage proteins (SSPs). In the model plant Arabidopsis as in most Brassicaceae species, a large embryo structure constitutes the major site of reserve deposition, but a thin endosperm tissue persists in mature seeds that also contain reserve compounds. In these seeds, SSPs and triacylglycerols represent the main reserve compounds, each type of compound accounting for roughly a third of mature seed dry weight. Seeds being mostly heterotrophic (despite a transient greening of the embryo observed during early maturation), synthesis of reserve compounds relies on the import of nutrients from the parent plant, in the form of sugars (mostly sucrose), amino acids, macro-elements (e.g. inorganic phosphate), and microelements such as iron or zinc. The bulk of the nutrients reaches the seed through the phloem. In Arabidopsis seeds that develop from anatropous ovules the vascular bundles terminate at the end of the funiculus in the chalazal seed coat. While the phloem is symplastically connected to the outer integument, zygotic tissues appear to be symplastically isolated. This symplastic isolation between seed tissues implies that nutrients must be transported across apoplastic borders to be ultimately delivered to the maturing embryo (Figure 3A). While the anatomy of Arabidopsis seeds has been well known for decades, our understanding of phloem unloading and nutrient transport has advanced only more recently thanks to the characterization of a range of transporters involved in either export to the apoplast or in subsequent re-import into an adjacent symplastic space [107]. Sucrose transport involves members of the SWEET [108] and SUC (SUCROSE-PROTON SYMPORTER) protein families [109], amino-acid export from the seed coat requires proton-dependent symporters of the UMAMIT (USUALLY MULTIPLE ACID MOVE IN AND OUT TRANSPORTER) family [110], while microelements are exported by different types of specialized transporters [107, 111].
Storage compound biosynthesis in maturing seeds of Arabidopsis. (A) Nutrient import from the parent plant through the vascular system and the seed coat. Provision of sucrose, amino acids, and micro- and macro-elements (ions) to the maturing embryo involves both symplastic transport in the maternal tissues of the seed and specialized transporters (e.g. SUC, SWEET, UMAMIT) for transport across apoplastic borders separating the different seed tissues. (B) Simplified overview of the metabolism associated with storage compound biosynthesis in embryo cells. AA, amino acid; AcCoA, acetyl-CoA; FA, fatty acid (C16 and C18); Gln, glutamine; ii, inner integument; MaCoA, malonyl-CoA; oi, outer integument; SSP, seed storage protein; SUC, sucrose; TAG, triacylglycerol; TCA cycle, tricarboxylic acid cycle; VLCFA, very long-chain fatty acid (C > 20).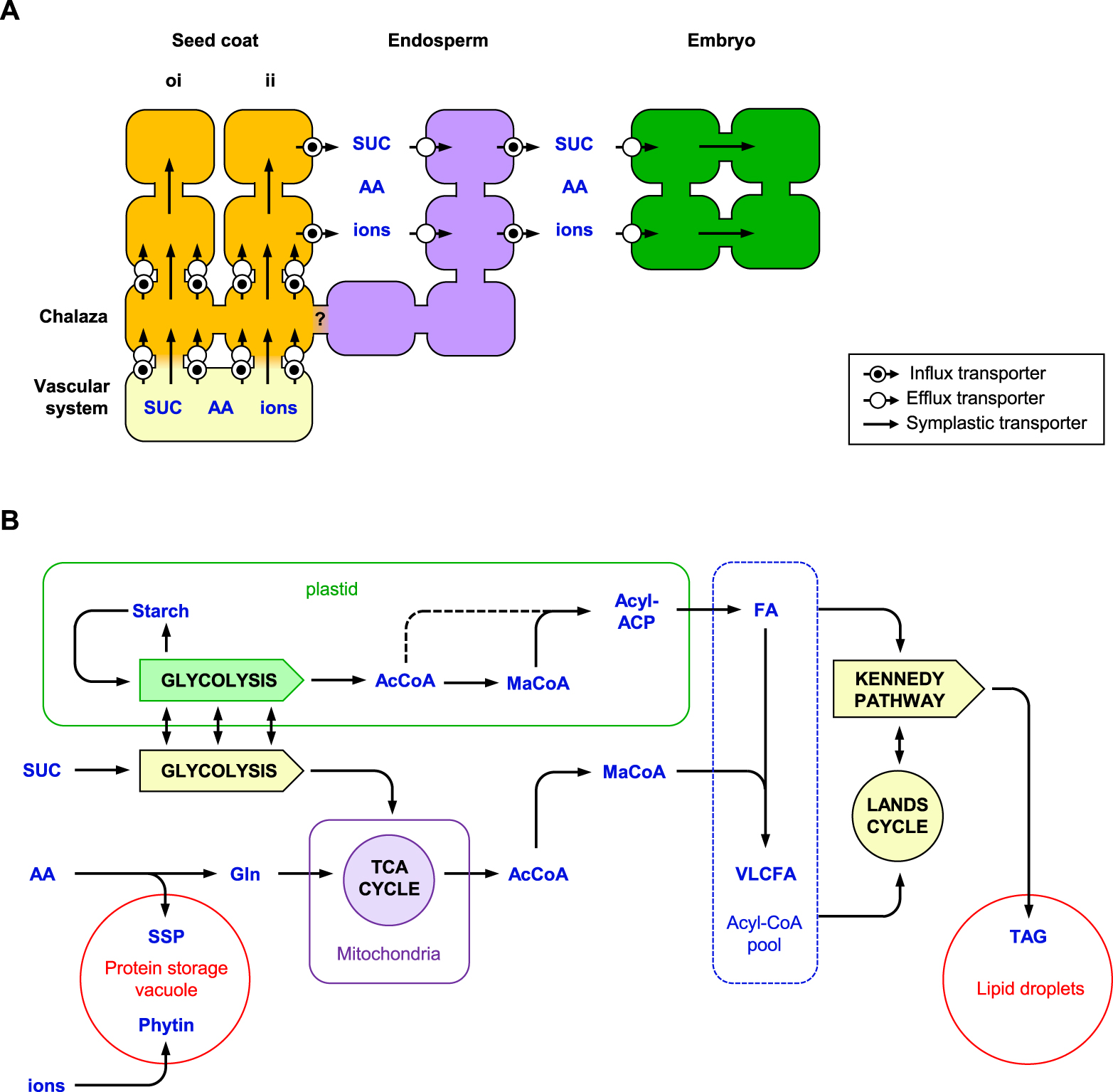
5.1. Fatty acids and lipids
In zygotic tissues, hexoses released by sucrose cleavage are converted to hexose-phosphates that can be metabolized through different pathways depending on the of type of storage compounds stored in seeds [112]. In the oleaginous seeds of Arabidopsis, end products of the glycolysis are further metabolized to acetyl-CoA and malonyl-CoA molecules that are used for de novo synthesis of fatty acids in the plastids (Figure 3B). A fatty acid synthase complex of type II produces and elongates saturated acyl chains bound to acyl carrier proteins (ACPs). 16:0 and 18:0 acyl chains are released from this complex that can be desaturated by stromal acyl-ACP desaturases (AADs) to form monounsaturated fatty acids [113]. After hydrolysis of the acyl groups by thioesterases, free fatty acids can be exported to the cytosol and activated to CoA esters. In the endoplasmic reticulum, the membrane-bound fatty acid elongase complex that proceeds via reactions analogous to those of de novo fatty acid synthesis and produces very-long-chain fatty acids comprising up to 24 carbon atoms can further elongate long-chain fatty acyl-CoAs. The enzymes responsible for the biosynthesis of polyunsaturated fatty acids found in seed oils are the microsomal fatty acid desaturases FAD2 and FAD3, which respectively convert 18:1 to 18:2, and 18:2 to 18:3, while theses fatty acids can in turn be esterified to phosphatidylcholine [114]. Triacylglycerols are composed of three fatty acyl chains esterified to a glycerol backbone. Their biosynthesis therefore requires the sequential acylation of glycerol-3-phosphate, which is derived from glycolytic intermediates, by acyltransferases of the Kennedy pathway. During the last decade however, studies have revealed that incorporation of fatty acids into triacylglycerols is much more complicated than just a linear pathway and involves various exchanges of acyl chains between different lipid pools [115]. Once synthesized, triacylglycerols are sequestered into discrete organelles called lipid droplets (LDs) formed of a core of triacylglycerols surrounded by a monolayer of phospholipids. Different categories of specialized proteins are embedded in the half-unit membrane of lipid droplets that prevent their coalescence during storage or facilitate oil breakdown after seed germination. Structural proteins associated with lipid droplets include oleosins and less abundant enzymes and aquaporins, among others [116].
5.2. Seed storage proteins
SSPs synthesized in zygotic tissues represent a source of carbon (C), nitrogen (N), and sulfur (S). In Arabidopsis, as in other Brassicaceae species, the predominant SSPs are the 12S globulins (also called cruciferins) and the 2S albumins (sometimes termed arabins). They represent 84% of protein species in these seeds and they are stored in protein storage vacuoles (PSVs) [117]. Less-abundant proteins stored in other cell compartments include oleosins and late embryogenesis-abundant (LEA) proteins. SSPs are initially synthesized as long precursors on the rough endoplasmic reticulum from amino acids directly imported from the parent plant or obtained through transamination reactions taking place in zygotic tissues. These precursors then undergo sequential post-translational cleavage catalyzed by vacuolar processing enzymes (VPEs) at PSVs or while in transit to PSVs [118]. Cleavage is necessary to release the α- and β-subunits comprising many SSPs before intra- and inter-chain disulfide bonds are established between these subunits to form complexes having a stable and compact three-dimensional structure. Moreover, limited truncations occurring successively at the C-terminal disordered region of 12S globulins have also been reported to yield a variety of polypeptides all derived from the same subunit. While the processing and trafficking of SSPs may differ between plant species, tissues, and developmental stages, these processes all rely on a highly dynamic and functionally specialized endomembrane system and involve the retromer complex [119, 120, 121]. Storage of SSPs also appears to be highly variable. In addition, mutations affecting the production of a given class of storage proteins were indeed shown to be compensated by an increased accumulation of other seed proteins, while the overexpression of transgenes encoding SSPs in seeds yielded unchanged amounts of total seed proteins [122]. Together, these results suggest that N supply rather than SSP metabolism regulates total N content in seeds. Also, PSVs do not only store protein reserves, these vacuolar compartments delimited by a lipid bilayer also contain minerals too. Intraorganellar inclusions termed globoids can be observed in PSVs that consists of phytin, a salt of phytic acid, and cations such as K+, Mg2+, and Ca2+ [123]. Seed iron can also be stored in globoids of the PSVs, or, to a lesser extent in plastid-localized ferritins that are able to store iron in a non-toxic form [124].
5.3. Reserve partitioning between seed compartments
While the diversity of seed storage compounds between species has long been documented, the continuous improvement of analytical techniques allows studies using smaller samples and the recent emergence of mass spectrometry imaging instruments has led to more detailed characterization of tissues, revealing heterogeneity of reserve composition between the two zygotic tissues of the seed, and even between different embryo regions [125, 126]. In the endosperm of Arabidopsis seeds for example, 12S globulins are underrepresented in the protein fraction [127] while triacylglycerols appear to be very rich in unusual ω-7 mono-unsaturated fatty acids that are at the limits of detection in the embryo [128]. These results invite to now investigate further seed metabolism at lower tissue scale, while integrating the influence of environmental factors (see below).
6. Seed specialized metabolites
Seeds accumulate a wide range of specialized metabolites (SMs), some of which being specific compared to roots, leaves or flowers [129, 130, 131, 132, 133]. SMs, which were previously defined as “secondary metabolites” are in fact largely involved in the interactions of plants with their environment and have specific and important developmental or physiological functions in seeds (Figure 4) [134, 135].
Environmental regulation of the seed Metabolome. The environmental regulation of seed specialized metabolites and reserve compounds. The impact of abiotic and biotic stresses on primary (proteins, lipids and fatty acids, C/N ratio) and specialized metabolism is shown. Legend: FA, fatty acids; SSP, seed storage proteins.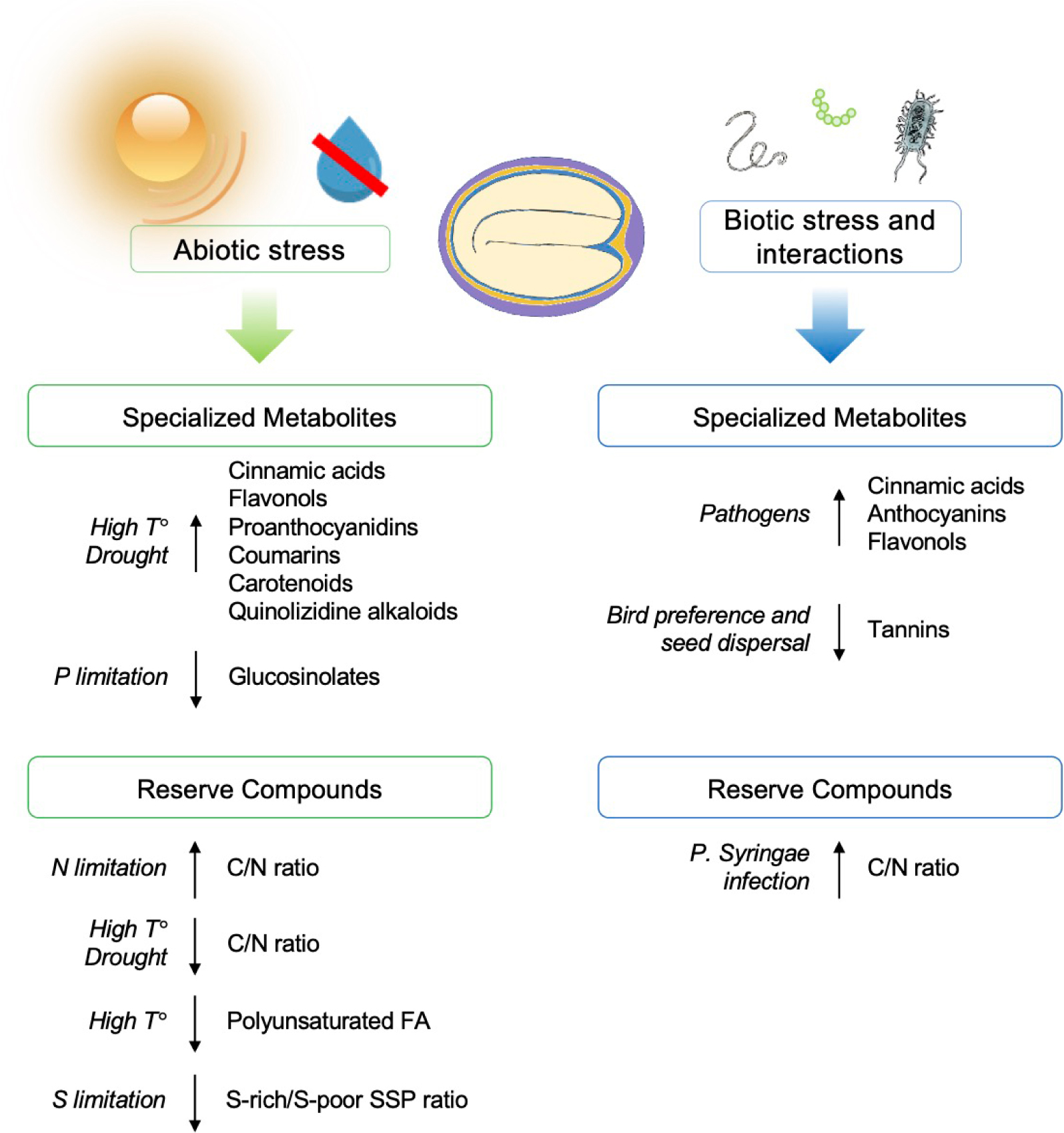
6.1. Functions of the seed specialized metabolites
Numerous flavonoids (for more details see above paragraph on seed coat differentiation), cinnamic acids, and coumarins constitute the first layer of seed defence against pathogen attacks [135]. Moreover, these SMs have a strong antioxidant activity and protect the seed coat and embryo from abiotic stresses, such as desiccation, high temperature or UV radiations that occur during seed maturation and/or aging [136]. In addition to their role against abiotic and biotic stresses, seed flavonoids also play a role in the regulation of seed longevity, dormancy and germination, hence impacting seedling vigour and crop yields [137]. Reduced seed dormancy has been observed in Arabidopsis mutants for flavonoid biosynthetic genes [105, 138]. Lastly, seeds constitute an important source of metabolite diversity for food and numerous SMs are important healthy or antinutritional compounds [85, 135]. In a global context of climate change and a demand for the development of more sustainable agriculture, these functions have led to increased interest in SMs for agroecological applications that range from sustainable pest control to food quality.
6.2. SM biosynthesis and diversity
SMs can be divided into three main classes: terpenes (more than 40,000 compounds), alkaloids and nitrogen containing molecules (about 20,000 compounds) and phenolic compounds or phenylpropanoids (more than 10,000 compounds) [135]. Phenylpropanoids are synthetized from shikimate and malonate pathways and are characterized by the presence of one or several hydroxylated aromatic rings, and multiple potential decorations (i.e. hydroxylation, glycosylation, acylation, methylation, and prenylation). Most abundant seed phenylpropanoids belong to the anthocyanins, flavonols, flavan-3-ols and proanthocyanidins (PAs; condensed tannins, which are polymers of flavan-3-ols) classes. Tannins are preferentially accumulated in the seed coats, which most likely has been an advantageous trait during evolution as it limits the consumption of seeds by animal prior to dispersal and germination [40]. Seed terpenoids are the precursors of several key metabolites, such as gibberellins and tocopherols (vitamin E). Terpenoids are classified according to the number of isoprene units (C5H8). Some examples are carvone and linalool in Carum carvi and/or Coriandrum sativum (monoterpenes), artemisinin in Taxus brevifolia (sesquiterpene), ginkgolides in Ginkgo biloba and (diterpenes), and saponin in Medicago truncatula (triterpene) [129, 139]. In addition to phenylpropanoids and terpenoids, alkaloids and N-containing compounds are accumulated to significant levels in seeds of several plant species. This class includes purine, tropane, quinolizidine and indole alkaloids [129]. Purine alkaloids, such as caffeine in Coffea arabica and theobromine in Theobroma cacao, are synthetized from nucleotides which may have allelopathic effect or induce antimicrobial activities [129]. Quinolizidine alkaloids are abundant in seeds of several legume species, such as Lupinus polyphyllus [140]. Given their toxicity, domestication led to the selection of legume varieties with reduced quinolizidine alkaloid contents. Other well-known N-containing metabolites are the glucosinolates (GLSs), that are present in the seeds of several Brassicaceae such as Arabidopsis, Camelina or Brassica species [42, 141, 142]. Despite being highly abundant in seeds, GLSs are synthesized in leaves (and/or roots) and then transported to seed tissues by GLUCOSINOLATE TRANSPORTERS (GTR1 and GTR2) [143, 144].
6.3. The impact of domestication on seed specialized metabolites
Domestication, often unintentionally modified the composition and amount of SMs in seeds of cultivated species compared to their wild relatives [39, 145, 146, 147, 148]. Domestication was mainly focused on physiological and visible traits, such as yield or taste sensory characteristics, and drastically modified the composition and amount of many plant and seed metabolites. Among the metabolic pathways that were altered by domestication, many have an undisputed beneficial health- and stress-related roles in crop plants [149]. For example, in most crop species domestication led to the selection of seeds with low tannin contents, since they are considered antinutritional compounds that can, for example, interact with proteins and reduce amino-acids availability [129, 150, 151]. The selection of plant species with reduced tannin levels may have inadvertently led to negative effects on the accumulation of other beneficial flavonoids for the plant and human health, such as the flavonols, by depleting the whole flavonoids pathway [81]. This is also the case for domesticated maize kernels that are characterized by lower flavonol levels compared to wild species [39]. In another example, it has been shown that, compared to the wild species, legume seeds of domesticated species accumulated lower levels of carotenoids [147].
7. Plasticity and environmental regulation of seed composition
In addition to genetic determinism and tissue distribution, the content and nature of storage compounds (including oil and proteins) and SMs are also shaped by the environment, and strongly impact the nutritional, agronomic or industrial quality of seeds (Figure 4). Many biotic and abiotic factors, such as drought or elevated temperatures can affect seed oil and protein composition [152, 153, 154, 155, 156], seed germination, and impact seedling vigour [157, 158].
7.1. Storage compounds
Nitrogen is an essential nutrient utilized for the biosynthesis of amino acids and proteins. SSP biosynthesis thus requires N that is assimilated, then distributed from source organs to developing sinks like seeds. The limitation of plant nitrate supply was shown to reduce N allocation in rosettes and stems while enhancing the remobilization of organic N for plant growth and reproduction [159]. Despite this increased N recycling, seeds produced by plants experiencing nitrate limitation have reduced N contents [160]. If N and C concentrations in seeds (used as indicators of SSP and oil contents) are differentially sensitive to nutrition depending on accessions, a general upward trend for N concentrations at high nitrate supplies is observed while, at the opposite, C concentrations are higher at low nitrate supplies. But SSPs also represent a S store and developing seeds are known to employ homeostatic mechanisms to balance N and S storage [122]. Seeds exhibit a remarkable capacity for maintaining the level of SSPs stored in conditions of varying S supply: under S-deficient conditions, S-rich proteins are suppressed while a concomitant increase in S-poor proteins is observed. In Arabidopsis, the amount of 2S albumins (At2S3) thus decreases under S-deficient conditions and the accumulation of the α- and β-subunits of At12S3 is suppressed [161]. Indeed, the At12S3 globulin is relatively rich in S-containing cysteine and methionine compared with At12S1, At12S2 and At12S4. The regulation of SSP composition in response to S-deficiency involves various transcriptional, translational and post-translational controls that have been studied in different plant systems [161].
Environmental stresses such as drought and heat have been reported to exert important constraints on productivity and yields while affecting seed composition in various oleaginous crops [162, 163]. Likewise, post-flowering drought dramatically affects seed development and reduces seed yield in Arabidopsis. What is more, seeds of Arabidopsis accessions experiencing drought during seed filling exhibit significantly reduced C contents that are only partially compensated by moderate increases in N [159]. In a somewhat similar manner, heat stress limits plant growth and fewer seeds are produced that also display decreased C/N ratios with respect to that of control plants. Various hypotheses have been put forward to explain the shift of this balance that include (i) a disequilibrium in the ratio of N available in the plants with respect to the few seeds produced [159], (ii) a negative effect of high temperatures on photosynthesis, and (iii) the transcriptional up-regulation of lipase-coding genes promoting triacylglycerol degradation in seeds [164].
Aside from the documented modification of the overall amount of oil stored in seeds, high temperatures also impact the fatty acid composition of these oils. An inverse relationship between temperature and the polyunsaturated fatty acid content of Arabidopsis seeds was reported [165]. For example, between 10 °C to 30 °C, the 18:1 content of seed triacylglycerol doubles while the 18:3 content halves [166]. If the response of Arabidopsis seeds is similar to that of several other oilseed crops such as oilseed rape, soybean, or sunflower, an opposite trend was described in seeds of oilseed rape exposed to high night temperature, suggesting to make a distinction between the effects of day heat and night heat[164]. But whatever the trend of the shift in polyunsaturated fatty acids, the response is largely controlled by the activities of the microsomal desaturases FAD2 and FAD3 on phosphatidylcholine, in turn leading to compositional changes in triacylglycerol composition. Different levels of regulations of these desaturases have been reported in the different species in which they have been studied that encompass modifications of substrate availability, transcriptional and translational controls, as well as modified protein turnover [166, 167]. Finally, biotic stresses also influence the accumulation of seed storage compounds. For example, Arabidopsis plants infected with an avirulent strain of Pseudomonas syringae able to activate the plant immune response display an increased C/N ratio reflecting a decrease in seed N content [159].
7.2. Specialized metabolites
The diversity and content of seed SMs is also affected by the environmental conditions. This has been exemplified above for soluble PC and quercetin (flavonol) deposition in Arabidopsis seed coat that can be modulated by environmental signals such as temperature [104, 105]. This is also the case for quinolizidine alkaloids, whose accumulation is enhanced in grains of some lupin species in response to high temperature and drought stress [168]. Despite little information is available, it has been also reported that glucosinolate content decreased in seeds of B. rapa plants subjected to potassium deficiency [169]. More recently, a large environmental induction and plasticity of SMs has been observed in seeds of Camelina sativa cultivated during five consecutive growing seasons [152, 153]. This work highlighted major effects of the environment on the stimulation of the seed specialized metabolome. In this work, flavonols, glucosinolates and alkaloids were identified as plastic metabolic categories, while flavan-3-ol amount was more stable among the five years of plant cultures. The authors also observed that seed SMs display a much higher environmental plasticity compared with oil and SSPs, other compounds related to seed quality, whose metabolism was moderately influenced by the environmental conditions. Nevertheless, much less is known about the diversity and plasticity of SM modifications (i.e. hydroxylation, glycosylation, methylation and acylation) in response to environmental variations.
8. Hormonal control of seed dormancy and germination
Seed dormancy is the temporary inability of a viable seed to undergo germination. This adaptative trait allows mature seeds to interrupt their life cycle and resume growth in the favourable season [1]. Dormancy is induced during seed development and/or after seed dispersal. It can be alleviated with time by environmental conditions such as light, temperature or nitrate, which are integrated by signalling networks involving hormones including ABA and GA and free radicals such as reactive oxygen species (ROS) and reactive nitrogen species (RNS) (Figure 5).
Interactions between ABA, ethylene and DOG1 signalling pathways in seed dormancy. ABA and ethylene interact at different levels in biosynthesis and signalling pathways as described in the text. This diagram summarizes recent findings on crosstalk of DOG1 with ABA and ethylene signalling downstream of LAFL master regulators of seed maturation, LEC1, ABI3, LEC2 and FUS3. ABI3 binds to the ODR1 promoter and represses its expression, thus releasing bHLH57 inhibition and promoting ABA biosynthesis and seed dormancy. A positive-feedback may occur by the stimulation of ABI3 by ABA. ABA binding to PYL receptor induces the formation of a complex with PP2C and inhibits phosphatase activity, allowing SnRK2 kinases to phosphorylate downstream targets and positively regulate dormancy. DOG1 interferes with ABA signalling by interacting with two PP2C, AHG1 and AHG3. In contrast to AHG3, AHG1 is resistant to PYL receptor binding, suggesting an alternate ABA-independent signalling pathway. DOG1 expression is activated by bZIP67 downstream of LEC1 and by ERF1 downstream of the ethylene receptor ETR1. The binding of DOG1 promoter by the complex of ERF12-TPL represses DOG1 expression and promotes the release of seed dormancy by ethylene.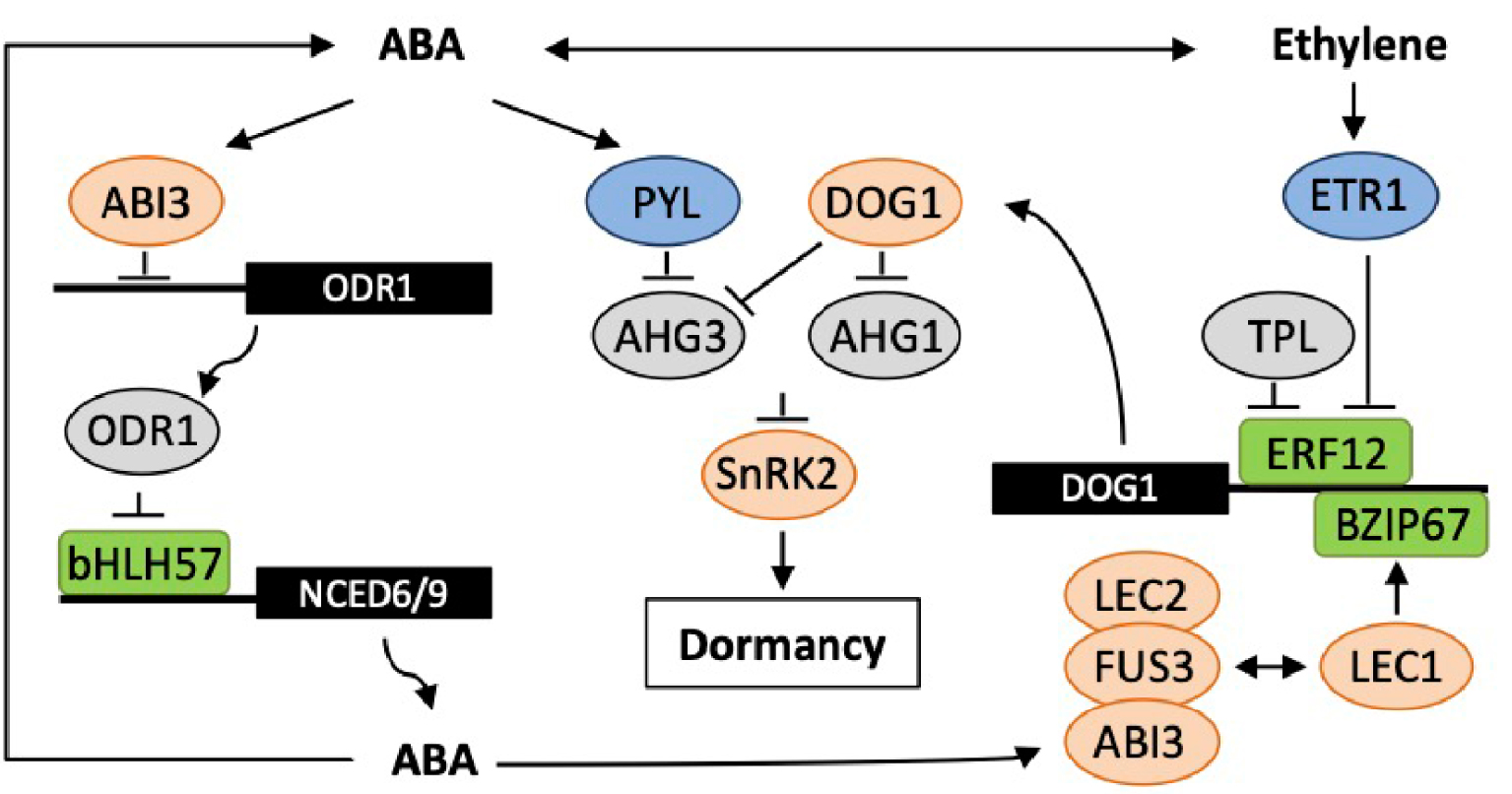
8.1. The key role of ABA in seed dormancy
The crucial role of ABA for the promotion of seed dormancy was deduced from seed phenotypes of ABA metabolism or signalling mutants in several species. The subsequent identification of the genes involved in ABA metabolism, transport, perception and signalling has extended our knowledge about molecular factors and interactive networks acting in the different tissues of the seed [170, 171]. ABA accumulation in developing Arabidopsis seeds has been shown to peak at mid-maturation and genetic studies demonstrated that ABA produced by zygotic tissues is responsible for dormancy induction. ABA levels decrease to low levels in mature dry seeds and further catabolism occurs in imbibed seeds [172]. ABA is formed after cleavage of carotenoid precursors and inactivated mainly by 8′-OH hydroxylation. Cleavage by the 9-cis-epoxycarotenoid dioxygenase (NCED) and catabolism by ABA 8′-hydroxylase (CYP707A) thus constitute key regulatory steps and both enzymes are encoded by multigene families. Fine-tuning of ABA levels that influences dormancy depth and is controlled by the expression of specific members of NCED and CYP707A families [173, 174, 175]. Notably, NCED5, NCED6 and NCED9 expression make a major contribution to dormancy induction during seed development and upon imbibition, expression of CYP707A2 is correlated with sustained higher ABA levels and germination inhibition in dormant seeds.
ABA is perceived by a family of ABA receptors encoded in Arabidopsis by 14 genes, named pyrabactin resistance/PYR-like/regulatory components of ABA receptor (PYR/PYL/RCAR). When ABA is present, these proteins sequester and inactivate clade A protein phosphatases 2C (PP2C), releasing the inhibition of a subset of three SNF1-related kinases 2 (SnRK2). SnRK2 kinases in turn can phosphorylate bZIP transcription factors of the ABA INSENSITIVE5 (ABI5)/ABA response elements (ABRE)-binding (AREB)/ABRE-binding factor (ABF) family, which bind to ABRE promoter sequences in ABA-inducible genes and contribute to repression of seed germination cutler [176, 177]. Besides ABA, DELAY OF GERMINATION 1 (DOG1) has been identified as a major regulator of seed dormancy, which strongly contributes to the natural variation of this trait, however despite intensive research, the molecular function of this heme-binding protein is not fully understood [171, 178, 179]. DOG1 protein accumulates during seed maturation and protein levels in dry seeds correlate well with the depth of dormancy in freshly-harvested seeds [180]. Nevertheless, DOG1 protein is still found in after-ripened seeds, when dormancy has been released, suggesting possible loss of activity by still unknown mechanisms. An important discovery concerning DOG1 function has been the identification of the interaction between DOG1 and the ABA signalling pathway (Figure 3). DOG1 has been shown to bind to and negatively affect the function of ABA HYPERSENSITIVE GERMINATION1 (AHG1) and AHG3, two clade A PP2C phosphatases [181, 182]. The double mutant ahg1 ahg3 exhibits a very deep dormancy indicating that AHG1 and AHG3 act redundantly in seeds to prevent dormancy release. Interestingly, AHG1 activity has been reported to be resistant to inhibition by PYR/PYL/RCAR receptor and thus able to repress SnRK2 activity in the presence of ABA. Seed dormancy could, therefore, be differentially regulated through two phosphorylation pathways, by AHG1 under the control of DOG1 only, and by AHG3 under the control of both ABA and DOG1 [181]. Heme-binding is essential for DOG1 function in seed dormancy, but it does not interfere with the interaction between DOG1 and AHG1 [182]. Since oxidative events are closely related to dormancy release, the presence of heme has been suggested to participate in redox state sensing. It was recently proposed that heme binding to oxygen could determine dormancy levels through the inactivation of DOG1 [171].
A recent study identified REVERSAL of RDO5 1 (ODR1) as a negative regulator of seed dormancy [183]. ODR1 has been shown to interact with the transcription factor bHLH57 preventing the activation of NCED gene expression (Figure 3). Arabidopsis ODR1 is the ortholog of rice Seed dormancy 4 (Sdr4) and thus has been also called Sdr4 like 1 (SFL1). Intriguingly, Arabidopsis odr1/sfl1 seeds are more dormant whereas rice sdr4 exhibits preharvest sprouting (precocious germination on the ear). Further evidence indicated that Arabidopsis ODR1/SFL1 would have a dual effect on dormancy, positive during early seed development and negative at late maturation stages, by modulating LAFL and DOG1 expression [184].
8.2. Interactions with other hormones
ABA and gibberellins (GA) have well-known antagonistic roles on embryo growth and endosperm weakening, which influence seed dormancy and germination in response to environmental signals. Knowledge about regulatory networks that differentially fine-tune the ABA/GA balance has been described in several recent reviews [170, 185, 186]. Important regulatory factors have been identified, such as SOMNUS (SOM) which functions in both light and heat responses and is controlled by master regulators of GA and ABA signalling, such as DELLA and ABI3 [187, 188]. DOG1 is also supposed to be indirectly involved in the ABA/GA balance through the down-regulation of GA catabolism [189]. For both SOM and DOG1, the downstream transcriptional regulators of hormone metabolism genes remain to be discovered. Interestingly, a transcription factor which belongs to the NIN-like protein (NLP) family involved in nitrate responses has been shown to directly bind to the CYP707A2 promoter suggesting a key function for NLP8 in nitrate signalling for the promotion of germination [190, 191]. Among hormones that synergistically or antagonistically interact with ABA [192, 193, 194, 195, 196], ethylene has multiple interplay with both ABA and DOG1 signalling pathways. Ethylene has a positive role in dormancy release by the negative regulation of ABA biosynthesis and signalling pathways and a positive interaction with GA pathways) [197, 198, 199]. Furthermore, the ethylene response factor ERF12 was recently shown to form a complex with the corepressor TOPLESS (TPL) and bind to the DOG1 promoter. These repressor complex functions downstream of the ethylene receptor ETHYLENE RESPONSE1 [200]. In addition to hormones, ROS are key players in dormancy alleviation and germination. While the implication of ROS in DOG1 function is still putative, their involvement in hormone signalling pathways is well documented [201, 202]. Nonetheless, many questions await further investigation concerning their production sites and mode of action [203, 204].
8.3. Environmental control
Environmental conditions, especially temperature, experienced by the mother plant during seed development have been widely reported to modulate dormancy depth. In Arabidopsis, the enhancement of dormancy by cool temperatures is correlated with an increased abundance of both DOG1 transcript and ABA levels in dry seeds [189]. To date, a limited number of regulatory factors have been identified. DOG1 expression has been shown to be directly activated by the transcription factor bZIP67 downstream of the master regulator of seed maturation LEC1, with bZIP67 protein abundance in dry seeds increasing at cool temperatures [205]. However, a more recent study suggested that the regulation of DOG1 by maternal temperature would be an indirect effect of cold temperature on DOG1 mRNA degradation [206]. In cold matured-dry seeds, the increase in ABA content correlates well with a decreased abundance in CYP707A2 transcripts [189]. Transcription factors, such as ABI4, MYB96 and bHLH57, have been shown to bind NCED or CYP707A promoters, but none of them has yet been linked to the maternal temperature-dependent control of ABA levels [207]. Nevertheless, the identification of the regulatory complex between the basic helix-loop-helix transcription factor bHLH57 and REVERSAL of RDO5 1 (ODR1) further strengthened the implication of LAFL genes in dormancy regulation by ABA and DOG1 [183]. Indeed, ODR1 gene expression has been shown to be directly repressed by ABI3. In turn, ODR1 negatively regulates NCED6 and NCED9 gene expression by its inhibitory interaction with bHLH57 that, in the absence of ODR1, directly binds to NCED promoters and positively regulates gene expression. ODR1 could, therefore, negatively regulate seed dormancy by affecting both ABA accumulation and signalling through the feedback regulation of ABI3 by ABA.
9. Post-translational modifications by free radicals during seed storage and germination
Reactive oxygen species (ROS) and reactive nitrogen species (RNS) incessantly produced as by-products of metabolism play significant roles in seed physiology. ROS (e.g. hydroxyl, superoxide radical and hydrogen peroxide) and RNS (e.g. nitric oxide, nitric dioxide, nitrous acid, and dinitrogen tetroxide) content changes in all phases of seed life cycle and influence seed dormancy, longevity, and vigour [202]. Studies carried out since 2010 illustrate that ROS and RNS are performing oxidative and nitrosative signalling to induce seed germination within an oxidative window level [201, 208, 209, 210]. ROS/RNS-mediated post-translational modifications (PTMs) such as carbonylation, oxidation of the sulfur amino acids, S-nitrosylation and nitration can promote seed germination.
9.1. Protein carbonylation
Protein oxidation by carbonylation is an irreversible and not repairable PTM. To avoid their toxic accumulation, carbonylated proteins are degraded through the action of the 20S proteasome in the cytosol. Oxidative attack of amino acyl moieties, such as Lys, Arg, Pro, and Thr, induces formation of carbonyl groups (aldehydes and ketones) on the side chains. Proteomic studies showed that massive protein oxidation occurs during Arabidopsis and pea seed maturation, storage and germination [78, 211, 212, 213, 214, 215]. These studies disclosed the existence of carbonylation of seed storage proteins (SSPs) in the mature dry seeds, which raised the hypothesis that such a PTM would help trigger their mobilization during germination by destabilizing the compact protein bodies, thus increasing their susceptibility toward proteolytic cleavage.
Proteomics revealed a new role for SSPs in dry Arabidopsis seeds during aging. Cruciferins (CRUs) are the most abundant SSPs in Arabidopsis and seeds of a triple mutant for three CRU isoforms (CRUa, CRUb, and CRUc) are more sensitive to artificial aging and their seed proteins are highly oxidized compared with wild-type seeds. These results confirm that oxidation is involved in seed deterioration and that SSPs buffer the seed from oxidative stress, thus protecting important proteins required for seed germination and seedling formation [216]. Carbonylation-ROS-dependent PTM of proteins-may be regarded as one of the important events in the process of aging in plants. It is estimated that during aging, starvation or disease of various organisms about 10% of the proteome is more prone to carbonylation [217]. In agreement, during the aging process in seeds, ROS were shown to induce the carbonylation of numerous proteins, which changes their functional properties and affects seed vigour. A recent work in wheat seeds disclosed the existence of a significant negative correlation between seed vigour and carbonyl protein content [210]. A similar behaviours was noted during long-term storage of beech seeds [218]. In rice seeds, results showed that although the germination rate of seeds only decreased by about 20% in early steps of seed aging, the carbonylation levels of 68 proteins markedly changed, indicating oxidative damage; again, the carbonylation level of seed-storage proteins increased [219]. Seeds of Arabidopsis lacking a functional NADP-MALIC ENZYME 1 (NADP-ME1) have reduced seed viability relative to the wild type and display higher levels of protein carbonylation. The results suggest that NADP-ME1 activity is required for protecting seeds against oxidation during dry storage [220]. In parallel, the carbonylation of many proteins involved in cellular and metabolic processes occurs during dormancy alleviation of sunflower [221] and upon imbibition of non-dormant Arabidopsis and pea seeds [213]. Also, it was hypothesized that impairment of some metabolic activities such as the oxidation of glycolytic enzymes could lead to the activation of the pentose phosphate pathway, thus providing reducing power promoting the germination potential. Although protein carbonylation is usually described as corresponding to deterioration factors, it surprisingly plays a positive role during after-ripening and germination of orthodox seeds. This may indicate the need for cellular recycling of proteins in the alleviation of seed dormancy and for germination [222].
9.2. Methionine oxidation
Due to their abundance and their chemical composition, proteins constitute a major target for ROS, especially concerning the amino acids methionine (Met) and cysteine (Cys) whose side chains contain a sulfur atom. The oxidation of Met leads to Met sulfoxide (MetO) formation, a redox type of PTM. This reaction can be reversed through the specific action of methionine sulfoxide reductases (MSR) [223]. Importantly, the MSR system has been shown to control seed longevity in Medicago, Arabidopsis [224], and Fagus sylvatica [225]. Then, a more severe oxidative attack of Met results in the irreversible formation of a sulfone derivative (Met sulfone). In a recent work carried out on mature dry seeds of Arabidopsis, twenty-four oxidized Met residues in 19 proteins identified by mass spectrometry were found to be surface exposed in these proteins [209]. In the three-dimensional environment of the oxidized Met, amino acid residues were detected that could be converted by oxidation (carbonylation) or by phosphorylation suggesting a possible interplay between Met oxidation and the other PTMs. An identification of the proteins oxidatively modified in Met residues revealed the finding that MetO-containing proteins are related to seed longevity. Moreover, this study disclosed the presence of MetO in Arabidopsis 12S globulins, which could indicate that, like carbonylation, MetO might induce structural changes in seed storage proteins and could interfere with their packaging during seed maturation and their mobilization during seed germination. Importantly, these changes might be reversible because MSRs were recently immunolocalized in protein storage vacuoles where SSPs are stored during seed maturation [225].
9.3. Protein S-nitrosylation
NO is a gaseous free radical involved in numerous signalization pathways both in animals and plants. There are three important NO-dependent modifications: metal nitrosylation, cysteine S-nitrosylation and tyrosine nitration [226]. S-nitrosylation of protein cysteine residues is believed to be the most important mechanism for transduction of bioactivity of NO in plants [227]. Several studies documented the role of RNS in regulating seed germination potential. During the first hours of post-imbibition of barley seeds, the levels of NO and the amounts of S-nitrosylated proteins increase [228, 229]. NO was shown to alleviate cell death through protein S-nitrosylation and transcriptional regulation during the aging of elm seeds [230]. Thus 80 S-nitrosylated proteins were identified, including glyceraldehyde 3-phosphate dehydrogenase, indicating that S-nitrosylation of this enzyme may accelerate cell death and seed deterioration during aging; similar results were obtained for Arabidopsis seeds [231]. It has been recently reported that elevated temperature (32 °C) inhibits Arabidopsis seed germination and triggers protein S-nitrosylation [232]. Under this temperature, the bHLH transcription factor LONG HYPOCOTYL IN FAR-RED1 (HFR1) is S-nitrosylated at Cyt164, resulting in its degradation and breakdown PIF1-HFR1 heterodimer to release PHYTOCHROME INTERACTING FACTOR 1 (PIF1) activity. PIF1 is a transcription factor targeting the expression of SOMNUS (SOM), a negative regulator of seed germination. Studies on several plant species have documented that NO breaks seed dormancy and promotes germination, notably in relation with hormone (ethylene, ABA) signalling networks [198, 212, 217, 233, 234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244].
One of the mechanisms of the crosstalk between ethylene and NO relies on activation of ethylene biosynthesis by NO, possibly through PTM of key enzymes such as ACC synthase and oxidase, by S-nitrosylation. In Arabidopsis seeds, the crosstalk of NO with ethylene signalling transcription factor EIN3 (Ethylene Insensitive 3) was shown to alleviate germination inhibition induced by high salt [239]. NO targets ABA INSENSITIVE5 (ABI5), a major regulator of ABA signalling, which illustrates the crosstalk between the NO and ABA pathways. NO negatively regulates ABI5 expression by modulating the group VII ethylene response factors (ERFVIIs) through the N-end rule pathway [245, 246]. A novel pathway, in which NO directly affects ABI5 protein stability, has been identified. KEEP ON GOING (KEG), a RING-type E3 ligase, mediates the direct regulation of ABI5 protein by NO. In this mechanism the thiol side chain (–SH) of the ABI5 cysteine 153 (Cys153) is subject to S-nitrosylation by NO, which results in the modified Cys153 with nitrosothiol (ABI5-SNO), triggering ABI5 ubiquitination by KEG and its subsequent destabilization by the 26S proteasome pathway [235]. When a mutation is introduced into the ABI5 protein to substitute Cys153 with Ser153, it does not affect the ABI5 function, such as dimer formation and ABA responsive element (ABRE) binding; however, S-nitrosylation and destabilization of ABI5 are abolished by this mutation. Thus, ABI5 serves as a NO sensor in seeds and seedlings.
9.4. Protein nitration
Protein tyrosine nitration consists of adding a nitro (–NO2) group to one of the two equivalent ortho carbons of the aromatic ring of the residues. Tyrosine nitration is considered as a selective process rather than a random one and can result in an alteration of diverse protein functions [247]. An increase in protein nitration is recognized as a suitable marker of nitro-oxidative stress that is frequently associated with oxidative stress under diverse environmental stress conditions [248]. A study carried out on sorghum embryonic axes revealed the appearance of several nitrated proteins after 24-h seed imbibition. Exogenous application of sodium nitroprussiate (SNP, a NO-donor) resulted in the mitigation of several oxidative damages but also in a significant increase in protein nitration. Consequently, nitration may be more than a biological marker of nitrosative stress and could participate in protein turnover, signal transduction and in plant hormone regulation [212, 249]. On this line, it is worth noting that NO may inhibit ABA signalling because NO-deficient plants are hypersensitive to this phytohormone [250]. The in vitro nitration of several ABA receptors such as PYR1 and PYL1 by SIN-1 has been described; the observed rapid decrease in ABA responsiveness due to PYR1 nitrated-mediated inactivation could trigger the ABA receptor degradation and thus control ABA signalling [251]. Proteomic analysis has shown that MoCo (the sulfurylated molybdenum co-factor that functions as co-factor for the abscisic aldehyde oxidase that converts abscisic aldehyde to ABA) is the target for the tyrosine nitration, inactivating ABA synthesis during germination. Thus, protein nitration might contribute to controlling the dormancy release and germination vigour [202].
10. A few conclusions and prospects
Given the importance of seed supplies to feed the world and the emergency for developing more sustainable agricultural practices (e.g. by limiting the use of synthetic chemicals), elucidating the molecular mechanisms underlying seed development, aging and germination constitutes more than ever a major scientific and societal challenge. This knowledge will be essential to guide breeding programs and develop new crop plants and varieties of agroeconomic interest designed to produce seeds with improved nutritional, agronomic orindustrial qualities, while maintaining or optimizing the general fitness and performance of crops. The successful integration of biostimulation and biocontrol strategies into plant breeding as well as technical seed production and seed enhancement technologies (such as seed coating and seed priming) is a major challenge. Old-fashioned treatments such as seed priming are currently being redesigned integrating biosolutions to improve seed vigour (i.e. rapid and synchronous germination) and to prevent damping-off by eliciting defence mechanisms during the seedling’s early establishment. These developments are necessary to protect and enhance seed and seedling performances and in order to satisfy societal and regulatory requirements that support the ban on the use of synthetic chemicals in the context of agroecological transitions.
Molecular genetic approaches with model species such as Arabidopsis remain well adapted and powerful tools for investigating these mechanisms, although they can present some limitations and drawbacks inherent to the small seed size and entanglement of the different tissues, or to limited genetic and biochemical diversity. Over the last decade, major technical advances have been made, that tackle some of these limitations. Tissue- or even cell-specific analyses can now be implemented and efficiently combined with various omic approaches and wild species can be directly investigated. In addition, efficient genetic approaches such as genome editing systems significantly facilitate cutting-edge research on crop plants and breeding for new traits of interest.
Likewise, the characterisation of seed diversity in wild and crop plants is now essential to understand what underlies species-specificities in seeds in terms of biochemical composition and resilience to adverse environmental conditions. This will contribute to the discovery of new molecules of interest for plant bio-protection, production of healthy food or renewable raw materials for bio-based industries. Advances in genome sequencing and functional genomics will facilitate the identification of key genes controlling corresponding biosynthetic pathways. However, the possible interplay existing between different metabolisms should be delt with care when trying to optimize the production of certain molecules of interest so as not to alter other seed or plant traits.
Currently, we are still far from having a complete understanding of the molecular and cellular mechanisms underlying seed development and germination, seed metabolism and physiological responses to challenging environments. Further research is required for the construction of a comprehensive and predictive spatio-temporal seed model. It will be crucial to elucidate the complex interactions existing between seed compartments, from both a signalling and a metabolic perspective. We also need to investigate seed metabolism at the cellular and intracellular levels, while integrating the influence of environmental factors.
Conflicts of interest
Authors have no conflict of interest to declare.
Acknowledgements
We wish to thank all our colleagues, technicians, engineers, students, post-docs and administrative staff who have contributed to the works presented in this review and apologize to those we haven’t been able to cite due to space limitations. We wish to warmly acknowledge the constant and strong support of Georges Pelletier and Michel Delseny to this work and for writing this review. We wish to thank Eric Postaire for correcting the review and writing the French version.
Version française
1. Introduction
La compréhension des mécanismes moléculaires qui sous-tendent le développement et le métabolisme des graines et qui permettent aux graines d’accumuler des composés de stockage, de mettre en pause la croissance de l’embryon et finalement de germer dans des conditions favorables, constitue un sujet important en biologie et en écologie végétales. Les graines ont contribué à l’immense succès des spermatophytes sur la terre (aujourd’hui, elles représentent 95 % des espèces végétales), permettant à la plupart d’entre eux de faire face à des conditions environnementales défavorables en interrompant leur cycle de vie et en reprenant leur croissance lorsqu’ils sont placés dans des conditions favorables [1]. En outre, les graines sont de la plus haute importance pour l’humanité. Elles fournissent plus de la moitié de l’apport mondial en protéines, huiles et amidon, minéraux ou vitamines, et constituent donc la principale source de nutrition humaine et d’alimentation animale [2]. Au cours des prochaines décennies, la population mondiale continuera d’augmenter et devrait atteindre environ 10 milliards d’habitants en 2050 [3]. Ce taux de croissance doit être accompagné d’une augmentation de la production alimentaire mondiale d’au moins 50 % [4]. En outre, les graines accumulent des composés carbonés de stockage tels que l’huile ou l’amidon, qui représentent une alternative aux chaînes carbonées fossiles en tant que précurseurs pour l’industrie chimique, ainsi que des composés à haute valeur ajoutée dont les applications en santé et en médecine ne cessent de se développer. Enfin, les semences constituent le principal vecteur d’amélioration des pratiques agronomiques et de gestion des ressources génétiques, deux facteurs clés pour le développement d’agricultures durables et la préservation de la biodiversité et de l’environnement. Ces différents besoins positionnent d’ores et déjà un marché mondial des semences commerciales à environ 60 milliards de dollars en 2020, et qui devrait atteindre plus de 100 milliards de dollars en 2031 (https://www.alliedmarketresearch.com/seed-market).
Michel Caboche a longtemps été un précurseur et un catalyseur de la recherche en génétique et physiologie végétales, y compris la biologie des semences [5]. Parmi les différents laboratoires qu’il a contribué à développer, le Laboratoire de Biologie des Semences créé en 1995, qui fait maintenant partie de l’Institut Jean-Pierre Bourgin (IJPB, INRAE-AgroParisTech), s’est concentré sur l’étude des mécanismes moléculaires et des gènes impliqués dans le contrôle du développement et du remplissage des graines, ainsi que dans la régulation de la dormance et de la germination des graines [6, 7, 8, 9]. L’analyse génétique du développement des graines a été initiée par l’isolement d’un grand nombre de mutants (Lepiniec et al. non publié) de la collection de mutants d’insertion d’ADN-T de Versailles [10, 11]. Cette approche génétique a conduit à la découverte d’une diversité d’acteurs impliqués dans la biosynthèse des réserves lipidiques comme l’acétyl-CoA carboxylase ACC1 [12] et la diacylglycérol acyltransférase TAG1 [13], dans la modification post-traductionnelle des protéines comme la glucosidase GCS1 [14], ou dans la régulation de la biosynthèse des flavonoïdes et la différenciation des téguments (les protéines « transparent testa » (TT)) [15, 16, 17, 18]. Ces approches génétiques, facilitées au fil du temps par la disponibilité toujours plus grande de lignées d’insertion d’ADN-T et de ressources associées, ont ouvert la voie à une étude systématique des facteurs de transcription exprimés dans les graines et de leurs réseaux de régulation qui est toujours en cours au laboratoire [19, 20].
En 2010, Michel Caboche a coordonné la préparation d’un manuscrit intitulé « Arabidopsis seed secrets unravelled after a decade of genetic and omics-driven research »[1]. L’objectif de cette revue était d’illustrer les progrès importants réalisés dans le domaine au cours de la dernière décennie et couvrait différents sujets, notamment le développement des graines (embryon, albumen et téguments), l’accumulation des réserves de graines, la dormance des graines et la germination. À cette époque, les outils « omiques » émergents, notamment l’inventaire des gènes déduit du séquençage du génome d’Arabidopsis thaliana, ont accéléré la découverte des fonctions biologiques associées aux graines. L’accès aux ressources « omiques » a permis des approches de génétique moléculaire et a attiré une grande partie de la communauté des semenciers à travailler avec l’espèce modèle Arabidopsis. Suite au décès prématuré de M. Caboche l’année dernière [5], nous avons décidé de mettre à jour cette revue en sa mémoire. Nous avons étendu la portée de l’article afin d’englober des recherches d’intérêt émergent telles que la caractérisation de la biodiversité des semences, les progrès récents dans l’analyse de la synthèse et des modifications des protéines et des métabolites spécialisés, et les études sur l’impact de l’environnement sur la qualité des semences. Les développements récents dans les approches à haut débit ont facilité la mise en œuvre d’études omiques dans un large spectre d’espèces, y compris de grandes cultures. La caractérisation de la diversité des semences chez les espèces sauvages et cultivées apparaît désormais essentielle pour comprendre ce qui fait l’originalité des semences en termes de composition biochimique, de tolérance aux conditions environnementales défavorables, de caractéristiques de germination et de compétition avec d’autres espèces. Aujourd’hui, il est possible de mener des recherches efficaces directement sur les espèces cultivées pour faire progresser les connaissances et adapter de nouveaux traits d’intérêt en utilisant des approches génétiques ciblées telles que les systèmes d’édition génomique basés sur le TILLING ou CRISPR.
2. (Gén)omique des semences
La recherche en biologie des semences a grandement bénéficié de divers outils et ressources génomiques d’Arabidopsis, notamment le séquençage du génome, la génétique inverse utilisant des bibliothèques d’insertion d’ADN-T et la bio-informatique [1]. Au cours de la dernière décennie, le développement des technologies à haut débit a conduit à l’établissement de ressources utiles pour les données (épi)génomiques, transcriptomiques ou protéomiques des plantes. Les outils bio-informatiques ont été continuellement améliorés pour faciliter l’accès et l’utilisation de ces immenses ensembles de données « omiques »[21]. Cependant, la plupart de ces approches « omiques » ont été initialement mises en œuvre sur des graines entières, qui sont composées de différents tissus (téguments, albumen et embryon), qui englobent eux-mêmes plusieurs types de cellules connues pour présenter des propriétés et des destins contrastés. En conséquence, ils sont caractérisés par des profils spécifiques d’expression génétique, de composition protéique et de contenu en métabolites. Jusqu’à très récemment, la caractérisation détaillée des types de tissus ou de cellules des graines a donc représenté un goulot d’étranglement critique pour la biologie des graines, en raison de la petite taille et de l’enchevêtrement de ces différents tissus et des inévitables contaminations croisées associées à leur dissection.
2.1. Omiques spécifiques aux tissus et aux cellules
Pour surmonter ces limitations, des outils spécifiques ont été développés, notamment le tri des cellules/noyaux par fluorescence (FACS/FANS), la purification des ribosomes par affinité (TRAP), l’isolement par marquage de noyaux de cellules spécifiques (INTACT) ou la microdissection [22, 23, 24, 25]. La microdissection assistée par laser (LAM) permet de récupérer avec précision des tissus spécifiques en fonction de leur morphologie ou de leur coloration préalable. Le couplage de la LAM à la RT-PCR quantitative ou aux puces à ADN a été réalisé avec succès avec différents tissus des graines [26, 27]. De nos jours, les technologies de séquençage de nouvelle génération (NGS) sont très sensibles et permettent la quantification de transcrits à faible abondance, même chez les espèces pour lesquelles aucune séquence génomique de référence n’est disponible. La combinaison du séquençage d’ARN à très faible abondance avec la microdissection laser est donc réalisable et a facilité des analyses précises et complètes des transcriptomes des cellules épidermiques par rapport aux cellules mésophiles dans les cotylédons des embryons d’Arabidopsis par exemple [24]. Plus récemment, le tri des noyaux marqués par fluorescence couplé au séquençage de l’ARNm de noyaux isolés a permis de construire un atlas d’expression génique pour les embryons d’Arabidopsis de stade précoce à une résolution cellulaire [28]. Néanmoins, ces techniques de plus en plus sensibles sont encore limitées par le besoin d’utiliser des protoplastes ou de passer par la transformation génétique avec des marqueurs spécifiques du type cellulaire, et par les biais liés à l’amplification des ARNm [29].
2.2. Intégration des données multi-omiques
Le développement de stratégies « multi-omiques » à haut débit, ainsi que l’évolution rapide des technologies d’analyse des données, ont amélioré notre compréhension de la biologie des semences. Les données multidimensionnelles de la -omique qui sont systématiquement étudiées dans les lots de semences par une analyse computationnelle utilisant des approches statistiques et bio-informatiques permettent l’identification de facteurs clés et des voies moléculaires, et ont conduit à la caractérisation d’un grand nombre de cibles potentielles pour les programmes de sélection ou les traitements des semences, afin d’améliorer leur qualité et leur résistance au stress. La combinaison de données multi-omiques a permis d’identifier des entités biologiques qui partagent des profils d’accumulation similaires dans différentes conditions physiologiques ou environnementales. Par exemple, des analyses approfondies des données transcriptomiques ont conduit à l’identification de groupes de gènes co-exprimés et impliqués dans des processus similaires tels que la physiologie des semences ou les réponses à des conditions environnementales défavorables. Un réseau spécifique aux graines, SeedNet (http://vseed.nottingham.ac.uk), a été développé pour fournir des modules de gènes associés à la dormance et à la germination des graines d’Arabidopsis [30, 31, 32]. Néanmoins, certaines études ont mis en évidence un défaut de corrélation entre l’abondance de l’ARNm et la synthèse des protéines, soulignant qu’une part importante des ARNm est traduite pendant la germination des graines [33]. Cette observation a été confirmée par des études récentes sur la régulation de la traduction dans les graines d’Arabidopsis par le profilage des polysomes [34, 35, 36]. En raison des régulations post-transcriptionnelles (par exemple, l’épissage ou l’édition) et post-traductionnelles (par exemple, la protéolyse ou les modifications post-traductionnelles, PTM), un seul gène peut donner naissance à une diversité d’isoformes de protéines qui peuvent différer par leur fonction, leur stabilité, leur localisation et leurs interactions (voir ci-après). Par exemple, des travaux récents basés sur les interactions protéine–protéine ont mis en évidence cette diversification fonctionnelle des protéines dans le système de signalisation de l’acide abscissique (ABA) [37], comme détaillé ci-après.
Outre les approches génomiques, transcriptomiques et protéomiques, l’amélioration de la sensibilité de la métabolomique ciblée et non ciblée et de l’analyse des données a permis de mieux caractériser l’état physiologique et la qualité des graines. La métabolomique, l’étude à grande échelle des métabolites primaires et spécialisés (SMs), est considérée comme la technique omique la plus proche du phénotype [38]. Au cours des dernières années, nous avons assisté à une augmentation du nombre d’études métabolomiques non ciblées et, dans une moindre mesure, ciblées, axées sur les acides gras et les lipides des graines, ainsi que sur la diversité et la régulation des acides aminés libres, en utilisant la métabolomique seule ou combinée avec d’autres techniques omiques [39, 40, 41, 42].
3. Développement de la graine
Le développement des graines peut être divisé en deux phases successives nommées embryogenèse et maturation [1, 43, 44]. Chez les angiospermes, la « double » fécondation du sac embryonnaire déclenche le développement de trois tissus génétiquement distincts comprenant les graines, les téguments (ou « testa ») d’origine maternelle provenant des deux téguments de l’ovule, l’albumen triploïde et l’embryon diploïde [1]. Au cours des dernières années, il a été démontré que la fécondation supprime le contrôle épigénétique négatif exercé par les protéines PcG (Polycomb Group) sur le développement de la graine [45]. La différenciation de l’enveloppe de la graine est initiée par la fécondation de l’ovule. L’albumen se développe avec une phase syncytiale suivie d’événements de cellularisation et de différenciation. Le zygote se divise en une cellule apicale et une cellule basale qui se divisent à leur tour et donnent naissance à un embryon et à son suspenseur, respectivement. Au stade dit « cœur », les principales structures de l’embryon d’Arabidopsis (c’est-à-dire les cotylédons, le méristème apical, l’hypocotyle et la racine embryonnaire) sont déjà établies, les divisions cellulaires commencent à s’atténuer et le programme de maturation est déclenché, ce qui entraîne l’accumulation de composés de stockage, la dessiccation des graines et l’acquisition de la dormance. La signalisation de l’auxine joue un rôle majeur pendant l’embryogenèse, tandis que l’ABA régule de multiples aspects de la maturation des graines, y compris l’accumulation des réserves et la dormance des graines (voir ci-dessous).
3.1. Croissance coordonnée de l’enveloppe de la graine, de l’albumen et de l’embryon
Le bon développement de la graine nécessite la croissance coordonnée des téguments, de l’albumen et de l’embryon et repose donc sur la communication entre ces différents compartiments [1, 46]. Par exemple, une croissance anormale des téguments affecte le développement de l’albumen et réduit la taille de la graine. Dans le même ordre d’idées, l’albumen fournit des signaux critiques pour le développement de l’embryon. Certains peptides riches en cystéine (EMBRYO SURROUNDING FACTOR, ESF) présents dans l’albumen sont impliqués dans le développement du suspenseur [47]. Récemment, il a été démontré qu’une voie de signalisation médiée par les peptides se déroule entre l’embryon et l’albumen et contrôle la formation de la cuticule de l’embryon, une couche protectrice de lipides de surface déposée au-dessus de son épiderme [48]. Le peptide TWISTED SEED1 (TWS1), produit par l’embryon [49], est activé par une subtilase de l’albumen, ce qui entraîne le renforcement et la protection de la cuticule de l’embryon [48]. Un autre exemple de ces interactions est la médiation du développement antagoniste du nucelle (tissu maternel) et de l’albumen par les facteurs de transcription MADS TRANSPARENT TESTA16 (TT16) et son paralogue GORDITA [50].
3.2. Le réseau LAFL et le contrôle du développement et de la maturation des graines
Les régulateurs transcriptionnels LAFL, à savoir LEC1 (LEAFY COTYLEDON1), LEC2 (LEAFY COTYLEDON2), ABI3 (ABSCISIC ACID INSENSITIVE3), et FUS3 (FUSCA3) chez Arabidopsis, sont nécessaires à la formation correcte des graines et semblent être conservés chez les angiospermes (Figure 1) [51]. LEC1 est un membre de la famille des protéines NF-YB (NF-YB9, sous unité HAP3 des facteurs de liaison CCAAT-box). ABI3, FUS3 et LEC2 appartiennent à une famille de facteurs de transcription (TFs) à domaine B3 spécifique aux plantes et sont donc également appelés régulateurs « AFL-B3 ». LEC1 et LEC2 sont les régulateurs maîtres du réseau, mais les quatre LAFL ont des fonctions partiellement redondantes et peuvent interagir pour former différents complexes régulateurs [52, 53]. Ces complexes contrôlent l’expression de gènes de structure ou régulateurs impliqués dans différentes voies, déclenchant et maintenant le développement et la maturation de l’embryon et réprimant la germination des graines [51, 54]. De nombreux gènes spécifiques aux graines qui participent à l’accumulation des composés de stockage, y compris ceux qui codent pour les oléosines, les globulines (par exemple la cruciférine 12S) et les albumines (par exemple les arabines 2S) par exemple, sont des cibles directes des LAFL [52]. De plus, plusieurs autres TFs agissent en aval ou en parallèle aux LAFLs pendant la maturation comme AGL15, ABI4, et différents bZIPs [51]. Un autre exemple est WRINKLED1 (WRI1), un membre de la famille des facteurs de transcription AP2/EREB, qui contrôle les voies de biosynthèse des acides gras en aval des LAFL [55]. L’activité de WRI1 est également régulée au niveau post-traductionnel [56]. Récemment, il a été démontré que WRI1 interagit avec BLISTER pour réguler la dynamique de la chromatine au niveau des promoteurs cibles [57]. Enfin, l’expression ectopique de LEC1 ou LEC2 dans les tissus végétatifs peut déclencher la formation d’embryons somatiques, ce qui implique que ces TF sont suffisants pour conférer une identité embryonnaire à des cellules. Conformément aux fonctions clés des gènes LAFL dans l’induction et le contrôle du développement de la graine, leur expression est étroitement contrôlée par de nombreux mécanismes transcriptionnels et chromatiniens, comme décrit plus en détail ci-dessous (Figure 1), tandis que les protéines LAFL sont soumises à des régulations post-traductionnelles [19, 58, 59].
Le réseau LAFL contrôle le développement et la maturation des graines. Les régulateurs maîtres LEC1 et LEC2 contrôlent à la fois le développement de l’embryon et la maturation de la graine. Les régulateurs B3, à savoir LEC2, FUS3 et ABI3 ont une fonction partiellement redondante dans le contrôle de la maturation des graines et l’acquisition de la tolérance à la dessiccation. Ils contrôlent directement et indirectement (par le biais d’autres régulateurs tels que WRI1 et différents bZIPs), l’expression de gènes impliqués dans ces processus. Le réseau de régulation des LAFLs et donc, le programme de développement des graines, est fortement réprimé au cours du développement végétatif par diverses régulations épigénétiques dont les complexes PRC1/PRC2. Enfin, les LAFLs induiraient indirectement leur propre répression à la fin de la phase de maturation.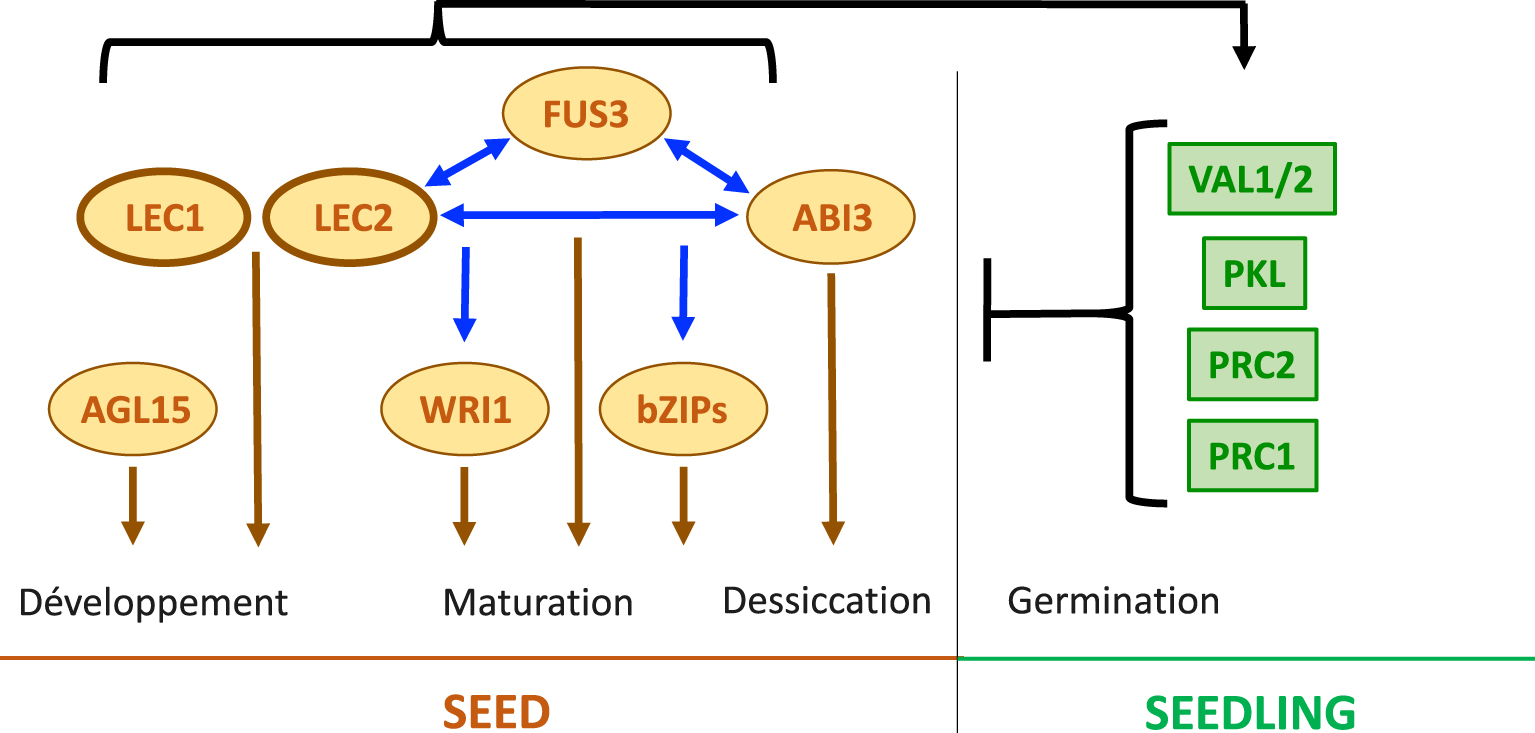
3.3. Les LAFL et les régulations chromatiniennes
Les ARNm LAFL sont détectés tôt au cours du développement de l’embryon d’Arabidopsis, l’expression de LEC1 et LEC2 atteignant un pic au stade du cœur du développement de l’embryon, tandis que ABI3 et FUS3 atteignent un pic environ une semaine plus tard, pendant la maturation [51]. Le contrôle hormonal du fonctionnement des LAFL est principalement exercé par l’auxine et l’ABA, agissant comme régulateurs positifs de l’embryogenèse et de la maturation, respectivement. En outre, les LAFL sont impliqués dans des boucles de rétroaction positives, ce qui est cohérent avec l’existence d’un seuil minimum de TFs LAFL « B3 » nécessaire à leur action [1]. L’importance de ces régulations est illustrée par la caractérisation fonctionnelle de plusieurs éléments cis-régulateurs positifs et négatifs dans les régions promotrices des gènes LEC2, ABI3 et FUS3 [19, 60, 61]. Une multitude de résultats ont démontré que les gènes LAFL sont réprimés dans les tissus végétatifs par de multiples voies, y compris des mécanismes chromatiniens avec les COMPLEXES REPRESSIFS POLYCOMBS 1 et 2 (PRC1 et PRC2) (Figure 1). PRC2 est une histone H3 lysine 27 méthyltransférase (H3K27me3) conservée au cours de l’évolution, tandis que PRC1 effectue la mono-ubiquitination de H2AK121 (H2AK121ub) [62]. Plusieurs résultats suggèrent que les LAFL peuvent agir non seulement comme des activateurs transcriptionnels directs, mais aussi comme des « facteurs pionniers », qui modifient de façon stable le paysage chromatinien en recrutant différents régulateurs [19, 63, 64, 65]. Ce rôle a été confirmé pour l’activation du gène FLOWERING LOCUS C (FLC) au cours du développement embryonnaire précoce [66, 67]. Les LAFL antagonisent VIVIPAROUS1/ABI3-LIKE1 (VAL1) et VAL2, deux TFs également de type B3 impliqués dans le recrutement des PRC au locus FLC [68, 69, 70, 71] ou le facteur de remodelage de la chromatine CHD3 PICKLE aux locus ABI3 et AGL15, pour réprimer la maturation des graines [72]. Il est intéressant de noter que FLC est impliqué dans la modulation de la dormance des graines en réponse à la température [73, 74].
De plus, LEC1 et FUS3 contrôlent également l’expression de PKL, VAL1, et RING1b (codant pour une sous-unité de PRC1) pendant la maturation tardive, qui réprime l’expression des gènes LAFL [75]. Dans cette lignée, LEC1 induit également l’expression de SEED DORMANCY 4-LIKE (SDR4L) conduisant à sa propre répression [76]. Ces derniers résultats suggèrent que les gènes LAFL pourraient avoir un rôle essentiel dans l’induction de la régulation par rétroaction négative de leur propre expression à la fin de la phase de maturation. Les modifications de la chromatine ainsi que les régulations transcriptionnelles exercées par les LAFL sont donc essentielles pour contrôler les transitions entre les différentes phases du développement de la graine, y compris la maturation de la graine.
4. Différenciation et fonctions de l’enveloppe de la graine
Les téguments maternels ou testa jouent un rôle essentiel dans la protection de l’albumen et de l’embryon contre les conditions environnementales défavorables pendant le stockage des graines sèches matures, et comme barrière physico-chimique régulant la germination et la dispersion des graines. Pour remplir ces fonctions importantes, les téguments d’Arabidopsis accumulent une batterie de métabolites spécialisés (SMs) tels que les flavonoïdes, des polysaccharides (mucilage) et des polyesters lipidiques, à travers des voies métaboliques qui sont étroitement régulées au niveau spatio-temporel [18, 77, 78] (Figure 2). Comme présenté ci-dessus, la différenciation des téguments est initiée par la fécondation de l’ovule [18], avec l’auxine produite par la cellule centrale fertilisée et exportée vers les tissus maternels libérant le blocage épigénétique exercé par les régulateurs du groupe Polycomb (PcG) [79]. Les téguments d’Arabidopsis comprennent cinq couches cellulaires superposées et entièrement développées qui subissent une spécialisation et une accumulation différentielle de SMs [18, 80]. La différenciation des téguments est achevée avant la fin de la maturation de l’embryon, les cellules subissant une mort cellulaire programmée, de sorte que les téguments matures sont un tissu mort.
Différenciation coordonnée des couches cellulaires accumulant les proanthocyanidines et les polysaccharides de mucilage dans le développement des téguments de la graine d’Arabidopsis. (A) Des réseaux de régulation transcriptionnelle simplifiés contrôlant la biosynthèse spatio-temporelle des proanthocyanidines (PCs) et du mucilage sont présentés. Les étapes du trafic vers les destinations subcellulaires finales, la vacuole et l’apoplaste respectivement, sont également présentées. Les protéines présentées dans le texte principal sont indiquées par leur abréviation en lettres majuscules roses et les noms des mutants correspondants en lettres minuscules bleues. Les flèches pointillées indiquent les étapes multiples. (B) Localisation histologique des proanthocyanines et du mucilage dans les téguments en développement. A gauche, représentation schématique d’une section de graine en développement détaillant les couches cellulaires tégumentaires. A droite, section de graine en développement avec du bleu de toluidine O colorant les proanthocyanidines (bleu) et le mucilage (rose), correspondant aux couleurs indiquées sur les couches cellulaires des téguments les plus internes et les plus externes de la représentation schématique, respectivement.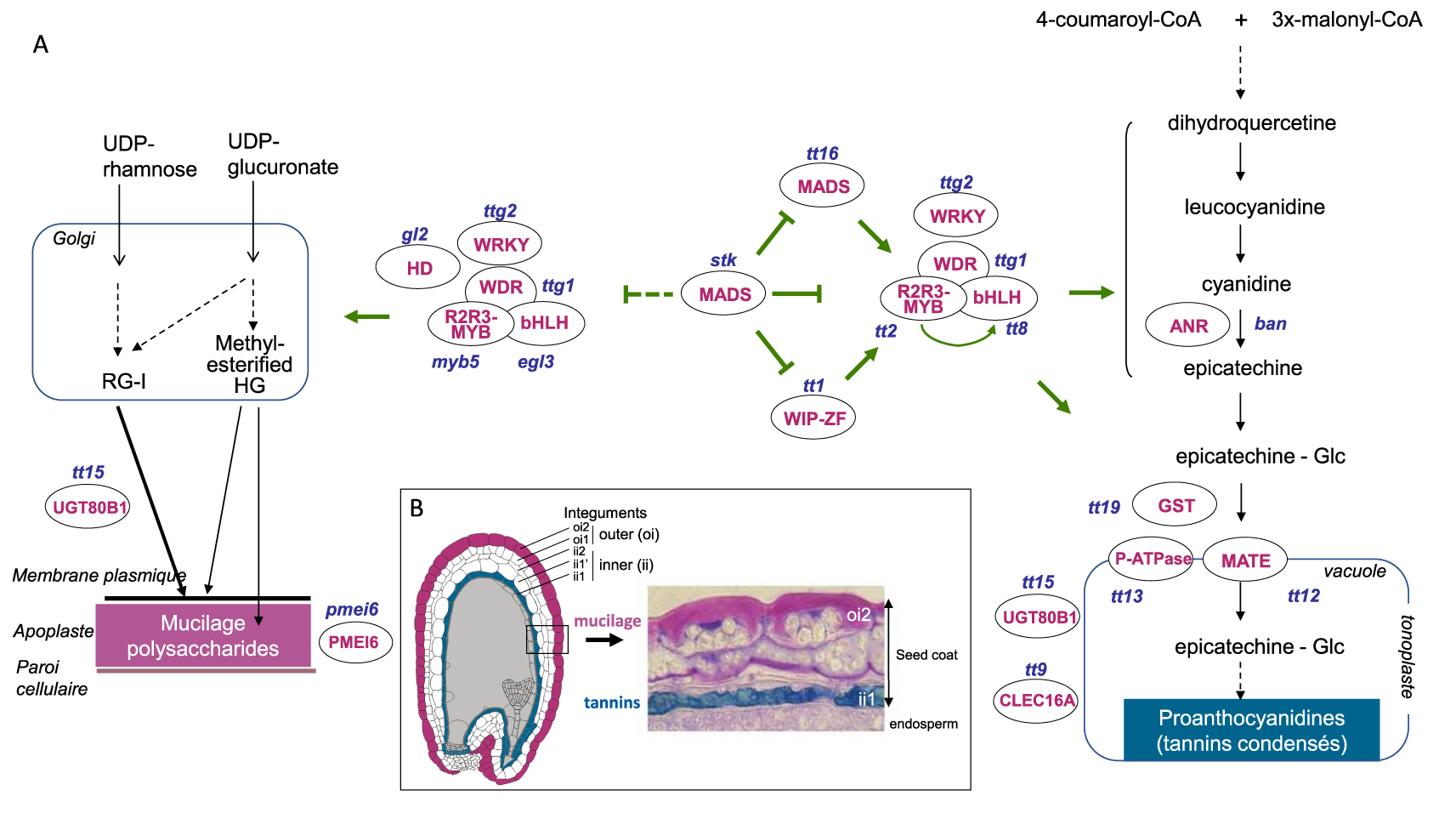
4.1. Biosynthèse des flavonoïdes
Dans les téguments d’Arabidopsis, les flavonoïdes sont constitués de proanthocyanidines (tanins condensés) de type procyanidines (PC) et de flavonols comprenant la quercétine 3-O-rhamnoside, le kaempférol-rhamnoside et l’isorhamnétine-rhamnoside [81]. Les PC et leurs monomères flavan-3-ol épicatéchine (EC) sont accumulés essentiellement dans la couche cellulaire la plus interne du tégument interne (également appelée endothélium), et dans quelques cellules du micropyle et de la chalaze. Ensuite, les téguments subissent une mort cellulaire programmée [18]. Alors que les flavonols peuvent être détectés dans l’ensemble des téguments de la graine, ils sont préférentiellement accumulés dans la couche cellulaire sous-épidermique du tégument externe. Les PCs sont présents spécifiquement dans la testa, alors que les flavonols sont également présents dans l’albumen et l’embryon [82].
Il est maintenant bien établi que la biosynthèse des flavonoïdes est catalysée par un métabolon enzymatique ancré à la face externe du réticulum endoplasmique. En revanche, les mécanismes de trafic par lesquels les divers produits finaux sont transportés vers leur destination subcellulaire finale n’ont pas été entièrement élucidés [83, 84]. De nombreux progrès dans ce domaine ont bénéficié de l’isolement et de la caractérisation fonctionnelle de mutants d’Arabidopsis affectés dans le métabolisme des PC, collectivement appelés mutants « transparent testa » (tt) en raison de la pigmentation modifiée des téguments [85]. Les PC sont initialement stockés dans la vacuole sous forme de composés incolores (Figure 2). Lors de la dessiccation de la graine, leur solubilité diminue en raison de l’oxydation par une polyphénol oxydase, la laccase TT10, ce qui colore en brun les téguments de la graine de type sauvage [18, 82, 86]. Un modèle actuel de la compartimentation vacuolaire des PC prévoit que les monomères d’EC sont glucosylés et que le l’EC-Glucose est transloquée dans la lumière de la vacuole par un transporteur secondaire TT12 MATE (Multidrug and Toxic Compound Extrusion) situé au niveau du tonoplaste [87, 88]. L’activation de TT12 serait assurée par la P3A-ATPase tonoplastique TT13 [89]. La glutathion S-transférase (GST) TT19 agirait alors comme une ligandine pour protéger l’EC-Glucose de la dégradation dans le cytosol pendant son transit via TT12 [90, 91]. De plus, la caractérisation moléculaire de TT9 comme une protéine putative de type C Lectin 16A-like (CLEC16A-like), qui appartient au complexe HOPS (HOmotypic fusion and Protein Sorting) impliqué dans la fusion vacuolaire, suggère fortement que le trafic vésiculaire est également impliqué dans la compartimentation vacuolaire du PC [92]. La façon dont les voies de transport/ligandine et de trafic vésiculaire coopèrent pour remplir cette fonction reste à établir. Il est intéressant de noter que TT9 est également impliquée dans le trafic des protéines de stockage des graines (SSP) [92]. Il est donc probable que la PC et les SSP partagent une partie de leur route de trafic vers la vacuole. Comme l’ont supposé Dixon et Sarnala [84], le système endomembranaire peut également détenir la clé du mystère de la condensation des monomères de flavan-3-ol, dont le mécanisme (enzymatique ou non) et le site subcellulaire ne sont toujours pas clairs.
4.2. Interaction entre la fonction et la production des polymères des téguments
Lors de la mort des cellules des téguments, les PA vacuolaires se lient aux composants de la paroi cellulaire et diffusent également dans la cuticule endothéliale, augmentant ainsi l’imperméabilité des téguments [93]. Comme cette cuticule est adjacente à l’albumen, elle diminue indirectement la perméabilité de l’albumen et renforce ainsi la dormance des graines [94]. La subérine déposée dans la couche cellulaire de l’épiderme de l’enveloppe et du hile a des effets similaires sur la perméabilité et la physiologie des graines [95]. La caractérisation de mutants tt15 affectés dans une stérol 3-β-glucosyltransférase a révélé que la composition des stérols glucosides est importante pour l’accumulation de PC, de cutine et de subérine [94, 96, 97], mais également pour l’accumulation de mucilage et de polysaccharides de la paroi dans les cellules de la couche externe des téguments de la graine [98]. Il a également été démontré que l’accumulation excessive de stérols a un impact sur la différenciation des téguments [99]. Enfin, les TFs TT1/WIP-Zinc Finger et TT16/MADS déterminent à la fois l’identité de l’endothélium, et donc indirectement le métabolisme des PA, et le dépôt de la cuticule endothéliale [100].
4.3. Régulation transcriptionnelle de l’accumulation des flavonoïdes et du mucilage dans les téguments des graines
Chez Arabidopsis, les protéines R2R3-MYB TT2 et MYB5 activent spécifiquement la biosynthèse des PA et du mucilage, respectivement, par leur interaction avec les protéines bHLH TT8 ou EGL3 et la protéine WDR TTG1, formant ainsi des complexes MBW similaires (Figure 2A) [20, 85]. Au cours des dernières décennies, les rôles clés des protéines MYB, bHLH et WDR conservées ont été caractérisés et constituent maintenant l’un des systèmes de régulation transcriptionnelle les mieux décrits chez les plantes [20, et références internes]. Les interactions directes ou indirectes de TTG1 avec TTG2/WRKY ajoutent un niveau de complexité supplémentaire à ce réseau de régulation [101]. De plus, SEEDSTICK (STK), une protéine de type MADS, régule négativement non seulement TT16 et le complexe MBW mais aussi directement BANYULS (BAN), le gène maître de la voie PA codant pour l’anthocyanidine réductase (ANR) qui catalyse la formation d’épicatéchine à partir de cyanidine [102]. Dans les cellules épidermiques de l’enveloppe, STK induit directement l’expression d’un inhibiteur de la pectine méthylestérase, PMEI6, qui joue un rôle clé dans la modification des propriétés de la paroi cellulaire la plus externe pour faciliter la libération des polysaccharides du mucilage [103]. Outre le développement, des signaux environnementaux tels que la température à laquelle les graines en développement sont exposées sur la plante mère peuvent moduler le dépôt de PC soluble et de quercétine (flavonol) [104, 105] et la spécificité tissulaire de facteurs de transcription tels que TT2 et MYB5 [106].
5. Composés de stockage des graines
Les graines accumulent des quantités importantes de composés de stockage pendant la phase de maturation. Selon l’espèce, il peut s’agir d’amidon, de β-glucanes, de lipides de stockage (triacylglycérols ou cires) ou de protéines de stockage des graines (SSP). Dans la plante modèle Arabidopsis comme chez la plupart des espèces de Brassicaceae, l’embryon constitue le site principal de dépôt des réserves, mais un mince albumen persiste dans les graines matures, qui contient également des composés de réserve. Dans ces graines, les SSP et les triacylglycérols représentent les principaux composés de réserve, chaque type de composé comptant pour environ un tiers du poids sec des graines matures. Les graines étant pour la plupart hétérotrophes (malgré un verdissement transitoire de l’embryon observé au début de la maturation), la synthèse des composés de réserve repose sur l’importation de nutriments de la plante mère, sous forme de sucres (principalement du saccharose), d’acides aminés, de macroéléments (par exemple, le phosphate inorganique) et de micro-éléments tels que le fer ou le zinc. La majeure partie des nutriments atteint la graine par le phloème. Dans les graines d’Arabidopsis, qui se développent à partir d’ovules anatropes, les faisceaux vasculaires se terminent à l’extrémité du funicule dans les téguments au niveau de la chalaze. Alors que le phloème est relié de manière symplastique aux téguments externes, les tissus zygotiques semblent être isolés de manière symplastique. Cet isolement symplastique entre les tissus de la graine implique que les nutriments doivent être transportés à travers les barrières apoplastiques pour être finalement délivrés à l’embryon en maturation (Figure 3A). Alors que l’anatomie des graines d’Arabidopsis est bien connue depuis des décennies, notre compréhension du déchargement du phloème et du transport des nutriments n’a progressé que plus récemment grâce à la caractérisation d’une série de transporteurs impliqués dans l’exportation vers l’apoplaste ou dans la réimportation ultérieure dans un espace symplastique adjacent [107]. Le transport du saccharose implique des membres des familles de protéines SWEET [108] et SUC (SUCROSE-PROTON SYMPORTER) [109], l’exportation des acides aminés à partir des téguments de la graine nécessite des symporteurs de la famille UMAMIT (USUALLY MULTIPLE ACID MOVE IN AND OUT TRANSPORTER) [110], tandis que les micro-éléments sont exportés par différents types de transporteurs spécialisés [107, 111].
Biosynthèse des composés de stockage dans les graines d’Arabidopsis en cours de maturation. (A) Importation des nutriments de la plante mère par le système vasculaire et les téguments de la graine. L’apport de saccharose, d’acides aminés et de micro- et macro-éléments (ions) à l’embryon en maturation implique à la fois un transport symplastique dans les tissus maternels de la graine et des transporteurs spécialisés (par exemple SUC, SWEET, UMAMIT) pour le transport à travers les frontières apoplastiques séparant les différents tissus de la graine. (B) Aperçu simplifié du métabolisme associé à la biosynthèse des composés de stockage dans les cellules embryonnaires. AA, acide aminé; AcCoA, acétyl-CoA; FA, acide gras (C16 et C18) ; Gln, glutamine ; ii, tégument interne; MaCoA, malonyl-CoA; oi, tégument externe; SSP, protéine de stockage de la graine; SUC, saccharose; TAG, triacylglycérol; TCA, cycle de l’acide tricarboxylique; VLCFA, acide gras à très longue chaîne (C > 20).
5.1. Acides gras et lipides
Dans les tissus zygotiques, les hexoses libérés par le clivage du saccharose sont convertis en hexose-phosphates qui peuvent être métabolisés par différentes voies en fonction du type de composés stockés dans les graines [112]. Dans les graines oléagineuses d’Arabidopsis, les produits finaux de la glycolyse sont ensuite métabolisés en molécules d’acétyl-CoA et de malonyl-CoA qui sont utilisées pour la synthèse de novo des acides gras dans les plastes (Figure 3B). Un complexe d’acides gras synthase de type II produit et allonge les chaînes acyles saturées liées aux protéines porteuses d’acyle (PPA). Les chaînes acyles 16:0 et 18:0 sont libérées de ce complexe et peuvent être désaturées par les acyl-ACP désaturases (AAD) stromales pour former des acides gras monoinsaturés [113]. Après hydrolyse des groupes acyles par les thioestérases, les acides gras libres peuvent être exportés vers le cytosol et activés en esters de CoA. Dans le réticulum endoplasmique, les acyl-CoAs gras à longue chaîne peuvent être encore allongés par le complexe d’acides gras élongase lié à la membrane. Ce complexe enzymatique procède par des réactions analogues à celles de la synthèse de novo des acides gras et produit des acides gras à très longue chaîne comprenant jusqu’à 24 atomes de carbone. Les enzymes responsables de la biosynthèse des acides gras polyinsaturés que l’on trouve dans les huiles de graines sont les désaturases microsomales des acides gras FAD2 et FAD3, qui transforment respectivement le 18:1 en 18:2 et le 18:2 en 18:3, ces acides gras pouvant à leur tour être estérifiés en phosphatidylcholine [114]. Les triacylglycérols sont composés de trois chaînes d’acyle gras estérifiées sur un squelette de glycérol. Leur biosynthèse nécessite donc l’acylation séquentielle du glycérol-3-phosphate, qui est dérivé des intermédiaires glycolytiques, par les acyltransférases de la voie de Kennedy. Cependant, au cours de la dernière décennie, des études ont révélé que l’incorporation des acides gras dans les triacylglycérols est beaucoup plus complexe qu’une simple voie linéaire et implique divers échanges de chaînes acyles entre différents pools lipidiques [115]. Une fois synthétisés, les triacylglycérols sont séquestrés dans des organites appelés gouttelettes lipidiques (LD) formées d’un noyau de triacylglycérols entouré d’une monocouche de phospholipides. Différentes catégories de protéines spécialisées sont intégrées dans la demi-membrane des gouttelettes lipidiques et limitent leur coalescence pendant le stockage ou facilitent la dégradation de l’huile après la germination des graines. Les protéines structurelles associées aux gouttelettes lipidiques comprennent les oléosines, des enzymes moins abondantes et les aquaporines, entre autres [116].
5.2. Protéines de stockage de la graine
Les SSP synthétisées dans les tissus zygotiques représentent une source de carbone (C), d’azote (N) et de soufre (S). Chez Arabidopsis, comme chez d’autres espèces de Brassicaceae, les SSP prédominantes sont les globulines 12S (également appelées cruciférines) et les albumines 2S (parfois appelées arabines). Elles représentent 84 % des espèces de protéines dans ces graines et sont stockées dans les vacuoles de stockage des protéines (PSV) [117]. Les protéines moins abondantes stockées dans d’autres compartiments cellulaires comprennent les oléosines et les protéines LEA (late embryogenesis-abundant). Les SSP sont initialement synthétisées sous forme de longs précurseurs dans le réticulum endoplasmique rugueux à partir d’acides aminés directement importés de la plante mère ou obtenues par des réactions de transamination ayant lieu dans les tissus zygotiques. Ces précurseurs subissent ensuite un clivage post-traductionnel séquentiel catalysé par des enzymes de transformation vacuolaire (EPV) lors de leur transit ou au niveau des PSV [118]. Le clivage est nécessaire pour libérer les sous-unités α et β composant de nombreuses SSP avant que des liaisons disulfures intra et inter-chaînes ne soient établies entre ces sous-unités pour former des complexes ayant une structure tridimensionnelle stable et compacte. De plus, des troncatures limitées se produisant successivement dans la région désordonnée C-terminale des globulines 12S ont également été rapportées pour produire une variété de polypeptides tous dérivés de la même sous-unité. Bien que la maturation et le trafic des SSP puissent différer selon les espèces végétales, les tissus et les stades de développement, ces processus reposent tous sur un système endomembranaire hautement dynamique et fonctionnellement spécialisé et impliquent le complexe rétromère [119, 120, 121]. Le stockage des SSP semble également être très variable. En outre, il a été démontré que les mutations affectant la production d’une classe donnée de protéines de stockage étaient compensées par une accumulation accrue d’autres protéines de la graine, tandis que la surexpression de transgènes codant pour les SSP dans les graines produisait des quantités inchangées de protéines totales de la graine [122]. Ensemble, ces résultats suggèrent que l’approvisionnement en N plutôt que le métabolisme des SSP régule la teneur totale en N des graines. De plus, les PSV ne stockent pas seulement des réserves de protéines, ces compartiments vacuolaires délimités par une bicouche lipidique contiennent également des minéraux. Des inclusions intra-organellaires appelées globoïdes peuvent être observées dans les PSV et sont constituées de phytine, un sel de l’acide phytique, et de cations tels que K+, Mg2+ et Ca2+ [123]. Le fer des graines peut également être stocké dans les globoïdes des PSV ou, dans une moindre mesure, dans les ferritines localisées dans les plastes qui sont capables de stocker le fer sous une forme non toxique [124].
5.3. Répartition des réserves entre les compartiments de la graine
Alors que la diversité des composés de stockage des graines entre les espèces est documentée depuis longtemps, l’amélioration continue des techniques analytiques permet des études utilisant des échantillons plus petits et l’émergence récente des instruments d’imagerie par spectrométrie de masse a conduit à une caractérisation plus détaillée des tissus, révélant une hétérogénéité de la composition des réserves entre les deux tissus zygotiques de la graine, et même entre les différentes régions de l’embryon [125, 126]. Dans l’albumen des graines d’Arabidopsis par exemple, les globulines 12S sont sous-représentées dans la fraction protéique [127] tandis que les triacylglycérols apparaissent très riches en acides gras ω-7 mono-insaturés inhabituels qui sont à la limite de la détection dans l’embryon [128]. Ces résultats invitent à approfondir maintenant le métabolisme des graines à une échelle tissulaire plus faible, tout en intégrant l’influence des facteurs environnementaux (voir ci-dessous).
6. Métabolites spécialisés (SMs) des graines
Les graines accumulent une large gamme de métabolites spécialisés dont certains sont spécifiques par rapport aux racines, feuilles ou fleurs [129, 130, 131, 132, 133]. Les SMs, qui étaient auparavant définis comme des « métabolites secondaires », sont en fait largement impliqués dans les interactions des plantes avec leur environnement et ont des fonctions développementales ou physiologiques spécifiques et importantes dans les graines (Figure 4) [134, 135].
Régulation environnementale du métabolome de la graine. La régulation environnementale des métabolites spécialisés des graines et des composés de réserve. L’impact des stress abiotiques et biotiques sur le métabolisme primaire (protéines, lipides et acides gras, rapport C/N) et spécialisé est illustré. Légende : FA, acides gras ; SSP, protéines de stockage des graines.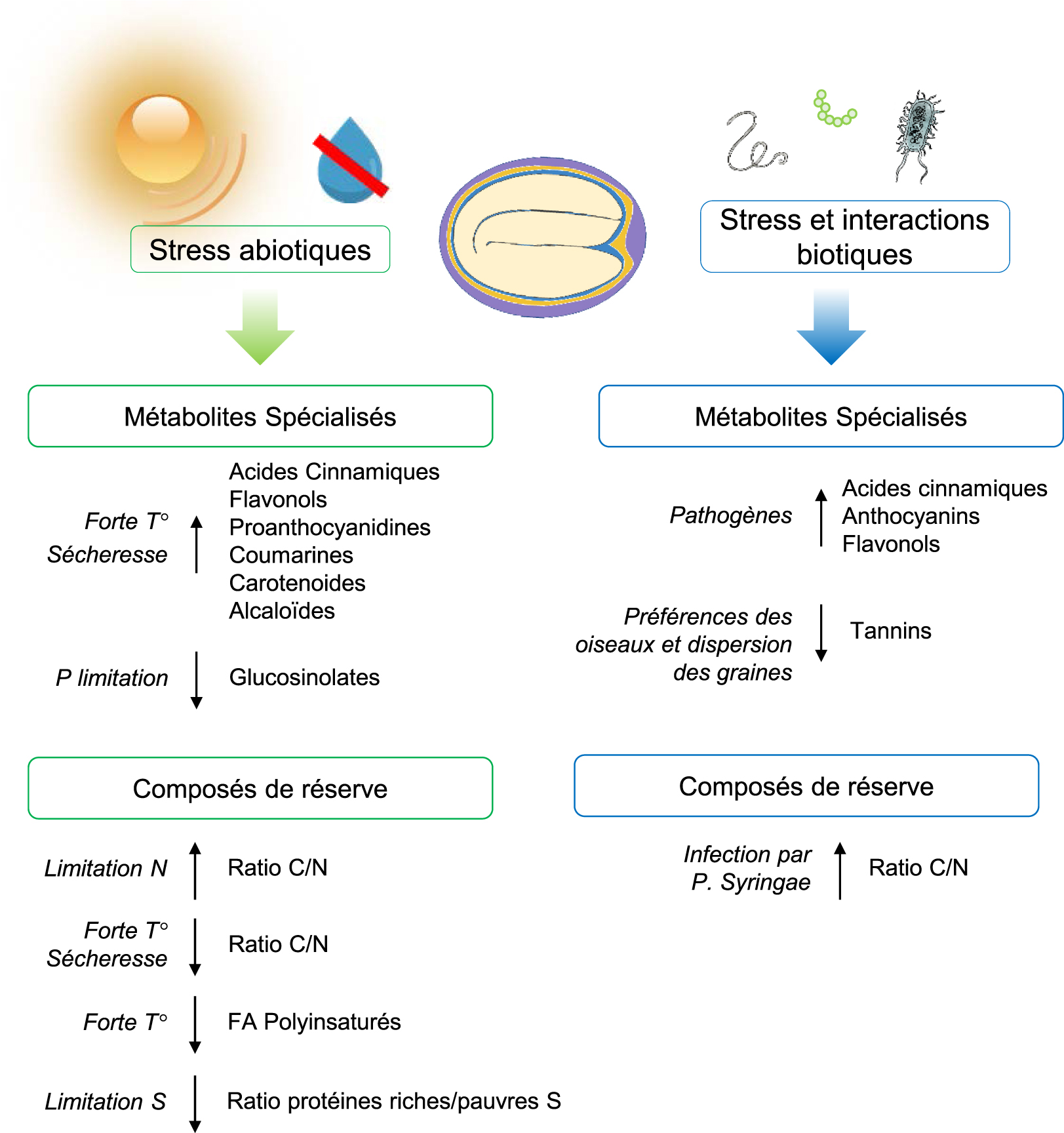
6.1. Fonctions des métabolites spécialisés des graines
De nombreux flavonoïdes (pour plus de détails, voir le paragraphe ci-dessus sur la différenciation des téguments), des acides cinnamiques et des coumarines constituent la première couche de défense des graines contre les attaques de pathogènes [135]. De plus, ces SMs ont une forte activité antioxydante et protègent les téguments et l’embryon des stress abiotiques, tels que la dessiccation, les hautes températures ou les radiations UV qui se produisent pendant la maturation et/ou le vieillissement des graines [136]. En plus de leur rôle contre les stress abiotiques et biotiques, les flavonoïdes des graines jouent également un rôle dans la régulation de la longévité, de la dormance et de la germination des graines, ce qui a un impact sur la vigueur des semis et le rendement des cultures [137]. Une dormance réduite des graines a été observée chez des mutants d’Arabidopsis pour les gènes de biosynthèse des flavonoïdes [105, 138]. Enfin, les graines constituent une source importante de diversité de métabolites pour l’alimentation et de nombreux SMs sont des composés sains ou antinutritionnels importants [85, 135]. Dans un contexte global de changement climatique et de demande pour le développement d’une agriculture plus durable, ces fonctions ont conduit à un intérêt accru pour les SMs pour des applications agroécologiques allant du contrôle durable des ravageurs à la qualité des aliments.
6.2. Biosynthèse et diversité des SMs
Les SMs peuvent être divisées en trois classes principales : les terpènes (plus de 40 000 composés), les alcaloïdes et les molécules azotées (environ 20 000 composés), et les composés phénoliques ou phénylpropanoïdes (plus de 10 000 composés) [135]. Les phénylpropanoïdes sont synthétisés à partir des voies du shikimate et du malonate et sont caractérisés par la présence d’un ou plusieurs cycles aromatiques hydroxylés, et de multiples décorations potentielles (c’est-à-dire hydroxylation, glycosylation, acylation, méthylation ou prénylation). Les phénylpropanoïdes les plus abondants dans les graines appartiennent aux classes des anthocyanines, des flavonols, des flavan-3-ols et des proanthocyanidines (PAs ; tannins condensés, qui sont des polymères de flavan-3-ols). Les tanins s’accumulent préférentiellement dans l’enveloppe des graines, ce qui a probablement été un trait avantageux au cours de l’évolution car il limite la consommation des graines par les animaux avant leur dispersion et leur germination [40]. Les terpénoïdes des graines sont les précurseurs de plusieurs métabolites clés, tels que les gibbérellines et les tocophérols (vitamine E). Les terpénoïdes sont classés en fonction du nombre d’unités isoprènes (C5H8). On peut citer la carvone et le linalool de Carum carvi et/ou Coriandrum sativum (monoterpènes), l’artémisinine de Taxus brevifolia (sesquiterpène), les ginkgolides de Ginkgo biloba et (diterpènes), et la saponine de Medicago truncatula (triterpène) [129, 139]. En plus des phénylpropanoïdes et des terpénoïdes, les alcaloïdes et les composés contenant de l’azote sont accumulés à des niveaux significatifs dans les graines de plusieurs espèces végétales. Cette classe comprend les alcaloïdes de purine, de tropane, de quinolizidine et d’indole [129]. Les alcaloïdes puriques, comme la caféine de Coffea arabica et la théobromine de Theobroma cacao, sont synthétisés à partir de nucléotides qui peuvent avoir un effet allélopathique ou induire des activités antimicrobiennes [129]. Les alcaloïdes de la quinolizidine sont abondants dans les graines de plusieurs espèces de légumineuses, comme le Lupinus polyphyllus [140]. Compte tenu de leur toxicité, la domestication a conduit à la sélection de variétés de légumineuses à teneur réduite en alcaloïdes quinolizidines. D’autres métabolites contenant de l’azote bien connus sont les glucosinolates (GLS), qui sont présents dans les graines de plusieurs Brassicaceae comme Arabidopsis, Camelina ou Brassica [42, 141, 142]. Bien qu’ils soient très abondants dans les graines, les GLS sont synthétisés dans les feuilles (et/ou les racines) et ensuite transportés vers les tissus des graines par les GLUCOSINOLATE TRANSPORTERS (GTR1 et GTR2) [143, 144].
6.3. L’impact de la domestication sur les métabolites spécialisés des graines
La domestication a souvent modifié involontairement la composition et la quantité de SMs dans les graines des espèces cultivées par rapport à leurs parents sauvages [39, 145, 146, 147, 148]. La domestication s’est principalement concentrée sur des traits physiologiques et visibles, tels que le rendement ou les caractéristiques sensorielles du goût, et a radicalement modifié la composition et la quantité de nombreux métabolites des plantes et des graines. Parmi les voies métaboliques qui ont été modifiées par la domestication, beaucoup ont un rôle bénéfique incontesté pour la santé et le stress des plantes cultivées [149]. Par exemple, chez la plupart des espèces cultivées, la domestication a conduit à la sélection de graines à faible teneur en tanins, car ceux-ci sont des composés antinutritionnels qui peuvent, par exemple, interagir avec les protéines et réduire la disponibilité des acides aminés [129, 150, 151]. La sélection d’espèces végétales à faible teneur en tanins peut avoir entraîné, par inadvertance, des effets négatifs sur l’accumulation d’autres flavonoïdes bénéfiques pour la plante et la santé humaine, tels que les flavonols, en appauvrissant l’ensemble de la voie des flavonoïdes [81]. C’est également le cas pour les grains de maïs domestiqués qui sont caractérisés par des niveaux de flavonols plus faibles que ceux des espèces sauvages [39]. Dans un autre exemple, il a été montré que les graines de légumineuses domestiquées accumulent des niveaux plus faibles de caroténoïdes que celles des espèces sauvages [147].
7. Plasticité et régulation environnementale de la composition des graines
Outre le déterminisme génétique et la distribution tissulaire, la teneur et la nature des composés de stockage (dont l’huile et les protéines) et des SM sont également façonnées par l’environnement et impactent fortement la qualité nutritionnelle, agronomique ou industrielle des graines (Figure 4). De nombreux facteurs biotiques et abiotiques, tels que le manque d’eau ou les températures élevées, peuvent affecter la composition en huile et en protéines des graines [152, 153, 154, 155, 156], la germination des graines et la vigueur des plantules [157, 158].
7.1. Composés de stockage
L’azote est un nutriment essentiel utilisé pour la biosynthèse des acides aminés et des protéines. La biosynthèse des SSP nécessite donc de l’azote qui est assimilé, puis distribué des organes sources aux puits de développement comme les graines. Il a été démontré que la limitation de l’approvisionnement en nitrate des plantes réduisait l’allocation de N dans les rosettes et les tiges tout en augmentant la remobilisation de N organique pour la croissance et la reproduction des plantes [159]. Malgré ce recyclage accru de l’azote, les graines produites par des plantes soumises à une limitation du nitrate ont des teneurs en azote réduites [160]. Si les concentrations de N et de C dans les graines (utilisées comme indicateurs des teneurs en SSP et en huile) sont différentiellement sensibles à la nutrition en fonction des accessions, on observe une tendance générale à l’augmentation des concentrations de N en cas d’apports élevés en nitrates, alors qu’à l’inverse, les concentrations de C sont plus élevées en cas d’apports faibles en nitrates. Mais les SSP représentent également un stock de S et les graines en développement sont connues pour employer des mécanismes homéostatiques pour équilibrer le stockage de N et de S [122]. Les graines présentent une capacité remarquable à maintenir le niveau des SSP stockées dans des conditions d’approvisionnement variable en S : dans des conditions de carence en S, les protéines riches en S sont supprimées tandis qu’une augmentation concomitante des protéines pauvres en S est observée. Chez Arabidopsis, la quantité d’albumines 2S (At2S3) diminue ainsi dans des conditions de carence en S et l’accumulation des sous-unités α et β de At12S3 est supprimée [161]. En effet, la globuline At12S3 est relativement riche en cystéine et méthionine contenant du S par rapport à At12S1, At12S2 et At12S4. La régulation de la composition des SSP en réponse à la carence en S implique divers contrôles transcriptionnels, traductionnels et post-traductionnels qui ont été étudiés dans différents systèmes végétaux [161].
Les stress environnementaux tels que la sécheresse et la chaleur exercent des contraintes importantes sur la productivité et les rendements et affectent la composition des graines de diverses cultures oléagineuses [162, 163]. De même, un stress hydrique post-floraison affecte dramatiquement le développement des graines et réduit le rendement chez Arabidopsis. De plus, les graines d’Arabidopsis qui subissent ce type de stress pendant le remplissage présentent des teneurs en C considérablement réduites, qui ne sont que partiellement compensées par des augmentations modérées de N [159]. De manière assez similaire, le stress thermique limite la croissance des plantes et produit moins de graines qui présentent également des rapports C/N réduits par rapport à ceux des plantes témoins. Diverses hypothèses ont été avancées pour expliquer la modification de cet équilibre, notamment (i) un déséquilibre entre l’azote disponible dans les plantes et le peu de graines produites [159], (ii) un effet négatif des températures élevées sur la photosynthèse, et (iii) l’induction de l’expression de gènes codant pour des lipases qui favorisent la dégradation du triacylglycérol dans les graines [164].
Outre la modification de la quantité globale d’huile stockée dans les graines, les températures élevées ont également un impact sur la composition en acides gras de ces huiles. Une relation inverse entre la température et la teneur en acides gras polyinsaturés des graines d’Arabidopsis a été rapportée [165]. Par exemple, entre 10 °C et 30 °C, la teneur en 18:1 du triacylglycérol des graines double alors que la teneur en 18:3 diminue de moitié [166]. Si la réponse des graines d’Arabidopsis est similaire à celle de différentes cultures oléagineuses comme le colza, le soja ou le tournesol, une tendance opposée a été décrite chez les graines de colza exposées à une température nocturne élevée, suggérant de faire une distinction entre les effets de la chaleur diurne et de la chaleur nocturne [164]. Mais, quelle que soit la tendance des modifications des acides gras polyinsaturés, la réponse est largement contrôlée par les activités des désaturases microsomales FAD2 et FAD3 sur la phosphatidylcholine, entraînant à leur tour des changements de composition du triacylglycérol. Différents niveaux de régulation de ces désaturases ont été rapportés dans les différentes espèces chez lesquelles elles ont été étudiées. Ces régulations comprennent des modifications de la disponibilité du substrat, des contrôles transcriptionnels et traductionnels, ainsi qu’une modification du renouvellement des protéines [166, 167]. Enfin, les stress biotiques influencent également l’accumulation des composés de stockage des graines. Par exemple, les plantes d’Arabidopsis infectées par une souche avirulente de Pseudomonas syringae capable d’activer leur réponse immunitaire présentent un rapport C/N plus élevé, reflétant une diminution de la teneur en N des graines [159].
7.2. Métabolites spécialisés
La diversité et le contenu des SMs des graines sont également affectés par les conditions environnementales. Cela a été illustré ci-dessus pour le dépôt des PCs solubles et de quercétine (flavonol) dans les téguments de la graine d’Arabidopsis qui peut être modulé par des signaux environnementaux tels que la température [104, 105]. C’est également le cas des alcaloïdes de la quinolizidine, dont l’accumulation est augmentée dans les grains de certaines espèces de lupin en réponse à une température élevée et à un stress hydrique [168]. Malgré le peu d’informations disponibles, il a également été rapporté que la teneur en glucosinolates diminue dans les graines de plantes B. rapa soumises à une carence en potassium [169]. Plus récemment, une forte induction environnementale et une plasticité des SMs ont été observées dans des graines de Camelina sativa cultivées pendant cinq saisons consécutives [152, 153]. Ce travail a mis en évidence des effets majeurs de l’environnement sur la stimulation du métabolome spécialisé de la graine. Dans ce travail, les flavonols, les glucosinolates et les alcaloïdes ont été identifiés comme des catégories métaboliques plastiques, tandis que la quantité de flavan-3-ol était plus stable durant les cinq années de cultures. Les auteurs ont également observé que les SMs des graines présentent une plasticité environnementale beaucoup plus élevée que l’huile et les SSP, d’autres composés liés à la qualité des graines, dont le métabolisme est modérément influencé par les conditions environnementales. Néanmoins, on en sait beaucoup moins sur la diversité et la plasticité des modifications des SM (c’est-à-dire l’hydroxylation, la glycosylation, la méthylation et l’acylation) en réponse aux variations environnementales.
8. Contrôle hormonal de la dormance et de la germination des graines
La dormance des graines est l’incapacité temporaire d’une graine viable à germer. Ce trait adaptatif permet aux graines matures d’interrompre leur cycle de vie et de reprendre leur croissance à la saison favorable [1]. La dormance est induite pendant le développement des graines et/ou après leur dispersion. Elle peut être atténuée avec le temps par des environnementaux tels que la lumière, la température ou les nitrates, qui sont intégrés par des réseaux de signalisation impliquant des hormones dont l’ABA et le GA et des radicaux libres tels que les espèces réactives de l’oxygène (ROS) et les espèces réactives de l’azote (RNS) (Figure 5).
Interactions entre les voies de signalisation ABA, éthylène et DOG1 dans la dormance des graines. L’ABA et l’éthylène interagissent à différents niveaux de leur biosynthèse et des voies de signalisation, comme décrit dans le texte. Ce diagramme résume les découvertes récentes sur l’interaction de DOG1 avec la signalisation de l’ABA et de l’éthylène en aval des régulateurs maitres de la maturation des graines les LAFLs : LEC1, ABI3, LEC2 et FUS3. ABI3 se lie au promoteur du gène ODR1 et réprime son expression, libérant ainsi l’inhibition de bHLH57 et favorisant la biosynthèse de l’ABA et la dormance des graines. Une rétroaction positive peut se produire par la stimulation de ABI3 par l’ABA. La liaison de l’ABA au récepteur PYL induit la formation d’un complexe avec PP2C et inhibe l’activité de la phosphatase, permettant aux kinases SnRK2 de phosphoryler les cibles en aval et de réguler positivement la dormance. DOG1 interfère avec la signalisation de l’ABA en interagissant avec deux PP2C, AHG1 et AHG3. Contrairement à AHG3, AHG1 est résistant à la liaison avec le récepteur PYL, ce qui suggère une autre voie de signalisation indépendante de l’ABA. L’expression de DOG1 est activée par bZIP67 en aval de LEC1 et par ERF1 en aval du récepteur de l’éthylène ETR1. La liaison du promoteur de DOG1 par le complexe ERF12-TPL réprime l’expression de DOG1 et favorise la levée de la dormance des graines par l’éthylène.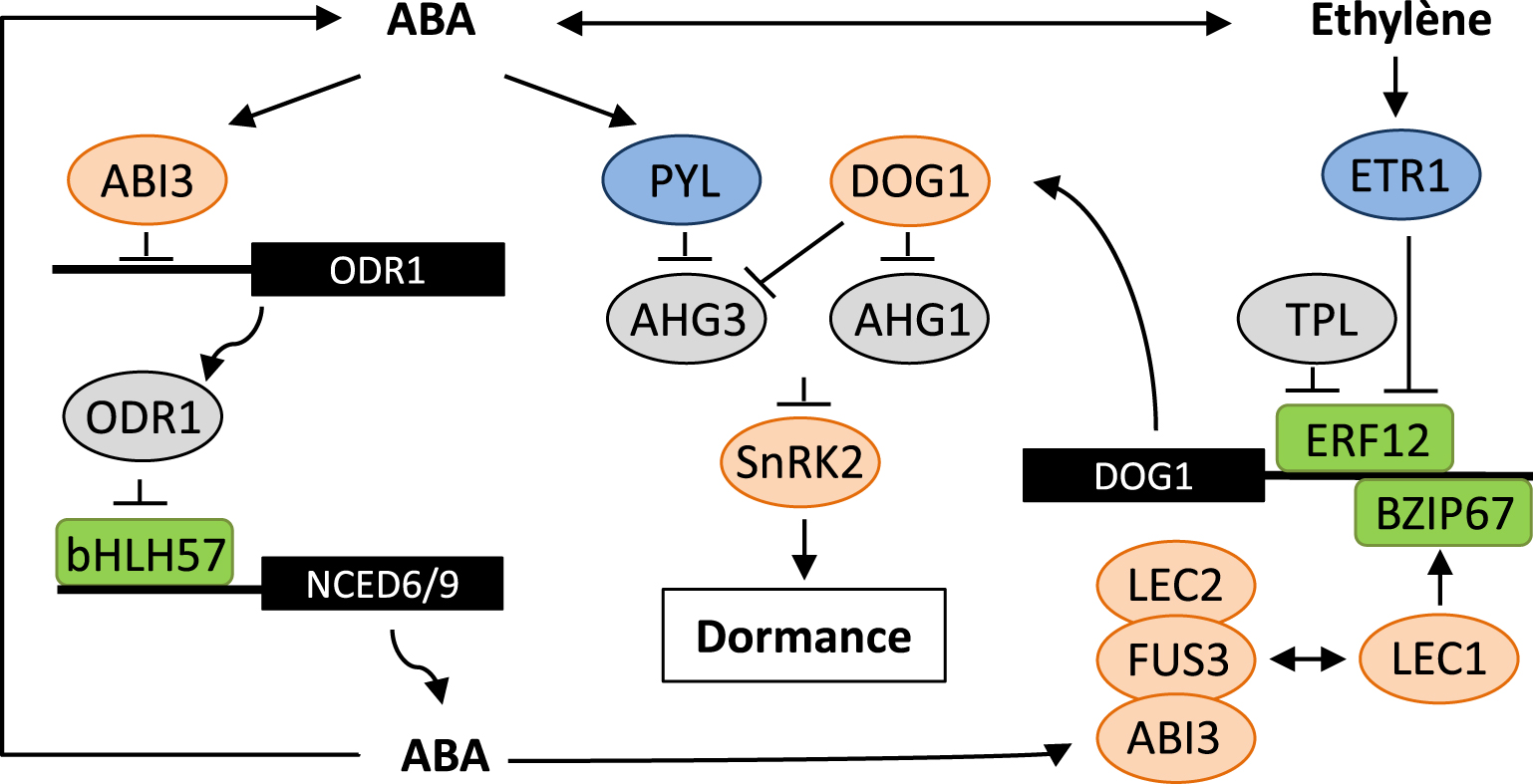
8.1. Le rôle clé de l’ABA dans la dormance des graines
Le rôle crucial de l’ABA dans l’induction de la dormance des graines a été déduit des phénotypes de graines de mutants du métabolisme ou de la signalisation de l’ABA chez plusieurs espèces. L’identification ultérieure des gènes impliqués dans le métabolisme, le transport, la perception et la signalisation de l’ABA a étendu nos connaissances sur les facteurs moléculaires et les réseaux interactifs agissant dans les différents tissus de la graine [170, 171]. Il a été démontré que l’accumulation d’ABA dans les graines d’Arabidopsis en développement atteint un pic à mi-maturation, et des études génétiques ont démontré que l’ABA produit par les tissus zygotiques est responsable de l’induction de la dormance. Les niveaux d’ABA diminuent dans les graines sèches matures et un catabolisme supplémentaire se produit dans les graines imbibées [172]. L’ABA est formé après clivage des précurseurs caroténoïdes et inactivé principalement par hydroxylation 8′-OH. Le clivage par la 9-cis-époxycaroténoïde dioxygénase (NCED) et le catabolisme par l’ABA 8′-hydroxylase (CYP707A) constituent donc des étapes régulatrices clés et les deux enzymes sont codées par des familles multigéniques. La régulation fine des niveaux d’ABA, qui influence la profondeur de la dormance, est contrôlé par l’expression de membres spécifiques des familles NCED et CYP707A [173, 174, 175]. Notamment, l’expression de NCED5, NCED6 et NCED9 apporte une contribution majeure à l’induction de la dormance pendant le développement de la graine et lors de l’imbibition. L’expression de CYP707A2 est corrélée à des niveaux d’ABA plus élevés et à l’inhibition de la germination dans les graines dormantes.
L’ABA est perçu par une famille de récepteurs de l’ABA codés chez Arabidopsis par 14 gènes, nommés pyrabactin resistance/PYR-like/regulatory components of ABA receptor (PYR/PYL/RCAR). En présence d’ABA, ces protéines séquestrent et inactivent les protéines phosphatases 2C de clade A (PP2C), libérant l’inhibition d’un sous-ensemble de trois kinases de type SNF1-related kinases 2 (SnRK2). Les kinases SnRK2 peuvent à leur tour phosphoryler les facteurs de transcription bZIP de la famille ABA INSENSITIVE5 (ABI5)/ABA response elements (ABRE)-binding (AREB)/ABRE-binding factor (ABF), qui se lient aux séquences promotrices ABRE dans les gènes inductibles par l’ABA et contribuent à la répression de l’inhibition de la germination des graines [176, 177]. Outre l’ABA, la protéine DELAY OF GERMINATION 1 (DOG1) a été identifiée comme un régulateur majeur de la dormance des graines et contribue fortement à la variation naturelle de ce caractère. Cependant, malgré des recherches intensives, la fonction moléculaire de cette protéine capable de se lier à un hème n’est pas entièrement comprise [171, 178, 179]. La protéine DOG1 s’accumule pendant la maturation des graines et les niveaux de protéine dans les graines sèches sont corrélés avec la profondeur de la dormance dans les graines fraîchement récoltées [180]. Néanmoins, la protéine DOG1 est toujours présente dans les graines après maturation, lorsque la dormance a été levée, ce qui suggère une possible perte d’activité par des mécanismes encore inconnus. Une découverte importante concernant la fonction de DOG1 a été l’identification de l’interaction entre DOG1 et la voie de signalisation de l’ABA (Figure 3). Il a été démontré que DOG1 se lie à et affecte négativement la fonction des protéines ABA HYPERSENSITIVE GERMINATION1 (AHG1) et AHG3, deux phosphatases PP2C [81, 182]. Le double mutant ahg1 ahg3 présente une dormance très profonde indiquant que AHG1 et AHG3 agissent de manière redondante dans les graines pour empêcher la levée de la dormance. Il est intéressant de noter que l’activité de AHG1 a été signalée comme étant résistante à l’inhibition par les récepteurs PYR/PYL/RCAR et donc capable de réprimer l’activité de SnRK2 en présence d’ABA. La dormance des graines pourrait donc être régulée de manière différentielle par deux voies de phosphorylation, par AHG1 sous le contrôle de DOG1 uniquement, et par AHG3 sous le contrôle de l’ABA et de DOG1 [181]. La liaison à l’hème est essentielle pour la fonction de DOG1 dans la dormance des graines, mais elle n’interfère pas avec l’interaction entre DOG1 et AHG1 [182]. Comme des événements oxydatifs sont étroitement liés à la levée de la dormance, il a été suggéré que la présence de l’hème participe à la détection de l’état redox. Ainsi, la liaison de l’hème à l’oxygène pourrait déterminer les niveaux de dormance par l’inactivation de DOG1 [171].
Une étude récente a identifié la protéine REVERSAL of RDO5 1 (ODR1) comme un régulateur négatif de la dormance des graines chez Arabidopsis [183]. Il a été démontré que ODR1 interagit avec le facteur de transcription bHLH57 empêchant l’activation de l’expression du gène NCED (Figure 3). ODR1 est l’orthologue de Seed dormancy 4 (Sdr4) du riz et a donc également été appelé Sdr4 like 1 (SFL1). Il est intriguant de constater que les graines d’Arabidopsis odr1/sfl1 sont plus dormantes alors que celles du riz sdr4 présentent une germination avant récolte (germination précoce sur l’épi). D’autres études suggèrent que ODR1/SFL1 aurait un double effet sur la dormance, positif au début du développement de la graine et négatif aux stades de maturation tardifs, en modulant l’expression des gènes LAFL et DOG1 [184].
8.2. Interactions avec d’autres hormones
L’ABA et les gibbérellines (GAs) ont des rôles antagonistes bien connus sur le développement de l’embryon et de l’albumen, qui influencent la dormance et la germination des graines en réponse à des signaux environnementaux. Les connaissances sur les réseaux de régulation qui ajustent de manière différentielle l’équilibre ABA/GA ont été décrites dans plusieurs revues récentes [170, 185, 186]. D’importants facteurs de régulation ont été identifiés, tels que SOMNUS (SOM) qui fonctionne à la fois dans les réponses à la lumière et à la chaleur et est contrôlé par des régulateurs de la signalisation des GAs et de l’ABA, tels que DELLA et ABI3 [187, 188]. DOG1 est également supposé être indirectement impliqué dans l’équilibre ABA/GA par la régulation négative du catabolisme des GAs [189]. Pour SOM et DOG1, les régulateurs transcriptionnels en aval des gènes du métabolisme hormonal restent à découvrir. Il est intéressant de noter qu’un facteur de transcription appartenant à la famille des protéines de type NIN (NLP), impliqué dans les réponses aux nitrates, se lie directement au promoteur de CYP707A2, ce qui suggère une fonction clé de NLP8 impliqués dans la signalisation des nitrates pour la promotion de la germination [190, 191]. Parmi les hormones qui interagissent de manière synergique ou antagoniste avec l’ABA [192, 193, 194, 195, 196], l’éthylène a de multiples interactions avec les voies de signalisation de l’ABA et de DOG1. L’éthylène joue un rôle positif dans la levée de la dormance par la régulation négative de la biosynthèse et des voies de signalisation de l’ABA et une interaction positive avec les voies liées aux GAs [197, 198, 199]. En outre, il a été récemment démontré que le facteur de réponse à l’éthylène ERF12 forme un complexe avec le corépresseur TOPLESS (TPL) et se lie au promoteur de DOG1. Ce complexe répresseur fonctionne en aval du récepteur de l’éthylène ETHYLENE RESPONSE1 [200]. En plus des hormones, les ROS sont des acteurs clés dans l’atténuation de la dormance et la germination. Alors que l’implication des ROS dans la fonction de DOG1 est encore putative, leur implication dans les voies de signalisation des hormones est bien documentée [201, 202]. Néanmoins, de nombreuses questions restent en suspens concernant leurs sites de production et leur mode d’action [203, 204].
8.3. Contrôle environnemental
Les conditions environnementales, en particulier la température, subies par la plante mère pendant le développement de la graine modulent la profondeur de la dormance. Chez Arabidopsis, le renforcement de la dormance par des températures fraîches est corrélé à une augmentation de l’abondance du transcrit DOG1 et des niveaux d’ABA dans les graines sèches [189]. Jusqu’à présent, un nombre limité de facteurs de régulation a été identifié. Il a été démontré que l’expression de DOG1 est directement activée par le facteur de transcription bZIP67 en aval du régulateur principal de la maturation des graines LEC1 et que l’abondance de la protéine bZIP67 dans les graines sèches augmente à des températures fraîches [205]. Cependant, une étude plus récente a suggéré que la régulation de DOG1 par la température maternelle serait également un effet indirect du froid sur la dégradation de l’ARNm de DOG1 [206]. Dans les graines sèches mûries au froid, l’augmentation de la teneur en ABA est bien corrélée avec une diminution de l’abondance des transcrits de CYP707A2 [189]. Il a été démontré que des facteurs de transcription, tels que ABI4, MYB96 et bHLH57, se lient aux promoteurs des gènes NCED ou CYP707A, mais aucun d’entre eux n’a encore été associé au contrôle des niveaux d’ABA en fonction de la température maternelle [207]. Néanmoins, l’identification du complexe régulateur avec les FTs bHLH57 et ODR1 a renforcé l’implication des gènes LAFL dans la régulation de la dormance par ABA et DOG1 [183]. En effet, il a été démontré que l’expression du gène ODR1 est directement réprimée par ABI3. A son tour, ODR1 régule négativement l’expression des gènes NCED6 et NCED9 par son interaction inhibitrice avec bHLH57 qui, en l’absence d’ODR1, se lie directement aux promoteurs des gènes NCED et induit leur expression. ODR1 pourrait donc réguler négativement la dormance des graines en affectant à la fois l’accumulation et la signalisation de l’ABA par la régulation d’ABI3 par l’ABA.
9. Modifications post-traductionnelles par les radicaux libres pendant le stockage et la germination des graines
Les espèces réactives de l’oxygène (ROS) et les espèces réactives de l’azote (RNS) produites en permanence comme sous-produits du métabolisme jouent un rôle important dans la physiologie des graines. La teneur en ROS (par exemple, les radicaux hydroxyle, superoxyde ou le peroxyde d’hydrogène) et en RNS (par exemple, l’oxyde nitrique, le dioxyde d’azote, l’acide nitreux et le tétroxyde de diazote) évolue à toutes les phases du cycle de vie des graines et influence la dormance, la longévité et la vigueur des graines [202]. Des études menées depuis 2010 montrent que les ROS et RNS effectuent une signalisation oxydative et nitrosative pour induire la germination des graines [201, 208, 209, 210]. Les modifications post-traductionnelles (PTMs) médiées par les ROS/RNS telles que la carbonylation, l’oxydation des acides aminés soufrés, la S-nitrosylation et la nitration peuvent favoriser la germination des graines.
9.1. Carbonylation des protéines
L’oxydation des protéines par carbonylation est une PTM irréversible. Pour éviter leur accumulation toxique, les protéines carbonylées sont dégradées par l’action du protéasome 20S dans le cytosol. L’attaque oxydative des acides aminés tels que Lys, Arg, Pro et Thr, induit la formation de groupes carbonyles (aldéhydes et cétones) sur les chaînes latérales. Des études protéomiques ont montré qu’une oxydation massive des protéines se produit pendant la maturation, le stockage et la germination des graines d’Arabidopsis et de pois [78, 211, 212, 213, 214, 215]. Ces études ont révélé l’existence de la carbonylation des protéines de stockage des graines (SSP) dans les graines sèches matures, ce qui a soulevé l’hypothèse qu’une telle PTM contribuerait à faciliter leur mobilisation pendant la germination en déstabilisant les corps protéiques compacts, augmentant ainsi leur susceptibilité au clivage protéolytique.
La protéomique a révélé un nouveau rôle pour les SSPs dans les graines sèches d’Arabidopsis pendant le vieillissement. Les cruciférines (CRU) sont les SSP les plus abondantes chez Arabidopsis et les graines d’un triple mutant pour trois isoformes de CRU (CRUa, CRUb et CRUc) sont plus sensibles au vieillissement artificiel. Leurs protéines sont fortement oxydées par rapport aux graines de type sauvage. Ces résultats confirment que l’oxydation est impliquée dans la détérioration des graines et que les SSP protègent la graine du stress oxydatif, protégeant ainsi d’autres protéines importantes nécessaires à la germination et à la formation des plantules [216]. La carbonylation peut donc être considérée comme l’un des événements importants du processus de vieillissement des plantes. On estime que pendant le vieillissement, une carence nutritionnelle ou une maladie, environ 10 % du protéome est plus enclin à la carbonylation [217]. De même, il a été démontré que pendant le processus de vieillissement des graines, les ROS induisent la carbonylation de nombreuses protéines, ce qui modifie leurs propriétés fonctionnelles et affecte la vigueur des graines. Un travail récent sur les graines de blé a révélé l’existence d’une corrélation négative significative entre la vigueur des graines et la teneur en protéines carbonylées [210]. Un comportement similaire a été observé lors du stockage à long terme de graines de hêtre [218]. Chez le riz, les résultats ont montré que, bien que le taux de germination des graines n’ait diminué que d’environ 20 % au cours des premières étapes du vieillissement, les niveaux de carbonylation de 68 protéines ont nettement changé, ce qui indique des dommages oxydatifs ; de même, le niveau de carbonylation des protéines de stockage des graines a augmenté [219]. Les graines d’Arabidopsis dépourvues de NADP-MALIC ENZYME 1 (NADP-ME1) fonctionnelle ont une viabilité réduite par rapport au type sauvage et présentent des niveaux de carbonylation des protéines plus élevés. Ces résultats suggèrent que l’activité de la NADP-ME1 est nécessaire pour protéger les graines contre l’oxydation pendant le stockage à sec [220]. Parallèlement, la carbonylation de nombreuses protéines impliquées dans les processus cellulaires et métaboliques se produit pendant la levée de la dormance du tournesol [221] et lors de l’imbibition de graines non dormantes d’Arabidopsis et de pois [213]. On a également émis l’hypothèse que l’altération de certaines activités métaboliques, comme l’oxydation des enzymes glycolytiques, pouvait conduire à l’activation de la voie des pentoses phosphates, fournissant ainsi un pouvoir réducteur améliorant le potentiel de germination. Bien que la carbonylation des protéines soit généralement décrite comme correspondant à des facteurs de détérioration, elle joue étonnamment un rôle positif pendant l’après-maturation et la germination des graines orthodoxes. Cela peut indiquer la nécessité du recyclage cellulaire des protéines dans l’atténuation de la dormance et pour la germination des graines [222].
9.2. Oxydation de la méthionine
De par leur abondance et leur composition chimique, les protéines constituent une cible majeure pour les ROS, notamment en ce qui concerne les acides aminés méthionine (Met) et cystéine (Cys) dont les chaînes latérales contiennent un atome de soufre. L’oxydation de résidus Met conduit à la formation de Met sulfoxyde (MetO). Cette réaction peut être inversée par l’action de méthionine sulfoxyde réductases (MSR) [223]. De manière importante, il a été démontré que le système MSR contrôle la longévité des graines chez Medicago, Arabidopsis [224] et Fagus sylvatica [225]. Une attaque oxydative plus sévère de Met entraîne la formation irréversible d’un dérivé sulfonique (Met sulfone). Dans un travail récent réalisé sur des graines sèches matures d’Arabidopsis, vingt-quatre résidus Met oxydés ont été identifiés dans 19 protéines par spectrométrie de masse [209]. Des résidus d’acides aminés convertis par oxydation (carbonylation) ou par phosphorylation ont été détectés dans l’environnement tridimensionnel des Met oxydés, ce qui suggère une interaction possible entre l’oxydation de résidus Met et d’autres PTMs. Une identification des protéines modifiées par oxydation des résidus Met a permis de découvrir que les protéines contenant MetO sont liées à la longévité des graines. De plus, cette étude a révélé la présence de MetO dans les globulines 12S d’Arabidopsis, ce qui pourrait indiquer que, comme la carbonylation, MetO pourrait induire des changements structurels dans les protéines de stockage des graines et pourrait interférer avec leur stockage pendant la maturation et leur mobilisation pendant la germination des graines. Il est important de noter que ces changements pourraient être réversibles car les MSRs ont été récemment immunolocalisées dans les vacuoles de stockage des protéines où les SSP sont stockées pendant la maturation des graines [225].
9.3. S-nitrosylation des protéines
Le NO est un radical libre gazeux impliqué dans de nombreuses voies de signalisation à la fois chez les animaux et les plantes. Il existe trois modifications importantes dépendantes du NO : la nitrosylation de centres métalliques, la S-nitrosylation des cystéines et la nitration des tyrosines [226]. La S-nitrosylation des résidus de cystéine des protéines est considérée comme le mécanisme le plus important du NO dans les plantes [227]. Plusieurs études ont documenté le rôle des RNS dans la régulation du potentiel de germination des graines. Dès les premières heures post-imbibition des graines d’orge, les niveaux de NO et les quantités de protéines S-nitrosylées augmentent [228, 229]. Le NO atténue la mort cellulaire par la S-nitrosylation des protéines et la régulation transcriptionnelle pendant le vieillissement des graines d’orme [230]. Ainsi, 80 protéines S-nitrosylées ont été identifiées, dont la glycéraldéhyde 3-phosphate déshydrogénase, indiquant que la S-nitrosylation de cette enzyme peut accélérer la mort cellulaire et la détérioration des graines au cours du vieillissement ; des résultats similaires ont été obtenus pour les graines d’Arabidopsis [231]. Il a été récemment signalé qu’une température élevée (32 °C) inhibe la germination des graines d’Arabidopsis et déclenche la S-nitrosylation des protéines [232]. Sous cette température, le facteur de transcription bHLH LONG HYPOCOTYL IN FAR-RED1 (HFR1) est S-nitrosylé à Cyt164, entraînant sa dégradation et la rupture de l’hétérodimère PIF1-HFR1 pour libérer l’activité du PHYTOCHROME INTERACTING FACTOR 1 (PIF1). PIF1 est un facteur de transcription ciblant l’expression de SOMNUS (SOM), un régulateur négatif de la germination. Des études sur plusieurs espèces végétales ont documenté que le NO rompt la dormance des graines et favorise la germination, notamment en relation avec des réseaux de signalisation hormonaux (éthylène, ABA) [198, 212, 217, 233, 234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244].
L’interaction entre l’éthylène et le NO repose sur l’activation de la biosynthèse de l’éthylène par le NO, probablement par le biais de PTM d’enzymes clés, comme l’ACC synthase et l’oxydase par S-nitrosylation. Dans les graines d’Arabidopsis, il a été démontré que l’interaction entre le NO et le facteur de transcription EIN3 (Ethylene Insensitive 3) atténue l’inhibition de la germination induite par une forte teneur en sel [239]. Le NO cible le FT ABA INSENSITIVE5 (ABI5), un régulateur majeur de la signalisation de l’ABA, ce qui illustre l’interaction entre les voies du NO et de l’ABA. Le NO régule négativement l’expression d’ABI5 en modulant l’activité de FTs de réponse à l’éthylène du groupe VII (ERFVII) par dégradation [245, 246]. Une nouvelle voie, dans laquelle le NO affecte directement la stabilité de la protéine ABI5, a été identifiée. La régulation directe de la protéine ABI5 par le NO est médiée par KEEP ON GOING (KEG), une ligase E3 de type RING. Dans ce mécanisme, la chaîne latérale thiol (–SH) de la cystéine 153 (Cys153) d’ABI5 est soumise à la S-nitrosylation par le NO, déclenchant l’ubiquitination d’ABI5 par KEG et sa déstabilisation ultérieure par la voie du protéasome 26S [235]. Lorsqu’une mutation est introduite dans la protéine ABI5 pour remplacer Cys153 par Ser153, elle n’affecte pas la fonction d’ABI5, telle que la formation de dimères et la liaison à l’ABA responsive element (ABRE) ; cependant, la S-nitrosylation et la déstabilisation d’ABI5 sont abolies par cette mutation. Ainsi, ABI5 sert de senseur de NO dans les graines et les plantules.
9.4. Nitration des protéines
La nitration des résidus tyrosines des protéines consiste à ajouter un groupe nitro (–NO2) à l’un des deux carbones ortho équivalents du cycle aromatique de ces résidus. La nitration de la tyrosine est considérée comme un processus sélectif plutôt qu’aléatoire et peut entraîner une altération de diverses fonctions protéiques [247]. Une augmentation de la nitration des protéines est reconnue comme un marqueur approprié du stress nitro-oxydatif qui est fréquemment associé au stress oxydatif dans diverses conditions de stress environnemental [248]. Une étude menée sur des axes embryonnaires de sorgho a révélé l’apparition de plusieurs protéines nitrées après l’imbibition des graines pendant 24 heures. L’application exogène de nitroprussiate de sodium (SNP, un donneur de NO) a permis d’atténuer plusieurs dommages oxydatifs mais a également entraîné une augmentation significative de la nitration des protéines. Par conséquent, la nitration pourrait être plus qu’un marqueur biologique du stress nitrosatif et pourrait participer au renouvellement des protéines, à la transduction des signaux et à la régulation des hormones végétales [212, 249]. Dans cette optique, il est intéressant de noter que le NO peut inhiber la signalisation de l’ABA car les plantes déficientes en NO sont hypersensibles à cette phytohormone [250]. La nitration in vitro de plusieurs récepteurs de l’ABA tels que PYR1 et PYL1 par SIN-1 a été décrite ; la diminution rapide observée de la réactivité de l’ABA due à l’inactivation de PYR1 par nitration pourrait déclencher la dégradation du récepteur de l’ABA et ainsi contrôler la signalisation de l’ABA [251]. Une analyse protéomique a montré que MoCo (le cofacteur de molybdène sulfuré qui fonctionne comme cofacteur pour l’aldéhyde oxydase abscissique qui convertit l’aldéhyde abscissique en ABA) est la cible de la nitration de tyrosine, ce qui inactive la synthèse de l’ABA pendant la germination. Ainsi, la nitration des protéines pourrait contribuer à contrôler la levée de la dormance et la vigueur de la germination [202].
10. Quelques conclusions et perspectives
Compte tenu de l’importance des réserves des graines pour nourrir le monde et de l’urgence de développer des pratiques agricoles plus durables (par exemple en limitant l’utilisation de produits chimiques de synthèse), l’élucidation des mécanismes moléculaires qui sous-tendent le développement, le vieillissement et la germination des graines constitue plus que jamais un enjeu scientifique et sociétal majeur. Ces connaissances seront essentielles pour orienter les programmes de sélection et développer de nouvelles plantes cultivées et variétés d’intérêt agroéconomique destinées à produire des graines aux qualités nutritionnelles, agronomiques ou industrielles améliorées, tout en maintenant ou en optimisant l’aptitude générale et les performances des cultures. L’intégration réussie de stratégies de biostimulation et de biocontrôle dans la sélection végétale ainsi que dans les technologies de production et d’amélioration des semences (telles que l’enrobage et le « priming » des semences) constitue un défi majeur. Les traitements traditionnels tels que le priming des semences sont en train d’être repensés en intégrant des biosolutions pour améliorer la vigueur des semences (c’est-à-dire une germination rapide et synchrone) et pour prévenir la fonte des semis en induisant des mécanismes de défense au début de l’établissement des semis. Ces développements sont nécessaires pour protéger et améliorer les performances des semences et des plantules et pour répondre aux exigences sociétales et réglementaires qui soutiennent l’interdiction de l’utilisation des produits chimiques de synthèse dans le contexte des transitions agroécologiques.
Les approches de génétique moléculaire avec des espèces modèles comme Arabidopsis restent des outils bien adaptés et puissants pour étudier ces mécanismes, bien qu’elles puissent présenter certaines limitations et inconvénients inhérents à la petite taille des graines et à l’enchevêtrement des différents tissus, ou à une diversité génétique et biochimique limitée. Au cours de la dernière décennie, des progrès techniques majeurs ont été réalisés, qui permettent de remédier à certaines de ces limitations. Des analyses spécifiques de tissus ou même de cellules peuvent maintenant être mises en œuvre et combinées efficacement avec diverses approches omiques, et les espèces sauvages peuvent être étudiées directement. En outre, des approches génétiques efficaces telles que les systèmes d’édition du génome facilitent considérablement la recherche de pointe sur les plantes cultivées et la sélection de nouveaux caractères d’intérêt.
De même, la caractérisation de la diversité des graines chez les plantes sauvages et cultivées est désormais essentielle pour comprendre ce qui sous-tend les spécificités des espèces en termes de composition biochimique et de résilience aux conditions environnementales défavorables. Cela contribuera à la découverte de nouvelles molécules d’intérêt pour la bioprotection des plantes, la production d’aliments sains ou de matières premières renouvelables pour les industries biosourcées. Les progrès du séquençage du génome et de la génomique fonctionnelle faciliteront l’identification des gènes clés contrôlant les voies de biosynthèse correspondantes. Cependant, les interactions possibles entre les différents métabolismes doivent être prises en compte lorsque l’on tente d’optimiser la production de certaines molécules d’intérêt afin de ne pas altérer d’autres caractéristiques des graines ou des plantes.
Actuellement, nous sommes encore loin d’avoir une compréhension complète des mécanismes moléculaires et cellulaires qui sous-tendent le développement et la germination des graines, leur métabolisme et les réponses physiologiques à des environnements difficiles. Des recherches supplémentaires sont nécessaires pour la construction d’un modèle spatio-temporel complet et prédictif des graines. Il sera crucial d’élucider les interactions complexes existant entre les différents compartiments de la graine, tant du point de vue de la signalisation que du métabolisme. Nous devons également étudier le métabolisme des graines aux niveaux cellulaire et même intracellulaire, tout en intégrant l’influence des facteurs environnementaux.
Conflit d’intérêt
Les auteurs n’ont aucun conflit d’intérêt à déclarer.
Remerciements
Nous tenons à remercier tous nos collègues, techniciens, ingénieurs étudiants, post-docs et personnel administratif qui ont contribué aux travaux présentés dans cette revue et nous nous excusons auprès de ceux que nous n’avons pas pu citer par manque de place. Nous tenons à remercier chaleureusement Georges Pelletier et Michel Delseny pour leur soutien constant et fort à ce travail et pour la rédaction de cette revue. Nous tenons à remercier Eric Postaire pour la correction de la revue et la rédaction de la version française.




 CC-BY 4.0
CC-BY 4.0