1. Non-homologous interchromosomal gene regulation
The organization of the genome inside the nucleus is central to gene regulation. The chromatin fiber is organized hierarchically, with most of the enhancer-gene promoter loops occurring in cis within self-interacting domains called Topologically Associating Domains (TADs). These TADs are further organized into active or repressed compartments of interactions. Finally, in most organisms, each chromosome occupies its own distinct territory in the nucleus [1]. This organization may give the impression that the regulation of a gene is controlled solely by its chromosomal environment. However, with the recent development of chromosome-conformation-capture and imaging technologies enabling the unbiased detection of proximity between genes located on different chromosomes, and their application to an increasing number of cell types and organisms, it is becoming apparent that interchromosomal gene interactions are more common than previously thought. Furthermore, a growing number of examples have demonstrated or suggested the functional importance of interchromosomal gene regulation in cell fate decisions or cellular functions [2, 3]. For example, in the mouse olfactory epithelium, only one olfactory receptor allele, out of ∼2400 candidate alleles, is expressed in each olfactory sensory neuron. These alleles are distributed throughout the mouse chromosomes and undergo a complex choreography to aggregate in the nuclear space and to select a single allele to be expressed [4].
Interchromosomal interactions are not restricted to multigene families, like the olfactory receptor genes. Physical associations also occur between a gene and a regulatory region located on different chromosomes. This is the case, for instance, in T-cells, where physical interactions occur between the promoter of the IFN-𝛾 gene, located on chromosome 10, and the regulatory region of the TH2 cytokine locus, located on chromosome 11 [5]. This interaction contributes to the regulation of cytokine expression in T cells and IFN-𝛾 production in response to viral infection. The aforementioned examples illustrate a specific class of interactions between non-homologous regions of the genome and that have been reviewed recently in Maass et al. [3].
2. Homologous interchromosomal gene regulation
Interchromosomal interactions can also occur between homologous genes (i.e., alleles) located on homologous chromosomes. Various manifestations of these interallelic interactions have been documented in different eukaryotic lineages, from fungi, plants, and animals [6]. The first case of interallelic interaction was reported by Ed Lewis in 1954 when he studied the regulation of the Hox gene Ultrabithorax (Ubx) in the fruitfly Drosophila melanogaster (D. melanogaster). Lewis described genetic complementation between the Ubx mutant alleles bx34e and Ubx1. Both alleles, when homozygous, cause a partial transformation of halteres into wings. However, trans-heterozygous flies (bx34e + ∕ + Ubx1) show partial complementation since the appendage transformation is less severe than in flies homozygous for either allele. In addition, Lewis showed that the genetic complementation of Ubx mutant alleles is compromised when the pairing—the alignment end-to-end—of homologous chromosomes is disrupted, suggesting that the alleles can only interact when they are physically close to one another. Lewis coined the term transvection to describe pairing-dependent interallelic complementation [7]. By extension, the term transvection has been used to describe proximity-dependent interallelic regulation.
Since the seminal work of Ed Lewis on Ubx regulation, many cases of transvection have been described for many genes in D. melanogaster, although usually using synthetic transgenes or intragenic complementation [8]. It has also been described in other organism, including yeast and mammals [9, 10]. Transvection can have a positive or negative effect on gene expression, indicating that different molecular mechanisms are involved. In Drosophila, as in all Diptera, homologous chromosomes are paired in all somatic cells during interphase [11]. This particular configuration presumably facilitates transvection throughout the genome [12, 13]. Although the D. melanogaster genome appears to be generally permissive for transvection, the physiological relevance of interallelic regulation in a wild-type context has long been questioned. However, several examples illustrate that it is crucial for the specification of certain cell types. We review a couple of these examples below.
3. Transvection controls photoreceptor diversity in the retina
In the D. melanogaster eye, each ommatidium (i.e., eye unit), contains eight photoreceptor cells named R1 to R8. R7 expresses one of the two possible light-detecting Rhodopsins, Rh3 or Rh4, defining two subtypes of ommatidia: pale (p, Rh3+) and yellow (y, Rh4+). The expression of the transcription factor Spineless is responsible for directing the R7 photoreceptor subtypes: when Spineless is present it activates the expression of Rh4 and represses the expression of Rh3, while in its absence Rh3 is expressed. The expression of spineless (ss) in R7 photoreceptors is stochastic, driving the formation of ∼70% yR7s and ∼30% pR7s. The expression pattern of ss, and therefore the ratio of pR7 and yR7 in the retina, is set by interactions between ss alleles. Different alleles or allelic combinations alter the ss expression pattern and, in turn, the ratio of the R7 subtypes. A regulatory element of the ss gene, acting as an insulator, is required for bringing two alleles in proximity, even if they are located on different chromosomes [14, 15]. This proximity between alleles presumably allows other regulatory regions of the locus to interact in trans to set the expression pattern of ss in the retina. Although the molecular details of the interallelic interactions are not well understood, this example shows that transvection is not necessarily dependent on chromosomal pairing per se, but the latter establishes the proximity between the alleles. Furthermore, it illustrates how transvection can contribute to a stochastic mechanism that generates cell fate diversity in the retina.
4. Transvection underlies sexual dimorphism
The D. melanogasteryellow (y) gene has long been a test case to study transvection, mostly using intragenic complementation [16]. Yet, whether regulatory interactions between y alleles, which can only occur in females since y is X-linked, contribute to sculpting y expression level or pattern in a wild type condition was unknown. y is required for the production of black pigment throughout the body and its expression pattern is sexually dimorphic in several tissues and body parts. We recently found that in D. biarmipes, a species closely related to D. melanogaster that harbors a male-specific wing pigmentation spot, the sexually dimorphic expression pattern of y in the wing is controlled by transvection. y expression in the wing is controlled by two neighboring enhancers located on the 5′ of the gene, the wing enhancer that drives uniform low expression, and the spot enhancer that drives high expression in the spot area (Figure 1A–C) [17]. In females, the activity of the spot enhancer—but not the wing enhancer—is silenced by transvection (Figure 1D–G). In hemizygous males, transvection cannot occur and the spot enhancer is active. Importantly, when the y locus is inserted on an autosome, at the same position on each homologous chromosome, the interallelic interaction can take place in both sexes. This shows that the silencing of the spot enhancer in females is not due to an X chromosome-specific regulatory mechanism but to the interaction between alleles. The transvection-dependent silencing of the spot enhancer requires the y intron, suggesting that it contains a regulatory element that is necessary for the interallelic interaction, or that, following the interaction, silences the spot enhancer, or both. Remarkably, another sexually dimorphic enhancer, active in the posterior abdominal segments of males and located nearby the spot enhancer, is not controlled by transvection but by the canonical sex determination hierarchy [18]. How two regulatory elements of the same gene, separated by only a few hundred base pairs, came to use completely different regulatory strategies to achieve similar male-specific expression raises intriguing mechanistic and evolutionary questions.
Sexually dimorphic regulation of y in the wing of D. biarmipes. The spot enhancer controls y expression in male wings by activating its transcription possibly through either a looping (A) or a linking (B) mechanism, resulting in a spotted expression pattern (C). In female wings, the spot enhancer activity is silenced by three possible mechanisms: a pairing-dependent looping topology of the locus that brings a repressor binding the intron sequence close to the the spot enhancer (D). Protein–protein interactions initiated at the intron could support the formation of a boundary between the spot and the wing enhancer, preventing the interaction between the spot enhancer and the yellow promoter (E). Finally, pairing of the yellow alleles could create a local microenvironment that recruits repressor proteins to block the activity of the spot enhancer (F). These hypotheses are not necessarily mutually exclusive. In the female wing, some cells randomly escape the spot enhancer silencing leading to dotted pattern of high levels of Yellow (G). The figure was created using Biorender.com.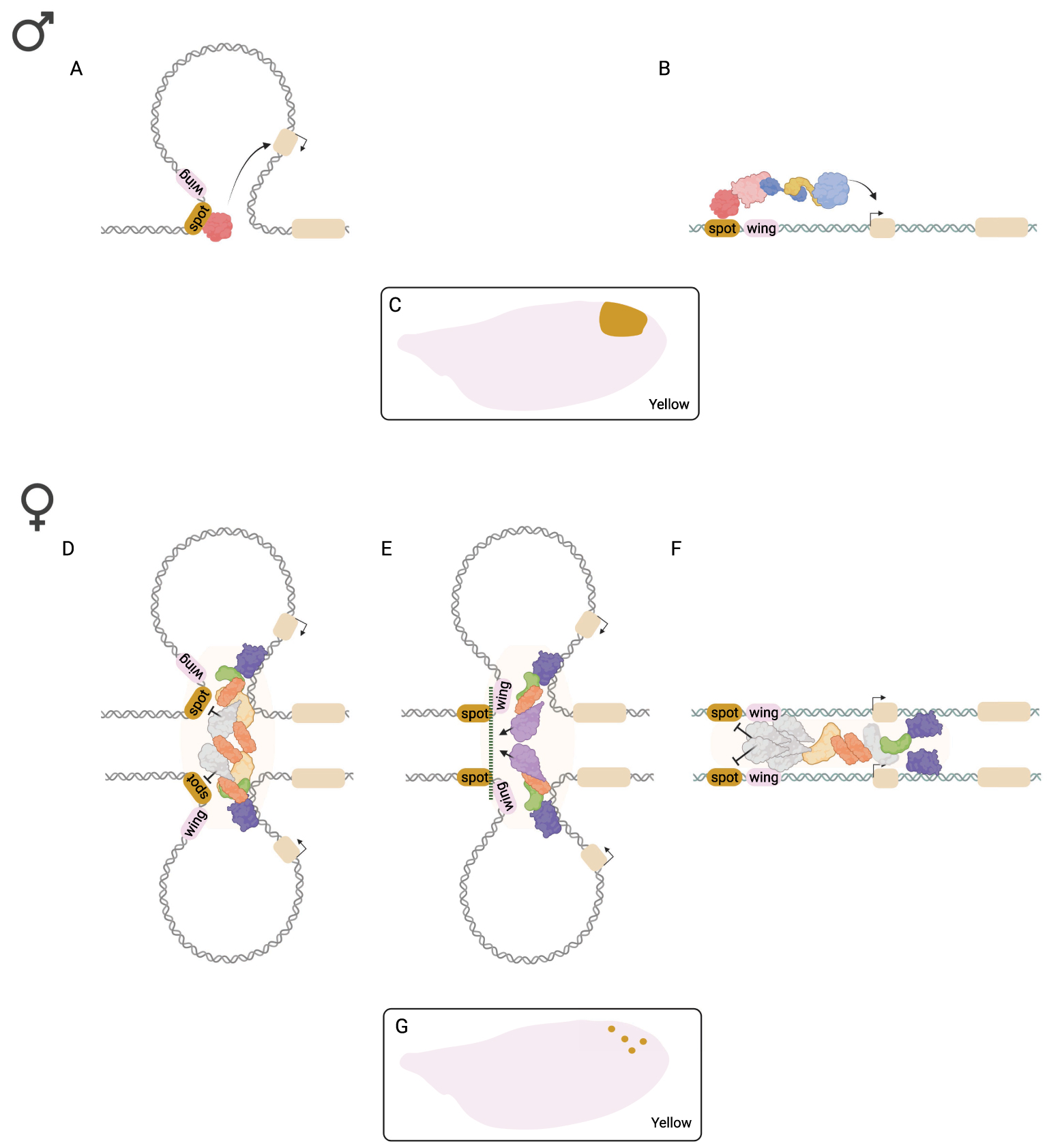
5. Elements of the mechanism of transvection
Transvection may have been confounded with chromosomal pairing, in particular in Drosophila where somatic homologous pairing is so prevalent and strongly associated with transvection. Yet, if transvection requires close proximity between alleles for functional interaction, chromosomal pairing is not necessarily the only way to bring two alleles near each other. So, the dissection of the mechanisms underlying transvection needs to distinguish the determinants of the physical association of homologous alleles from the regulatory events following this association that subsequently impact gene regulation. In the following sections, we will discuss what is known about each of these two steps, mostly using Drosophila studies.
5.1. Homologous somatic pairing establishes the physical association of homologous alleles
In 1908 Nettie M. Stevens observed that homologous chromosomes are paired in several Diptera species [11]. In contrast to the homologous pairing that is being observed during meiosis, which is required for chromosome segregation and recombination, the raison d’être of homologous pairing in somatic tissues observed during interphase is unknown. In D. melanogaster the pairing of homologous chromosomes appears progressively during embryogenesis. The initiation of homologous somatic pairing takes place around the same time when TADs are also formed in the last syncytial nuclear cycles, leading to a higher degree of pairing observed in nuclear cycle 14, when cellularization has not yet occurred [19, 20]. The whole process coincides with the maternal-to-zygotic transition. Live-imaging data revealed the dynamics of homologous somatic pairing in the early embryo, showing that homologous loci are separated by 2.2 μm before pairing, and by only 0.4 μm after pairing has occurred. Furthermore, this analysis reveals that once established, homolog pairing is overall highly stable, and characterized by small dynamic changes in the distance between homologous loci [21].
A prevalent model for the initiation of pairing is that some button loci, regions of high pairing affinity distributed throughout the chromosomes, find their counterparts on homologous chromosomes through a random exploration in the nuclear space [15, 21]. Spreading of pairing then occurs by a “zippering” mechanism from button to button along the length of the chromosome [21].
Haplotype-resolved Hi-C studies revealed that the D. melanogaster chromosomes are organized into regions of tight pairing and regions of loose pairing [22, 23]. The extent to which regions of tight and loose pairing are identical between different cell types is not known, although the buttons that drive pairing appear to contribute to different extents between different cell types [15].
The button model can explain the initiation and the spreading of the pairing between homologous chromosomes. It is conceivable that most individual button pairs provide low interaction energy that on its own is not sufficient to drive pairing and that the density of buttons along the length of the chromosome is an important determinant of pairing dynamics [21]. Ectopic pairing, between non-homologous chromosomes, can be induced in Drosophila between relatively large chromosomal transgenes (∼100 kb) and their homologous regions, suggesting that pairing between any homologous sequences requires a substantial number of “weak” buttons [15]. At the other end of the affinity spectrum, some short regions (Fab-7, Mcp) are sufficient to mediate pairing, even when inserted on non-homologous chromosomes [24, 25], suggesting that different buttons may have different affinities. Some situations appear intermediate between the high-affinity regions and the “weak” individual buttons. For instance, we have shown that homologous copies of y, which is a fairly compact gene (∼10 kb), can still physically associate and functionally interact even if they are separated by 5 Mb on the same chromosome, but they cannot interact when they are inserted on non-homologous chromosomes [18]. Altogether, these results suggest that physical interaction between homologous regions is driven by buttons that have different affinities and densities, conferring weak to strong pairing capacity to each region depending on their distribution along the length of the chromosomes.
5.2. Trans-acting factors and insulators
Whether the same factors that establish and/or maintain pairing also directly affect transvection is not known. It is also not known whether there are generic factors that directly affect all cases of transvection, or whether distinct factors are associated with distinct loci. Several factors have been described that affect transvection in Drosophila by influencing homologous somatic pairing. Initially, two factors, Su(Hw) and Top2, were shown to promote homologous somatic pairing [26, 27]. Surprisingly, Cap-H2, a subunit of the Condensin II complex, was shown to antagonize somatic pairing, indicating that the pairing state of a nucleus may be the output of the balance between pairing and anti-pairing factors [28]. Genetic screens in D. melanogaster embryos and cells identified more than 100 genes that promote or inhibit homologous somatic pairing. This gene set was enriched for genes implicated in the cell cycle control, suggesting a link between chromosome organization and cell cycle progression [29, 30].
Many of the factors that upon deregulation affect transvection are insulator proteins. Insulator proteins are a diverse class of chromatin architectural proteins that bind to or are associated with insulator DNA, originally identified as DNA segments that block the communication between an enhancer and a promoter when placed between them. Also, insulators have been shown to pair in cis, bringing two elements located on either side of them closer together. More generally, insulators, and the protein complexes that bind to them, play a central role in regulating chromatin organization, from local loops to higher-order chromatin structures [31]. Most importantly, different combinations of insulator proteins appear to be enriched in buttons such as CTCF, GAF, BEAF-32, Cp190, and Mod(mdg4), which are also enriched in TAD boundaries, suggesting that homologous pairing and TAD formation might be related phenomena [15]. In addition, regions of tight pairing are enriched for architectural and insulator proteins [23].
Transgenes containing the gypsy retrotransposon (which contains an insulator sequence), Wari, 1A2, or Fab-8 insulators can support transvection at many sites in the genome, also suggesting that support for transvection may be a general feature of insulators [32, 33]. Visualization of interactions between homologous regions at the Abd-B locus [34, 35] or using transgenes containing an enhancer from the snail locus indicates that the role of insulators is to stabilize the interaction between homologous alleles [36], consistent with their overrepresentation in button loci [15]. Interactions between different types of insulators have been shown to support transvection [33]. These insulators seem to be bound by the same factors suggesting that the combination of these factors drives these interactions.
How precisely DNA insulators and insulator proteins influence transvection is unclear. For instance, Mod(mdg4), a classical insulator co-factor, is required for transvection-dependent negative regulation of the yspot enhancer in the wing [18]. However, what Mod(mdg4) does to y is unknown. One possibility is that it could be involved in topological changes at the y locus, by either bridging a repressor binding the intron and the spot enhancer (Figure 1D), or by forming a strong boundary between the spot and the wing enhancers that would preserve the wing activity and block the interaction between the spot and the promoter (Figure 1E). Further investigation of this system, and others, will be necessary to better understand how insulator DNA sequences and architectural proteins contribute to transvection.
5.3. Transvection might create discrete nuclear microenvironments
Homologous enhancers typically compete for accessing the promoter, and usually, the cis-interaction (enhancer and promoter on the same chromosome) is favored over the trans-interaction (enhancer and promoter on the different chromosomes). However, when the loci are paired the cis-competition might be reduced. Indeed, Lim and colleagues have observed using live-imaging of locus-resolved transcription that a single shared enhancer has equal access to both promoters when the loci are paired [36]. This result is consistent with the notion that tight association of homologous loci might create a local nuclear microenvironment in which transcription factors and co-factors are enriched and shared between homologous enhancers. Nuclear environments can be a positive or a negative effect on enhancer activity and gene expression, depending on the factors they recruit. Applying this model to the regulation of the yspot enhancer, one can imagine that the interaction between the two alleles creates a local microenvironment that facilitates the cooperative recruitment of factors repressing the spot activity (Figure 1F).
6. Conclusions and perspectives
Although transvection was described almost 70 years ago, its significance for gene expression in physiological contexts has long remained an open question. We have now several examples establishing that transvection plays a key role in controlling gene expression patterns in Drosophila, arguing for the relevance of this gene regulatory mechanism. Beyond Drosophila genetics, transvection and transvection-related phenomena may also be more common than currently appreciated. The conceptual association of transvection with homologous chromosome pairing may have led to the misconception that transvection is a Drosophila-specific phenomenon. The absence of homologous chromosome pairing may disfavor interallelic interactions, but these interactions can nevertheless occur transiently and affect gene regulation. An illustration of this comes from the regulation of Oct4, which is highly expressed in embryonic stem cells in mammals. During development, the Oct4 alleles come closer, leading to Oct4 repression, which marks the exit from the pluripotent state [10]. This example suggests that there must be other situations in mammals where interactions between alleles influence gene expression.
How interchromosomal gene regulation works remains a vast open question. We suggest that there might be mechanistic continuum between homologous and non-homologous interchromosomal gene regulation, and that general principles underlie these different phenomena. Transvection and transvection-like phenomena rely on proximity between interacting genes. Yet, how close loci need to be to functionally interact is largely unknown. The most accurate measurements of the distance between homologous loci (but not necessarily functionally interacting) in the Drosophila embryo show that they are separated by 0.4 μm [21]. This is still a considerable distance to bridge for transcription factors binding the regulatory region of one allele and to act on the homologous allele. Further measurements of the distance and associated dynamics of functionally interacting alleles are needed as well as an assessment of how these properties leave their footprint on gene expression. It is also crucial to understand the consequences of allelic pairing on locus positioning in the nucleus or on topological changes at the locus. Using haplotype-resolved chromosome-conformation-capture technologies and super-resolution imaging will certainly help us to better characterize what happens when homologous alleles or regulatory regions come close, and how they influence each other.
Acknowledgements
CCG acknowledges funding from the European Molecular Biology Organization (297-2021). BP acknowledges funding from European Research Council under the European Union’s Seventh Framework Programme (FP/2007-2013)/ERC grant agreement 615789, and support from the CNRS.
Conflicts of interest
Authors have no conflict of interest to declare.
French version
1. Régulation génique interchromosomique non homologue
L’organisation du génome à l’intérieur du noyau est centrale pour la régulation des gènes. La fibre chromatinienne est organisée de manière hiérarchique, la plupart des boucles formées par l’interaction entre des séquences activatrices (« enhancers » et le promoteur d’un gène se trouvant en cis dans des domaines d’auto-interaction appelés domaines d’association topologique (TAD). Ces TAD sont ensuite organisés en compartiments actifs ou inactifs. Enfin, chez la plupart des organismes, chaque chromosome occupe un territoire distinct dans le noyau [1]. Cette organisation peut donner l’impression que la régulation d’un gène est contrôlée uniquement par son environnement chromosomique en cis-. Cependant, avec le développement récent des technologies de capture de la conformation des chromosomes et d’imagerie permettant la détection non biaisée de la proximité entre des gènes situés sur des chromosomes différents, et leur application à un nombre croissant de types de cellules et d’organismes, il devient évident que les interactions génétiques interchromosomiques sont plus courantes qu’on ne le pensait. En outre, un nombre croissant d’exemples a démontré ou suggéré l’importance fonctionnelle de la régulation interchromosomique des gènes dans les décisions relatives au destin cellulaire ou aux fonctions cellulaires [2, 3]. Par exemple, dans l’épithélium olfactif de la souris, un seul allèle de récepteur olfactif, sur ∼2400 allèles candidats, est exprimé dans chaque neurone sensoriel olfactif. Ces allèles sont répartis sur l’ensemble des chromosomes de la souris et participent à une chorégraphie complexe pour s’agréger dans l’espace nucléaire et sélectionner un seul allèle à exprimer [4].
Les interactions interchromosomiques ne sont pas limitées aux familles multigéniques, comme les gènes des récepteurs olfactifs. Des associations physiques se produisent également entre un gène et une région régulatrice située sur des chromosomes différents. C’est le cas, par exemple, dans les lymphocytes T, où des interactions physiques se produisent entre le promoteur du gène IFN-𝛾, situé sur le chromosome 10, et la région régulatrice du locus des cytokines TH2, située sur le chromosome 11 [5]. Cette interaction contribue à la régulation de l’expression des cytokines dans les lymphocytes T et à la production d’IFN-𝛾 en réponse à une infection virale. Ces exemples illustrent une classe spécifique d’interactions entre des régions non homologues du génome, qui ont été passées en revue par Maass et al. [3].
2. Régulation interchromosomique des gènes homologues
Des interactions interchromosomiques peuvent également se produire entre des gènes homologues (c’est-à-dire des allèles d’un même gène) situés sur des chromosomes homologues. Diverses manifestations de ces interactions interalléliques ont été documentées dans différentes lignées eucaryotes, des champignons aux plantes en passant par les animaux [6]. Le premier cas d’interaction interallélique a été signalé par Ed Lewis en 1954 lorsqu’il a étudié la régulation du gène Hox Ultrabithorax (Ubx) chez Drosophila melanogaster (D. melanogaster). Lewis a décrit la complémentation génétique entre les allèles mutants Ubxbx34e et Ubx1. Chacun des deux allèles, à l’état homozygote, provoque ne transformation partielle des haltères en ailes. Cependant, les mouches trans-hétérozygotes (bx34e + ∕ + Ubx1) montrent une complémentation partielle puisque la transformation des appendices est moins sévère que chez les mouches homozygotes pour l’un ou l’autre des allèles. De plus, Lewis a montré que la complémentation génétique des allèles mutants Ubx est compromise lorsque l’appariement — l’alignement bout à bout — des chromosomes homologues est perturbé, ce qui suggère que les allèles ne peuvent interagir que lorsqu’ils sont physiquement proches les uns des autres. Lewis a inventé le terme transvection pour décrire la complémentation interallélique dépendant de l’appariement [7]. Par extension, le terme transvection a été utilisé pour décrire la régulation interallélique dépendante de la proximité.
Depuis les travaux précurseurs d’Ed Lewis sur la régulation d’Ubx de nombreux cas de transvection ont été décrits pour de nombreux gènes chez D. melanogaster, bien qu’utilisant généralement des transgènes synthétiques ou une complémentation intragénique [8]. La transvection a également été décrite dans d’autres organismes, notamment chez la levure et les mammifères [9, 10]. La transvection peut avoir un effet positif ou négatif sur l’expression des gènes, ce qui indique que différents mécanismes moléculaires sont impliqués. Chez la drosophile, comme chez tous les diptères, les chromosomes homologues sont appariés dans toutes les cellules somatiques pendant l’interphase [11]. Cette configuration particulière facilite vraisemblablement la transvection tout au long du génome le génome [12, 13]. Bien que le génome de D. melanogaster semble être généralement permissif pour la transvection, la pertinence physiologique de la régulation interallélique dans un contexte de type sauvage a longtemps été remise en question. Cependant, plusieurs exemples montrent qu’elle est cruciale pour la spécification de certains types de cellules. Nous passons en revue quelques-uns de ces exemples ci-dessous.
3. La transvection contrôle la diversité des photorécepteurs dans la rétine
Dans l’œil de D. melanogaster, chaque ommatidie (c’est-à-dire l’unité oculaire) contient huit cellules photoréceptrices nommées R1 à R8. R7 exprime l’une des deux rhodopsines photoréceptrices possibles, Rh3 ou Rh4, définissant deux sous-types d’ommatidies : pâle (p, Rh3+) et jaune (y, Rh4+). L’expression du facteur de transcription Spineless est responsable de l’orientation du destin des sous-types de photorécepteurs R7 : lorsque Spineless est présent, il active l’expression de Rh4 et réprime l’expression de Rh3, alors qu’en son absence Rh3 est exprimé. L’expression de spineless (ss) dans les photorécepteurs R7 est stochastique, conduisant à la formation de ∼70% yR7s et ∼30% pR7s. Le profil d’expression de ss, et donc le ratio entre pR7 et yR7 dans la rétine, est fixé par les interactions entre les allèles ss. Différents allèles ou combinaisons d’allèles modifient le profil d’expression de ss et, à son tour, le ratio des sous-types R7. Un élément régulateur du gène ss, agissant comme un isolateur, est nécessaire pour amener deux allèles à proximité, même s’ils sont situés sur des chromosomes différents [14, 15]. Cette proximité entre les allèles permet vraisemblablement à d’autres régions régulatrices du locus d’interagir en trans pour définir le profil d’expression de ss dans la rétine. Bien que les détails moléculaires des interactions interalléliques ne soient pas bien compris, cet exemple montre que la transvection ne dépend pas nécessairement de l’appariement chromosomique en soit. Cependant, l’appariement chromosomique facilite la transvection en rapprochant les allèles homologues. En outre, il illustre de quelle manière la transvection peut contribuer à un mécanisme stochastique qui génère la diversité du destin cellulaire dans la rétine.
4. Rôle de la transvection dans la régulation sexuellement dimorphique de l’expression des gènes du chromosome X
Le gène yellow (y) de D. melanogaster a longtemps été un cas d’école pour étudier la transvection, principalement en utilisant la complémentation intragénique [16]. Cependant, on ne savait pas si les interactions régulatrices entre les allèles de y, qui ne peuvent se produire que chez les femelles puisque y est lié au chromosome X, contribuent à réguler le niveau ou le profil d’expression de y dans la condition sauvage. y est nécessaire à la production de pigment noir dans tout le corps et son profil d’expression est sexuellement dimorphique dans plusieurs tissus et parties du corps. Nous avons récemment découvert que chez D. biarmipes, une espèce étroitement apparentée à D. melanogaster dont les mâles possèdent une tache de pigmentation au bout des ailes, le profil d’expression sexuellement dimorphique de y dans l’aile est contrôlé par transvection. L’expression de y dans l’aile est contrôlée par deux séquences régulatrices voisines de type « enhancers » situées en 5’ du gène : l’enhancer de l’aile (ou « wing ») qui conduit à une faible expression uniforme dans toute l’aile, et l’enhancer « de la tâche » (ou « spot ») qui entraîne une forte expression dans la zone de l’aile où la tâche de pigmentation se formera (Figure 1A–C) [17]. Chez les femelles, l’activité de l’enhancer spot — mais pas celle de l’enhancer wing — est réduite au silence par transvection (Figure 1D–G). Chez les mâles hémizygotes, qui n’ont qu’un seul chromosome X, la transvection ne peut pas se produire et l’enhancer spot est actif. Il est important de noter que lorsque le locus y est inséré sur un autosome (un chromosome non sexuel), à la même position sur chaque chromosome homologue, l’interaction interallélique peut avoir lieu dans les deux sexes. Cela montre que la répression de l’enhancer spot chez les femelles n’est pas due à un mécanisme de régulation spécifique au chromosome X, ou spécifique aux femelles, mais bien à l’interaction entre les allèles. L’inactivation de l’enhancer spot dépendante de la transvection nécessite l’intron du gène y, ce qui suggère qu’il contient un élément régulateur nécessaire à l’interaction interallélique, ou qui, après l’interaction, éteint l’enhancer spot, ou les deux. Il est remarquable qu’un autre enhancer sexuellement dimorphique, actif dans les segments abdominaux postérieurs des mâles, et situé à proximité de l’enhancer spot, ne soit pas contrôlé par la transvection mais par la hiérarchie canonique de détermination du sexe [18]. La façon dont deux éléments régulateurs du même gène, séparés par seulement quelques centaines de paires de bases, en sont venus à utiliser des stratégies de régulation complètement différentes pour obtenir une expression similaire spécifique aux mâles, soulève des questions intrigantes sur le plan mécanique et évolutif.
Régulation sexuellement dimorphique du gène yellow dans l’aile de D. biarmipes. L’enhancer spot contrôle l’expression de y dans les ailes mâles en activant sa transcription possiblement par la formation d’une boucle (A) ou un mécanisme de liaison (B), résultant en un profil d’expression qui préfigure la forme du motif de pigmentation (ou spot) (C). Dans les ailes femelles, l’activité de l’enhancer spot est réduite au silence par trois mécanismes possibles : une topologie en boucle du locus dépendant de l’appariement des chromosomes homologues qui rapproche l’enhancer spot d’un répresseur lié à la séquence de l’intron (D). Des interactions protéine–protéine initiées au niveau de l’intron pourraient favoriser la formation d’une frontière entre l’enhancer spot et l’enhancer wing, empêchant l’interaction entre l’enhancer spot et le promoteur de y (E). Enfin, l’appariement des allèles de y pourrait créer un microenvironnement local qui recrute des protéines répressives pour bloquer l’activité de l’enhancer spot (F). Ces hypothèses ne sont pas nécessairement mutuellement exclusives. Dans l’aile femelle, certaines cellules échappent de manière aléatoire à l’extinction de l’enhancer spot, ce qui conduit à un motif poivre et sel de niveaux élevés de y (G). La figure a été créée à l’aide de Biorender.com.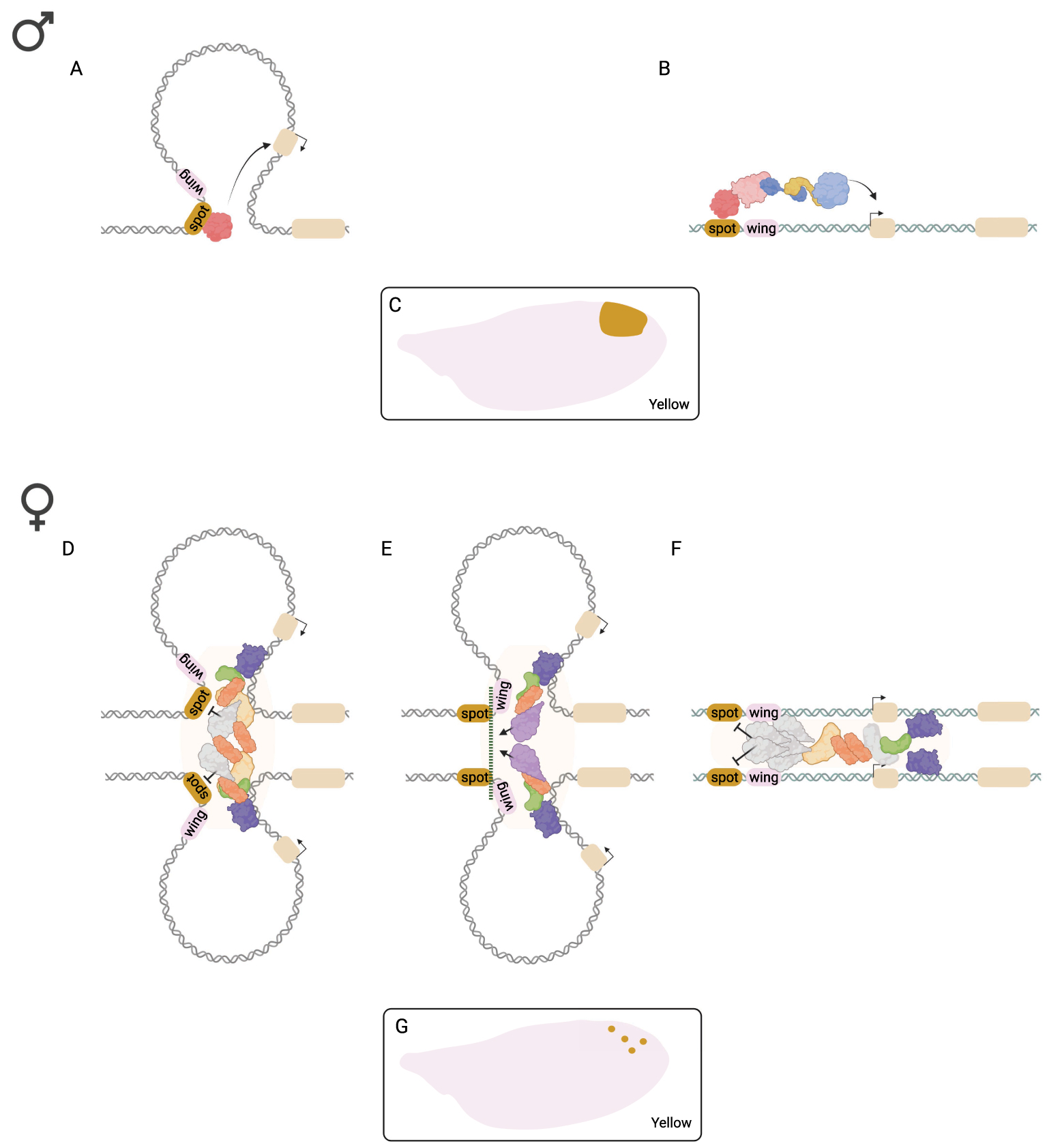
5. Éléments mécanistiques de la transvection
La transvection a pu être confondue avec l’appariement chromosomique, en particulier chez la drosophile où l’appariement complet entre chromosomes homologues est si fortement associé à la transvection. Pourtant, si la transvection nécessite une proximité étroite entre les allèles pour une interaction fonctionnelle, l’appariement chromosomique complet n’est pas nécessairement le seul moyen de rapprocher deux allèles. Ainsi, la dissection des mécanismes sous-jacents à la transvection doit distinguer les déterminants de l’association physique d’allèles homologues des événements de régulation qui suivent cette association et qui ont un impact sur l’expression des gènes. Dans les sections suivantes, nous discuterons de ce que l’on sait de chacune de ces deux étapes, en nous appuyant principalement sur des études sur la drosophile.
5.1. L’appariement somatique homologue établit l’association physique d’allèles homologues
En 1908, Nettie M. Stevens a observé pour la première fois que les chromosomes homologues sont complètement appariés chez plusieurs espèces de diptères [11]. Contrairement à l’appariement homologue observé pendant la méiose, qui est nécessaire à la ségrégation et à la recombinaison des chromosomes, la raison d’être de l’appariement homologue dans les tissus somatiques observé pendant l’interphase est inconnue. Chez D. melanogaster, l’appariement des chromosomes homologues apparaît progressivement au cours de l’embryogenèse. L’initiation de l’appariement somatique homologue a lieu à peu près au même moment où les TAD apparaissent, dans les derniers cycles nucléaires syncytiaux, ce qui conduit à un degré d’appariement plus élevé observé dans le cycle nucléaire 14 [19, 20]. L’ensemble du processus coïncide avec l’activation du génome zygotique. Les données d’imagerie sur tissu vivant ont révélé la dynamique de l’appariement somatique homologue dans l’embryon précoce, montrant que les loci homologues sont séparés de 2,2 μm avant l’appariement, et de seulement 0,4 μm après l’appariement. De plus, cette analyse révèle qu’une fois établi, l’appariement homologue est globalement très stable, et caractérisé par de faibles changements dynamiques de la distance entre les loci homologues [21].
Le modèle le plus accepté pour l’initiation de l’appariement est que certaines régions de la chromatine forment des sortes de boutons, des régions de haute affinité d’appariement distribuées le long des chromosomes. Elles trouvent leurs homologues sur les chromosomes homologues par une exploration aléatoire dans l’espace nucléaire [15, 21]. La propagation de l’appariement se produit alors par un mécanisme de « fermeture éclair » d’un bouton à l’autre sur toute la longueur du chromosome [21].
Les études de capture de conformation de la chromatine de type Hi-C tenant compte des haplotypes ont révélé que les chromosomes homologues de D. melanogaster sont organisés en régions d’appariement serré et régions d’appariement plus lâche [22, 23]. On ne sait pas dans quelle mesure les régions d’appariement serré et d’appariement lâche sont identiques entre les différents types de cellules, bien que les boutons semblent avoir des contributions à l’appariement variable selon les types cellulaires [15].
Le modèle de boutons peut expliquer l’initiation et la propagation de l’appariement entre chromosomes homologues. Il est possible que la plupart des paires de boutons individuels fournissent une faible énergie d’interaction qui, à elle seule, n’est pas suffisante pour entraîner l’appariement de tout le chromosome et que la densité des boutons sur la longueur du chromosome soit un déterminant majeur de la dynamique d’appariement [21]. L’appariement ectopique, entre chromosomes non homologues, peut être induit chez la drosophile entre des transgènes chromosomiques relativement grands (∼100 kb) et leurs régions homologues, ce qui suggère que l’appariement entre n’importe quelle séquence homologue nécessite un nombre important de boutons « faibles » [15]. À l’autre extrémité du spectre d’affinité, certaines régions courtes (Fab-7, Mcp) suffisent à assurer l’appariement, même lorsqu’elles sont insérées sur des chromosomes non homologues [24, 25], ce qui suggère que des boutons diffèrent par leurs affinités. Certaines situations semblent intermédiaires entre les régions à haute affinité et les boutons individuels « faibles ». Par exemple, nous avons montré que les copies homologues de y, qui est un gène assez compact (∼10 kb), peuvent toujours s’associer physiquement et interagir fonctionnellement même si elles sont séparées par 5 Mb sur le même chromosome, mais elles ne peuvent pas interagir lorsqu’elles sont insérées sur des chromosomes non homologues [18]. L’ensemble de ces résultats suggère que l’interaction physique entre les régions homologues est orchestrée par des séquences boutons qui ont des affinités et des densités différentes, conférant une capacité d’appariement faible à forte à chaque région en fonction de leur distribution sur la longueur des chromosomes.
5.2. Facteurs de transvection et isolateurs
On ne sait pas si les mêmes facteurs qui établissent et/ou maintiennent l’appariement affectent aussi directement la transvection. On ne sait pas non plus s’il existe des facteurs génériques qui affectent directement tous les cas de transvection, ou si des facteurs distincts sont associés à des loci distincts. Plusieurs facteurs ont été décrits pour leur rôle dans la transvection chez la drosophile via l’appariement des chromosomes homologues. Initialement, il a été montré que deux facteurs, Su(Hw) et Top2, favorisaient l’appariement des chromosomes dans les cellules somatique [26, 27]. De manière surprenante, il a été démontré que Cap-H2, une sous-unité du complexe Condensin II, s’oppose à l’appariement somatique, indiquant que l’état d’appariement des chromosomes dans un noyau est le résultat de l’équilibre entre des facteurs qui promeuvent ou au contraire empêchent l’appariement [28]. Des cribles génétiques dans des embryons et des cellules de D. melanogaster ont permis d’identifier plus de 100 gènes qui favorisent ou inhibent l’appariement des chromosomes homologues. Certains de ces gènes sont impliqués dans le contrôle du cycle cellulaire, ce qui suggère un lien entre l’organisation chromosomique et la progression du cycle cellulaire [29, 30].
Un grand nombre des facteurs qui, lorsqu’ils sont dérégulés, affectent la transvection sont des protéines isolatrices. Les protéines isolatrices sont une classe diversifiée de protéines architecturales de la chromatine qui se lient ou sont associées aux séquences d’ADN isolatrices, identifiées à l’origine comme des segments d’ADN qui bloquent la communication entre un enhancer et son promoteur lorsqu’ils sont placés entre eux. Il a également été démontré que les séquences isolatrices peuvent s’apparier en cis, rapprochant ainsi leurs séquences voisines. Plus généralement, les séquences isolatrices, et les complexes protéiques qui s’y lient, jouent un rôle central dans la régulation de l’organisation de la chromatine, des boucles locales aux structures chromatiniennes d’ordre supérieur [31]. Plus important encore, différentes combinaisons de protéines isolatrices semblent être enrichies au niveau des boutons, telles que CTCF, GAF, BEAF-32, Cp190 et Mod(mdg4), qui sont également enrichies dans les limites des TAD, ce qui suggère que l’appariement homologue et la formation de TAD pourraient être des phénomènes liés [15]. En outre, les régions d’appariement serrées sont enrichies en protéines architecturales et isolatrices [23].
Les transgènes contenant le rétrotransposon gypsy (qui contient une séquence isolatrice), ou bien les séquences isolatrices Wari, 1A2 ou Fab-8 peuvent favoriser la transvection à de nombreux endroits du génome, ce qui suggère qu’une fonction générale de ces séquences est de faciliter la transvection [32, 33]. La visualisation des interactions entre les régions homologues au locus Abd-B [34, 35], ou en utilisant des transgènes contenant un enhancer du gène snail, indique que le rôle des séquences isolatrices est de stabiliser l’interaction entre les allèles homologues [36], ce qui est cohérent avec leur surreprésentation dans les séquences boutons [15]. Il a été démontré que les interactions entre différents types de séquences isolatrices favorisent la transvection [33]. Ces séquences isolatrices semblent être liées par les mêmes facteurs, ce qui suggère que la combinaison de ces facteurs est à l’origine de ces interactions.
La manière précise dont les séquences d’ADN isolatrices et les protéines qui s’y attachent influencent la transvection n’est pas claire. Par exemple, Mod(mdg4), un cofacteur d’isolateur classique, est nécessaire pour la régulation négative dépendante de la transvection du gène y dans l’aile [18]. Cependant, on ne sait pas ce que Mod(mdg4) fait précisément. Une possibilité est qu’il soit impliqué dans des changements topologiques au locus y, soit en établissant un pont entre un répresseur liant l’intron et l’enhancer spot (Figure 1D), soit en formant une frontière forte entre l’enhancer spot et l’enhancer wing qui préserverait l’activité dans l’aile et bloquerait l’interaction entre le spot et le promoteur (Figure 1E). Une étude plus approfondie de ce système, et d’autres, sera nécessaire pour mieux comprendre comment les séquences d’ADN isolatrices et les protéines architecturales contribuent à la transvection.
5.3. La transvection pourrait créer des microenvironnements nucléaires distincts
Les enhancers homologues sont généralement en compétition pour accéder aux promoteurs de chaque allèle, et habituellement, l’interaction cis (enhancer et promoteur sur le même chromosome) est favorisée par rapport à l’interaction trans (enhancer et promoteur sur des chromosomes homologues différents). Cependant, lorsque les loci sont appariés, la concurrence en cis peut être réduite. En effet, Lim et ses collègues ont observé, en utilisant l’imagerie sur tissu vivant, qu’un seul enhancer partagé a un accès égal aux deux promoteurs lorsque les loci sont appariés grâce à l’action des séquences d’ADN isolatrices [36]. Ce résultat est cohérent avec l’idée que l’association étroite de loci homologues peut créer un microenvironnement nucléaire local dans lequel les facteurs de transcription et les cofacteurs sont enrichis et partagés entre les enhancers homologues. Les environnements nucléaires peuvent avoir un effet positif ou négatif sur l’activité des enhancers et l’expression des gènes, en fonction des facteurs qu’ils recrutent. En appliquant ce modèle à la régulation de l’enhancer spot de y, on peut imaginer que l’interaction entre les deux allèles crée un microenvironnement local qui facilite le recrutement coopératif de facteurs réprimant l’activité de ce enhancer (Figure 1F).
6. Conclusions et perspectives
Bien que la transvection ait été décrite il y a près de 70 ans, sa signification pour l’expression des gènes dans des contextes physiologiques est longtemps restée une question ouverte. Nous disposons maintenant de plusieurs exemples établissant que la transvection joue un rôle clé dans le contrôle des profils d’expression génique chez la drosophile, ce qui plaide en faveur de la pertinence de ce mécanisme de régulation génique. Au-delà de la génétique de la drosophile, la transvection et les phénomènes apparentés peuvent également être plus courants qu’on ne le pense actuellement. L’association conceptuelle de la transvection avec l’appariement des chromosomes homologues peut avoir conduit à l’idée erronée que la transvection est un phénomène spécifique à la drosophile. L’absence d’appariement de chromosomes homologues peut défavoriser les interactions interalléliques, mais ces interactions peuvent néanmoins se produire de manière transitoire et affecter la régulation des gènes. Une illustration en est donnée par la régulation d’Oct4, qui est fortement exprimé dans les cellules souches embryonnaires chez les mammifères. Au cours du développement, les allèles homologues d’Oct4 se rapprochent, conduisant à la répression d’Oct4, qui marque la sortie de l’état pluripotent de ces cellules [10]. Cet exemple suggère qu’il doit exister d’autres situations chez les mammifères où les interactions entre allèles influencent l’expression des gènes.
Le fonctionnement de la régulation génique interchromosomique reste un champ de recherche largement ouvert. Nous suggérons qu’il pourrait y avoir un continuum mécanistique entre la régulation interchromosomique des gènes homologues et non-homologues, et que des principes généraux sous-tendent ces différents phénomènes. La transvection et les phénomènes apparentés reposent sur la proximité entre les gènes en interaction. Pourtant, on ignore encore dans quelle mesure les loci doivent être proches pour interagir fonctionnellement. Les mesures les plus précises de la distance entre des loci homologues (mais pas nécessairement en interaction fonctionnelle) dans l’embryon de drosophile montrent qu’ils sont séparés de 0,4 μm [21]. Cela reste une distance considérable à franchir pour que les facteurs de transcription se lient à la région régulatrice d’un allèle et agissent sur l’allèle homologue. D’autres mesures de la distance et de la dynamique associée des allèles en interaction fonctionnelle sont nécessaires, ainsi qu’une évaluation de la manière dont ces propriétés laissent leur empreinte sur l’expression des gènes. Il est également crucial de comprendre les conséquences de l’appariement allélique sur le positionnement du locus dans le noyau ou sur les changements topologiques au niveau du locus. L’utilisation de technologies de capture de la conformation des chromosomes tenant compte des haplotypes et l’imagerie à super-résolution nous aideront certainement à mieux caractériser ce qui se passe lorsque des allèles ou des régions régulatrices homologues se rapprochent, et comment ils s’influencent mutuellement.
Remerciements
CCG remercie l’EMBO pour son financement (297-2021). BP remercie l’ERC pour son financement dans le cadre du septième programme-cadre de l’Union européenne (FP/2007-2013)/accord de subvention 615789, ainsi que le CNRS pour son soutien.
Conflit d’intérêt
Les auteurs n’ont aucun conflit d’intérêt à déclarer.




 CC-BY 4.0
CC-BY 4.0