1. Introduction
Malaria is an acute debilitating illness that is caused by parasitic protozoans from the genus Plasmodium that are transmitted by Anopheles mosquitoes. According to the latest report of the World Health Organization (WHO), the disease accounted for 241 million cases and 627,000 deaths worldwide in 2020. The estimated number of malaria deaths stood at 627,000 in 2020 (an increase of 69,000 deaths over the previous year). Sub Saharan African countries continue to carry a disproportionately high share of the global malaria burden, with 95% of all malaria cases and 96% of deaths. Children under 5 years of age accounted for about 80% of all malaria deaths in this region [1].
Among the species responsible for human infection, Plasmodium vivax is the most geographically widespread and the leading cause of disease outside Sub Saharan Africa, in Southeast Asia, South America and Northeast Africa [2]. P. vivax has a complex life cycle consisting of the cyclical infection of a human host and a female Anopheline mosquito vector. Following the bite of an infected mosquito, sporozoites migrate through the dermis into capillaries, reach the liver and invade hepatocytes [3]. While most sporozoites undertake an asexual maturation into schizonts, some of them diverge to form quiescent hypnozoites. These hypnozoites can emerge weeks or months after the primary infection and give rise to relapsing episodes of malaria [4]. When schizonts eventually rupture, they release merozoites that migrate into the bloodstream and invade reticulocytes. They develop into rings, trophozoites and then schizonts [2]. Again, schizonts rupture and release new merozoites out of the infected reticulocytes to invade healthy ones. This results in a continuous round of asexual multiplications increasing the number of parasites [5]. Alternatively, some parasites differentiate into male or female gametocytes. When they infect new mosquitoes, they undergo fertilization inside the midgut and produce ookinetes that mature into oocysts. These oocysts release sporozoites that migrate to salivary glands and can be inoculated into new hosts by female mosquitoes during a bloodmeal. Thereby, P. vivax life cycle is perpetuated (Figure 1) [6].
P. vivax life cycle. The life cycle of P. vivax involves the cyclical infection of a Anopheline mosquito vector and a human host. When a malaria-infected mosquito takes a blood meal, it injects sporozoites into the human host. They migrate through the dermis to the liver and invade hepatocytes. They mature into tissue schizonts and establish a stock of latent hypnozoites. When schizonts rupture, they release merozoites that migrate to the bloodstream and invade reticulocytes. They develop into rings, trophozoites and blood-stage schizonts. When the schizonts rupture, they lyse the infected reticulocytes and release new merozoites. A small proportion of the parasite population differentiates into male and female gametocytes. When a mosquito takes a new blood meal, it ingests gametocytes. In the midgut, the microgametocytes and macrogametocytes fertilize, undertake sexual division, and transform into ookinetes that mature into oocysts. When the oocysts rupture, they release sporozoites that migrate to the salivary glands to perpetuate the life cycle. Figure created with BioRender.com.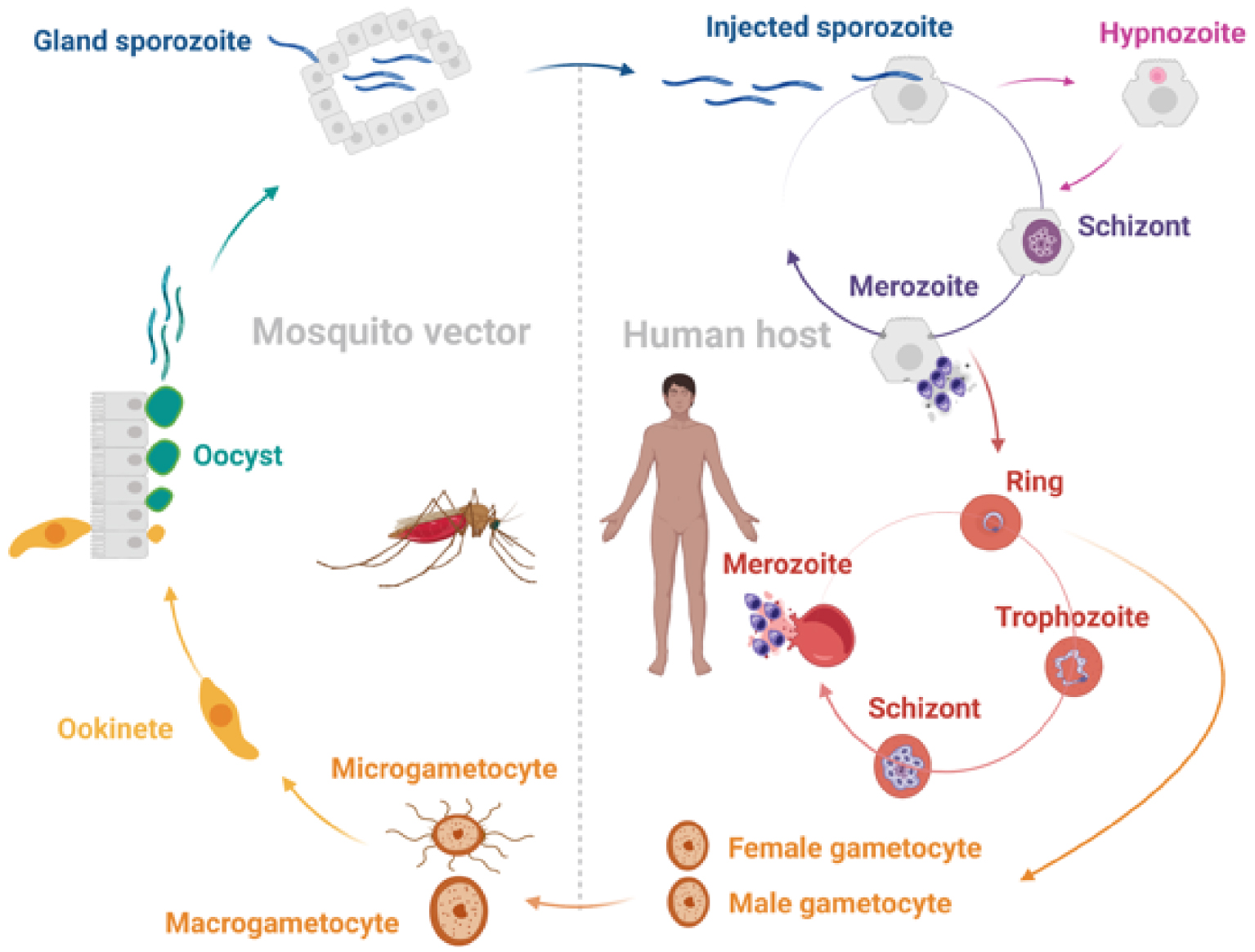
While it may be cryptic, P. vivax is the most widely distributed cause of malaria across the globe [7]. Indeed, its ability to develop at low temperatures (18–25 °C) provides potential for infectivity and transmission in both tropical and temperate areas [8]. Interestingly, the intra-erythrocytic cycle of P. vivax is characterized by low parasitemia and asynchrony, which contribute to missed diagnoses [9]. Low parasitemia, commonly observed in P. vivax infection, is probably due to the unique tropism of P. vivax merozoites for reticulocytes and the specific physiological and/or metabolic needs of the parasite for blood-stage growth and survival [2]. In absence of biological evidence, a model for invasion by P. vivax has been proposed, based on observations during red blood cell invasion by P. falciparum or P. knowlesi merozoites. It suggests that during the invasion process, P. vivax expresses adhesins to bind receptors on the surface of reticulocytes and establish an initial and reversible attachment. This results in the deformation of the reticulocyte membrane and the apical reorientation of merozoites. Then, they activate organelles and release invasion-related proteins to form a tight and irreversible junction [10]. The merozoites are propelled by an actin–myosin motor and the junction moves from the apical to posterior pole. That forms the parasitophorous vacuole in which the parasites will develop after the invasion pore and reticulocyte membrane are re-sealed [11, 12] (Figure 2 and Table 1 for details).
P. vivax invasion mechanisms within Duffy-positive reticulocytes. (1) Primary attachment & Reorientation—when P. vivax merozoites are released in the blood circulation, the primary attachment involves PvMSPs. Other proteins such as PvTRAg56.2 and PvGAMA may play a role in the adhesion and stabilization of the parasite. (2) Ligand binding & Receptors—the PvDBP is gradually coupled to DARC (region II) to create a stable heterotetramer consisting of two PvDBP molecules and two DARC molecules. The recognition and selection of reticulocytes for invasion are mediated by PvRBPs, notably through the interactions PvRBP2b-CD71 and PvRBP2a-CD98. In absence of biological evidence and based on observations during red blood cell invasion by P. falciparum or P. knowlesi merozoites, we suppose that many P. vivax proteins including PvMSPs, PvRBSA and other proteins may play a role to ensure P. vivax apical reorientation. (3) Tight junction & Engagement—P. vivax merozoites penetrate the reticulocytes via glideosome activity. Proteins such as PvAMA1 and PvRON might lead either directly or indirectly to the formation of a tight and irreversible junction and mediate invasion. Several P. vivax surface proteins may be rapidly degraded whereas others such as PvTRAg36.6 and PvETRAMP may be expressed to form a parasitophorous vacuole and support the development of the parasite. Of note, the P. vivax merozoite invasion mechanisms described here are so far largely limited to a comparison with P. falciparum and P. knowlesi invasion homologs and a handful of known P. vivax invasion gene families. The role of P. vivax ligands present in purple bold font have been confirmed, while the others present in black font are only suspected. Figure created with BioRender.com.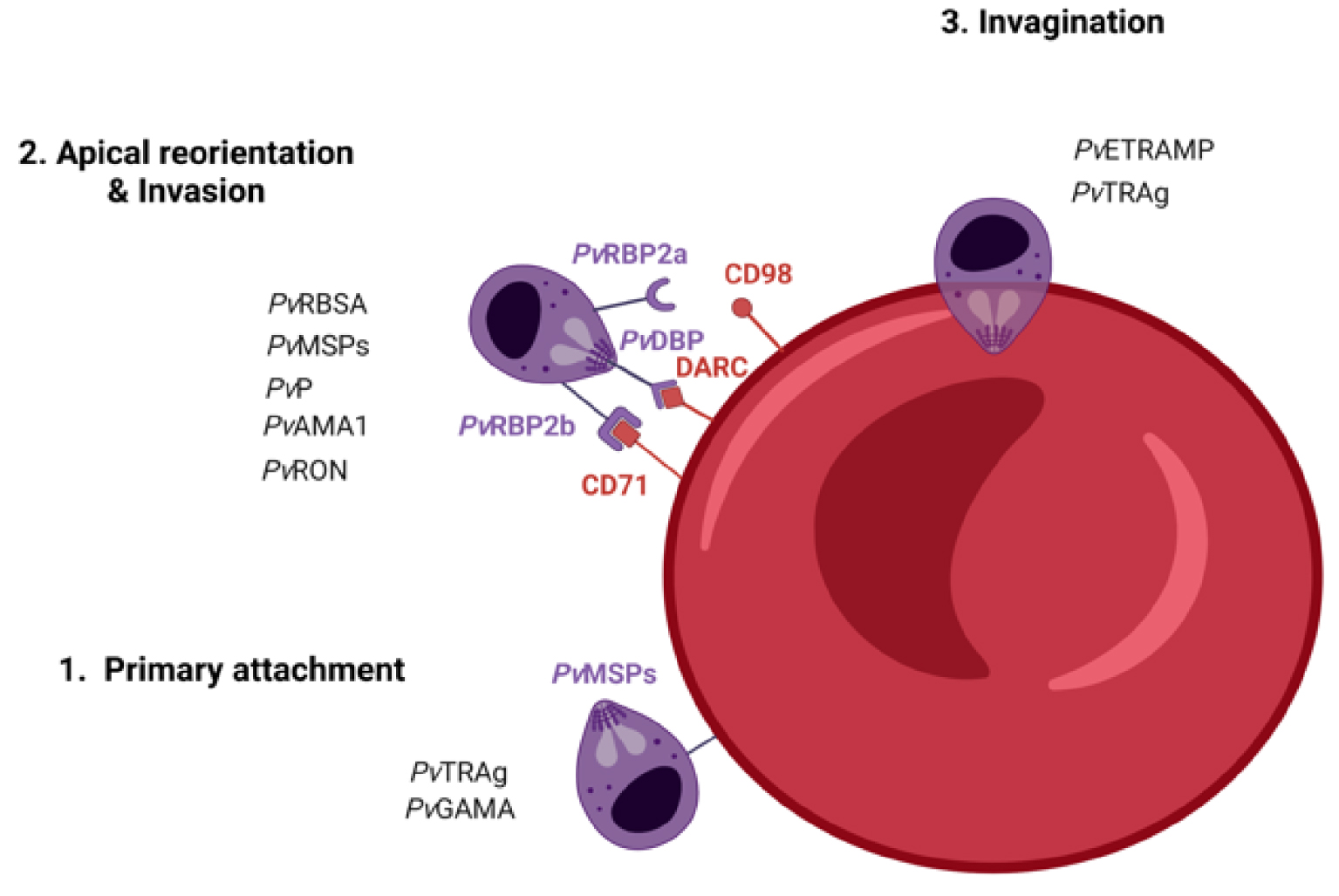
For many years, scientists have sought to identify the key host-parasite interactions leading to the invasion of human reticulocytes by P. vivax. Between 1920 and 1960, clinical and field trials carried out with individuals of African or African American ancestry demonstrated that they were highly resistant to vivax malaria when challenged with human blood or mosquitoes infected with limited numbers of P. vivax strains [13, 14, 15, 16]. Following the identification of the Duffy blood groups [17, 18], it was established that the natural resistance to vivax infection was related to the absence of Duffy antigen receptors for chemokines (DARC) on the surface of human erythrocytes [19]. The molecular basis of Duffy-negativity was later demonstrated as a single point mutation in the GATA1 (tct >cct) sequence located in the 5′ promoter region of the ackr1 gene [20, 21]. Seminal works with controlled experimental infections of volunteers through sporozoite challenges [19] and in vitro studies using P. knowlesi (a zoonotic malaria parasite closely related to P. vivax) as a model [22] subsequently confirmed that DARC was required as a receptor for P. vivax merozoites to invade erythrocytes. In parallel, the parasite ligand that specifically binds DARC was identified as P. vivax Duffy binding protein (PvDBP) [23]. Hence, the scientific paradigm by which P. vivax exclusively invades Duffy-positive erythrocytes through the interaction between PvDBP and DARC was established and vivax malaria was assumed to be seldom transmitted or even absent in regions where Duffy-negativity is predominant such as Sub Saharan Africa [19]. However, over the last two decades, many studies have reported cases of P. vivax infection within Duffy-negative individuals from Sub Saharan countries such as Angola [24], Benin [25], Cameroon [26, 27], Equatorial Guinea [24, 28], Ethiopia [29], Kenya [30], Madagascar [31], Mali [32], Mauritania [33] and Sudan [34]. Although such unexpected observations are still scarce, they raise questions about the actual invasion mechanisms used by P. vivax to infect Duffy-negative individuals. They also challenge common assumptions such as the central role played by the PvDBP-DARC interaction in blood-stage infections, the limit of detection of current diagnostic tools and the evolution of host-pathogen interactions [35]. At present, we do not know whether these cases represent a pre-existing phenomenon that was overlooked in the past or a newly evolved DARC-independent invasion pathway that could lead to an expansion of vivax malaria within Duffy-negative populations [36].
Because the collection of field samples from remote endemic areas is difficult, time demanding and expensive, research has long been restricted to direct sampling from patient donors or the study of closely related Plasmodium species such as P. knowlesi [37, 38] or P.cynomolgi [39]. Furthermore, the development of reproducible long-term in vitro culture methods is still impaired by P. vivax low parasitemia and strong tropism for reticulocytes [40].
Nevertheless, recent omics technologies have revolutionized vivax malaria research with the development of innovative and affordable techniques enabling a more comprehensive characterization of P. vivax genes, transcripts, and proteins. This review aims to provide broad insights into P. vivax invasion mechanisms enabled by genomics, transcriptomics, and proteomics and to illustrate the importance of integrated multi-omics studies.
2. P. vivax molecular mechanisms of invasion
In 2004, the P. vivax genome sequencing project provided the first reference sequence P. vivax Salvador-1 (Sal-1) using a whole genome shotgun strategy [41, 42]. In the last twenty years, a few additional field and clinical P. vivax isolates have been sequenced through microarray, high-depth sequencing, or whole genome sequencing. All these sequences are publicly available on online database such as PlasmoDB (https://plasmodb.org/plasmo/app) and are regularly annotated. Overall, the analysis of these P. vivax sequences has contributed to the identification of potential invasion-related genes.
List of P. vivax invasion-related ligands characterized by omics technologies
| Step | Pv ligands | Accession numbers | Role (confirmed/ potential) | Erythrocyte receptor | Vaccine candidates (blood stage) | Evidence from | ||
|---|---|---|---|---|---|---|---|---|
| Genomics | Transcriptomics | Proteomics | ||||||
| Primary attachment & reorientation | MSP1 | PVP01_0728900 | Confirmed | Yes | pvmsp1 is highly polymorphic (SNPs) [43] | Among all genes, pvmsp1 is one of the most expressed gene in P. vivax schizonts [44] | Based on the study of homologous PfMSPs in P. falciparum, it has been hypothesized that PvMSP1 may recognize a cogent receptor on the surface of reticulocytes [45] | |
| MSP7 | PVP01_1219500, PVP01_1219700, PVP01_1219400, PVP01_1219600, PVP01_1220300, PVP01_1220300, PVP01_1220400, PVP01_1219900 | Potential | pvmsp7 is highly polymorphic (SNPs) [46]. pvmsp3 presented copy number variations ranging from 1–4 copies | pvmsp7 transcript peaks during the late trophozoite-schizont transition [47]. pvmsp3 transcripts were detected in very late schizonts [5]. Among all genes, pvmsp9 is one of the most expressed gene in P. vivax schizonts [44] | Based on homology with PfMSP7, PvMSP7 may be a specific ligand for the receptor P-selectin [48] | |||
| GAMA | PVP01_0505600 | Potential | pvgama displays a size polymorphism [49] | Functional assays: PvGAMA may play a role in the adhesion of P. vivax merozoites to reticulocytes [49] and binds to Band3 and ANK1 [50] | ||||
| MSP1P | PVP01_0728800 | Potential | PvMSP1P may play an important role in the adhesion of P. vivax merozoites to reticulocytes through interaction with its C-terminal region [43] | |||||
| TRAg56.2 | PVX_088850 | Potential | The pvtrag gene family is seldom polymorphic [51] | Single cell RNA sequencing: different expression levels for different members of the pvtrag gene family [5] | Immunoprecipitation and mass spectrometry: PvTRAg56.2 seems to co-localize with PvMSP1 and interact with PvMSP7 to play a stabilizing role [52] | |||
| Ligand bindings | DBP | PVP01_0623800 | Confirmed | DARC | Yes | pvdbp is highly polymorphic (SNPs, CNVs) [53] | pvdbp gene amplification was associated with an increase in messenger RNA (mRNA) level, suggesting a potential increase in PvDBP protein level [54] | Binding and functional assays |
| EBP | PVP01_0102300 | Potential | pvebp is highly polymorphic but less than pvdbp (SNPs, CNVs) [55] | pvebp transcripts were found to be highly expressed during P. vivax schizont stage [55, 56] | PvEBPII binds preferentially immature Duffy-positive reticulocytes, but a minimal binding was observed in immature Duffy-negative reticulocytes [57, 58] . | |||
| RBP2b | PVP01_0800700 | Confirmed | CD71 | pvrbp2a has conserved regions but is relatively polymorphic [59, 60] | Ex vivo culture system: the interaction PvRBP2b-CD71 plays a crucial role in the recognition of reticulocytes [61] | |||
| RBP2a | PVP01_1402400 | Confirmed | CD98 | pvrbp2a has conserved regions but is relatively polymorphic [59, 60] | Binding assays: CD98 is the cogent receptor of PvRBP2a and the interaction PvRBP2a-CD98 is involved in the recognition of reticulocytes [62] | |||
| RBSA | PVP01_0004240 | Potential | pvrbsa presents less genetic diversity than other P. vivax merozoite surface genes [63] | PvRBSA specifically binds to Duffy-positive reticulocytes, but its cogent receptor is unknown [63] | ||||
| P12 | PVP01_1136400 | Potential | The PvP protein family is expressed on the surface of P. vivax merozoites and may play a role in the invasion of reticulocytes [64] | |||||
| Tight junction formation | AMA1 | PVP01_0934200 | Potential | Yes | Functional assays: conserved functions for PfAMA1 and PvAMA1 [65]. May be involved with PvRON2 in the formation of the tight junction [66] | |||
| RON | PVP01_0916600 | Potential | Identification of pvron4 by homology to pfron4 and pkron4 in P. vivax VCG-1 [67] | pvron4 presents a similar transcription pattern to the one reported for pfron4 [67], pvron5 transcripts were found in schizonts [66] | Indirect immunofluorescence and immunoassays: PvRON4 may be expressed in P. vivax VCG-1 apical pole and may participate in the formation of a tight junction [67]. PvRON5 has a marked binding preference for reticulocytes and is thus suspected to be involved in the invasion process [66] | |||
| ETRAMP | PVP01_0532300 | Potential | PvETRAMP could be involved in the formation of parasitophorous vacuole | |||||
| TRAg36.6 | PVX_112690 | Potential | DNA sequencing: the pvtrag gene family is seldom polymorphic [51] | Single cell RNA sequencing: different expression levels for different members of the pvtrag gene family [5] | Immunoprecipitation and mass spectrometry: PvTRAg36.6 seems to colocalize with PvETRAMP to form and/or maintain the parasitophorous vacuole membrane [52] | |||
| Unknown function | MSA180 | PVP01_0814200 | Potential | Among all genes, pvmsa180 is one of the most expressed gene in P. vivax schizonts [44] | ||||
| P92 | PVP01_1107900 | Potential | ||||||
| RAMA | PVP01_0107500 | Potential | Among all genes, pvrama is one of the most expressed gene in P. vivax schizonts [44] | |||||
| RBP1a | PVP01_0701200 | Potential | DNA sequencing: genetic diversity of pvrbp1a seems to be low [68] | pvrbp1 transcripts were found to be highly expressed during P. vivax schizont stage [55, 56] | Native PvRBP1 and PvRBP2 may function as an adhesive protein complex [69] | |||
| RBP1b | PVP01_0701100 | Potential | DNA sequencing: genetic diversity of pvrbp1b seems to be low [68] | pvrbp1 transcripts were found to be highly expressed during P. vivax schizont stage [55, 56] | Native PvRBP1 and PvRBP2 may function as an adhesive protein complex [69] | |||
| RBP2c | PVP01_0534300 | Potential | DNA sequencing: pvrbp2c is highly polymorphic [51, 70, 59] | |||||
| RBP1p1 (partial) | ? | Potential | DNA sequencing: identification of the partial gene pvrbp1p1 | |||||
| RBP2p1 (partial) | PVP01_0534400 | Potential | DNA sequencing: identification of the partial gene pvrbp2p1 | |||||
| RBP2p2 (partial) | PVX_101590 | Potential | DNA sequencing: identification of the partial gene pvrbp2p2 | |||||
| RBP2d (pseudo) | PVP01_1471400 | Potential | DNA sequencing: identification of the pseudogene pvrbp2d | |||||
| RBP2e (pseudo) | PVP01_0700500 | Potential | DNA sequencing: identification of the pseudogene pvrbp2e | |||||
| RBP3 (pseudo) | PVP01_1469400 | Potential | DNA sequencing: identification of the pseudogene pvrbp3 | |||||
| RhopH3 | PVP01_0703800 | Potential | Among all genes, pvrhoph3 is one of the most expressed gene in P. vivax schizonts [44] | |||||
| RON5 | KP026121 | Potential | The pvron5 transcript is found in schizonts in P. vivax VCG-1 [66] | Binding, Western blot and immunofluorescence assays: PvRON5 is expressed in P. vivax apical pole and preferentially binds reticulocytes [66] | ||||
Accession numbers starting from PVP01 are from PvP01 reference strain while accession numbers starting from PVX are from Sal-I reference strain (https://plasmodb.org/plasmo/app). Accession numbers starting from KP are from VCG-1 reference strain (https://www.ncbi.nlm.nih.gov).
2.1. The crucial role of pvdbp gene in the invasion of Duffy-positive reticulocytes
The P. vivax Duffy Binding Protein (pvdbp) gene encodes the invasion-related ligand P. vivax Duffy Binding Protein (PvDBP) [71]. The pvdbp gene displays coding sequences for a N-terminal signal peptide domain, a conserved cysteine-rich Duffy binding domain referred to as region II, another conserved cysteine-rich domain referred to as region VI, followed by a transmembrane domain and a C-terminal cytoplasmic tail [72]. Based on current high throughput sequencing data, our appreciation of genetic diversity is less and less limited by the nature and/or density of markers studied along the genome. Notably, the recent publication of more than two hundred P. vivax genomes showed that the pvdbp gene sequences were highly polymorphic irrespective of their geographic origin. Interestingly, many non-synonymous single nucleotide polymorphisms (SNPs) were found in the DARC binding domain region II (PvDBPII) [53]. The diversity and localization of these SNPs suggest that the pvdbp gene may be under differential selective pressure due to antibody responses and its function in receptor-binding and invasion [51].
In addition, copy number variations (CNVs) at the pvdbp locus were found to be one of the most common gene expansions to occur across multiple alleles worldwide [73]. While the detection of CNVs relied, in the past, on labour intensive and costly techniques such as in situ hybridization, recent advances in high throughput sequencing enabled their direct detection based on sequencing data. Notably, whole genome sequencing approaches revealed two distinct duplications in parasites from Madagascar and Cambodia. In Madagascar, P. vivax isolates from Duffy-positive and Duffy-negative patients exhibited a common duplication with two copies of pvdbp next to each other [74]. In Cambodia, a different duplication of the pvdbp gene was commonly detected in P. vivax isolates from Duffy-positive patients. Furthermore, the proportion of P. vivax isolates carrying the duplication was found to be significantly higher in homozygous Duffy-positive patients than in heterozygous Duffy-positive patients who are expected to exhibit a lower expression of DARC on the surface of their reticulocytes. However, the latter analysis could not completely rule out that patients could carry multiple P. vivax clones while the estimation of CNVs by quantitative PCR can only reflect the major clone [53]. More recently, a geographical survey demonstrated that these two distinct duplications of pvdbp were circulating in Asia, South America, and East Africa. While Duffy negativity is prevalent in some regions, duplications of the pvdbp gene were also largely found in regions where Duffy negativity was almost absent [51]. In contrast, a concurrent genomic analysis showed that no duplication of pvdbp was found in one hundred eighty-two P. vivax isolates from eleven endemic countries. However, it was noticed that this discrepancy was probably due to the primers used for CNV validation by PCR [75].
Despite the specificity and distribution of the duplications of pvdbp, there was no evidence that P. vivax genomes bypassed the barrier of Duffy negativity through this genetic variation. Larger expansions of pvdbp were also detected worldwide. They clustered separately but presented similar patterns. High haplotype diversity was also observed between the copies while the 5′ and 3′ flanking sequences displayed moderate diversity [76]. In the Asia-Pacific region, sequencing of more than two hundred P. vivax isolates from Duffy-positive patients revealed the common presence of two to three copies of the pvdbp gene [73]. In Ethiopia, sequences of two P. vivax isolates from Duffy-negative patients exhibited the presence of three and eight copies of the pvdbp gene [57]. Later on, another common expansion of the pvdbp gene, quite similar to those previously described in Madagascar and Cambodia but ranging from three to five copies, was also detected [51]. These findings have therefore allowed to hypothesize that the expansion of pvdbp may allow for mutation arising in the second copy. Such expansions may facilitate the binding to an alternative erythrocytic receptor with a lower affinity for PvDBP ligand [57]. However, there is no evidence of localized-selected CNVs that would enhance invasion efficiency and induce selective pressure. Since the invasion of Duffy-positive erythrocytes depends on the interaction between PvDBP and DARC, it is likely that the expansions of pvdbp may be driven by genetic variations in DARC or inhibitory antibody responses to PvDBPII [75]. Nonetheless, it is unclear whether these expansions do provide an enhanced ability to the parasite to invade Duffy-negative erythrocytes.
2.2. The potential role of pvebp gene in an alternative invasion pathway
The P. vivax Erythrocyte Binding Protein (pvebp) gene was discovered through de novo assembly of one P. vivax isolate from Cambodia. It encodes the putative P. vivax Erythrocyte Binding Protein (PvEBP) ligand and is present in most P. vivax genomes. The organization of the pvebp gene is similar to the pvdbp gene with an N-terminal signal peptide, a Duffy binding like domain referred to as region II, another cysteine-rich domain referred to as region VI followed by a transmembrane domain and cytoplasmic tail [55]. Despite these similarities, pvebp appears to cluster separately from all known Plasmodium Duffy binding protein like genes. DNA sequencing of ten P. vivax genomes from Asia, South Pacific, Africa, Central and South America showed that pvebp is polymorphic though less diverse than pvdbp. Indeed, the pvebp gene displayed only 11 polymorphic sites in 2412 positions while the pvdbp gene displayed 32 polymorphic sites in 3213 positions for the same samples. Most of the SNPs detected were non-synonymous [55]. Furthermore, the genomic analysis of two hundred nineteen P. vivax isolates from Duffy-positive patients in Cambodia and Madagascar confirmed that pvebp was less diverse than pvdbp (12 vs. 33 alleles observed from the same isolates) but presented only non-synonymous mutations indicating the existence of a strong selective pressure. Indeed, eleven non-synonymous point mutations were found in pvebp sequences and led to twelve different alleles. Some SNPs were observed in both countries whereas others were specific to Cambodia and Madagascar. These SNPs were distributed across the Duffy binding like domain [53]. Just as for pvdbp, the diversity and distribution of SNPs in pvebp reflected the likely existence of a selective pressure in the pvebp gene though it seemed to operate at a lower level than in the pvdbp gene [58].
The genomic analysis of P. vivax isolates from Cambodia, Madagascar and Ethiopia also revealed the existence of copy number variations in the pvebp gene. In Madagascar, pvebp expansions were observed, ranging from two to five copies. A bimodal distribution of parasites with a specific population presenting more than three copies was found. However, no significant difference in susceptibility to vivax infection was observed between homozygous and heterozygous Duffy-positive individuals. In Cambodia, pvebp expansions ranged from one to two copies. The proportion of P. vivax isolates with multiple gene copies was more important in high transmission areas [53]. Overall, a high proportion of P. vivax isolates with multiple gene copies was found in Madagascar where Duffy-negativity and Duffy-positivity coexist as compared to Cambodia where Duffy-negativity is virtually absent. In contrast, no pvebp expansion was found in Ethiopia where Duffy-negativity is also frequent [51]. Considering the similarities observed between the pvdbp and pvebp genes, the polymorphism and expansion of pvebp may correlate with the expression of a new ligand interacting with erythrocyte membrane proteins other than DARC and therefore contribute to an alternative invasion pathway [55]. However, no evidence that such variations confer any abilities to infect Duffy-negative individuals have been provided so far.
2.3. The role of pvrbp and pvrbsa genes in reticulocyte selectivity
The P. vivax Reticulocyte Binding Protein (pvrbp) gene family was identified based on the annotation of adhesive ligands of P. vivax genome [69]. Genome sequencing of several P. vivax isolates identified eleven members in the pvrbp gene family including five full-length genes (pvrbp1a, pvrbp1b, pvrbp2a, pvrbp2b and pvrbp2c), three partial genes (pvrbp1p1, pvrbp2p1 and pvrbp2p2) and three pseudogenes (pvrbp2d, pvrbp2e and pvrbp3) [77]. The pvrbp genes are constituted of two exons with one exon encoding a signal peptide at the N-terminal region and another exon encoding a transmembrane domain at the C-terminal region [56]. Recent investigations reported that their genetic diversity seems to be generally low, except for the pvrbp2 genes [68]. The analysis of P. vivax isolates from China, Thailand and Ethiopia confirmed that pvrbp2c was highly polymorphic [51, 70, 59]. Although pvrbp2a and pvrbp2b presented highly conserved regions, the genomic analysis of P. vivax isolates from Papua New Guinea and Thailand showed that they were also relatively polymorphic [59, 60]. As observed for the pvdbp and pvebp genes, non-synonymous SNPs clustered around three main sites including the reticulocyte binding domain. This may reflect the functional importance of this binding site and the existence of a selective pressure [60].
Furthermore, CNVs affecting both pvrbp2a and pvrbp2b were observed in two P. vivax isolates from Thailand. While a strain presented duplication of both genes (pvrbp2a and pvrbp2b), the other strain had two copies of pvrbp2b only [59]. These findings suggest that the pvrbp genes may encode ligands involved in the recognition of target cells. The P. vivax reticulocyte binding surface antigen (pvrbsa) gene has also been identified as a gene potentially involved in the recognition of reticulocytes though it displays less genetic diversity than other P. vivax merozoite surface genes [63].
2.4. The potential role of pvmsp genes in primary attachment
The P. vivax merozoite surface protein (pvmsp) gene family encodes multiple antigenic determinants that are suspected to mediate the primary attachment between P. vivax merozoites and human reticulocytes [11]. These pvmsp genes are highly polymorphic. In particular, pvmsp1 [43], pvmsp3 [43, 78] and pvmsp7 [46] exhibit numerous non-synonymous SNPs indicating that they may be under selective immune pressure [51]. Analysis of P. vivax isolates from Ethiopia revealed that pvmsp3 presented copy number variations ranging from one to four copies. Interestingly, the expansions of pvdbp and pvmsp3 were often observed as tandem copies. The functional relevance of this expansion of copy numbers is still not known.
2.5. The other genes and their potential role in P. vivax invasion pathway
Other P. vivax genes are suspected to be involved in the invasion process. For instance, the P. vivaxtryptophan-rich antigen (pvtrag) gene family is thought to encode thirty-six proteins that can bind to human erythrocytes [79]. The genomic analysis of P. vivax strains from Ethiopia revealed that the pvtrag gene family seems to bear only a few genetic polymorphisms [51]. Similarly, the P. vivax glycosylphosphatidylinositol-anchored micronemal antigen (pvgama) gene encodes the P. vivax glycosylphosphatidylinositol-anchored micronemal antigen (PvGAMA) that binds erythrocytes regardless of Duffy antigen status [43]. This protein may play a role in the adhesion of P. vivax merozoites to its target cells [49]. Finally, the P. vivaxrhoptry neck protein (pvron) gene family encodes for P. vivaxrhoptry neck proteins (PvRON) that may participate in the formation of the tight junction as demonstrated for the P. falciparum homologs [67].
Genomic technologies clearly contributed to refine vivax malaria research by reducing the cost of sequencing as well as improving the quality and availability of sequences. However, only a few hundred P. vivax genomes have been sequenced so far. Field and clinical isolates present low parasitemia and high human DNA contamination that still make the acquisition of sufficient amount of high-quality parasite DNA difficult. Yet, the available genomic sequences have enabled the identification of a few potential invasion-related genes. Single nucleotide polymorphisms and copy number variations found in these genes may lead to structural differences and result in selective pressures, but the functional consequences of these variations remain unknown. Analysis of P. vivax genomes should be combined with the identification of their transcriptomic profiles and characterization of their putative proteins to detect specific signatures, which may provide insights into function. But the lack of in vitro culture methods for P. vivax makes functional analysis difficult.
3. Transcription of P. vivax invasion-related genes
Constant advances of high throughput sequencing associated with the recent ability to mature one cycle ex vivo isolates have opened the way to transcriptomic studies. The profiling of P. vivax blood stage transcriptome through microarray and RNA sequencing (bulk and at single cell level) is progressively unveiling new experimental abilities and elucidating the transcription, function, and regulation of invasion-related genes.
3.1. Stage-specific expression of P. vivax invasion-related genes
Generating high quality transcriptomes across multiple P. vivax isolates is essential to explore the changes in gene expression throughout the intraerythrocytic cycle and leading to invasion. Recently, the single cell transcriptomics profiling in P. vivax isolates from bloodstream infections of Aotus and Saimiri monkeys revealed that most of the genes had a stage-specific expression and were strongly over-expressed, in contrast to housekeeping genes [5]. Given that most clinical infections were asynchronous, the analysis of P. vivax transcriptomes identified distinct populations. Rings and early trophozoites mainly expressed genes involved in transcription and translation whereas late trophozoites and schizonts possibly expressed invasion-related genes [5]. For instance, the pvebp and pvrbp1 transcripts were found to be highly expressed during P. vivax schizont stage [55, 56]. Similarly, the pvmsp3 transcripts were detected in very late schizonts [5] and the pvmsp7 transcript generally peaked during the late trophozoite-schizont transition, when merozoites are being formed [47]. In the primate model-adapted P. vivax strain (VCG-1), the pvron5 transcripts were found in schizonts [66]. Moreover, RNA sequencing experiments conducted with ex vivo P. vivax isolates from Brazil showed a wide variation in the expression of genes that were suspected to be involved either in the host-parasite interaction, reticulocyte selectivity or invasion. In particular, pvmsp1, pvmsp9, P. vivaxmerozoite surface antigen 180 (pvmsa180), P. vivaxrhoptry-associated membrane antigen (pvrama) and P. vivaxrhoptry H3 (pvrhoph3) were the most expressed among all genes [44]. The expression of invasion-related genes in late schizonts implies the key importance of merozoite development and reticulocyte invasion for P. vivax survival.
3.2. Selective expression of P. vivax invasion-related genes
The current P. vivax genome contains many gene families that were defined based on sequence homology and conservation of amino acid domains. Single-cell RNA sequencing analyses demonstrated that selective gene expression may be due to its specific role or additional functions. While most of the pvmsp genes are expressed during late developmental stages, pvmsp3 was found to be exclusively expressed in very late schizonts suggesting its role in the invasion process. More surprisingly, a pvmsp7 like gene appeared to be exclusively expressed in gametocytes implying a functional difference [5]. Comparable results were reported using clinical P. vivax isolates from Thailand. While the expression of most of the pvmsp7 genes was associated with schizonts, the pvmsp7a and pvmsp7f genes maintained high expression levels throughout the entire intraerythrocytic cycle. Such findings suggest that these paralogs may have additional functions besides their established role on the surface of merozoites [47]. Similarly, different expression levels were observed among members of the pvtrag gene family but their significance is unknown [5]. Despite their sequence homology, members of a gene family may also have different functions at specific time points [5]. Finally, a directional and amplification-free transcriptome (DAFT) sequencing of four P. vivax isolates from Cambodia revealed that transcription may be a selective process where the parasites would select particular members of a gene family in response to the host [80]. The authors showed that genes that were the most variably expressed among isolates physically clustered along chromosomal regions. For instance, the pvmsp3 locus presented many paralogs which were likely to be of functionally redundant thus increasing antigenic diversity and enhancing immune evasion during the invasion process. Within clusters, gene expression appears to arise as if parasites were choosing the gene version to be expressed within a given host [80]. Overall, these findings reflect a developmental regulation and functional differentiation within specific gene families.
3.3. Amplification of P. vivax invasion-related genes
In Plasmodium species, gene amplification may provide a simple way to increase gene expression and thereby changing invasion phenotypes. Previous genomic studies demonstrated that some P. vivax strains had amplification of invasion-related genes. Notably, the pvdbp gene is highly variable in the number of gene copies. For instance, transcriptomic profiling of schizonts from Cambodian P. vivax isolates revealed that the pvdbp gene amplification was associated with an increase in messenger RNA (mRNA) level, suggesting a potential increase in Pv DBP protein level [54]. Given that PvDBP protein plays a crucial role in the invasion of Duffy-positive reticulocytes, it was hypothesized that the parasites capable of infecting Duffy-negative reticulocytes may have been selected for their ability to use multiple copies of pvdbp in order to bind an alternative receptor [69]. However, P. vivax strains with multiple copies of pvdbp have also been commonly detected in regions where Duffy-negativity is rare. On another note, even though pvebp presents many similarities with pvdbp, there is no evidence that its amplification leads to increased mRNA and/or protein levels [53]. Thus, the functional consequences of invasion-related gene expansion are uncertain and require further investigations with a high number of P. vivax isolates.
3.4. A complex and dynamic regulation system
Transcriptomic data from individual parasites suggested that P. vivax exhibited a tightly regulated transcriptional cascade during its intraerythrocytic cycle [81]. Investigations showed that co-expressed genes presented conserved motifs, and potential sites for regulatory binding proteins [82]. For instance, long 5′ and 3′ untranslated transcribed regions (UTRs) were observed in genes suspected to be involved in the invasion and the formation of parasitophorous vacuole [80]. In addition, variations of 5′-UTRs seemed to lead to a differential selection of transcription start sites (TSS) reflecting the dispersed patterns of transcription that could arise [83]. The presence of bidirectional promoters to regulate the expression of adjacent gene pairs has also been frequently observed. Lastly, post-transcriptional modifications such as chromatin structure, non-coding transcripts and alternative splicing may play an important role in regulation, but the mechanisms remain unclear. Notably, the putative spatial relationship between genes which are variably expressed suggests that chromatin structure and access may be important for gene regulation [83]. Thus, P. vivax has an elusive and complex regulatory network that may govern invasion-related genes and thereby allow the parasite to thrive within infected human reticulocytes.
Transcriptomics provides precious insights into the dynamics of P. vivax invasion-related genes. It is likely that P. vivax schizonts selectively express specific genes to initiate the invasion process. However, the small sample sizes, lack of reliable in vitro culture methods and paucity of constantly expressed genes complicate the identification of new transcripts and regulatory motifs. In the future, single-cell profiling and long read sequencing of P. vivax isolates from geographically distinct regions should be combined with proteomic approaches to assess the full diversity of invasion-related ligand expression.
4. P. vivax cellular invasion mechanisms
Proteomics data for P. vivax infections are minimal due to the limited access to biological material and lack of robust in vitro culture system. The list of proteins expressed in P. vivax merozoites has thus not been clearly defined. This has generated critical knowledge gaps in the understanding of P. vivax invasion mechanisms. Given that the basic structure of merozoites is similar across Plasmodium species, most of the researchers based the investigation of P. vivax invasion mechanisms on the hypothesis that the parasite may use the same steps despite having different cell tropism and engaging different receptor-ligand interactions.
4.1. Primary attachment
The invasion process may be initiated by the reversible attachment of P. vivax merozoites to reticulocytes. This binding may be mediated by P. vivax Merozoite Surface Proteins (PvMSPs), which may form large complexes constituted of glycophosphatidylinositol anchored proteins and transmembrane proteins covering the surface of P. vivax merozoites. Based on the study of homologous PfMSPs in P. falciparum, it has been hypothesized that PvMSP1 may recognize a cogent receptor on the surface of reticulocytes. The C-terminal sub-unit (MSP-142) could be cleaved and most of the complex may be shed, leaving only a small C-terminal sub-unit (MSP-119) on the surface. Similarly, its paralog PvMSP1P may play an important role in the adhesion of P. vivax merozoites to reticulocytes through interaction with its C-terminal region [43]. Furthermore, it has been hypothesized that PvMSP7 may be a specific ligand for the receptor P-selectin which acts as a cell adhesion molecule [48] and that PvMSP1 and PvMSP7 could bind together to form a multi-protein complex that enhances invasion efficiency [47].
Other ligands may be involved in this step, but their ability to trigger attachment or abrogate recognition by the host immune system are unknown. For instance, the PvTRAg56.2 protein seems to co-localize with PvMSP1 on the merozoite surface and interact with PvMSP7 to play a stabilizing role [52]. In the same vein, the micronemal PvGAMA protein may bind to specific receptors on the surface of reticulocytes and thus play a role in the adhesion of P. vivax merozoites to their target host cells [65]. Indeed, the proteomic analysis of ankyrin 1 (ANK1) and Band 3 by various biochemical approaches, including liquid chromatography-mass spectrometry, showed that the fragment 2 of the membrane binding domain of ANK1 and the extracellular loop 5 of Band 3 bind to PfGAMA and PvGAMA. In addition, anti-Band 3-P5 antibodies block the binding of PfGAMA to erythrocytes [50]. Furthermore, field and laboratory studies showed that P. vivax exposure induce robust IgG responses to PvGAMA in natural exposure [84, 85] and that anti PvGAMA antibodies inhibit in vitro P. knowlesi invasion and growth [86], although knockout of the pkgama gene had a minimal effect on P. knowlesi growth. Thus, the primary attachment of P. vivax merozoites to reticulocytes or erythrocytes involves specific and reversible interactions to guarantee an efficient encounter.
4.2. Apical reorientation
Following primary attachment, P. vivax may undergo apical reorientation which triggers various ligand-receptor interactions. As P. vivax merozoites have a strong tropism for reticulocytes (0.2–2% of the total erythrocyte population), the chances to encounter a cell permissive to invasion are low. Thus, it was hypothesized that a host cell selection step occurs during apical reorientation to prevent the merozoites from invading non-target cells [87]. Based on the study of homologous proteins in other Plasmodium species, the PvRBP protein family is believed to mediate P. vivax tropism for reticulocytes. Previous works showed that native PvRBP1 and PvRBP2 may function as an adhesive protein complex [69] and PvRBP2a and PvRBP2b can bind reticulocytes independently of their Duffy phenotype [60]. In 2015, an ex vivo study confirmed that P. vivax merozoites preferentially target immature reticulocytes expressing the marker CD71 [61]. In 2018, another study showed that CD71 was the cogent receptor for the ligand PvRBP2b [88]. Analyses by cryo-electron microscopy, immunoprecipitation and fluorescence resonance energy transfer confirmed that the binding of PvRBP2b to CD71 is specific and directly proportional to the level of CD71 expression [89]. Finally, functional studies identified the marker CD98, expressed on the surface of immature reticulocytes, as the cogent receptor of PvRBP2a [62]. Both interactions PvRBP2b—CD71 and PvRBP2a—CD98 seem to drive the strict tropism of P. vivax for reticulocytes. Their action may be independent or combined, suggesting the potential existence of alternative invasion pathways. Apart from the PvRBP proteins, the P. vivax reticulocyte binding surface antigenic adhesin (PvRBSA) may also play a role in the recognition of reticulocytes. A recent study showed that the recombinant PvRBSA (rPvRBSA) binds specifically to Duffy-positive reticulocytes. But its cogent receptor is still unknown [63]. In addition to their adhesive function, PvMSP proteins may also help in the recognition of reticulocytes. Indeed, recent investigations showed that peptides from PvMSP1 have higher binding activity to reticulocytes than erythrocytes [45]. Although the mechanisms remain unclear, the recognition of immature reticulocytes by P. vivax merozoites seems to be governed by PvRBP2a and/or PvRBP2b, potentially in conjunction with PvRBSA and PvMSPs. The PvRBP proteins may also trigger subsequent steps of invasion such as the modulation of erythrocyte cytoskeleton, assembly of the tight junction or signalling for the secretion of rhoptry proteins.
4.2.1. Invasion
The apical reorientation is followed by the formation of a tight and irreversible junction between P. vivax merozoites and reticulocytes. Within Duffy-positive individuals, in absence of evidence, we do not know whether the P. vivax Duffy Binding Protein (PvDBP) localizes to the tight junction between merozoites surface and the receptor Duffy Antigen Receptor for Chemokines (DARC). Indeed, for P. falciparum, PfEBA175 (a P. falciparum erythrocyte-binding ligand) binds Glycophorin A to trigger release of PfRH5 and other rhoptry proteins, but PfEBA175 itself does not participate in junction formation. The PvDBP binding site is monomeric in the absence of DARC but dimerizes upon receptor engagement and forms a homodimer with the N-terminal extracellular domain of DARC [60]. The formation of the tight junction may also be facilitated by the secretion of the ligand P. vivax Apical Membrane Antigen 1 (PvAMA1) to the merozoite surface, the injection of P. vivax rhoptry neck protein (PvRON) complexes to the reticulocyte surface and their binding. Notably, it has been shown that PvRON5 has a marked binding preference for reticulocytes and is thus suspected to be involved in the invasion process [66]. Finally, the expression of various members of the P. vivax 6-cysteine protein (PvP) family on the merozoite surface is also thought to play a role in this step [64].
Within Duffy-negative individuals, the mechanisms and factors involved in the formation of the tight junction are unknown, but several hypotheses are currently discussed (Figure 3). In Madagascar, the evidence of vivax infections in Duffy-negative patients correlated with the existence of a common pvdbp gene duplication. This supported the hypothesis that the pvdbp gene amplification may enhance the binding of the PvDBP protein to another receptor with lower affinity. However, such an alternative receptor has not been identified yet. Further P. vivax strains presenting multiple pvdbp copies are frequently found in endemic zones where Duffy-negativity is rare [53]. A second hypothesis is that the PvEBP ligand may work as an alternate ligand for the invasion of Duffy-negative reticulocytes. Indeed, PvEBP appears to share structural similarities with PvDBP such as a Duffy binding site and a C-terminal domain with conserved cysteine positions, and likely similar functions [55]. Based on these structural similarities, a recombinant receptor binding site for PvEBP (rPvEBPII) was produced. Results of binding assays showed that PvEBPII binds preferentially immature Duffy-positive reticulocytes, but a minimal binding was observed in immature Duffy-negative reticulocytes. In contrast, no binding was observed for mature erythrocytes [57, 58]. Recently, a functional study performed on P. vivax isolates from Ethiopia confirmed that PvEBPII can bind both Duffy-positive and Duffy-negative reticulocytes though at a lower frequency than PvDBP. This finding indicate that PvEBPII may not have the same binding capacities as PvDBPII despite their structural similarities [51]. As the cogent receptor of PvEBP is still unknown, it is unclear whether this potential alternate ligand plays a role in the invasion of Duffy-negative reticulocytes. Finally, it was recently alleged that Duffy-negative reticulocytes may express low and/or transient levels of DARC on the surface of their reticulocytes. Preliminary data suggest that DARC may be expressed on the surface of erythroid precursor cells from Duffy-negative individuals but require to be confirmed [90].
Hypothetical P. vivax invasion mechanisms within Duffy-negative reticulocytes. Hypothesis 1. P. vivax merozoites invade Duffy-negative reticulocytes through the interaction between the ligand PvDBP and the receptor DARC implying that Duffy-negative reticulocytes may express DARC at low and/or transient level. Hypothesis 2. P. vivax merozoites invade Duffy-negative reticulocytes through the interaction between the ligand PvDBP and an unknown receptor expressed on the surface of Duffy-negative reticulocytes. Hypothesis 3. P. vivax merozoites invade Duffy-negative reticulocytes through the interaction between the ligand PvEBP and an unknown receptor expressed on the surface of Duffy-negative reticulocytes. Hypothesis 4. P. vivax merozoites invade Duffy-negative reticulocytes through the interaction between an unknown ligand expressed on the surface of P. vivax merozoites and an unknown receptor expressed on the surface of Duffy-negative reticulocytes. Figure created with BioRender.com.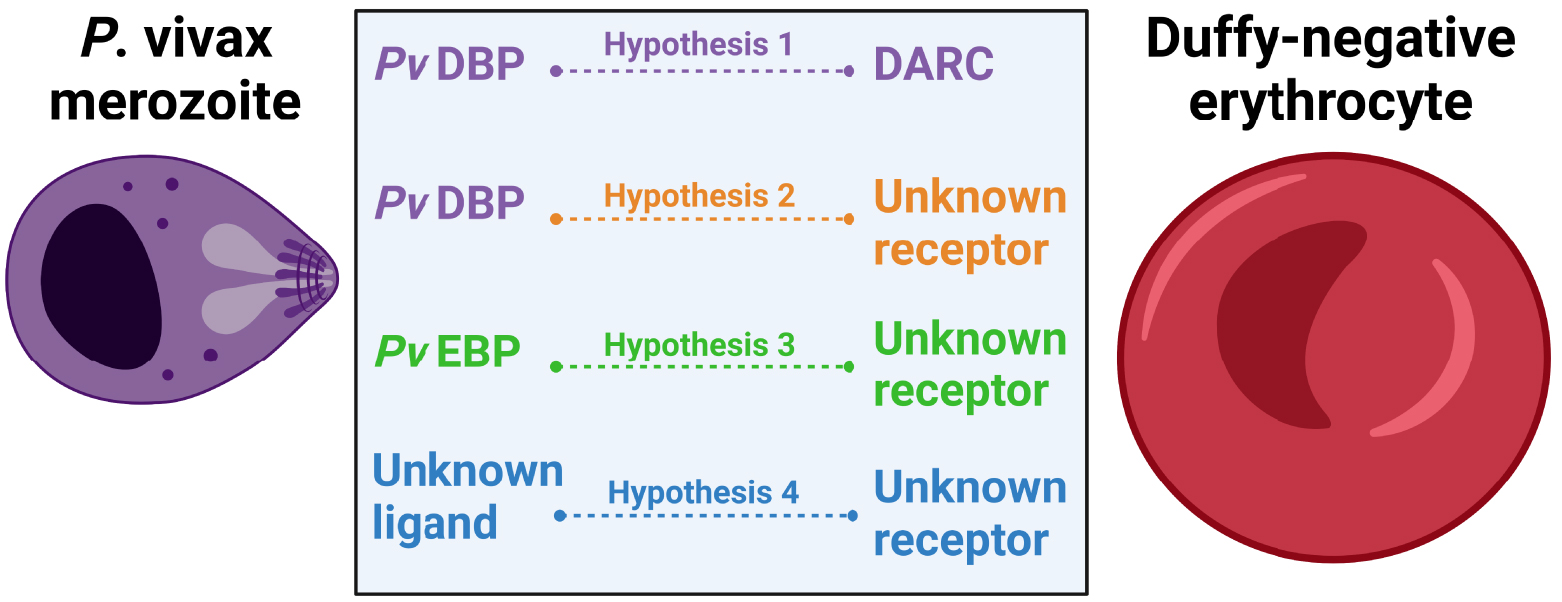
4.3. Invagination
Once P. vivax merozoites are irreversibly committed to invasion, they can penetrate the reticulocytes via glideosome activity and seal themselves within the parasitophorous vacuole. Although these mechanisms remain unclear, it seems that PvTRAg36.6 and P. vivax early transcribed membrane protein (PvETRAMP) colocalize in the apical region of the merozoites to form and/or maintain the parasitophorous vacuole membrane [52].
While proteomic technologies are not yet fully exploited, they seem essential to answer long-standing questions about P. vivax invasion mechanisms. The constant improvement of techniques (two-dimensional gel electrophoresis, mass spectrometry, yeast two-hybrids screens, etc.), high-throughput approach, computational prediction programs and data processing capabilities offers great promises to study the expression, interaction, and modification of P. vivax invasion-related ligands. However, proteomic data alone may be insufficient to establish a fine analysis of the invasion process and propose testable models. They require to be integrated into a more global and fundamental knowledge of P. vivax biology [91].
5. Conclusion
For a long time, vivax malaria has been considered a benign infection. Consequently, research on P. vivax received lower priority from researchers, policy makers and funding agencies. In addition, the limited access to biological material and lack of robust in vitro culture systems hampered the development of functional studies.
To address this issue, scientists have considered evidence from homologous protein families in other Plasmodium species and produced recombinant proteins to study P. vivax invasion-related ligands. But these methods have not fully provided comprehensive information on the actual functions of native ligands. In fact, the critical question for vivax malaria research cut across several disciplines. Indeed, genomics technologies shed some light on P. vivax genetic diversity. But they may not be sufficient to understand the functional consequences of these variations. While transcriptomic technologies are powerful tools to characterize P. vivax stage-specific and highly regulated transcripts, data analysis is limited by the small sample sizes and paucity in constantly expressed genes. However, recent applications of single-cell technologies have enabled to profile both liver-stage parasites and host cells to explore host-parasite interactions [92] and this technology could be applied to study host factors in the reticulocyte invasion pathway. In addition to omics studies, various in vivo models have been utilized for studying P. vivax invasion pathways. Non-human primate models (NHP) have been widely used in the past, despite their inherent limitations such as ethical concerns and the variations of host susceptibility to infection with P. vivax field isolates, necessitating adaption before replication, likely modifying the biological features of the adapted strains [55]. More recently, efforts have been made to develop humanized mouse systems as models to replicate in vivo P. vivax infections [93]. Although encouraging, this highly fragile and expensive system still remains in its infancy and needs further refinement to allow continuous maintenance of asexual blood stage infection and develop genetically modified P. vivax parasites [94].
In this context, proteomic technologies focus on deciphering the various interactions between P. vivax invasion-related ligands and cogent receptors. Nevertheless, they fail to provide a general picture of P. vivax invasion mechanisms, especially explaining how P. vivax is capable to invade Duffy-negative reticulocytes. Thus, the development of integrated and complementary multi-omics studies together with in vitro invasion studies should allow us to better understand the biology of this parasite and support the future implementation of prevention, control, and eradication strategies worldwide.
Conflicts of interest
Authors have no conflict of interest to declare.
Acknowledgements
This review was supported by a grant overseen by the French National Research Agency (VIPeRs, ANR-18-CE15-0026), by the Institut Pasteur, Paris, the Laboratoire d’Excellence (LabEx) “French Parasitology Alliance for Health Care” (ANR-11-15 LABX-0024-PARAFRAP) and the University of Strasbourg through the Programme IdEX 2022 (Attractivité — Dotation d’accueil des professeurs d’université recrutés en 2021).
Version française
1. Introduction
Le paludisme est une maladie débilitante aiguë causée par des parasites protozoaires du genre Plasmodium qui sont transmis par des moustiques Anophèles femelles. Selon le dernier rapport de l’Organisation mondiale de la santé (OMS), cette maladie a été responsable de 241 millions de cas et de 627,000 décès dans le monde en 2020. Le nombre estimé de décès dus au paludisme s’élevait à 627,000 en 2020 (soit une augmentation de 69,000 décès par rapport à l’année précédente). Les pays d’Afrique subsaharienne continuent de supporter une part disproportionnée de la charge mondiale du paludisme, avec 95% de tous les cas de paludisme et 96% des décès. Les enfants de moins de 5 ans représentent environ 80% de tous les décès dus au paludisme dans cette région [1].
Parmi les espèces responsables de l’infection humaine, Plasmodium vivax est la plus répandue géographiquement et la principale cause de maladie en dehors de l’Afrique subsaharienne, en Asie du Sud-Est, en Amérique du Sud et en Afrique du Nord-Est [2]. P. vivax présente un cycle de vie complexe consistant en l’infection cyclique d’un hôte humain et d’un moustique vecteur anophèle femelle. Après la piqûre d’un moustique infecté, les sporozoïtes migrent à travers le derme dans les capillaires, atteignent le foie et envahissent les hépatocytes [3]. Alors que la plupart des sporozoïtes entreprennent une maturation asexuée en schizontes, certains d’entre eux divergent pour former des formes quiescentes, les hypnozoïtes. Ces hypnozoïtes sont capables d’émerger des semaines voire des mois après la primo-infection pour donner lieu à des épisodes récurrents de paludisme [4]. Lorsque les schizontes finissent par se rompre, ils libèrent des mérozoïtes qui migrent dans la circulation sanguine et envahissent les réticulocytes. Ils se transforment en anneaux, en trophozoïtes, puis en schizontes [2]. Les schizontes se rompent et libèrent de nouveaux mérozoïtes qui vont envahir les réticulocytes sains. Il en résulte un cycle continu de multiplications asexuées, ce qui augmente progressivement le nombre de parasites dans la circulation sanguine [5]. Par ailleurs, certains parasites se différencient en gamétocytes mâles ou femelles. Lorsqu’un moustique prélève son repas sanguin chez un hôte infecté, les gamétocytes entreprennent une fécondation à l’intérieur de l’intestin moyen du moustique et produisent des ookinètes qui se transforment en oocystes. Ces oocystes libèrent ensuite des sporozoïtes qui migrent vers les glandes salivaires pour être inoculés chez de nouveaux hôtes par les moustiques femelles lors d’un repas sanguin. Ainsi, le cycle de vie de P. vivax se perpétue (Figure 1) [6].
Cycle de vie de P. vivax. Le cycle de vie de P. vivax implique l’infection cyclique d’un moustique vecteur Anophèle et d’un hôte humain. Lorsqu’un moustique infecté par P. vivax prend un repas sanguin, il injecte des sporozoïtes dans l’hôte humain. Ces sporozoïtes migrent à travers le derme jusqu’au foie et envahissent les hépatocytes. Ils mûrissent en schizontes tissulaires et établissent un stock d’hypnozoïtes latents. Lorsque les schizontes se rompent, ils libèrent des mérozoïtes qui migrent vers la circulation sanguine et envahissent les réticulocytes. Ils se développent en anneaux, en trophozoïtes et en schizontes intra-érythrocytaires. Lorsque les schizontes se rompent, ils lysent les réticulocytes infectés et libèrent de nouveaux mérozoïtes. Une petite proportion de la population parasitaire se différencie en gamétocytes mâles et femelles. Lorsqu’un moustique prend un nouveau repas sanguin, il ingère des gamétocytes. Dans l’intestin moyen, les microgamétocytes et les macrogamétocytes se fécondent, entreprennent une division sexuelle et se transforment en ookinètes qui mûrissent en oocystes. Lorsque les oocystes se rompent, ils libèrent des sporozoïtes qui migrent vers les glandes salivaires pour perpétuer le cycle de vie. Figure créée avec BioRender.com.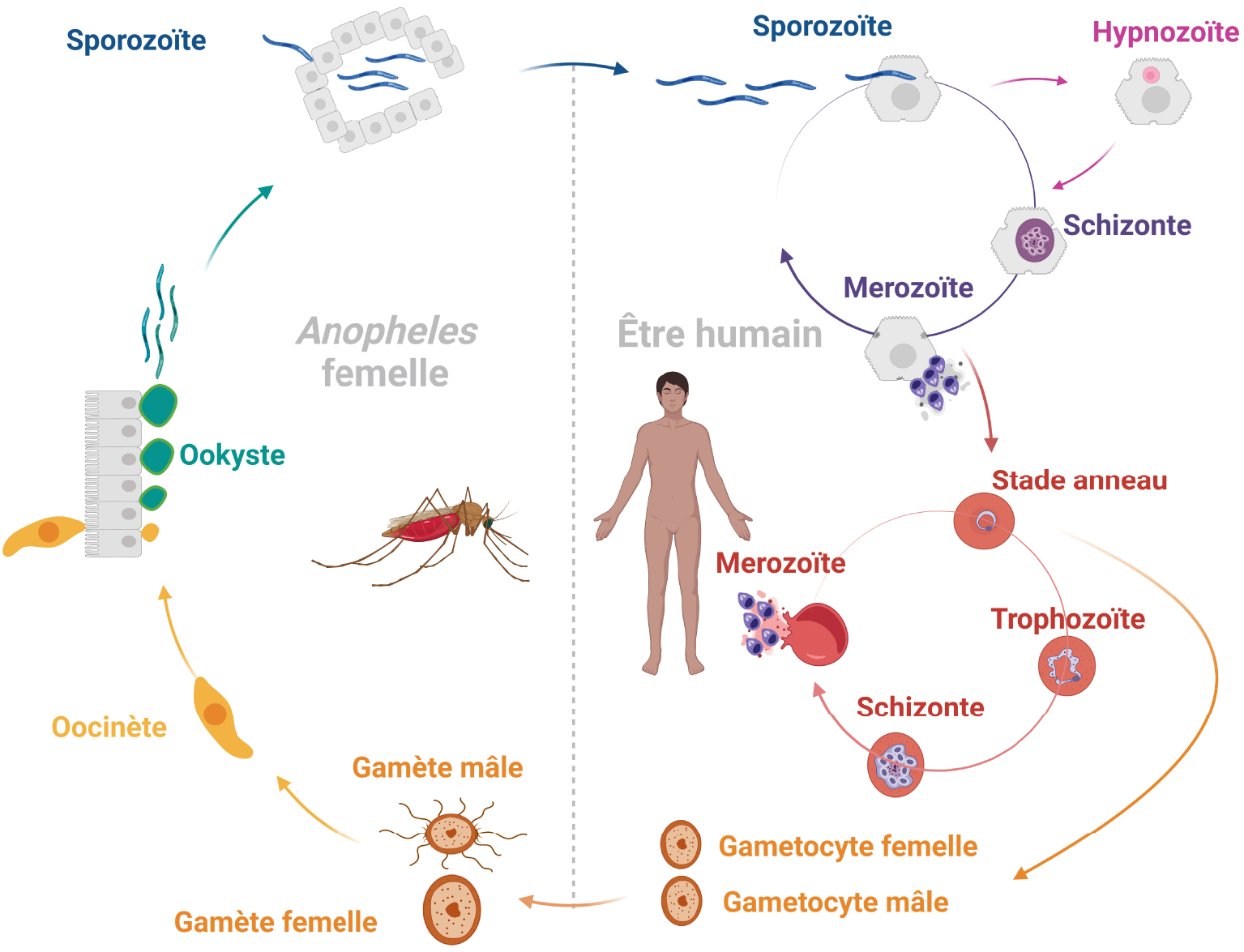
Bien que cryptique, P. vivax est la cause de paludisme la plus répandue dans le monde [7]. En effet, sa capacité à se développer à basse température (18–25 °C) lui confère un potentiel d’infectivité et de transmission dans les zones tropicales et tempérées [8]. Par ailleurs, son cycle intra-érythrocytaire est caractérisé par une faible parasitémie et une asynchronie, ce qui contribue probablement à des erreurs de diagnostic (faux négatifs) [9]. En effet, la faible parasitémie, couramment observée dans les infections à P. vivax, est probablement due au fort tropisme des mérozoïtes de P. vivax pour les réticulocytes et aux besoins physiologiques et/ou métaboliques spécifiques des stades sanguins du parasite pour sa croissance et sa survie [2]. En l’absence de solides preuves biologiques, un modèle d’invasion par P. vivax a été proposé, sur la base des observations faites lors de l’invasion d’érythrocytes humains par les mérozoïtes de P. falciparum ou P. knowlesi. Ce modèle suggère qu’au cours du processus d’invasion, P. vivax exprime des adhésines qui se lieraient à des récepteurs présents à la surface des réticulocytes et établiraient une fixation initiale et réversible. Cela entraînerait la déformation de la membrane des réticulocytes et la réorientation apicale des mérozoïtes. Ensuite, ils activeraient des organelles et libéreraient des protéines pour former une jonction serrée et irréversible [10]. Les mérozoïtes seraient propulsés par un moteur actine–myosine et la jonction se déplacerait du pôle apical au pôle postérieur. Cela formerait la vacuole parasitophore dans laquelle les parasites se développeraient après la fermeture du pore d’invasion et de la membrane réticulocytaire [11, 12] (Figure 2 et Tableau 1 pour les détails).
Mécanismes d’invasion de P. vivax dans les réticulocytes Duffy-positifs. (1) Attachement primaire et réorientation — Lorsque les mérozoïtes de P. vivax sont libérés dans la circulation sanguine, la fixation primaire implique les protéines PvMSPs. D’autres protéines telles que PvTRAg56.2 et PvGAMA pourraient jouer un rôle dans l’adhésion et la stabilisation du parasite. (2) Liaison au ligand et récepteurs — Le ligand PvDBP est progressivement couplé au récepteur DARC (région II) pour créer un hétérotétramère stable constitué de deux molécules de PvDBP et de deux molécules de DARC. La reconnaissance et la sélection des réticulocytes pour l’invasion sont médiées par les protéines PvRBPs, notamment par les interactions PvRBP2b-CD71 et PvRBP2a-CD98. En l’absence de preuves biologiques et sur la base des observations faites pendant l’invasion des érythrocytes par les mérozoïtes de P. falciparum ou de P. knowlesi, nous supposons que de nombreuses protéines de P. vivax, y compris les PvMSPs, les PvRBSAs et autres, pourraient jouer un rôle dans la réorientation apicale de P. vivax. (3) Jonction serrée et engagement — Les mérozoïtes de P. vivax pénètrent dans les réticulocytes via l’activité du glidéosome. Des protéines telles que PvAMA1 et PvRON pourraient conduire directement ou indirectement à la formation d’une jonction serrée et irréversible et servir de médiateurs à l’invasion. Plusieurs protéines de surface de P. vivax pourraient être rapidement dégradées alors que d’autres, comme PvTRAg36.6 et PvETRAMP, pourraient être exprimées pour former une vacuole parasitophore et favoriser le développement du parasite. Il est important de noter que les mécanismes d’invasion des mérozoïtes de P. vivax décrits ici sont jusqu’à présent largement limités à une comparaison avec les homologues d’invasion de P. falciparum et P. knowlesi et une poignée de familles de gènes d’invasion de P. vivax connues. Le rôle des ligands de P. vivax présents en caractères gras violets a été confirmé, tandis que les autres présents en caractères noirs sont seulement suspectés. Figure créée avec BioRender.com.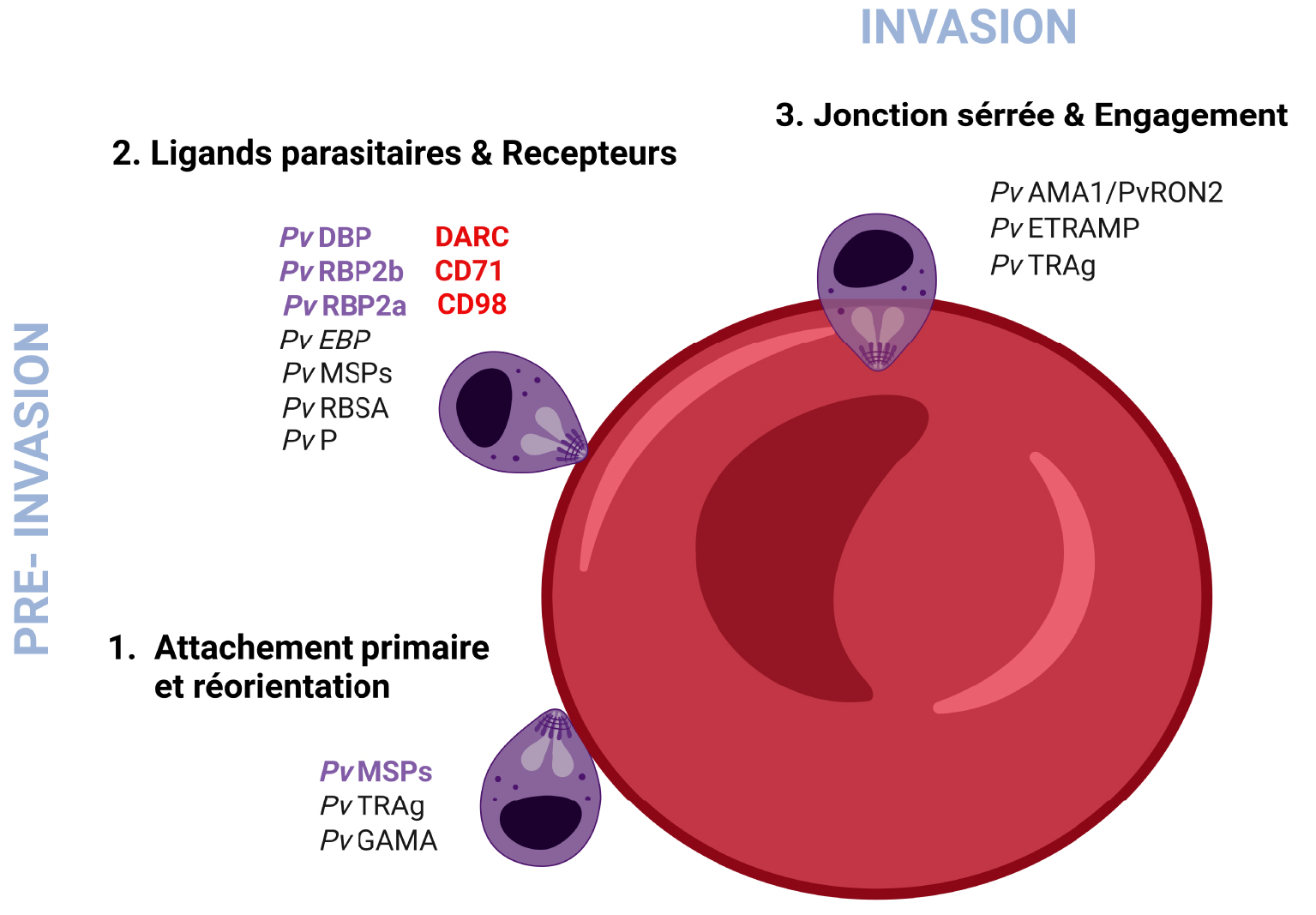
Pendant longtemps, les scientifiques ont cherché à identifier les principales interactions hôte-parasite conduisant à l’invasion des réticulocytes humains par P. vivax. Entre 1920 et 1960, des essais cliniques et de terrain ont été menés sur des individus d’origine africaine ou afro-américaine. Ces essais ont démontré que ces individus étaient hautement résistants au paludisme à P. vivax lorsqu’ils étaient exposés à du sang humain ou à des moustiques infectés par un nombre limité de souches de P. vivax [13, 14, 15, 16]. Après l’identification des groupes sanguins Duffy [17, 18], il a été établi que la résistance naturelle à l’infection à P. vivax était liée à l’absence de la protéine Duffy Antigen Receptor for Chemokines (DARC) à la surface des érythrocytes humains [19]. La base moléculaire de l’absence d’expression de l’antigène Duffy à la surface des globules rouges a ensuite été démontrée comme étant due une mutation ponctuelle unique dans la séquence GATA1 (tct >cct) située dans la région promotrice 5′ du gène ackr1 [20, 21]. Des travaux novateurs portant sur des infections expérimentales contrôlées de volontaires à l’aide de sporozoïtes de P. vivax [19] et des études in vitro utilisant P. knowlesi (un parasite responsable du paludisme zoonotique étroitement apparenté à P. vivax) comme modèle [22] ont ensuite confirmé que l’interaction entre DARC et les mérozoïtes de P. vivax était cruciale pour permettre l’invasion des réticulocytes. Parallèlement, le ligand du parasite qui se lie spécifiquement à DARC a été identifié comme étant la protéine P. vivax Duffy Binding Protein (PvDBP) [23]. Ainsi, le paradigme scientifique selon lequel P. vivax envahit exclusivement les érythrocytes Duffy-positifs par le biais de l’interaction entre PvDBP et DARC était établi. Il a également été supposé que le paludisme à P. vivax était rarement transmis ou même absent dans les régions où la proportion d’individus Duffy-négatifs est prédominante comme en Afrique subsaharienne [19]. Cependant, au cours des deux dernières décennies, de nombreuses études ont rapporté des cas d’infection à P. vivax chez des individus Duffy-négatifs dans des pays subsahariens comme l’Angola [24], le Bénin [25], le Cameroun [26, 27], la Guinée équatoriale [24, 28], l’Ethiopie [29], le Kenya [30], Madagascar [31], le Mali [32], la Mauritanie [33] et le Soudan [34]. Bien que ces observations soient encore parcellaires, elles soulèvent des interrogations quant aux mécanismes d’invasion utilisés par P. vivax pour infecter les individus Duffy-négatifs. Elles remettent également en question certaines hypothèses telles que le rôle central et unique joué par l’interaction PvDBP-DARC dans l’invasion des réticulocytes, la limite de détection des outils de diagnostic actuels et l’évolution des interactions hôte-pathogène [35]. A l’heure actuelle, nous ne savons toujours pas si ces cas représentent un phénomène préexistant qui a été négligé dans le passé ou une nouvelle voie d’invasion indépendante de DARC, qui pourrait potentiellement conduire à une expansion du paludisme à P. vivax au sein des populations Duffy-négatives [36].
La collecte d’échantillons de terrain dans des zones endémiques éloignées étant difficile, longue et coûteuse, la recherche s’est longtemps limitée à l’échantillonnage direct de patients donneurs ou à l’étude d’espèces de Plasmodium étroitement apparentées telles que P. knowlesi [37, 38] ou P. cynomolgi [39]. En outre, le développement de méthodes de culture in vitro reproductibles est encore entravé par la faible parasitémie de P. vivax et son fort tropisme pour les réticulocytes humains [40].
Néanmoins, les récentes technologies dites « omiques » ont révolutionné la recherche sur le paludisme à P. vivax avec le développement de techniques innovantes et abordables permettant une caractérisation plus complète des gènes, des transcrits et des protéines de P. vivax. Cette revue a non seulement pour but de fournir un aperçu général des mécanismes d’invasion de P. vivax grâce aux techniques génomiques, transcriptomiques et protéomiques mais également d’illustrer l’importance de la complémentarité de ces approches.
2. Mécanismes moléculaires d’invasion de P. vivax
En 2004, le projet de séquençage du génome de P. vivax a fourni la première séquence de référence P. vivax Salvador-1 (Sal-1) en utilisant une stratégie de séquençage du génome entier [41, 42]. Au cours des vingt dernières années, plusieurs autres isolats de terrain et cliniques de P. vivax ont été séquencés par microarray, séquençage profond ou séquençage du génome entier. Toutes ces séquences sont disponibles publiquement sur des bases de données en ligne telles que PlasmoDB (https://plasmodb.org/plasmo/app) et sont régulièrement annotées. Globalement, l’analyse de ces séquences de P. vivax a contribué à l’identification de gènes potentiellement liés à l’invasion.
Liste des ligands liés à l’invasion de P. vivax caractérisés par les technologies omiques
| Etape | Ligands parasitaires | Numéro d’accession | Rôle (confirmer/ potentiel) | Recepteur érythrocytaire | Candidats vaccins (stade sanguin) | Preuves biologiques obtenues par des études | ||
|---|---|---|---|---|---|---|---|---|
| Genomiques | Transcriptomiques | Proteomiques | ||||||
| Primary attachment & reorientation | MSP1 | PVP01_0728900 | Confirmer | Oui | pvmsp1 est fortement polymorphe (mutations) [43] | Parmi tous les gènes, pvmsp1 est un des plus exprimé au stade schizonte [44] | Par homologie avec les protéines PfMSPs chez P. falciparum, il a été émis comme hypothèse que PvMSP1 pourrait reconnaitre un recepteur situé à la surface des réticulocytes [45] | |
| MSP7 | PVP01_1219500, PVP01_1219700, PVP01_1219400, PVP01_1219600, PVP01_1220300, PVP01_1220300, PVP01_1220400, PVP01_1219900 | Potentiel | pvmsp7 est fortement polymorphe (mutations) [46]. pvmsp3 presente un nombre de copie amplifiée allant de 1 à 4 copies | Les ARN messagers de pvmsp7 transcript sont les plus élevés au stade trophozoïte âgé-schizonte [47]. Les ARN messagers de pvmsp3 sont détectés au stade schizonte âgé [5]. Parmi tous les gènes, pvmsp9 est un des plus exprimé au stade schizonte [44] | Par homologie avec les protéines PfMSP7 chez P. falciparum, PvMSP7 serait un ligand spécifique des récepteurs P-selectin [48] | |||
| GAMA | PVP01_0505600 | Potentiel | pvgama présente un polyphisme de taille [49] | Etudes fonctionnelles: PvGAMA pourait jouer un rôle dans l’adhésion des mérozoïtes de P. vivax aux réticulocytes [49] et se lier aux protéines Band3 et ANK1 [50] , | ||||
| MSP1P | PVP01_0728800 | Potentiel | PvMSP1P pourait jouer un rôle dans l’adhésion des mérozoïtes de P. vivax aux réticulocytes grâce à une interaction avec sa région C terminale [43] | |||||
| TRAg56.2 | PVX_088850 | Potentiel | la famille des gènes pvtrag est peu polymorphe [51] | Séquencage à l’échelle cellulaire: des niveaux d’expression des ARN messagers différents ont été observés entre les membres des gènes de la famille pvtrag [5] | Immunoprecipitation et spectrométrie de masse: PvTRAg56.2 semble se co-localiser avec PvMSP1 et interagir avec PvMSP7 et jouer un rôle stabilisateur [52] | |||
| Ligand bindings | DBP | PVP01_0623800 | Confirmer | DARC | Oui | pvdbp est fortement polymorphe (mutations et variation du nombre de copie de gène) [53] | L’amplification du gène pvdbp serait associée à une augmentation de l’expression des ARN messagers, suggérant une augmentation de l’expression de la protéine PvDBP [54] | Etudes de liaision et étdues fonctionnelles |
| EBP | PVP01_0102300 | Potentiel | pvebp est fortement polymorphe mais moins que pvdbp (mutations et variation du nombre de copie de gène) [55] | Les ARN messagers de pvebp sont fortement exprimés au stade schizonte [55, 56] | PvEBPII se lie préférentiellement aux réticulocytes Duffy-positifs immatures. La liaision avec les réticulocytes Duffy-négatifs immatures est minime [57, 58]. | |||
| RBP2b | PVP01_0800700 | Confirmer | CD71 | Malgrè l’existence de régions conservées, pvrbp2a est relativement polymorphe [59, 60] | Culture ex vivo: l’intéraction PvRBP2b-CD71 joue une rôle crucial dans la réconnaissance des réticulocytes [61] | |||
| RBP2a | PVP01_1402400 | Confirmer | CD98 | Malgrè l’existence de régions conservées, pvrbp2a est relativement polymorphe [59, 60] | Etudes de liaison: CD98 est le recepteur de la protéine PvRBP2a de P. vivax et l’intéraction entre PvRBP2a-CD98 est impliquée dans la reconnaisance des réticulocytes [62] | |||
| RBSA | PVP01_0004240 | Potentiel | pvrbsa presente une diversité génétique réduite par rapport aux autres gènes codant pour des protéines de surfance des mérozoïtes de P. vivax [63] | PvRBSA se lie spécificiquement aux réticulocytes Duffy-positifs, mais son recepteur n’est pas connu [63] | ||||
| P12 | PVP01_1136400 | Potentiel | Les protéines de la famille PvP sont exprimées à la surface des mérozoïtes de P. vivax et ils pourraient jouer un rôle dans l’invasion des réticulocytes [64] | |||||
| Tight junction formation | AMA1 | PVP01_0934200 | Potentiel | Oui | Etudes fonctionnelles: La fonction de PvAMA1 semble similaire à celle de PfAMA1 [65] et PvAMA1 pourrait être impliquée avec RON2 dans la formation de la jonction serrée [66] , | |||
| RON | PVP01_0916600 | Potentiel | pvron4 a été indentifié par homologie avec pfron4 et pkron4 dans la souche VCG-1 de P. vivax [67] | pvron4 présente un profil de transcription similaire à celui de pfron4 [67]. pvron5 est expimé au stade schizonte [66] | Immunofluorescence indirecte et immunoassays: PvRON4 serait exprimée au niveau du pôle apical de P. vivax (souche VCG-1) et pourrait participer à la formation de la jonction serrée [67]. PvRON5 présente une préférence de liaision avec les réticulocytes et est donc fortement suspecté d’être impiqué dans le processus d’invasion des réticulocytes [66]. | |||
| ETRAMP | PVP01_0532300 | Potentiel | ETRAMP serait impliqué dans la formation de la vacuole parasitophore, | |||||
| TRAg36.6 | PVX_112690 | Potentiel | Séquencage de l’ADN: la famille de gène pvtrag est rarement polymorphe [51] | Séquencage à l’échelle cellulaire: des niveaux d’expression des ARN messagers différents ont été observés entre les membres des gènes de la famille pvtrag [5] | Immunoprecipitation et spectrométrie de masse: PvTRAg36.6 semble se co localiser avec PvETRAMP pour former et/ou maintenir l’intergrité de la membrane de la vacuole parasitophore [52] | |||
| Unknown function | MSA180 | PVP01_0814200 | Potentiel | Parmi tous les gènes, pvmsa180 est un des plus exprimé au stade schizonte [44] | ||||
| P92 | PVP01_1107900 | Potentiel | ||||||
| RAMA | PVP01_0107500 | Potentiel | Parmi tous les gènes, pvrama est un des plus exprimé au stade schizonte [44] | |||||
| RBP1a | PVP01_0701200 | Potentiel | Séquencage de l’ADN: la diversité génétique de pvrbp1a semble réduite [68] | Les ARN messagers de pvrbp1 sont fortement exprimés au stade schizonte [55, 56]. | Les protéines PvRBP1 et PvRBP2 natives pourraient être impliquées dans un complexe protéique d’adhesion [69] | |||
| RBP1b | PVP01_0701100 | Potentiel | Séquencage de l’ADN: la diversité génétique de pvrbp1b semble réduite [68] | Les ARN messagers de pvrbp1 sont fortement exprimés au stade schizonte [55, 56]. | Les protéines PvRBP1 et PvRBP2 natives pourraient être impliquées dans un complexe protéique d’adhesion [69] | |||
| RBP2c | PVP01_0534300 | Potentiel | Séquencage de l’ADN: pvrbp2c est fortement polymorphe [51, 70, 59] | |||||
| RBP1p1 (partial) | Potentiel | Séquencage de l’ADN: identification partielle du gène pvrbp1p1 | ||||||
| RBP2p1 (partial) | PVP01_0534400 | Potentiel | Séquencage de l’ADN: identification partielle du gène pvrbp2p1 | |||||
| RBP2p2 (partial) | PVX_101590 | Potentiel | Séquencage de l’ADN: identification partielle du gène pvrbp2p2. | |||||
| RBP2d (pseudo) | PVP01_1471400 | Potentiel | Séquencage de l’ADN: identification du pseudogène pvrbp2d. | |||||
| RBP2e (pseudo) | PVP01_0700500 | Potentiel | Séquencage de l’ADN: identification du pseudogène pvrbp2e. | |||||
| RBP3 (pseudo) | PVP01_1469400 | Potentiel | Séquencage de l’ADN: identification du pseudogène pvrbp3. | |||||
| RhopH3 | PVP01_0703800 | Potentiel | Parmi tous les gènes, pvrhoph3 est un des plus exprimé au stade schizonte [44] | |||||
| RON5 | Potentiel | Les ARN messagers de pvron5 sont fortement exprimés au stade schizonte dans la souche VCG-1 [66] | Etudes de liaison, Western blot et tests d’immunofluorescence: PvRON5 serait exprimée au niveau du pôle apical de P. vivax (souche VCG-1) and présente une préférence de liaision avec les réticulocytes [66] | |||||
Les numéros d’accession commençant par PVP01 proviennent de la souche de référence PvP01, tandis que les numéros d’accession commençant par PVX proviennent de la souche de référence Sal-I (https://plasmodb.org/plasmo/app). Les numéros d’accession commençant par KP proviennent de la souche de référence VCG-1 (https://www.ncbi.nlm.nih.gov).
2.1. Le rôle crucial du gène pvdbp dans l’invasion des réticulocytes Duffy-positifs
Le gène P. vivax Duffy binding protein (pvdbp) code pour le ligand PvDBP [71]. Il présente des séquences de codage pour un domaine peptidique N-terminal, un domaine d’attachement Duffy riche en cystéines (PvDBPII), un autre domaine riche en cystéines (PvDBPVI), un domaine transmembranaire et une queue cytoplasmique C-terminale [72]. Sur la base des données actuelles de séquençage à haut débit, notre appréciation de la diversité génétique est de moins en moins limitée par la nature et/ou la densité des marqueurs génomiques étudiés. Notamment, la publication récente de plus de deux cents génomes de P. vivax a montré que les séquences du gène pvdbp étaient hautement polymorphes, quelle que soit leur origine géographique. Il est intéressant de noter que de nombreux polymorphismes nucléotidiques simples (SNP) non synonymes ont été trouvés dans le domaine PvDBPII [53]. La diversité et la localisation de ces SNPs suggèrent que le gène pvdbp pourrait subir une pression sélective différentielle en raison des réponses des anticorps et de sa fonction dans la liaison aux récepteurs et l’invasion [51].
En outre, les variations du nombre de copies (CNV) au niveau du locus pvdbp se sont révélées être l’une des expansions génétiques les plus courantes, avec des allèles multiples [73]. Alors que la détection des CNVs reposait dans le passé, sur des techniques coûteuses et nécessitant une main-d’œuvre importante comme l’hybridation in situ, les progrès récents du séquençage à haut débit ont permis leur détection directe à partir des données de séquençage. Notamment, les approches de séquençage du génome entier ont révélé deux duplications distinctes chez les souches P. vivax circulant à Madagascar et au Cambodge. À Madagascar, les isolats de P. vivax provenant de patients Duffy-positifs et Duffy-négatifs présentaient une duplication commune avec deux copies de pvdbp l’une à côté de l’autre [74]. Au Cambodge, une duplication différente du gène pvdbp a été couramment détectée dans les isolats de P. vivax provenant de patients Duffy-positifs. De plus, la proportion d’isolats de P. vivax portant la duplication s’est avérée être significativement plus élevée chez les patients homozygotes Duffy-positifs que chez les patients hétérozygotes Duffy-positifs qui devraient présenter une plus faible expression de DARC à la surface de leurs réticulocytes. Cependant, cette dernière analyse n’a pas permis d’exclure complètement que les patients puissent être porteurs de plusieurs clones de P. vivax, alors que l’estimation des CNVs par PCR quantitative ne reflète que le clone principal [53]. Récemment, une étude géographique a démontré que les deux duplications distinctes du gène pvdbp circulaient en Asie, en Amérique du Sud et en Afrique de l’Est. Alors que la Duffy-négativité est prévalente dans certaines régions, les duplications du gène pvdbp ont également été largement trouvées dans des régions où la Duffy-négativité était presque absente [51]. En revanche, une analyse génomique simultanée a montré qu’aucune duplication de pvdbp n’a été trouvée dans cent quatre-vingt-deux isolats de P. vivax provenant de onze pays endémiques. Cependant, il a été remarqué que cette divergence était probablement due aux amorces utilisées pour la validation des CNVs par PCR [75].
Malgré la spécificité et la distribution des duplications du gène pvdbp, rien n’indique que les génomes de P. vivax aient contourné la barrière de la Duffy-négativité grâce à cette variation génétique. Des expansions plus importantes de pvdbp ont également été détectées dans le monde entier. Elles se regroupent séparément mais présentent des schémas similaires. Une grande diversité d’haplotypes a également été observée entre les copies, tandis que les séquences flanquantes 5′ et 3′ présentaient une diversité modérée [76]. Dans la région Asie-Pacifique, le séquençage de plus de deux cents isolats de P. vivax provenant de patients Duffy-positifs a révélé la présence commune de deux à trois copies du gène pvdbp [73]. En Ethiopie, les séquences de deux isolats de P. vivax provenant de patients Duffy-négatifs ont montré la présence de trois et huit copies du gène pvdbp [57]. Par la suite, une autre expansion commune du gène pvdbp, assez similaire à celles précédemment décrites à Madagascar et au Cambodge mais allant de trois à cinq copies, a également été détectée [51]. Ces résultats suggèrent que l’expansion du gène pvdbp pourrait permettre la survenue de mutations dans la deuxième copie du gène. De telles expansions pourraient faciliter la liaison à un autre récepteur érythrocytaire ayant une affinité plus faible pour le ligand PvDBP [57]. Cependant, il n’y a aucune preuve de l’existence de CNVs localisés et sélectionnés qui amélioreraient l’efficacité de l’invasion et induiraient une pression sélective. Étant donné que l’invasion des érythrocytes Duffy-positifs dépend de l’interaction entre PvDBP et DARC, il est probable que les expansions du gène pvdbp soient dues à des variations génétiques de DARC ou à des réponses à des anticorps inhibant l’attachement au domaine PvDBPII [75]. Néanmoins, il ne semble pas que ces expansions donnent au parasite une capacité accrue pour envahir les érythrocytes Duffy-négatifs.
2.2. Le potentiel rôle du gène pvebp dans une voie d’invasion alternative
Le gène P. vivax erythrocyte binding protein (pvebp) a été découvert grâce à l’assemblage de novo d’un isolat de P. vivax du Cambodge. Il code pour un ligand putatif de la protéine P. vivax Erythrocyte Binding Protein (PvEBP) et est présent dans la plupart des génomes de P. vivax. L’organisation du gène pvebp est similaire à celle du gène pvdbp avec un peptide signal N-terminal, un domaine d’attachement Duffy PvEBPII semblable au domaine PvDBPII, un autre domaine riche en cystéines PvEBPVI, un domaine transmembranaire et une queue cytoplasmique C-terminale [55]. Malgré ces similitudes, le gène pvebp semble se regrouper séparément de tous les gènes possédant un domaine d’attachement Duffy-binding like. Le séquençage de l’ADN de dix génomes de P. vivax provenant d’Asie, du Pacifique Sud, d’Afrique, d’Amérique centrale et d’Amérique du Sud a montré que le gène pvebp est polymorphe bien que moins divers que le gène pvdbp. En effet, le gène pvebp ne présente que 11 sites polymorphes sur 2412 positions alors que le gène pvdbp présente 32 sites polymorphes sur 3213 positions pour les mêmes échantillons. La plupart des SNPs détectés étaient non synonymes [55]. De plus, l’analyse génomique de deux cent dix-neuf isolats de P. vivax provenant de patients Duffy-positifs au Cambodge et à Madagascar a confirmé que le gène pvebp était moins diversifié que le gène pvdbp (12 vs. 33 allèles observés à partir des mêmes isolats) et ne présentait que des mutations non synonymes indiquant l’existence d’une forte pression sélective. En effet, onze mutations ponctuelles non synonymes ont été trouvées dans les séquences pvebp et ont conduit à douze allèles différents. Certains SNPs ont été observés dans les deux pays alors que d’autres étaient spécifiques au Cambodge et à Madagascar. Ces SNPs étaient distribués dans le domaine d’attachement [53]. Tout comme pour le gène pvdbp, la diversité et la distribution des SNPs dans le gène pvebp reflètent l’existence probable d’une pression sélective, bien qu’elle semble opérer à un niveau plus faible [58].
L’analyse génomique des isolats de P. vivax du Cambodge, de Madagascar et d’Éthiopie a également révélé l’existence de variations du nombre de copies dans le gène pvebp. À Madagascar, des expansions du gène pvebp ont été observées, allant de deux à cinq copies. Une distribution bimodale des parasites avec une population spécifique présentant plus de trois copies a été trouvée. Cependant, aucune différence significative de susceptibilité à l’infection à P. vivax n’a été observée entre les individus homozygotes et hétérozygotes Duffy-positifs. Au Cambodge, les expansions de pvebp variaient d’une à deux copies. La proportion d’isolats de P. vivax présentant des copies multiples du gène était plus importante dans les zones de forte transmission [53]. Dans l’ensemble, une proportion élevée d’isolats de P. vivax avec des copies de gènes multiples a été trouvée à Madagascar où la Duffy-négativité et la Duffy-positivité coexistent par rapport au Cambodge où la Duffy-négativité est pratiquement absente. En revanche, aucune expansion de pvebp n’a été trouvée en Éthiopie où la Duffy-négativité est également fréquente [51]. Compte tenu des similitudes observées entre les gènes pvdbp et pvebp, le polymorphisme et l’expansion du gène pvebp pourraient être corrélés à l’expression d’un nouveau ligand interagissant avec des protéines membranaires érythrocytaires autres que DARC et contribuer ainsi à une voie d’invasion alternative [55]. Cependant, aucune preuve que ces variations confèrent des capacités à infecter des individus Duffy-négatifs n’a été fournie jusqu’à présent.
2.3. Le rôle des gènes pvrbp et pvrbsa dans la sélectivité des réticulocytes
La famille de gènes P. vivax reticulocyte binding protein (pvrbp) a été identifiée sur la base de l’annotation des ligands adhésifs du génome de P. vivax [69]. Le séquençage du génome de plusieurs isolats de P. vivax a identifié onze membres de la famille pvrbp, dont cinq gènes complets (pvrbp1a, pvrbp1b, pvrbp2a, pvrbp2b et pvrbp2c), trois gènes partiels (pvrbp1p1, pvrbp2p1 et pvrbp2p2) et trois pseudogènes (pvrbp2d, pvrbp2e et pvrbp3) [77]. Les gènes pvrbp sont constitués de deux exons, l’un codant pour un peptide signal dans la région N-terminale et l’autre pour un domaine transmembranaire dans la région C-terminale [56]. Des études récentes ont montré que leur diversité génétique semble être généralement faible, sauf pour les gènes pvrbp2 [68]. L’analyse des isolats de P. vivax de Chine, de Thaïlande et d’Éthiopie a confirmé que le gène pvrbp2c était hautement polymorphe [51? , 59]. Bien que pvrbp2a et pvrbp2b présentent des régions hautement conservées, l’analyse génomique des isolats de P. vivax de Papouasie-Nouvelle-Guinée et de Thaïlande a montré qu’ils étaient également relativement polymorphes [59, 60]. Comme observé pour les gènes pvdbp et pvebp, les SNPs non synonymes se sont regroupés autour de trois sites principaux, dont le domaine de liaison aux réticulocytes. Cela peut refléter l’importance fonctionnelle de ce site de liaison et l’existence d’une pression sélective [60].
De plus, des CNVs affectant à la fois pvrbp2a et pvrbp2b ont été observés dans deux isolats de P. vivax de Thaïlande. Alors qu’une souche présentait une duplication des deux gènes (pvrbp2a et pvrbp2b), l’autre souche avait deux copies de pvrbp2b seulement [59]. Ces résultats suggèrent que les gènes pvrbp peuvent coder des ligands impliqués dans la reconnaissance de cellules cibles. Le gène P. vivax reticulocyte binding surface antigen (pvrbsa) a également été identifié comme un gène potentiellement impliqué dans la reconnaissance des réticulocytes, bien qu’il présente moins de diversité génétique que les autres gènes de surface des mérozoïtes de P. vivax [63].
2.4. Le rôle potentiel des gènes pvmsp dans l’attachement primaire
La famille de gènes P. vivax merozoite surface protein (pvmsp) code pour de multiples déterminants antigéniques qui sont suspectés être les médiateurs de l’attachement primaire entre les mérozoïtes de P. vivax et les réticulocytes humains [11]. Ces gènes pvmsp sont hautement polymorphes. En particulier, pvmsp1 [43], pvmsp3 [43, 78] et pvmsp7 [46] présentent de nombreux SNPs non synonymes indiquant qu’ils peuvent être soumis à une pression immunitaire sélective [51]. L’analyse des isolats de P. vivax d’Éthiopie a révélé que pvmsp3 présentait des CNVs allant d’une à quatre copies. Par ailleurs, il est intéressant de noter que les expansions de pvdbp et pvmsp3 ont souvent été observées en tandem. La pertinence fonctionnelle de cette expansion du nombre de copies n’est pas encore connue.
2.5. Les autres gènes et leur rôle potentiel dans la voie d’invasion de P. vivax
D’autres gènes de P. vivax sont suspectés d’être impliqués dans le processus d’invasion. Par exemple, la famille des gènes P. vivax tryptophan rich antigen (pvtrag) coderait pour trente-six protéines qui peuvent se lier aux érythrocytes humains [79]. L’analyse génomique de souches de P. vivax en Éthiopie a révélé que la famille de gènes pvtrag ne semble porter que quelques polymorphismes génétiques [51]. De même, le gène P. vivax glycosylphosphatidylinositol-anchored micronemal antigen (pvgama) code pour l’antigène micronémique ancré dans le glycosylphosphatidylinositol de P. vivax (PvGAMA) qui se lie aux érythrocytes indépendamment du statut Duffy [43]. Cette protéine peut jouer un rôle dans l’adhésion des mérozoïtes de P. vivax à leurs cellules cibles [49]. Enfin, la famille de gènes P. vivax rhoptry neck protein (pvron) code pour les protéines P. vivax Rhoptry Neck (PvRON) qui pourraient participer à la formation de la jonction serrée comme cela a été démontré pour les homologues de P. falciparum [67].
Les technologies génomiques ont clairement contribué à affiner la recherche sur le paludisme à P. vivax en réduisant le coût du séquençage ainsi qu’en améliorant la qualité et la disponibilité des séquences. Cependant, seules quelques centaines de génomes de P. vivax ont été séquencés jusqu’à présent. Les isolats de terrain et cliniques présentent une faible parasitémie et une forte contamination par l’ADN humain, ce qui rend toujours difficile l’acquisition d’une quantité suffisante d’ADN parasitaire de bonne qualité. Pourtant, les séquences génomiques disponibles à ce jour ont permis l’identification de quelques gènes potentiellement liés à l’invasion. Les polymorphismes de nucléotides simples (SNPs) et les variations du nombre de copies (CNVs) trouvés dans ces gènes peuvent conduire à des différences structurelles et entraîner des pressions sélectives. Cependant, les conséquences fonctionnelles de ces variations restent inconnues. L’analyse des génomes de P. vivax devrait être combinée à l’identification de leurs profils transcriptomiques et à la caractérisation de leurs protéines putatives afin de détecter des signatures spécifiques, susceptibles de fournir des informations sur leur fonction. Mais l’absence de méthodes robustes permettant de cultiver in vitroP. vivax rend l’analyse fonctionnelle difficile.
3. Transcription des gènes d’invasion de P. vivax
Les progrès constants du séquençage à haut débit associés à la capacité récente de maturer des isolats de P. vivax par culture ex vivo pendant un cycle ont ouvert la voie aux études transcriptomiques. Le profil transcriptomique des stades sanguins de P. vivax via l’utilisation de microarrays et d’un séquençage ARN (à l’échelle de la population ou de l’individu) dévoile progressivement de nouvelles capacités expérimentales et permet d’élucider la transcription, la fonction et la régulation des gènes liés à l’invasion.
3.1. Expression spécifique des gènes liés à l’invasion
La génération de transcriptomes de haute qualité à travers l’utilisation de multiples isolats de P. vivax est essentielle pour explorer les changements dans l’expression des gènes tout au long du cycle intra-érythrocytaire. Récemment, les profils transcriptomiques d’isolats de P. vivax d’infections sanguines provenant de singes Aotus et Saimiri ont révélé que la plupart des gènes de P. vivax avaient une expression spécifique à un stade parasitaire et étaient fortement surexprimés, contrairement aux gènes domestiques [5]. Étant donné que la plupart des infections cliniques étaient asynchrones, l’analyse des transcriptomes de P. vivax a permis d’identifier des populations distinctes. Les anneaux et les trophozoïtes précoces exprimaient principalement des gènes impliqués dans la transcription et la traduction, tandis que les trophozoïtes tardifs et les schizontes exprimaient des gènes potentiellement liés à l’invasion [5]. Par exemple, les transcrits pvebp et pvrbp1 ont été trouvés fortement exprimés pendant le stade schizonte de P. vivax [? 56]. De même, les transcrits pvmsp3 ont été détectés dans les schizontes très tardifs [5] et le transcrit pvmsp7 a généralement atteint un pic pendant la transition trophozoïte tardif vers schizonte, lorsque les mérozoïtes sont formés [47]. Dans la souche de P. vivax adaptée au modèle primate (VCG-1), les transcrits pvron5 ont été trouvés dans les schizontes [66]. De plus, des expériences de séquençage ARN menées avec des isolats de P. vivax cultivés ex vivo provenant du Brésil ont montré une grande variation dans l’expression des gènes suspectés d’être impliqués dans l’interaction hôte-parasite, la sélectivité des réticulocytes ou l’invasion. En particulier, pvmsp1, pvmsp9, P. vivax merozoite surface antigen 180 (pvmsa180), P. vivax rhoptry-associated membrane antigen (pvrama) et P. vivax rhoptry H3 (pvrhoph3) étaient les plus exprimés parmi tous les gènes [44]. L’expression des gènes liés à l’invasion dans les schizontes tardifs implique l’importance du développement des mérozoïtes et de l’invasion des réticulocytes pour la survie de P. vivax.
3.2. Expression sélective des gènes liés à l’invasion de P. vivax
Le génome actuel de P. vivax contient de nombreuses familles de gènes qui ont été définies sur la base de l’homologie de séquence et de la conservation des domaines d’acides aminés. Les analyses de séquençage RNA à l’échelle de la cellule (single cell) ont démontré que l’expression sélective des gènes peut être due à leur rôle spécifique ou à des fonctions supplémentaires. Alors que la plupart des gènes pvmsp sont exprimés au cours des stades de développement tardifs, on a constaté que pvmsp3 était exclusivement exprimé dans les schizontes très tardifs, ce qui suggère son rôle dans le processus d’invasion. Étonnamment, un gène similaire à pvmsp7 s’est avéré être exprimé exclusivement dans les gamétocytes, ce qui implique une différence fonctionnelle [5]. Des résultats comparables ont été rapportés en utilisant des isolats cliniques de P. vivax provenant de Thaïlande. Alors que l’expression de la plupart des gènes pvmsp7 était associée aux schizontes, les gènes pvmsp7a et pvmsp7f ont maintenu des niveaux d’expression élevés pendant tout le cycle intra-érythrocytaire. Ces résultats suggèrent que ces paralogues peuvent avoir des fonctions supplémentaires en plus de leur rôle établi à la surface des mérozoïtes [47].
De même, des niveaux d’expression différents ont été observés parmi les membres de la famille de gènes pvtrag, mais leur signification est inconnue [5]. Malgré leur homologie de séquence, les membres d’une famille de gènes peuvent également avoir des fonctions différentes à des moments spécifiques [5]. Enfin, un séquençage du transcriptome sans amplification et directionnel (DAFT) de quatre isolats de P. vivax du Cambodge a révélé que la transcription peut être un processus sélectif dans lequel les parasites sélectionneraient des membres particuliers d’une famille de gènes en réponse à l’hôte [80]. Les auteurs ont montré que les gènes dont l’expression variait le plus entre les isolats étaient physiquement regroupés le long de régions chromosomiques. Par exemple, le locus pvmsp3 présente de nombreux paralogues qui étaient susceptibles d’être fonctionnellement redondants, augmentant ainsi la diversité antigénique et améliorant l’évasion immunitaire pendant le processus d’invasion. Au sein des clusters, l’expression des gènes semble se produire comme si les parasites choisissaient la version du gène à exprimer chez un hôte donné [80]. Globalement, ces résultats reflètent une régulation du développement et une différenciation fonctionnelle au sein de familles de gènes spécifiques.
3.3. Amplification des gènes liés à l’invasion de P. vivax
Chez les espèces de Plasmodium, l’amplification des gènes peut constituer un moyen simple d’augmenter le niveau d’expression des gènes et de modifier ainsi les phénotypes d’invasion. Des études génomiques antérieures ont démontré que certaines souches de P. vivax présentaient une amplification des gènes liés à l’invasion. Notamment, le gène pvdbp est très variable dans le nombre de copies du gène. Par exemple, le profil transcriptomique de schizontes d’isolats cambodgiens de P. vivax a révélé que l’amplification du gène pvdbp était associée à une augmentation de l’expression d’ARN messager (ARNm), ce qui suggère une augmentation potentielle du niveau de la protéine PvDBP [54]. Étant donné que la protéine PvDBP joue un rôle crucial dans l’invasion des réticulocytes Duffy-positifs, on a émis l’hypothèse que les parasites capables d’infecter les réticulocytes Duffy-négatifs ont pu être sélectionnés pour leur capacité à utiliser des copies multiples de pvdbp afin de se lier à un récepteur alternatif [69]. Cependant, des souches de P. vivax avec des copies multiples de pvdbp ont également été couramment détectées dans des régions où la Duffy-négativité est rare. Par ailleurs, même si le gène pvebp présente de nombreuses similitudes avec le gène pvdbp, rien ne prouve que son amplification entraîne une augmentation des niveaux d’ARNm et/ou de protéines [53]. Ainsi, les conséquences fonctionnelles de l’expansion des gènes liés à l’invasion sont incertaines et nécessitent des investigations supplémentaires avec un nombre élevé d’isolats de P. vivax.
3.4. Un système de régulation complexe et dynamique
Les données transcriptomiques des parasites suggèrent que P. vivax présente une cascade transcriptionnelle étroitement régulée pendant son cycle intra-érythrocytaire [81]. Des études ont montré que les gènes co-exprimés présentaient des motifs conservés et des sites potentiels pour des protéines de liaison régulatrices [82]. Par exemple, de longues régions transcrites non traduites (UTR) 5′ et 3′ ont été observées dans des gènes suspectés être impliqués dans l’invasion et la formation de la vacuole parasitophore [80]. De plus, les variations des 5′-UTRs semblent conduire à une sélection différentielle des sites de début de transcription (TSS) reflétant la variation du niveau d’expression des gènes [83]. La présence de promoteurs bidirectionnels pour réguler l’expression de paires de gènes adjacents a également été fréquemment observée. Enfin, les modifications post-transcriptionnelles telles que la structure de la chromatine, les transcrits non codants et l’épissage alternatif peuvent jouer un rôle important dans la régulation, mais les mécanismes restent encore mal compris. Notamment, la relation spatiale présumée entre les gènes qui sont exprimés de manière variable suggère que la structure et l’accès à la chromatine peuvent être importants pour la régulation des gènes [83]. Ainsi, P. vivax dispose d’un réseau de régulation insaisissable et complexe qui peut régir l’expression des gènes liés à l’invasion et permettre ainsi au parasite de se développer dans les réticulocytes humains infectés.
La transcriptomique fournit des informations précieuses sur la dynamique des gènes liés à l’invasion de P. vivax. Il est probable que les schizontes de P. vivax expriment sélectivement des gènes spécifiques pour initier le processus d’invasion. Cependant, la petite taille des échantillons, le manque de méthodes de culture in vitro fiables et la dynamique d’expression des gènes de ménage compliquent l’identification de nouveaux transcrits et de motifs régulateurs. À l’avenir, l’exploration des profils transcriptomiques à l’échelle cellulaire et le séquençage à longue lecture d’isolats de P. vivax provenant de régions géographiquement distinctes devraient être combinés à des approches protéomiques pour évaluer toute la diversité de l’expression des ligands liés à l’invasion.
4. Mécanismes cellulaires d’invasion de P. vivax
Les données protéomiques concernant les infections à P. vivax sont minimes en raison de l’accès limité au matériel biologique et au manque de système robuste permettant de cultiver in vitro le parasite. La liste des protéines exprimées par les mérozoïtes de P. vivax n’a donc toujours pas été clairement définie. Cela a généré des lacunes importantes dans la compréhension des mécanismes d’invasion de P. vivax. Étant donné que la structure de base des mérozoïtes est similaire chez toutes les espèces de Plasmodium, la plupart des chercheurs ont basé l’étude des mécanismes d’invasion de P. vivax sur l’hypothèse que le parasite établit les mêmes étapes malgré un tropisme cellulaire différent et des interactions récepteur-ligand différentes.
4.1. Attachement primaire
Le processus d’invasion semble être initié par l’attachement réversible des mérozoïtes de P. vivax aux réticulocytes. Cette fixation peut être médiée par les protéines de surface des mérozoïtes de P. vivax (PvMSP), qui peuvent former de grands complexes constitués de protéines ancrées au glycophosphatidylinositol et de protéines transmembranaires couvrant la surface des mérozoïtes de P. vivax. Sur la base de l’étude des protéines de surface des mérozoïtes de P. falciparum (PfMSP) homologues aux PvMSPs de P. vivax, on a émis l’hypothèse que la protéine PvMSP1 pourrait reconnaître un récepteur à la surface des réticulocytes humains. La sous-unité C-terminale (MSP-142) de PvMSP1 pourrait être clivée et la plus grande partie du complexe pourrait être éliminée, laissant seulement une petite sous-unité C-terminale (MSP-119) à la surface. De même, son paralogue PvMSP1P pourrait jouer un rôle important dans l’adhésion des mérozoïtes de P. vivax aux réticulocytes par interaction avec sa région C-terminale [43]. De plus, on a émis l’hypothèse que la protéine PvMSP7 pourrait être un ligand spécifique au récepteur P-sélectine qui agit comme une molécule d’adhésion cellulaire [48]. De même, la protéine PvMSP1 et la protéine PvMSP7 pourraient se lier ensemble pour former un complexe afin d’augmenter l’efficacité de l’invasion [47].
D’autres ligands pourraient être impliqués dans cette étape, mais leur capacité à déclencher l’attachement ou à abroger la reconnaissance par le système immunitaire de l’hôte est inconnue. Par exemple, la protéine PvTRAg56.2 semble se localiser avec la protéine PvMSP1 à la surface du mérozoïte et interagir avec la protéine PvMSP7 pour jouer un rôle stabilisateur [52]. De la même façon, la protéine micronémale PvGAMA pourrait se lier à des récepteurs spécifiques à la surface des réticulocytes et ainsi jouer un rôle dans l’adhésion des mérozoïtes de P. vivax à leurs cellules hôtes cibles [65]. En effet, l’analyse protéomique de la protéine ankyrine 1 (ANK1) et de la protéine Band 3 par diverses approches biochimiques incluant la chromatographie liquide-spectrométrie de masse, a montré que le fragment 2 du domaine de liaison à la membrane de l’ANK1 et la boucle extracellulaire 5 de Band 3 se lient à la protéine PfGAMA et à la protéine PvGAMA. En outre, les anticorps anti-Band 3-P5 bloquent la liaison PfGAMA aux érythrocytes [50]. Par ailleurs, des études sur le terrain et en laboratoire ont montré que l’exposition à P. vivax induit des réponses IgG robustes à PvGAMA dans le cadre d’une exposition naturelle [84, 85] et que les anticorps anti-PvGAMA inhibent l’invasion et la croissance in vitro de P. knowlesi [86], bien que le knock-out du gène pkgama ait un effet minime sur la croissance de P. knowlesi. Ainsi, l’attachement primaire des mérozoïtes de P. vivax aux réticulocytes ou aux érythrocytes implique des interactions spécifiques et réversibles permettant la réorientation apicale.
4.2. Réorientation apicale
Après la fixation primaire, il se pourrait que P. vivax subisse une réorientation apicale qui déclenche diverses interactions ligand-récepteur. Comme les mérozoïtes de P. vivax ont un fort tropisme pour les réticulocytes (qui représentent entre 0,2% et 2% de la population érythrocytaire totale), les chances de rencontrer une cellule permissive à l’invasion sont faibles. On a donc émis l’hypothèse qu’une étape de sélection de la cellule hôte se produisait pendant la réorientation apicale pour empêcher les mérozoïtes d’envahir des cellules non ciblées [87]. D’après l’étude de protéines homologues chez d’autres espèces de Plasmodium, on pense que la famille de protéines PvRBP est responsable du tropisme de P. vivax pour les réticulocytes. Des travaux récents ont montré que les protéines PvRBP1 et PvRBP2 natives peuvent fonctionner comme un complexe protéique adhésif [69] et que les protéines PvRBP2a et PvRBP2b peuvent se lier aux réticulocytes indépendamment de leur phénotype Duffy [60]. En 2015, une étude ex vivo a confirmé que les mérozoïtes de P. vivax ciblent préférentiellement les réticulocytes immatures exprimant le marqueur CD71 [61]. En 2018, une autre étude a montré que CD71 était le récepteur du ligand PvRBP2b [88]. Des analyses par microscopie cryo-électronique, immunoprécipitation et transfert d’énergie par résonance de fluorescence ont confirmé que la liaison de PvRBP2b à CD71 était spécifique et directement proportionnelle au niveau d’expression de CD71 [89]. Enfin, des études fonctionnelles ont identifié le marqueur CD98, exprimé à la surface des réticulocytes immatures, comme étant le principal récepteur du PvRBP2a [62]. Les deux interactions PvRBP2b - CD71 et PvRBP2a - CD98 semblent être à l’origine du tropisme strict de P. vivax pour les réticulocytes. Leur action pourrait être indépendante ou combinée, ce qui suggère l’existence potentielle de voies d’invasion alternatives. Outre les protéines PvRBP, l’adhésine antigénique de surface liant les réticulocytes de P. vivax (PvRBSA) pourrait également jouer un rôle dans la reconnaissance des réticulocytes. Une étude récente a montré que la protéine PvRBSA recombinante (rPvRBSA) se lie spécifiquement aux réticulocytes Duffy-positifs. Mais son récepteur est encore inconnu [63]. En plus de leur fonction adhésive, les protéines PvMSPs peuvent également contribuer à la reconnaissance des réticulocytes. En effet, des études récentes ont montré que les peptides de la protéine PvMSP1 ont une activité de liaison plus élevée avec les réticulocytes qu’avec les érythrocytes [45]. Bien que les mécanismes ne soient pas encore clairs, la reconnaissance des réticulocytes immatures par les mérozoïtes de P. vivax semble être gouvernée par PvRBP2a et/ou PvRBP2b, potentiellement en conjonction avec la protéine PvRBSA et les protéines PvMSPs. Les protéines PvRBPs pourraient également déclencher des étapes ultérieures de l’invasion, telles que la modulation du cytosquelette érythrocytaire, l’assemblage de la jonction serrée ou la signalisation de la sécrétion de protéines PvRhops.
4.3. Invasion
La réorientation apicale pourrait être suivie par la formation d’une jonction serrée et irréversible entre les mérozoïtes de P. vivax et les réticulocytes. Chez les individus Duffy-positifs, en l’absence de preuves, nous ne savons pas si la protéine PvDBP se localise à la jonction serrée entre la surface des mérozoïtes et le récepteur DARC. En effet, pour P. falciparum, PfEBA175 (un ligand de liaison aux érythrocytes de P. falciparum) se lie à la Glycophorine A pour déclencher la libération de PfRH5 et d’autres protéines PfRhops. Mais PfEBA175 lui-même ne participe pas à la formation de la jonction. Le site de liaison de PvDBP est monomérique en l’absence de DARC mais se dimérise lors de l’engagement du récepteur et forme un homodimère avec le domaine extracellulaire N-terminal de DARC [60]. La formation de la jonction serrée pourrait également être facilitée par la sécrétion du ligand P. vivax Apical Membrane Antigen 1 (PvAMA1) à la surface du mérozoïte, l’injection de complexes de la protéine PvRON à la surface des réticulocytes et leur interaction. Il a notamment été démontré que PvRON5 a une préférence marquée pour les réticulocytes et est donc soupçonné d’être impliqué dans le processus d’invasion [66]. Enfin, l’expression de divers membres de la famille de la protéine vivax 6-cystéine (PvP) à la surface du mérozoïte jouerait également un rôle dans cette étape [64].
Chez les individus Duffy-négatifs, les mécanismes et les facteurs impliqués dans la formation de la jonction serrée restent inconnus, mais plusieurs hypothèses sont actuellement discutées (Figure 3). À Madagascar, la mise en évidence d’infections à P. vivax chez des patients Duffy-négatifs a été corrélée à l’existence d’une duplication commune du gène pvdbp. Ceci a conforté l’hypothèse selon laquelle l’amplification du gène pvdbp pourrait favoriser la liaison de la protéine PvDBP à un autre récepteur de moindre affinité. Cependant, un tel récepteur alternatif n’a pas encore été identifié. De plus, les souches de P. vivax présentant de multiples copies de pvdbp sont fréquemment trouvées dans les zones endémiques où la Duffy-négativité est rare [53]. Une deuxième hypothèse est que le ligand PvEBP pourrait fonctionner comme un ligand alternatif pour l’invasion des réticulocytes Duffy-négatifs. En effet, le ligand PvEBP semble partager des similitudes structurelles avec le ligand PvDBP, comme un site d’attachement Duffy-binding like et un domaine C-terminal avec des positions de cystéine conservées, et probablement des fonctions similaires [55]. Sur la base de ces similarités structurelles, un site de liaison au récepteur recombinant pour PvEBP (rPvEBPII) a été produit. Les résultats des essais de liaison ont montré que PvEBPII se lie préférentiellement aux réticulocytes immatures Duffy-positifs, mais une liaison minimale a été observée dans les réticulocytes immatures Duffy-négatifs. En revanche, aucune liaison n’a été observée pour les érythrocytes matures [57, 58]. Récemment, une étude fonctionnelle réalisée sur des isolats de P. vivax provenant d’Éthiopie a confirmé que PvEBPII peut se lier aux réticulocytes Duffy-positifs et Duffy-négatifs, mais à une fréquence plus faible que PvDBP. Cette découverte indique que le domaine PvEBPII n’a peut-être pas les mêmes capacités de liaison que le domaine PvDBPII malgré leurs similitudes structurelles [51]. Le récepteur principal de PvEBP étant encore inconnu, on ne sait pas si ce ligand alternatif potentiel joue un rôle dans l’invasion des réticulocytes Duffy-négatifs. Enfin, il a été récemment évoqué que les réticulocytes Duffy-négatifs peuvent exprimer transitoirement des niveaux faibles de DARC à la surface de leurs réticulocytes. Des données préliminaires suggèrent que DARC pourrait être exprimé à la surface des cellules précurseurs érythroïdes des individus Duffy-négatifs, mais cela doit encore être confirmé [90].
Mécanismes hypothétiques d’invasion de P. vivax dans les réticulocytes Duffy-négatifs. Hypothèse 1: Les mérozoïtes de P. vivax envahissent les réticulocytes Duffy-négatifs par l’interaction entre le ligand PvDBP et le récepteur DARC, ce qui implique que les réticulocytes Duffy-négatifs peuvent exprimer DARC à un niveau faible et/ou transitoire. Hypothèse 2: Les mérozoïtes de P. vivax envahissent les réticulocytes Duffy-négatifs par l’interaction entre le ligand PvDBP et un récepteur inconnu exprimé à la surface des réticulocytes Duffy-négatifs. Hypothèse 3: Les mérozoïtes de P. vivax envahissent les réticulocytes Duffy-négatifs par l’interaction entre le ligand PvEBP et un récepteur inconnu exprimé à la surface des réticulocytes Duffy-négatifs. Hypothèse 4: Les mérozoïtes de P. vivax envahissent les réticulocytes Duffy-négatifs par l’interaction entre un ligand inconnu exprimé à la surface des mérozoïtes de P. vivax et un récepteur inconnu exprimé à la surface des réticulocytes Duffy-négatifs. Figure créée avec BioRender.com.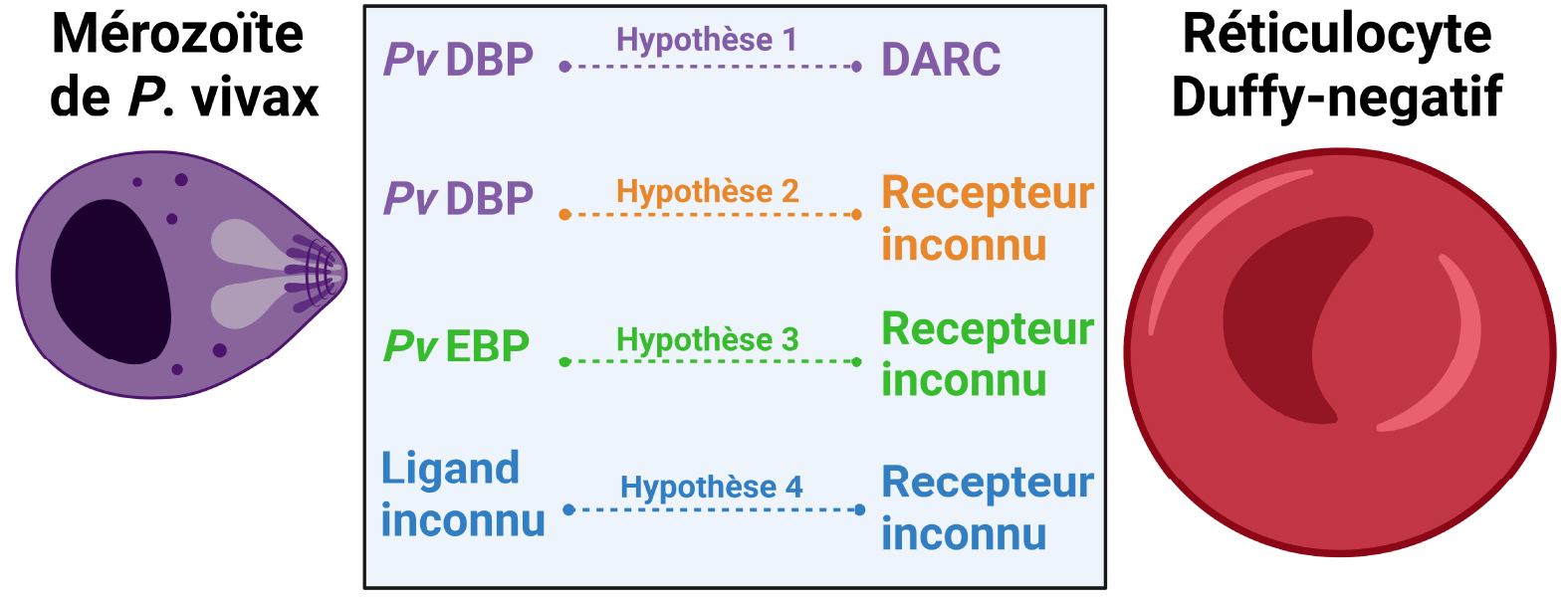
4.4. Invagination
Une fois que les mérozoïtes de P. vivax sont engagés de manière irréversible dans le processus d’invasion, ils pourraient pénétrer dans les réticulocytes via l’activité du glidéosome et se sceller dans la vacuole parasitophore. Bien que ces mécanismes ne soient pas clairs, il semble que la protéine PvTRAg36.6 et la protéine P. vivax Early Transcribed Membrane Protein (PvETRAMP) soient colocalisés dans la région apicale des mérozoïtes pour former et/ou maintenir la membrane de la vacuole parasitophore [52].
Bien que les technologies protéomiques ne soient pas encore pleinement exploitées, elles semblent essentielles pour répondre à des questions concernant les mécanismes d’invasion de P. vivax. L’amélioration constante des techniques (électrophorèse sur gel bidimensionnelle, spectrométrie de masse, criblage à deux hybrides de levure, etc.), l’approche à haut débit, les programmes de prédiction computationnelle et les capacités de traitement des données offrent d’énormes possibilités pour étudier l’expression, l’interaction et la modification des ligands de P. vivax. Cependant, les données protéomiques seules semblent être insuffisantes pour établir une analyse fine du processus d’invasion et proposer des modèles testables. Elles doivent être intégrées dans une connaissance plus globale et fondamentale de la biologie de P. vivax [91].
5. Conclusion
Pendant longtemps, le paludisme à P. vivax a été considéré comme une infection bénigne. Par conséquent, la recherche sur P. vivax a reçu une priorité moindre de la part des chercheurs, des décideurs politiques et des agences de financement. En outre, l’accès limité au matériel biologique et le manque de systèmes robustes pour la culture in vitro ont entravé le développement d’études fonctionnelles.
Pour résoudre ce problème, les scientifiques ont pris en compte les données provenant de familles de protéines homologues chez d’autres espèces de Plasmodium et ont produit des protéines recombinantes pour étudier les ligands de P. vivax potentiellement impliqués dans le processus d’invasion. Mais ces méthodes n’ont pas permis d’obtenir des informations complètes sur les fonctions réelles des ligands natifs. En fait, la question cruciale pour la recherche sur le paludisme à P. vivax recoupe plusieurs disciplines. En effet, les technologies génomiques apportent un certain éclairage sur la diversité génétique des souches P. vivax. Mais elles ne sont peut-être pas suffisantes pour comprendre les conséquences fonctionnelles de ces variations. Alors que les technologies transcriptomiques sont des outils puissants pour caractériser les transcriptions hautement régulées et spécifiques à chacun des stades de développement de P. vivax, l’analyse des données reste limitée par l’insuffisance d’échantillons et la difficulté d’étudier les gènes de ménage à expression constante. Cependant, de récentes applications des technologies dites « single-cell » ont permis de produire des profils transcriptomiques à la fois de parasites au stade hépatique et des cellules hôtes pour explorer les interactions hôte-parasite [92]. Il est donc possible que cette technologie soit appliquée à l’étude des facteurs de l’hôte dans la voie d’invasion des réticulocytes humains. En plus des études omiques, divers modèles in vivo ont été utilisés pour étudier les voies d’invasion de P. vivax. Les modèles de primates non humains (PNH) ont été largement utilisés dans le passé, malgré leurs limites inhérentes telles que les problèmes éthiques et les variations de la sensibilité de l’hôte à l’infection par des isolats de terrain de P. vivax nécessitant une adaptation avant la réplication, ce qui modifie probablement les caractéristiques biologiques des souches adaptées [55]. Plus récemment, des efforts ont été entrepris pour développer des systèmes de souris humanisées comme modèles pour reproduire in vivo les infections à P. vivax [93]. Bien qu’encourageants, ces systèmes très fragiles et coûteux n’en sont qu’à leurs débuts et doivent être perfectionnés pour permettre le maintien continu d’une infection à P. vivax asexuée au stade sanguin et le développement de parasites P. vivax génétiquement modifiés [94].
Dans ce contexte, les technologies protéomiques se concentrent sur le décryptage des diverses interactions entre les ligands impliqués dans le processus d’invasion de P. vivax et leurs récepteurs spécifiques. Néanmoins, elles ne parviennent pas à fournir une vision générale des mécanismes d’invasion de P. vivax, notamment en expliquant comment le parasite est capable d’envahir les réticulocytes Duffy-négatifs. Le développement d’études multi-omiques intégrées et complémentaires ainsi que les études d’invasion in vitro devraient nous permettre de mieux comprendre la biologie de ce parasite et soutenir le développement de stratégies de prévention, de contrôle et d’éradication dans le monde entier.
Conflit d’intérêt
Les auteurs déclarent ne pas avoir de conflit d’intérêt.
Remerciements
Cette revue a été soutenue par une subvention de l’Agence Nationale de la Recherche (VIPeRs, ANR-18-CE15-0026), par l’Institut Pasteur, Paris, le Laboratoire d’Excellence (LabEx) « Alliance Française de Parasitologie pour la Santé » (ANR-11-15 LABX-0024-PARAFRAP) et l’Université de Strasbourg à travers le Programme IdEX 2022 (Attractivité — Dotation d’accueil des professeurs d’université recrutés en 2021).




 CC-BY 4.0
CC-BY 4.0