1. L’immunité innée et les virus, une relation antagoniste
L’une des premières lignes de défense physiologique de l’hôte est l’immunité antivirale innée, comprenant la production d’effecteurs antiviraux capables de limiter la réplication virale dans les cellules. En réponse, des virus capables de contourner, échapper ou contrer ces effecteurs, en produisant des protéines antagonistes, sont sélectionnés au cours du temps. Lorsque le virus est pathogène, cette relation antagoniste entraîne une « course aux armements génétiques » où les deux entités co-évoluent pour survivre [1, 2]. Il en résulte des signatures d’adaptations génétiques (sélection positive) dans le génome de l’hôte et du virus. Ces signatures d’adaptations peuvent se manifester sous forme d’excès de mutations non-synonymes (qui modifient les acides aminés), d’évènements de recombinaison, ou de duplications/pertes de gènes qui altèrent notamment l’interface virus–hôte [3]. Elles sont ainsi des témoins indirects de la pathogénicité de virus ayant circulé ou circulant chez les hôtes. Chaque hôte ayant sa propre histoire évolutive avec les virus, les signatures peuvent être spécifiques à l’espèce hôte et se traduire par des différences fonctionnelles majeures dans la réponse antivirale. Ainsi, ces innovations génétiques peuvent non seulement expliquer la variabilité de la susceptibilité entre espèces hôtes, mais aussi constituer des barrières naturelles aux transmissions inter-espèces [4, 5]. Appréhender l’évolution de ces interfaces moléculaires virus–hôtes est donc une approche originale et robuste pour caractériser les déterminants de la spécificité des interactions virus–hôte, et décrypter les facteurs immunitaires de l’hôte qui sous-tendent les événements de transmissions inter-espèces.
2. Les chauves-souris, un modèle de choix
Dans ce contexte, les chauves-souris (ordre des Chiroptères) sont un modèle de choix car elles hébergent une grande diversité de virus, dont plusieurs ont déjà émergé chez l’humain et les animaux domestiques, tels que les virus MERS-CoV et Nipah [6]. Par ailleurs, un certain nombre de caractéristiques écologiques et biologiques contribuent à renforcer leur rôle de réservoirs de certains pathogènes viraux.
Notamment, les chiroptères constituent le deuxième ordre de mammifères le plus diversifié après les rongeurs, représentant près de 20 % de la biodiversité. Seuls mammifères capables de vol actif, ils sont également remarquables par leur abondance et leur distribution ubiquiste, leur longévité importante par rapport à leur taille (jusqu’à plus de 30 ans selon l’espèce), leur capacité migratoire sur des centaines de milliers de kilomètres et leur mode de vie varié (colonies multi spécifiques ou vie en solitaire) et souvent grégaire. Si des études récentes suggèrent que les chauves-souris ne seraient pas des réservoirs « spéciaux » tel qu’admis pendant des années, leur richesse spécifique [7, 8], couplée à ces caractéristiques écologiques font de ces mammifères d’excellents réservoirs d’agents zoonotiques [9].
D’autre part, des études expérimentales suggèrent que les chauves-souris hébergent certains virus sans présenter de symptômes apparents [10]. Ces caractéristiques ont récemment suscité des efforts considérables pour caractériser les mécanismes immunitaires de ces mammifères volants. L’hypothèse maintenant communément admise est une balance entre tolérance et résistance immunitaire. En effet, des adaptations spécifiques aux chauves-souris ont été depuis peu mises en évidence au niveau de l’inflammasome, notamment dans les gènes codant les protéines NLRP3, STING, et IRF3 [11, 12, 13], atténuant les effets néfastes liés à l’accumulation des composés oxydants en lien avec la consommation énergétique requise par le vol, et réduisant l’inflammation excessive. Ces adaptations contribueraient à augmenter la tolérance virale. Néanmoins, avec 60 millions d’années de divergence [14], les chauves-souris ont co-évolué avec une grande diversité d’agents pathogènes viraux. Par conséquent, des changements adaptatifs en réponse aux virus pourraient également contribuer à contrôler efficacement les infections virales [6]. Par exemple, un certain nombre d’effecteurs antiviraux chez les chauves-souris portent des signatures de pression de sélection, notamment dans des protéines antivirales majeures de l’immunité innée, telles que l’enzyme d’édition de l’ARNm des Apolipoprotéines B, les protéines de type polypeptide catalytique (APOBECs), la protéine « tetherin », la protéine transmembranaire 3 induite par l’interféron (IFITM3) [15, 16, 17]. Malgré ces avancées, une grande partie de la diversification fonctionnelle de l’immunité innée des chauves-souris reste à élucider.
3. La protéine kinase R, un effecteur antiviral puissant et à large spectre
Afin de déchiffrer comment les chauves-souris se sont adaptées à leurs virus, et comprendre l’impact fonctionnel de ces adaptations passées, nous avons récemment caractérisé la diversification d’une protéine antivirale majeure, la protéine kinase R (PKR). Conservée chez l’ensemble des vertébrés, PKR est un effecteur antiviral contre un large spectre de familles virales à ADN et à ARN, telles que les Poxviridae, les Herpesviridae et les Orthomyxoviridae [18]. Au cours d’une infection, PKR détecte l’ARN double brin viral, ce qui conduit à son activation et à la phosphorylation d’un substrat cellulaire, la protéine EIF2α (eukaryotic initiation factor 2 alpha), induisant ainsi l’arrêt de la traduction des protéines cellulaires et virales. L’importance de PKR dans l’immunité innée est notamment soulignée par les multiples stratégies adoptées par les virus pour contourner, bloquer ou échapper à la fonction de PKR [19]. Un exemple bien documenté est celui des Poxvirus, virus circulant chez un grand nombre de mammifères, y compris les chauves-souris. Ces virus produisent une protéine antagoniste, appelée K3, agissant par mimétisme du substrat EIF2α et inhibant ainsi l’action de PKR [20]. Au cours de l’évolution, ces interactions antagonistes entre PKR et les protéines virales ont évolué dans des dynamiques de courses aux armements génétiques chez les primates [21, 22]. Chez les chauves-souris, la trajectoire évolutive de PKR était jusqu’alors inconnue.
Dans l’étude que nous avons récemment réalisée [23], nous avons combiné analyses évolutives et expérimentations fonctionnelles dans un contexte cellulaire, pour caractériser la diversification fonctionnelle de PKR chez les chiroptères. Pour ce faire, la séquence codante du gène a été analysée chez 33 espèces de chauves-souris, représentant 60 millions d’années d’évolution. En particulier, 14 espèces appartenant à dix familles ont été échantillonnées et nouvellement séquencées.
4. La PKR des chauves-souris a évolué sous fortes pressions de sélection exercées par d’anciens poxvirus
Les analyses évolutives ont mis en évidence que PKR a évolué sous fortes pressions de sélection chez les chiroptères. De plus, en comparant l’évolution de la PKR des chauves-souris à celle d’autres mammifères, notre étude a révélé que ces changements adaptatifs étaient plus fréquents chez les chauves-souris, et que la localisation des sites adaptatifs était remarquablement différente entre mammifères. Alors que la plupart des sites sous sélection positive sont concentrés dans le domaine kinase de la PKR chez les primates, les artiodactyles et les rongeurs, ces sites sont dispersés sur l’ensemble de la PKR de la chauve-souris (Figure 1A). La plupart de ces sites et régions de PKR correspondent à des interfaces connues entre la PKR humaine et des antagonistes viraux, tels que les protéines NS1 du virus de la grippe, TRS1 des cytomégalovirus ou NS5A des hepacivirus [24, 25], dont les homologues sont présents chez les virus de chauves-souris. Ces résultats suggèrent que les patrons observés résulteraient de courses aux armements génétiques entre la PKR des chauves-souris et des virus pathogènes et refléteraient donc des pressions exercées par différentes épidémies anciennes.
PKR a évolué sous pression de sélection chez les chauves-souris (Modifiée de [23]). (A) Sites sous sélection positive dans la PKR des mammifères. Le graphe représente les probabilités postérieures de sélection positive (axe y) pour chaque codon (axe x). Les barres rouges indiquent les sites identifiés par deux modèles évolutif. Les chiffres entre parenthèses représentent le nombre total d’espèces analysées dans chaque ordre de mammifères. (B) Approche méthodologique des tests fonctionnels dans les levures permettant de déterminer la sensibilité de PKR à l’antagonisme de K3. Dans ces tests, la protéine virale K3 est co-exprimée avec PKR. Lorsque la protéine K3 est incapable de bloquer la fonction de PKR (par une interaction directe protéine–protéine), PKR est alors fonctionnelle et inhibe la traduction dans les levures, ce qui se traduit par un arrêt de la croissance des levures. Dans les cas où la protéine K3 est capable d’interagir avec PKR pour bloquer sa fonction, la traduction n’étant pas inhibée, la croissance des levures est préservée permettant la réplication virale. (C) Tests fonctionnels révélant que les PKRs de différentes espèces de chauves-souris diffèrent dans leur capacité à résister à l’antagonisme par les K3 de eptesipoxvirus (EPTV), de la vaccine (VACV) et de la variole (VARV). (D) Test fonctionnels de deux mutants de PKRs permettant d’identifier les déterminants génétiques qui contribuent aux différences fonctionnelles entre les PKRs. (E) Alignement multiple et comparaison de la séquence de la protéine K3 entre des poxvirus divergents. Les couleurs indiquent les variations de site entre les séquences par rapport à la séquence consensus. (F) Tests fonctionnels de la capacité d’antagonisme de la protéine K3 du virus eptesipox et du mutant K3δCter (δ85–120 aa). (G) Représentation du locus canonique du gène EIF2AK2/PKR chez les chauves-souris, les primates, les rongeurs, les carnivores et les artiodactyles. Les flèches de couleur unie représentent les gènes EIF2AK2, la flèche rayée montre le pseudogène EIF2AK2 et les flèches blanches sont des gènes synténiques adjacents. Les gènes dupliqués EIF2AK2 chez M. myotis et M. velifer sont situés en tandem dans le génome, tandis que le pseudogène est localisé en dehors du locus canonique. (H) Tests d’activité luciférase évaluant la fonction cellulaire des PKR dupliquées et leur capacité à échapper à des antagonistes viraux dans des cellules humaines. Les expériences comparent l’activité d’une protéine luciférase dans des cellules transfectées avec ou sans PKR humaine, ou avec les PKR de Myotis myotis (PKR1 ou PKR2), en présence ou en absence des antagonistes viraux : des antagonistes viraux : E3 de VACV, et NS5A du HCV de chauves-souris (souches O. martiensseni et P. macrotis), NS5A du HCV humain (JFH1), NS1 de l’IAV humain, TRS1 du CMV humain, et E3 d’EPTV. Lorsque PKR est fonctionnelle, elle inhibe la traduction, ce qui entraîne une réduction de la production de luciférase et une diminution de la lumière émise. Les variations d’activité luciférase permettent de déterminer la capacité des différentes PKR à résister à l’inhibition par ces antagonistes viraux.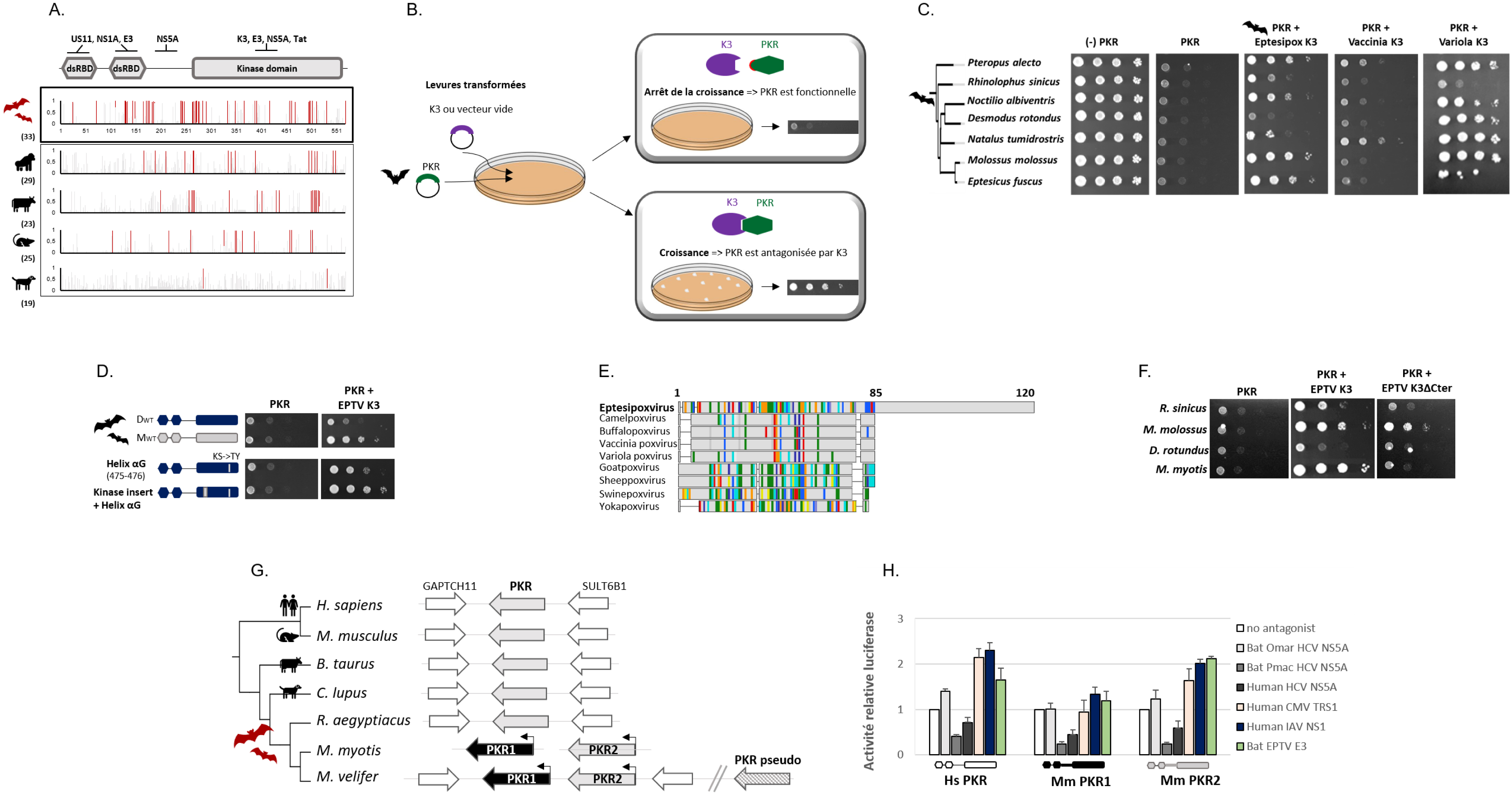
Pour tester cette hypothèse, la PKR de différentes espèces de chauve-souris ont été exprimées dans des cellules de levures permettant de tester (i) leurs fonctions cellulaires (i.e., capacité à bloquer la traduction) et (ii) leur sensibilité à l’antagoniste K3 de divers poxvirus, y compris les K3 d’un poxvirus isolé chez les chauves-souris (Eptesipoxvirus, EPTV), du virus de la vaccine (VACV), et de la variole (VARV) (Figure 1B). Cette approche a révélé que les PKR diffèrent dans leur capacité à échapper aux antagonistes viraux d’une manière spécifique à l’espèce hôte et au virus (Figure 1C). Ces résultats suggèrent que d’anciens poxvirus ont été des moteurs importants dans l’adaptation de la PKR des chauves-souris, et que leur coévolution a façonné des interfaces spécifiques entre PKR et K3 chez les chiroptères. La caractérisation de cette interface, tant du côté de l’hôte que du côté du virus, a permis d’identifier dans la PKR des chauves-souris une nouvelle région d’interaction avec la protéine K3 d’EPTV (Figure 1D–F). Du côté de l’hôte, nous avons caractérisé le rôle synergétique deux régions localisées dans le domaine kinase de PKR (l’hélice αG et l’insert), qui pourrait être spécifique aux chiroptères (Figure 1D). Du côté du virus, l’alignement des séquences K3 de différents poxvirus a révélé une insertion d’acides aminés spécifique à la protéine K3 du virus EPTV qui est indispensable pour bloquer la fonction des PKR des chauves-souris (Figure 1E,F). D’autres études seront importantes pour déterminer si d’autres poxvirus de chauves-souris ont évolué vers des changements adaptatifs similaires et pour déchiffrer l’implication fonctionnelle dans la pathogénicité et l’épidémiologie des poxvirus.
5. Des duplications ont façonné la diversification de PKR chez les espèces du genre Myotis
D’autres formes de changements génomiques peuvent être adaptatives durant les courses aux armements entre le virus et l’hôte. En particulier, la duplication et la recombinaison des gènes sont des mécanismes participant à la diversification du répertoire antiviral des mammifères [26, 27]. En étudiant l’évolution du gène qui code la protéine PKR, nous avons découvert qu’au-delà des substitutions, la duplication de gènes a contribué à l’évolution de PKR chez les chiroptères du genre Myotis. Alors que tous les autres mammifères étudiés possèdent un seul gène codant pour la protéine PKR, plusieurs espèces de chauves-souris du genre Myotis expriment au moins deux copies de PKR (Figure 1G). Cela indique une histoire évolutive complexe, marquée par une duplication ancienne de PKR, suivie de duplications indépendantes spécifiques à chaque espèce.
Afin de déterminer l’impact fonctionnel de tels changements, nous avons testé la fonction des copies de PKR, leur sensibilité à divers antagonistes viraux et leur capacité à bloquer la réplication de deux virus divergents (virus de la vaccine et de la stomatite vésiculaire). Ces approches fonctionnelles ont révélé que les copies de PKR étaient capables d’arrêter la synthèse des protéines, mais différaient significativement dans leur capacité à échapper aux antagonistes viraux de virus à ARN et ADN (NS1 du virus influenza A, TRS1 du cytomegalovirus et E3 du virus EPTV) (Figure 1H). Ces résultats indiquent que la duplication de PKR a contribué à diversifier les mécanismes d’échappement à l’antagonisme viral, et que certains virus, tel qu’influenza, pourraient être des moteurs de cette diversification. Par ailleurs, les tests d’infection ont révélé que les copies de PKR sont capables de réduire la réplication virale, mais se distinguent dans leur capacité à restreindre la réplication du virus de la vaccine, et ce, malgré une expression comparable des copies de PKR. Par conséquent, l’expansion de PKR chez les Myotis a conduit à la production d’au moins deux effecteurs antiviraux fonctionnels avec une spécialisation potentielle de leur fonction.
6. Conclusions
En résumé, notre étude [23] a montré que des épidémies virales ont façonné le génome des chauves-souris et a apporté des connaissances cruciales sur la diversification fonctionnelle du répertoire antiviral des chauves-souris. Il a été suggéré que la tolérance immunitaire plutôt qu’un contrôle viral accru joue un rôle clé dans l’immunité des chauves-souris [11, 13, 28]. Nous avons cependant montré que, les changements adaptatifs dans les PKR des chauves-souris augmentent leur fonction antivirale et leur capacité à échapper à l’inhibition par les virus, ce qui soutient un renforcement adaptatif des défenses antivirales chez certaines espèces. Ceci est en accord avec plusieurs études rapportant un taux d’évolution accéléré dans les facteurs de restriction des chauves-souris, indiquant une défense accrue contre l’infection virale [15, 29, 30, 31]. Chaque espèce ayant sa propre histoire d’exposition virale, des communautés virales spécifiques ont certainement conduit à une adaptation spécifique de l’immunité antivirale de chaque espèce, soulignant la nécessité d’inclure de multiples espèces dans les études fonctionnelles comparatives. En conclusion, si l’atténuation de la réponse inflammatoire pourrait être commune aux chauves-souris, des adaptations spécifiques, en réponse à d’anciennes épidémies virales, pourraient avoir façonné l’immunité innée de chaque espèce. D’autres études fonctionnelles et mécanistiques sont maintenant nécessaires pour (i) déchiffrer la fonction, les mécanismes et voies de signalisation de la PKR dupliquée, (ii) déterminer s’il y a une spécialisation dans la réponse antivirale ou les virus ciblés et (iii) comprendre les coûts et bénéfices associés à cette duplication.
Déclaration d’intérêts
Les auteurs ne travaillent pas, ne conseillent pas, ne possèdent pas de parts, ne rec˛oivent pas de fonds d’une organisation qui pourrait tirer profit de cet article, et n’ont déclaré aucune autre affiliation que leurs organismes de recherche.
Financements
Ce travail a été soutenu par le CNRS et par le LABEX ECOFECT (ANR-11-LABX-0048) de l’Université de Lyon, dans le cadre du programme « Investissements d’Avenir » (ANR-11-IDEX-0007) de l’Agence Nationale de la Recherche (ANR). SJ, DP et LE sont soutenues par l’Agence nationale de la recherche française, dans le cadre de la subvention ANR-20-CE15-0020-01 (projet « BATantiVIR »). SJ est également soutenue par le CNRS et la Fondation L’Oréal-Unesco « For Women In Science ». LE est soutenue par le CNRS et par des subventions de l’Agence française de recherche sur le VIH et les maladies infectieuses émergentes ANRS/MIE (nos ECTZ19143 et ECTZ118944). DP est soutenue par le CNRS, le Fonds européen de développement régional (FEDER) et l’ANR EBOFAC.
Remerciements
Nous remercions particulièrement l’ensemble des co-auteurs et co-autrices qui ont contribué à l’article original, ainsi que toutes les personnes qui nous ont aidé de près ou de loin.





 CC-BY 4.0
CC-BY 4.0