1. Introduction
La biologie superpose deux conceptions bien différentes de ce qu’est la vie. D’une part, sous sa forme la plus couramment comprise, elle exploite ce qu’on sait de la chimie. D’autre part elle fait apparaître, sous le nom de génétique, un monde abstrait plus difficile à saisir pour la majorité, celui des règles de l’hérédité. L’une ou l’autre de ces deux façons de voir, la biochimie ou la génétique, fournit un cadre conceptuel et expérimental qui permet de valider, ou non, les hypothèses cherchant à rendre compte de ce qu’est la vie. Le consensus – généralement non explicite – est que pour qu’une démonstration biologique soit acceptable il faut qu’elle mette en œuvre une approche analytique utilisant des expériences biochimiques où tous les éléments sont identifiés et compris. Depuis plus d’un demi-siècle, ce consensus est illustré par le développement d’un nouveau domaine des sciences de la vie, la biologie moléculaire, qui implique qu’une démonstration purement génétique – où la seule modification d’un caractère héréditaire au sein d’un organisme est utilisée pour établir la preuve de ce qu’on cherche à montrer – n’est pas acceptée comme suffisamment probante par la majorité des expérimentateurs. C’est dans ce contexte qu’est apparue une nouvelle discipline, la biologie synthétique. L’idée sous-jacente est de transposer à l’organisme vivant entier le recours nécessaire à une démonstration biochimique, en allant au-delà de la simple méthode analytique. Pour cela, il suffirait de combiner en un tout fonctionnel, défini par ses caractères biochimiques, les deux entités qui définissent la cellule vivante, à savoir un acide nucléique jouant le rôle de programme génétique et un « châssis » constitué de toute la machinerie permettant l’expression de ce programme, au sein d’une enveloppe semi-perméable permettant à la cellule d’interagir avec son environnement.
En raison des réussites spectaculaires de la biologie moléculaire et du rôle central qu’y jouent les acides nucléiques, la plupart des chercheurs, oubliant le châssis pourtant nécessaire, ont limité leur vision de la biologie synthétique à la conception et la synthèse d’acides nucléiques ou d’analogues jouant le rôle de programme génétique. Cette vue restrictive repose sur la généralisation du transfert par conjugaison de morceaux de chromosomes, et même de chromosomes entiers d’une espèce à une autre, connus depuis les débuts de la biologie moléculaire [1]. Depuis la fin des années 2000, on sait en effet construire puis transplanter des gènes ou des génomes synthétiques directement au sein de cellules modèles [2]. Ce sous-ensemble expérimental constitue la très grande majorité des expériences étiquetées comme relevant de la biologie synthétique. C’est donc là que la construction d’entités destinées à usage malveillant est la plus critique.
À ce jour, la majorité des efforts concernant l’utilisation de châssis pour la biologie synthétique se limite à la simplification du génome d’espèces existantes pour les rendre compatibles avec la mise en place de réseaux génétiques synthétiques (voir par exemple [3, 4, 5, 6]). Pourtant, ne prendre en compte que le programme génétique et sa synthèse artificielle revient à ignorer la contribution centrale des châssis cellulaires, qui comportent les agents responsables de l’apparente « animation » de la chimie biologique [7]. Cela n’est compris que par une très petite minorité d’expérimentateurs. Les défis scientifiques et techniques à relever pour synthétiser de novo un châssis fonctionnel sont tels que seuls quelques rares laboratoires ont cherché à réaliser cet exploit [8], et ils restent encore très loin d’y parvenir. Cette difficulté est principalement due au fait qu’il ne suffit pas de mettre ensemble tous les composants reconnus d’un châssis modèle pour qu’il soit fonctionnel [9]. Par chance, l’étude des propriétés nécessaires à réunir pour permettre cette création montre que ce domaine est mal adapté à un usage qui soit contrôlable par des opérateurs malfaisants. Nous limitons donc notre discussion à la synthèse de gènes et de génomes, qui reste un domaine particulièrement préoccupant.
2. Ce qu'est la biologie synthétique
La mode, hélas, domine souvent le choix des thèmes de recherche. C’est que, pour avoir une chance raisonnable de développer des travaux toujours coûteux, il faut procéder à la manière des agences de publicité et montrer qu’on participe à un courant porteur. Cette contrainte omniprésente, qui a remplacé la curiosité naguère à l’origine de la plupart des découvertes importantes, conduit à l’utilisation de termes qui attirent les financeurs. « Biologie synthétique » en fait partie. Pour comprendre ce que recouvre le domaine en cause, il faut comprendre ce que le fait même de nommer signifie.
2.1. Nommer n'est pas sans conséquences
Umberto Eco l’a souligné dans son livre, Le Nom de la Rose, nous sommes nominalistes et pour nous l’essence d’une chose tient dans la pureté de son nom : Stat rosa pristina nomine, nomina nuda tenemus. Ce n’est pas innocent, car nommer dirige la pensée vers tout un contexte, celui de l’usage du nom en cause. Les véritables tenants de la démarche scientifique l’ont bien compris et utilisent des racines grecques pour nommer ce qui relève de la science [10], rappelant ainsi à tous son origine. Or, la discipline que nous appelons biologie synthétique recouvre en réalité deux domaines bien différents qui relèvent chacun d’une pratique recouvrant à la fois le savoir académique et ses applications. Ces pratiques consistent à créer des processus qui sont soit,
- non naturels avec des composants naturels (il s’agit alors d’une simple extension du génie génétique et cela explique que pour beaucoup la biologie synthétique n’ait rien d’original) ;
soit,
- naturels avec des composants non naturels.
Cette dichotomie disparaît lorsque ces processus sont combinés en assemblages divers. Cela brouille l’image de la biologie synthétique qui évolue alors en parallèle avec un domaine très flou, celui de la « biologie des systèmes », et renforce l’ambiguïté du terme « système » très utilisé par la pensée confuse qui fait un usage immodéré du terme « complexe », dont le sens est aussi bien « si compliqué qu’il est impossible de le comprendre », que « qui implique un ordre particulièrement élaboré et par là mystérieux ». Physicien, Murray Gell-Mann avait essayé de remédier à cette ambiguïté délétère en remplaçant le latin complex par le grec plectics pour s’écarter des amalgames induits par l’usage abusif du terme [11]. Dans un effort distinct, Victor de Lorenzo et moi-même avons tenté de réunir les deux domaines sous le nom de « biologie symplectique ». Nous n’avons pas eu plus de succès que Gell-Mann dont l’article, curieusement, n’est apparu en ligne que longtemps après son écriture, juste au moment où, indépendamment, nous avancions la même idée [12]. Ce point d’histoire souligne l’ambivalence qui affecte toutes les discussions mettant en cause les usages, bons ou mauvais, de la biologie synthétique.
2.2. Brève histoire du domaine
Ce qui est au goût du jour est l’aboutissement d’une histoire, parfois ancienne, mais souvent ignorée des créateurs ou des usagers de la mode. Or, de plus en plus répandue, cette ignorance conduit non seulement à réinventer la roue, mais aussi, parce que nous avons perdu la mémoire, à construire des modes faites d’accrétion d’entités incompatibles entre elles. Cette faille conceptuelle rend les conséquences du développement de la biologie synthétique, qu’elles soient bonnes ou mauvaises, difficiles non seulement à évaluer mais aussi à maîtriser correctement.
Parce que c’est le titre d’un ouvrage qui a eu en son temps un succès certain, on fait parfois référence au livre de Stéphane Leduc (1853-1939), La Biologie synthétique, publié en 1912 [13], pour présenter ce nouveau domaine. Son contenu mériterait une analyse approfondie car il illustre remarquablement ce à quoi conduit l’usage abusif de l’analogie, très utilisée par la mode, en particulier lorsqu’il s’agit d’analogie de formes. Ce qui est présenté comme nouveau aujourd’hui est en réalité déjà présent en 1970 dans un commentaire de James Danielli (1911-1984) [14], détaillé dans une revue qui se préoccupe des conséquences négatives des applications de la science, le Bulletin of the Atomic Scientists [15] :
Nous pouvons aujourd’hui facilement distinguer trois âges dans la science qui constitue la biologie moderne : L’âge de l’observation —> L’âge de l’analyse —> L’âge de la synthèse. […] [...] L’âge de la synthèse n’en est qu’à ses débuts, mais il est clairement perceptible. Au cours de la dernière décennie (1960-70), nous avons assisté aux premières synthèses d’une protéine, d’un gène, d’un virus, d’une cellule et de souris allophéniques. Rien d’aussi spectaculaire n’a jamais été vu auparavant en biologie. Jusqu’à présent, les sélectionneurs de plantes et d’animaux étaient capables de créer des espèces virtuellement nouvelles, et ce à un rythme de l’ordre de 104 fois supérieur à celui des processus évolutifs moyens. Un nouvel accroissement de ce rythme se profile à l’horizon. Pour ce faire, nous avons besoin de quelques « premières » supplémentaires : (1) pouvoir synthétiser un chromosome à partir de gènes et d’autres macromolécules idoines ; (2) pouvoir insérer un chromosome dans une cellule ; ou, plutôt que (1) et (2), pouvoir (3) insérer des gènes dans une cellule d’une autre manière ; (4) nous devons aussi apprendre à mettre sous les mécanismes de contrôle de la cellule l’ensemble des gènes qui y sont importés, de sorte qu’ils ne s’emballent pas. Aucun de ces problèmes ne semble être d’une difficulté exceptionnelle.
Dans cet article de 1972, Danielli nous met en garde, comme nous le faisons à nouveau un demi-siècle plus tard :
Au fur et à mesure de cette évolution, il pourrait être nécessaire de veiller à ce que ces organismes ne puissent pas vivre en dehors de l’environnement industriel particulier dont ils ont besoin, et même d’inclure des mécanismes d’autodestruction pour réduire leur viabilité dans la biosphère naturelle.Toutefois, ces mécanismes d’autodestruction, et tous les autres mécanismes nocifs pour une espèce, ne doivent être introduits qu’après une étude théorique et expérimentale particulièrement rigoureuse, en raison de la possibilité, toujours présente chez les systèmes génétiques, que le processus pathogène passe d’une espèce à l’autre et engendre ainsi de nouveaux organismes pathogènes fulminants.
Peu après, en 1974, le nom anglais « synthetic biology » est proposé par Wacław Szybalski [16], pour définir ce qui allait devenir le génie génétique, fondé sur l’accès récent à la détermination expérimentale de la séquence des gènes. Mais, par un effet de cette mode qui fait fi de l’histoire, c’est l’année 2004 qu’on considère souvent comme le début de cette discipline, avec la construction par Drew Endy d’un génome artificiel du bactériophage T7 où les signaux de contrôle de l’expression des gènes viraux étaient réécrits de façon compréhensible par l’homme [17]. Et, dès 2005, ces expériences purement académiques ont été suivies par la reconstruction du génome du virus de la grippe pandémique de 1918, une expérience irréfléchie qui ouvrait un espace innovant pour des applications malveillantes [18]. La synthèse chimique de gènes et de génomes s'est alors généralisée. Cela a naturellement suscité de sérieuses réserves en raison du danger évident de voir des agents pathogènes reconstitués par des acteurs malveillants, mais sans que des mesures contraignantes pour poser des limites ne soient mises en place [19]. L'Europe n'a pas été en reste. Dès avril 2006 s’est tenu à Airlie en Virginie un premier atelier mixte entre l’Europe et les États-Unis destiné à identifier les recherches en cours dans le domaine de la biologie synthétique et leurs conséquences pour la société, y compris en termes de biosécurité : "Synthetic Biology and Synthetic Genomes" (voir le résumé présenté en encadré). Aucune contrainte n’a cependant été imposée pour réguler l’usage possible des techniques de la biologie synthétique conduisant à des applications à double usage.
2.3. Principes de la biologie synthétique
Pour le distinguer du simple génie génétique, les créateurs de ce nouveau domaine, spécifique des sciences de la vie, soulignent quatre principes :
Résumé de l'atelier États-Unis-CE sur la biologie synthétique, Airlie House, 24–25 avril 2006
Coprésidents : Maurice Lex (UE) et Marvin Stodolsky (Doe)
Sessions :
- Recherche biologique, renforcement par les outils de la biologie synthétique ;
- Les nouveaux domaines de la R&S rendus possibles par la biologie synthétique ;
- Leçons déjà apprises dans le secteur des ELSI (ethical, legal and social issues), avec une discussion élargie sur les nouveautés apportées par la biologie synthétique ;
- Les nouvelles constructions génétiques, les génomes et les ressources disponibles ;
- Discussions sur les questions de protection contre l'utilisation abusive des ressources disponibles de la biologie synthétique.
La biologie synthétique n'a pas le même sens pour tous, y compris pour les biologistes moléculaires engagés dans des sciences qui peuvent relever de la biologie « de synthèse ».
Les travaux présentés dans la section 1 comprenaient des discussions sur la « structure en domaines » des chromosomes bactériens, et l'utilisation des codons dans leurs génomes pour la mettre en évidence (Danchin, Institut Pasteur, Paris, France) ; le rôle (et l'utilisation) de facteurs de transcription natifs et artificiels (fragments d'anticorps à chaîne unique, scFv, qui peuvent imiter des facteurs de régulation) pour réguler à la baisse l'expression de lacZ (de Lorenzo, CSIC, Madrid, Espagne) ; la synthèse, à partir d'oligonucléotides synthétisés chimiquement, du chromosome minimal de Mycoplasma genitalium (H. Smith, Venter Institute) et l'utilisation de chromosomes artificiels humains (HAC) générés à l'aide d'une méthode de clonage spéciale basée sur les réions centromériques humaines, pour introduire des gènes codant des protéines de son choix dans des cellules humaines (Larionov, NCI).
Lors de la session 2, Drew Endy, du MIT, l'un des principaux acteurs de la biologie synthétique, a parlé du Catalogue de bio-briques (http://parts.mit.edu) et des premières tentatives de construction de fonctionnalités en les utilisant. Stelling (ICS, Zurich, Suisse) a évoqué les défis auxquels est confrontée la biologie synthétique (et ses différences avec le génie génétique), notamment le manque de robustesse, des « unités » biologiques mal caractérisées (jusqu'à présent), l'environnement cellulaire bruité dans lequel ils doivent fonctionner, le manque d'isolation par rapport à d'autres influences (cytoplasmiques), et le manque de compréhension de l'intégration dans les systèmes. S. Panke (IPE, Zurich) a parlé d’expériences visant à construire des chemins métaboliques artificiels existant et fonctionnant indépendamment de l’environnement cellulaire, en commençant par la voie de la glycolyse d’Escherichia coli. D’autres exposés ont porté sur la conception de novo de protéines et la modification de la fonction de catalyseurs bactériens.
La session 3 était consacrée aux implications sociétales. Joyce Tait (Innogen Center, Édimbourg, Écosse) a abordé les questions internationales liées à la gouvernance de la biologie synthétique, notamment les processus de découverte scientifique et d’innovation, les aspects politiques et réglementaires et les préoccupations exprimées par le public. L’opinion internationale est influencée par les controverses de longue date concernant les organismes génétiquement modifiés (OGM) et il existe une crainte du public qui n’est pas apaisée par les efforts actuels d’éducation et de sensibilisation et qui pourrait être exacerbée par des affirmations trop enthousiastes sur les avantages de la recherche et les délais de mise en œuvre, ainsi que par des arguments contre la nécessité d’une réglementation. Shannon Nesby-Odell (CDC, Atlanta) a parlé de la biosécurité aux États-Unis en se concentrant principalement sur la construction des installations, l’équipement de sécurité et les bonnes pratiques de laboratoire comme moyens de réduire la probabilité d’une dissémination accidentelle. Elle a aussi évoqué la nécessité de messages clairs et concis mettant l’accent sur les avantages de la technologie, de promouvoir les défenseurs de cette dernière (par exemple au Congrès) et d’adopter une approche équilibrée des politiques. Les politiques de surveillance devraient être proactives plutôt que réactives et impliquer la communauté des chercheurs. Il convient d’adopter une approche à plusieurs niveaux, impliquant des instruments juridiques et la participation de la communauté, afin de minimiser les perceptions négatives injustifiées. Elle a souligné la nécessité d’une autorité exécutive, y compris d’une surveillance internationale harmonisée, afin de protéger la technologie contre les allégations d’intérêts particuliers et a noté que les actions de l’industrie pouvaient involontairement entraver le progrès. Dan Drell (DOE, remplaçant de dernière minute de Mildred Cho de Stanford) a évoqué les anciens programmes ELSI sur les implications éthiques, juridiques et sociales, notant que l’intégration des implications sociétales dans la haute technologie était toujours un défi, en particulier lorsque ceux qui mènent des enquêtes sur les implications sociales ne veillent pas à justifier l’importance de ces études pour le succès de la science. Les avantages doivent être expliqués avec beaucoup de franchise et sans exagération. De nombreuses questions se poseront (et se posent déjà) au-delà des préoccupations immédiates en matière de sécurité et de santé. Il faut aussi reconnaître que les grandes décisions sur ce qu’il convient de faire (et de ne pas faire) avec les nouvelles technologies sont prises par la société dans son ensemble, et non par les scientifiques agissants seuls.
La discussion ouverte qui a suivi les questions soulevées lors de la session 3 a révélé la nervosité de la communauté scientifique envers l’idée de surveillance réglementaire et la possibilité que de mauvais choix réglementaires puissent réduire notre capacité à réagir rapidement aux agents infectieux naturels ou artificiels ou à saisir de nouvelles occasions de développer des produits d’intérêt général. Les scientifiques européens ont estimé que la surveillance réglementaire dans l’UE était déjà suffisante pour couvrir les questions incluses dans l’accord de Berkeley. Toutefois, à d’autres moments de la discussion, la nécessité d’une capacité de mise en œuvre a été soulignée, ainsi que la nécessité d’identifier les expériences préoccupantes au fur et à mesure qu’elles se produisent. Dans ce domaine complexe, la législation, la surveillance des agences et les codes de conduite professionnels sont tous nécessaires. Il ne s’agit pas seulement d’un défi pour les biologistes – de nombreuses disciplines universitaires sont concernées. En outre, la question de l’établissement de normes est au moins aussi importante que la réglementation pour régir les processus d’innovation dans le domaine de la biologie synthétique, et il sera plus facile d’établir des règles sur les normes lorsque, comme c’est le cas aujourd’hui, les acteurs concernés sont relativement peu nombreux.
La session 4, le deuxième jour, a commencé avec George Church (Harvard) qui a parlé de son travail sur la synthèse d’oligonucléotides à l’échelle du micron et du traitement des erreurs de la polymérase qui peuvent en compromettre le succès. Santi (UCSF) a parlé de son travail de construction de gènes de polycétide synthase modifiés afin d’élaborer de nouvelles polycétide synthases en les assemblant à partir de modules plus petits. Dans l’ensemble, il a utilisé plus de 2,5 millions de pb de nouvelles séquences de gènes PKS pour valider son approche de l’identification de nouveaux ensembles multi-modules productifs, ce qui a donné lieu à une approche combinatoire pour la production de polycétides sur commande. M. Mulligan (Blue Heron Technologies) a parlé des tentatives de son entreprise pour industrialiser la synthèse des gènes. Compte tenu de l’expansion rapide de cette technologie, la faisabilité d’une régulation en dehors d’installations centralisées est peut-être un vain espoir.
Bien que la biologie synthétique ne soit pas encore une activité clairement définie, allant de la synthèse de l’ADN à l’ingénierie des protéines, l’atelier a mis en évidence le potentiel croissant de la synthèse des « unités » fonctionnelles des systèmes vivants et donc de leur reconfiguration, à la fois pour étudier leur fonctionnement et pour essayer de créer de nouvelles capacités. Ces travaux sont très prometteurs et devraient permettre de développer de nombreux avantages pour la santé humaine et la société, mais ils soulèvent aussi des inquiétudes quant à la manière dont ils pourraient être utilisés et détournés. Des travaux impressionnants sont en cours des deux côtés de l’Atlantique et la poursuite des conversations et des échanges d’informations sera nécessaire et bénéfique.
- D’abord, un principe d’abstraction applique aux entités canoniques des cellules les lois connues de la vie pour en concevoir d’analogues, mais de nature différente, que cela concerne les éléments utilisés dans les constructions ou leurs mises en relation (architectures ou contrôles régulateurs).
Cette idée, illustrée en 1957 par Fred Hoyle dans son livre Le Nuage noir, lui a permis d’imaginer une vie fondée sur une physique tout autre que celle que connaît la biologie [20]. Au-delà de cette vue romancée, le projet REPRAP (REPlicating RApid Prototyper, https://reprap.org) vise à créer une imprimante laser 3D qui se reproduise. La machine produit la plupart de ses propres composants, mais il lui manque des éléments cruciaux : un programme (gestion de l’information), une ligne d’assemblage (gestion du temps et de l’espace), et des fonctions spécifiques comme la lubrification. Ce n’est donc pas une réelle illustration de ce qu’on attend d’une biologie de synthèse. On notera ici que ce projet vise à synthétiser une contrepartie du châssis, en oubliant le programme, alors que les efforts dominants de biologie synthétique font l’inverse. L’objectif ultime, bien sûr, serait de faire la synthèse et du châssis et du programme.
Pour notre propos, l’intérêt des approches qui utilisent des maillons élémentaires différents de ceux de la chimie du vivant est qu’elles peuvent être vues comme le moyen d’éviter les conséquences néfastes d’accidents ou encore de l’usage malveillant de la biologie synthétique, nommée xénobiologie dans ce contexte [21]. Il faut toutefois souligner que, l’accès aux matériaux de base et aux équipements étant de plus en plus facile, le contrôle de leur usage devient de plus en plus difficile. Le risque d’accident et les dangers d’un mésusage sont de ce fait augmentés. À titre d’exemple, la situation peut être comparée à celle des imprimantes 3D qui peuvent être facilement utilisées pour produire illégalement des armes à des fins criminelles [22]. Les procédés de synthèse et d’expression de gènes étant de plus en plus largement accessibles à un large éventail d’acteurs, la possibilité d’un encadrement efficace est illusoire ; leur mise en œuvre à des fins malveillantes ne peut donc être exclue. - Un deuxième principe repose sur la standardisation des procédés et des composants. Il voit la biologie synthétique comme appartenant au domaine de l’ingénierie. Cela implique de respecter des normes où la construction des édifices artificiels harmonise et organise de façon rationnelle les matériaux et les principes de base de la biologie moléculaire sous la forme de programmes exprimés dans un châssis. Pour commencer, ce châssis dérive d’organismes existants qu’on imagine naïvement entièrement caractérisés (ce qui suppose une connaissance qui, en réalité, n’est ni complète ni universellement accessible). On remarque ici que mettre à disposition du public général des éléments standardisés rend la construction de circuits synthétiques très facile. Il s’agit là encore d’un caractère qui peut être utilisé à mauvais escient.
- Le troisième principe concerne l’objectif majeur de la biologie synthétique. Il vise notamment, à partir du raisonnement de l’ingénieur, à reconstituer un organisme vivant de façon à comprendre ce qu’est la vie et découvrir les entités manquantes. Les acteurs du domaine aiment à rappeler ce que disait Feynman : « Ce que je ne peux créer, je ne le comprends pas » (Figure 1).

Sur le tableau noir de Feynman au moment de sa mort. https://digital.archives.caltech.edu/collections/Images/1.10-29/.
- Enfin, le quatrième principe prend en compte la maxime de Dobzhansky : « rien en biologie n'a de sens, si ce n'est à la lumière de l'évolution » [23]. Les organismes synthétiques vont évoluer, et il faut utiliser rationnellement la sélection naturelle pour produire des variants des fonctions retenues comme importantes en combinant conception et évolution pour en diriger l’adaptation aux conditions choisies [24].
Une conséquence constante de l’utilisation de l’évolution d’organismes synthétiques, choisie ou subie, doit être soulignée : l’évolution n’a aucune raison a priori d’aller dans le sens souhaité par l’homme.C’est une particularité essentielle à comprendre si l’on considère les applications de la biologie synthétique, qui, même en principe sans danger, peuvent évoluer vers une forme néfaste. Tout organisme, y compris éloigné des formes existantes, apte à survivre dans un environnement naturel sans aide humaine, évoluera par lui-même, échappant à ses auteurs et les transformant en apprentis sorciers. Ce risque est bien compris et pris en compte par les structures de gouvernance universitaires, gouvernementales et même industrielles, mais il est susceptible d’être négligé par ignorance par un certain nombre d’acteurs, surtout s’ils sont malintentionnés.
2.4. Synthèse chimique de gènes et de génomes
À l’origine longue et difficile, et longtemps limitée à la synthèse d’oligonucléotides, la synthèse de gènes est possible commercialement depuis bien des années. Synthétiser quelques kilobases d’ADN est courant et la technologie s’améliore constamment. Ces progrès techniques ont une importance cruciale pour le développement de la fraction « naturelle » de la biologie synthétique. Cela concerne essentiellement des suites de chaînons élémentaires (« biobriques ») destinés à des usages industriels : nouvelles voies métaboliques ou amélioration des modes de régulation de l’expression de gènes pour telle ou telle application, ou pour introduire des variants des nucléotides canoniques dans les génomes [25]. Ces synthèses peuvent aussi servir à recréer des génomes entiers, en particulier hélas ceux d’organismes pathogènes dangereux, comme nous l’avons vu pour la grippe de 1918, mais aussi les poxvirus [26, 27]. Il est même possible d’utiliser ces techniques pour accroître leur virulence [28]. Par ailleurs, pour les génomes de pathogènes naturels, une petite quantité d’une construction initiale est facilement amplifiée. Un nouvel acteur, longtemps resté dans l’obscurité, l’intelligence artificielle (AI), est venu accroître le pouvoir d’invention malfaisante fondé sur le nombre immense des données génomiques [29]. En effet, spécialement sous sa forme générative, l’IA sait manipuler le texte des programmes génétiques et y incorporer les conséquences possibles de l’expression des gènes impliqués dans le pouvoir pathogène des bactéries ou des virus [30]. Ce danger doit être souligné au moment où l’évolution d’un de ces virus, mPox, cause une épidémie mondiale [31]. Cela est d’autant plus inquiétant que ces synthèses d’acides nucléiques peuvent désormais être effectuées dans des laboratoires artisanaux.
Dans le domaine militaire, on doit noter cependant que la guerre biologique n’a que peu d’intérêt stratégique sur le champ de bataille. Cela a été illustré au cours de l’invasion de la Chine par le Japon avec le programme de l’Unité 731 commencé en 1932 et achevé en 1945 [32], où les armes biologiques utilisées contre la population chinoise (Yersinia pestis, Bacillus anthracis, Vibrio cholerae, agents du typhus…) ont provoqué des pertes dans l’armée japonaise. Par exemple, l’usage de V. cholerae répandu en 1942 dans les puits au sud de Shanghai a tué des centaines de soldats japonais et interrompu les attaques japonaises sur le terrain. Mais la propagation des maladies a, pour les sociétés humaines, bien d’autres effets qu’une action directe sur la santé. Cela a été observé à nouveau au cours de l’analyse récente des conséquences économiques et sociales de la pandémie de SARS-CoV-2 [33]. Elles sont considérables. Il ne fait aucun doute qu’une agression biologique intentionnelle, qu’elle s’inscrive dans un contexte militaire ou terroriste, aurait les mêmes effets, qui seraient probablement amplifiés par un climat de panique lié au contexte stratégique ou politique.
Tout cela rend critiques non seulement la synthèse chimique des acides nucléiques longs, mais aussi les techniques de transplantation des génomes [34]. La réplication de l’ADN dans des hôtes choisis est simple dès qu’elle ne conduit pas à son expression (chromosomes artificiels YAC dans la levure pour le programme de séquençage du génome humain [35], fragments du chromosome de Mycoplasma mycoides dans la levure [36], le génome d’une cyanobactérie dans B. subtilis [37], etc.), ce qui permet d’obtenir toutes sortes de constructions nouvelles afin de les transplanter ensuite dans des châssis où elles seront actives. Cependant, cette transplantation reste encore une étape difficile à contrôler pour des génomes longs. Ce n’est pas le cas des virus pour lesquels les techniques de transfection sont monnaie courante [38]. L'utilisation, habituelle en virologie, de la synthèse génomique à base de ribozymes auto-excisables utilisée par Liang et al. [39] pour la génération de virus difficiles à multiplier est désormais généralisée [40], certainement sans le moindre contrôle. Il existe bien une mise en garde relayée par les médias de masse [41], qui donne des idées pour contrôler les laboratoires de synthèse ou les appareils permettant cette synthèse, mais sa mise en place dans le monde entier paraît malheureusement illusoire [42].
2.5. Usages non conventionnels des acides nucléiques de synthétiques en biologie synthétique
Il est courant qu’une invention technique destinée à un usage spécifique soit utilisée, voire détournée, à d’autres fins. C’est le cas de la synthèse de l’ADN, technique indispensable à la biologie synthétique. Son succès a donné lieu à une multitude d’usages dont les dérives néfastes potentielles ne sont pas encore bien explorées. Certaines des applications de la biologie synthétique en cours de développement sont tout à fait non conventionnelles et originales. En voici quelques exemples.
2.5.1. Calcul miniaturisé hautement parallèle
À condition d’être dans l’eau, les acides nucléiques forment des structures comprenant des sections en double hélice faites de brins dont la séquence est complémentaire. En effet, l’augmentation de l’entropie de la solution due à la contrainte imposée à la structure de l’eau lorsque les bases des nucléotides sont accessibles à ce solvant aux propriétés physiques uniques conduit à cette propriété géométrique remarquable. La cinétique de formation des doubles hélices est rapide, car elle rééquilibre très vite le réseau des protons de l’eau, et cela à longue distance. Cette caractéristique, qui est strictement liée à la séquence des nucléotides, a suscité un grand intérêt pour la réalisation de calculs hautement parallèles [43]. Par exemple, le problème classique, NP-complet, qui vise à trouver des sous-ensembles (cliques) de sommets tous adjacents deux à deux dans un graphe, souvent formulé comme la recherche d’une « clique maximale », a été résolu au moyen de techniques nécessitant la synthèse d’un groupe d’oligonucléotides correspondant à l’ensemble des cliques à six sommets, puis de mettre en œuvre le processus de sélection impliquant la formation d’hélices les associant spécifiquement les uns aux autres [44].
Plus récemment, l’ADN a été utilisé pour créer des analogues du calcul neuromimétique en utilisant des enzymes actifs sur l’ADN, dont les poids et les biais sont réglables, et qui sont assemblés dans des architectures multicouches [45]. Ces ensembles logiques sont assemblés dans des gouttelettes de la taille d’une cellule. Cette puissance de calcul et cette miniaturisation extrême ouvrent la voie à l’interrogation et à la gestion de systèmes moléculaires au contenu complexe. Utiliser de l’ADN plutôt qu’un ordinateur pour un objectif malveillant peut être le moyen de le dissimuler. Il faut tenir compte, cependant, des erreurs introduites dans la construction des oligonucléotides, de même que du temps nécessaire à leur construction, ce qui a nui à la généralisation de l’usage de l’ADN pour le calcul. Cet impact peut être réduit par la redondance, en multipliant les constructions en principe identiques. Cela ne convient pas au calcul, mais en revanche c’est parfait pour l’archivage.
2.5.2. Un nouveau type d'écriture et l'archivage des données massives
Beaucoup d’archives, parfois rassemblées dans d’immenses bâtiments, témoignent de l’histoire passée. Leur objectif est de maintenir la mémoire d’un très grand nombre de faits, peut-être intéressants un jour, mais ne demandant pas un accès immédiat. Faites de textes ou d’images, elles sont facilement encodées en écriture binaire. Plutôt que de conserver les originaux, on a donc très vite utilisé des supports variés, bandes magnétiques ou disques optiques, pour réduire leur espace de conservation. Cependant, la durée de vie de ces supports est très courte, souvent beaucoup moins d’un siècle, bien plus courte que ce qu’on attend pour des archives importantes. Au contraire, l’ADN est très stable. On le sait lorsqu’on reconstitue le génome d’espèces vivantes disparues depuis des milliers d’années [46]. Comme l’ADN se comporte comme le support matériel d’un texte écrit dans un alphabet à quatre lettres, il est facile d’imaginer y encoder n’importe quelle suite de symboles sous la forme d’une séquence de nucléotides, stockable dans un volume extrêmement petit. On peut aussi faire plusieurs copies indépendantes du même texte, ce qui réduit les conséquences négatives des erreurs ou du vieillissement et permet, même après un très long temps, de reconstituer le texte initial. Les Archives nationales de France ont ainsi conservé sous forme d’ADN double brin le texte numérisé de la Déclaration des droits de l’homme et du citoyen de 1789 et de la Déclaration du droit de la femme et de la citoyenne de 1791 [47]. Nous verrons plus loin qu’au-delà de la création d’un nouveau mode de communication de messages ou d’images, cet usage de l’ADN pour transmettre des informations utiles peut malheureusement être détourné à des fins néfastes.
2.5.3. Origami et nanoboîtes d'ADN
L’utilisation rationnelle des sous-unités de l’ADN polymérase et d’autres enzymes qui modifient la molécule d’ADN permet de l’assembler dans une variété de formes, selon un procédé semblable à celui de la construction, classique au Japon, d’origamis [48]. Pour construire un origami, on replie un long ADN simple brin au moyen de multiples brins complémentaires plus petits choisis par l’expérimentateur et jouant le rôle d’agrafes là où ils s’apparient en double brin à une région du brin principal. Ce pliage permet de créer des nano-architectures biocompatibles, prévisibles et statiques, ou des nanodispositifs dynamiques dont la géométrie est conçue rationnellement, et dont l’adressabilité spatiale est précise. Des peptides, des protéines, des aptamères, des sondes fluorescentes, des nanoparticules, etc., peuvent y être facilement intégrés, avec une précision nanométrique. Mis en route par des stimuli chimiques ou physiques, les nanodispositifs dynamiques peuvent, en étant contrôlés par différents composants moléculaires ou stimuli externes, passer d’une conformation à une autre ou se déplacer de manière autonome. Ils fournissent ainsi des outils puissants pour la biodétection intelligente ou l’administration de médicaments [49]. Des analogues de l’ADN comme les acides nucléiques peptidiques (peptide nucleic acids, PNA) ont aussi été proposés pour la fabrication d’origamis formant des nanostructures destinées à des usages similaires [50]. Comme dans le cas de la majorité des approches nanotechnologiques, il n’est pas facile d’éviter les possibilités d’un double usage (voir par exemple [51]).
L’ensemble de ces approches est résumé plus loin (Tableau 1) avec quelques exemples des constructions mentionnées. Certains, dont l’usage peut être détourné, sont décrits dans les quelques paragraphes qui suivent.
Quelques exemples de l’usage de la synthèse de programmes génétiques.
| Thème | Exemples | Références |
|---|---|---|
| Minimiser le génome | Mycoplasma mycoides JCVI-syn 3.0 | [52] |
| Escherichia coli | [53] | |
| Bacillus Subtilis | [54] | |
| Pseudomonas putida | [55] | |
| Reconstruire ce qui est connu | Bactériophage phiX174 | [56] |
| Influenza H1N1 grippe de 1918 | [18] | |
| Mycoplasma genitalium | [57] | |
| Réécrire en simplifiant | Bactériophage T7 | [17] |
| Génome synthétique de la levure (Sc2.0) | [58, 59] | |
| Mycoplasma mycoides JCVI-syn 3.0 | [36] | |
| Escherichia coli | [60] | |
| Réécrire le programme génétique ou le métabolisme en innovant | HCV | [39] |
| Flux de carbone optimisé | [61] | |
| Acide artémisique | [62] | |
| Biobriques | [63] | |
| Construire des gènes ancestraux | Anhydrase carbonique | [64] |
| Diterpène cyclase | [65] | |
| Protéines thermostables | [66] | |
| Polyphosphate kinase | [67] | |
| Changer le squelette du support de l'hérédité | l-threofuranosyl nucleic acid (TNA) | [68] |
| Acides nucléiques peptidiques (PNA) | [69] | |
| Glycol acides nucléiques (GNA) | [70] | |
| Xéno acides nucléiques (XNAs) | [71] | |
| Changer les bases des acides nucléiques | 2-aminoadénine (Z-genomes) | [72] |
| N1-méthyl-pseudouridine | [73] | |
| 5-chlorouracile, 7-désazaadénine, etc. | [74] | |
| Paires de bases hydrophobes artificielles (UBPs) | [75] | |
| Créer des applications nouvelles | Biologie synthétique fonctionnelle | [76] |
| Détecteur d'explosifs | [77] | |
| International Genetically Engineered Machine (iGEM) | [78] | |
| Changer le code génétique | Traduction avec des acides aminés non canoniques | [79] |
| Réécriture du génome d'Escherichia coli | [80] | |
| Changer les acides aminés in vivo | Norleucine, sélénométhionine | [81] |
| 4-fluorotryptophane | [82] | |
| ARN synthétiques pseudomessagers | ARN non immunogène pour la vaccination | [83] |
| ARN synthétique pour l'expression des protéines à haut niveau | [84, 85] | |
| Synthétiser des polymères non peptidiques avec à séquence définie | Flexizymes | [86] |
2.6. Les ARN synthétiques non canoniques peuvent mimer les ARN messagers
L’épisode toujours en cours de la pandémie de COVID-19 a mis au jour une application remarquable des acides nucléiques de synthèse. Dans cette application, vacciner avec un ARN synthétique stimule l’immunité adaptative des mammifères, habituellement dirigée contre des antigènes directement administrés, par une action qui se développe en deux temps. D’abord, c’est l’expression d’un ARN synthétique mimant un ARN messager dans le cytoplasme de cellules du système immunitaire qui dirige la synthèse d’un antigène, dont l’action, ensuite, déclenche la réponse immune.
Pourquoi n’est-ce pas possible avec des ARN naturels ? Pour se défendre rapidement des infections, spécialement virales, la réponse immune a évolué via l’émergence d’une immunité innée capable de reconnaître un certain nombre de signatures universellement présentes chez les agents pathogènes, comme la présence d’un génome. C’est ainsi que se sont imposés des récepteurs spécifiques reconnaissant les acides nucléiques libres, spécialement l’ARN sous sa forme faite des quatre ribonucléotides canoniques. Dès qu’un ARN naturel se lie à l’un de ces récepteurs, comme les récepteurs cytoplasmiques RIG-I-like ou l’un des récepteurs Toll-like impliqués dans cette reconnaissance [87], les gènes de cytokines qui assurent une réponse antivirale voient leur synthèse déclenchée [88]. On a donc observé très tôt que ce type d’ARN est reconnu et éliminé par le système de défense naturelle, mais que ce n’est pas le cas des ARN cellulaires.
L’un des mécanismes associés identifiés récemment permet à la cellule de discriminer entre l’ARN cellulaire naturel et l’ARN viral [89] en dissipant de l’énergie via une activité ATPase dont on comprend progressivement mieux les facteurs déclenchants [90]. On a par ailleurs remarqué que les extrémités des acides nucléiques de la cellule sont modifiées de façon spécifique (le plus souvent une coiffe à l’extrémité 5′ et un poly A à l’extrémité 3′ pour les ARN messagers) et que ces modifications sont différentes chez les virus. Tout cela permet que l’ARN génomique des virus soit spécifiquement reconnu par le système immunitaire, mais on n’en sait pas encore assez pour pouvoir synthétiser des ARN formés de nucléotides canoniques pouvant être utilisés comme vaccins. Une autre raison qui a contribué à les écarter des approches médicales est que ces ARN sont des molécules particulièrement fragiles, sensibles à l’activité des ribonucléases, universellement présentes. Enfin, sous l’action d’enzymes comme les transcriptases inverses, ils peuvent produire des copies ADN susceptibles de s’intégrer au génome de la cellule hôte comme dans le cas du VIH, ce qu’il faut bien sûr éviter. Pour ces raisons, bien qu’on ait depuis très longtemps cherché à utiliser de l’ARN naturel pour produire des ARN interférents ou encore de l’ARN messager destiné à produire des protéines vaccinantes, cette approche n’a pas eu le succès espéré.
Faute d’une connaissance suffisante pour leur utilisation courante, la recherche s’est tournée vers la synthèse d’acides nucléiques comprenant des nucléosides modifiés [91], spécialement la pseudouridine (Ψ) ou la N1-méthyl-pseudouridine (N1MeΨ) qui altèrent peu la structure générale de la molécule [92].
Ces analogues naturels de nucléosides canoniques sont connus depuis longtemps dans certains ARN stables, ARN de transfert et ARN ribosomiques [93, 94]. Lorsqu’ils remplacent l’uridine dans les ARN messagers, l’expérience montre qu’ils sont traduits, ouvrant un champ nouveau pour l’usage d’ARN synthétique en tant que matrice permettant la synthèse de protéines vaccinantes [25]. Comme ils échappent à l'immunité innée [73], ce sont d’excellents candidats pour leur utilisation via leur expression au sein de cellules immunocompétentes [95]. De fait, cette application de la biologie synthétique a eu un retentissement considérable quand il est apparu qu’un ARN synthétique contenant ces nucléosides modifiés donnait un vaccin très efficace contre le coronavirus responsable de la pandémie COVID-19 [96] (Figure 2).
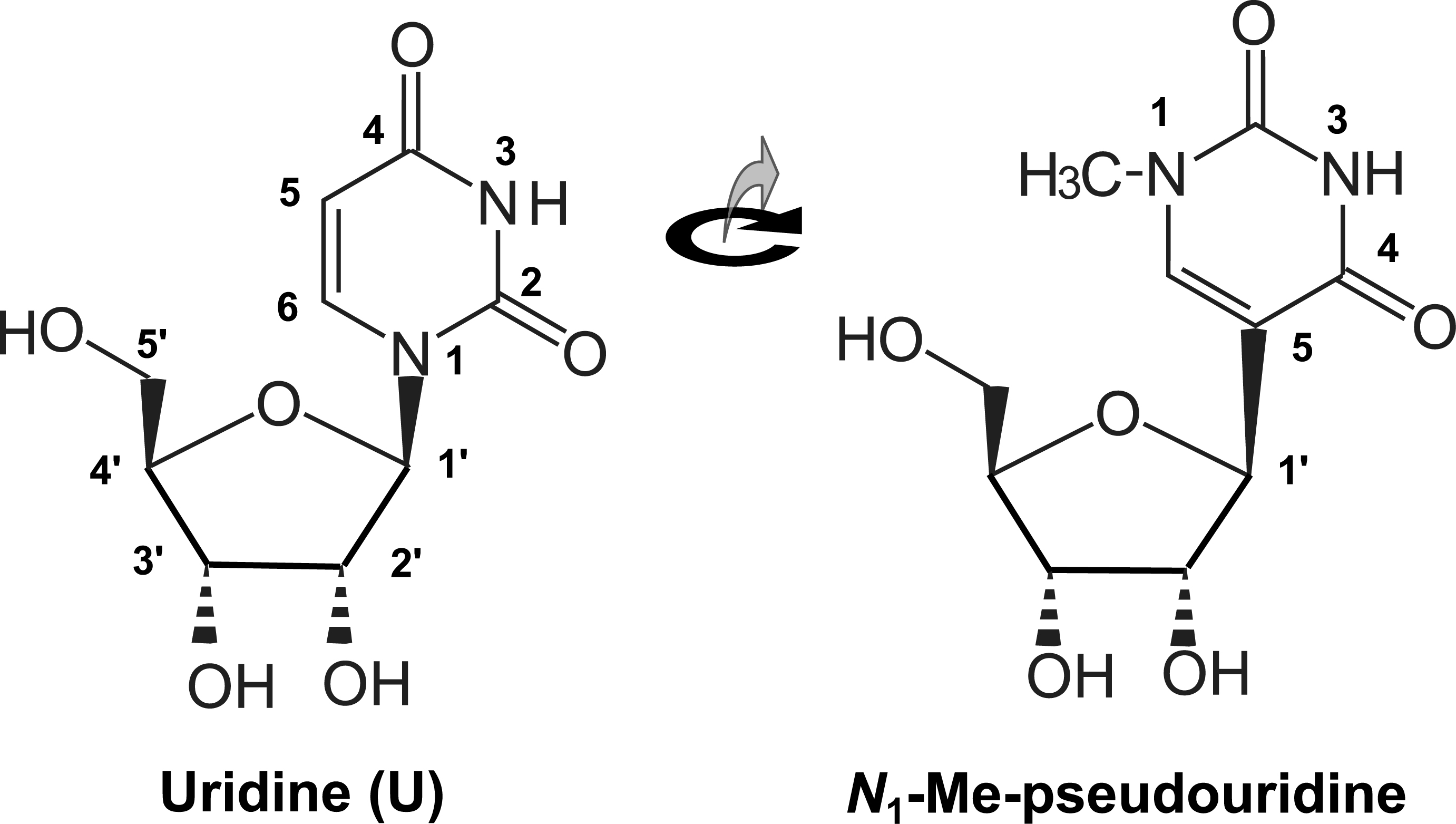
Pseudouridine et méthylpseudouridine ont une forme très voisine. Cependant, la liaison canonique entre l’azote 1 de la base et le carbone 1′ du ribose est plus courte que la liaison entre ce même carbone et ce qui est alors le carbone 5 de la base. Cela affecte la rapidité et surtout la fidélité du décodage au cours de la traduction de l’ARN synthétique.
Sachant qu’on ne pouvait pas connaître le détail des événements moléculaires au cours de l’expression d’un ARN synthétique chez l’animal, la généralisation de cette vaccination peut être vue comme une expérience en vraie grandeur sur des centaines de millions de personnes. Nous en savons donc aujourd’hui beaucoup plus. Le mouvement d’un ARN dans le ribosome au cours de sa traduction est sensible à la séquence de nucléotides traduite par groupe de trois (codons) qui se succèdent sans se recouvrir, établissant un cadre de lecture bien déterminé. En fonction du contexte, la molécule d’ARN peut glisser et moduler l’ajustement de ce cadre de lecture en avant ou en arrière. Cette propriété est utilisée par les virus pour diriger la synthèse de plusieurs protéines codées par la même séquence de nucléotides, mais dans un cadre de lecture différent. C’est ainsi que le SARS-CoV-2 produit deux protéines différentes essentielles pour sa réplication, Orf1a (Nsp11) et Orf1b (Nsp12). La deuxième résulte du glissement du génome viral d’un nucléotide en arrière dans la suite de nucléotides 5′– UUUAAAC-3′, ce qui revient, pour la deuxième protéine, à lire le dernier nucléotide, C, deux fois (décalage -1) [97]. Il s’agit donc là d’un « changement de cadre de lecture programmé » [98], processus souvent rencontré, mais très régulé, dans l’expression des gènes.
Cependant, lorsqu’ils se substituent aux ARN messagers, les ARN de synthèse modifient la fidélité et la vitesse de leur décodage par le ribosome. L’expérience a établi récemment que cela affecte le cadre de lecture de leur traduction en fonction de la séquence où se trouvent les nucléotides non canoniques. On commence à en connaître les conséquences. L’incorporation de N1MeΨ dans l’ARNm entraîne un glissement du cadre de lecture ribosomique d’un nucléotide vers l’avant en certains endroits de la séquence. Cela a été observé in vitro, mais aussi in vivo après la vaccination, comme on le constate chez la souris et chez l’homme avec la présence d’anticorps contre les produits traduits à partir d’une séquence décalée de +1 lors de la traduction de l’ARN synthétique codant la spicule virale vaccinante [65]. Il s’agit donc d’une « vaccination » accidentelle contre un antigène involontairement présent. Cela aurait pu avoir un effet adjuvant, donc positif [99], et n’a eu aucun effet négatif autant qu’on puisse en juger. Il faut noter d’ailleurs qu’un processus semblable, mais en des endroits différents du génome, se développe au cours de la traduction normale du génome viral. Pour contrôler ce processus dont on ignore les conséquences, il semble utile de remplacer les codons contenant certains résidus N1MeΨ responsables de ces glissements par des synonymes, de façon à réduire la production de produits codés dans la phase décalée [101]. D’un autre côté, cette observation conduit à imaginer que ce phénomène puisse être utilisé pour produire des variants toxiques fondés sur ce principe. Cela doit être pris en compte dans la réglementation future autorisant ces familles de vaccins.
2.7. Au-delà de la synthèse de protéines non canoniques, synthèse de polymères à motifs programmables
Réaliser la synthèse de polymères de séquence programmable à partir de maillons élémentaires variés est d’un intérêt industriel considérable. Au-delà de l’introduction in vivo d’analogues des acides aminés canoniques dans une construction synthétique reproduisant la synthèse des protéines (Tableau 1), c’est in vitro que cette approche est la plus prometteuse, spécialement avec des motifs de base chimiquement éloignés des acides aminés. Cela est réalisé par l’invention remarquable des « flexizymes » dans le laboratoire de Hiroaki Suga à Tokyo pour la synthèse de polymères non répétitifs [86], révolution déjà proposée par Danielli il y a un demi-siècle dans l'article cité plus haut :
La fabrication de polymères de séquence définie présente un énorme potentiel pour l’industrie biologique. Il y a peu d’espoir que l’industrie actuelle des polymères puisse développer les techniques existantes pour produire une large gamme de polymères de séquence définie. Or les cellules produisent une grande variété de polypeptides et des polynucléotides de séquence définie depuis deux milliards d’années. Même limité, l’accès à une telle polyvalence au niveau industriel élargirait notre horizon au-delà de ce que nous pouvons concevoir aujourd’hui [15].
Les flexizymes sont des ribozymes synthétiques destinés à charger une grande variété d’acides aminés non naturels ou d’autres précurseurs de polymères sur les ARN de transfert. Cela permet de reprogrammer le code génétique in vitro en réassignant les codons correspondant aux acides aminés naturels à des résidus non naturels, et ainsi de réaliser la synthèse de polypeptides non naturels ou même de polymères éloignés des polypeptides dont la séquence peut être apériodique. Ces polymères dont la séquence est définie par l’expérimentateur sont d’intérêt académique ou industriel [102]. Il est bien sûr possible d’utiliser ces techniques pour l’expression de programmes génétiques synthétiques permettant, par exemple, de produire des toxines ou des précurseurs chimiques de composés dangereux difficiles à obtenir autrement. Il s’agit donc bien de technologies dont le « double usage » est possible en fonction du contexte scientifique ou industriel. La mise en œuvre de ces techniques relève d’une « analyse de risque » préalable qui devrait être réalisée au cas par cas.
3. Un futur à explorer, le châssis, avec au cœur de la biologie synthétique une catégorie négligée de la physique, l’information
Pour terminer cette brève analyse de l’état le plus courant de la biologie de synthèse et de ses applications, il faut prendre en compte la machine, souvent oubliée. Ce « châssis » définit un intérieur et un extérieur, produit une copie semblable à lui-même, et permet l’expression d’un programme (un « livre de recettes »), qui dirige la synthèse d’une réplique (une copie exacte) de lui-même. Au cours de ce processus, le métabolisme transfère l’information de façon récursive à partir du programme et gère les flux de matière et d’énergie. Mais il faut souligner ici un caractère original souvent méconnu, comme on le voit dans l’usage indifférencié du terme : la reproduction n’est pas la réplication. Cela est démontré dans l’expérience où le génome d’une espèce est transplanté dans une autre espèce [103]. Le programme se réplique (produit une copie exacte), alors que le châssis se reproduit (produit une copie approximative) [104], ce qui implique un ensemble de propriétés qui s’écartent fortement de la logique de standardisation et explique le désintérêt apparent des chercheurs pour la création de châssis synthétiques.
3.1 . Gestion de l'information dans la cellule
Par exemple, le châssis comprend inévitablement des entités âgées, sensibles au temps, dont l'âge est contraint par leurs propriétés physico-chimiques dans l'environnement de la cellule. La gestion de ce phénomène suppose, comme d’ailleurs pour les machines humaines, l’existence d’une maintenance. Mais ce processus, rarement pris en compte dans les réflexions de la biologie synthétique, présente la caractéristique unique d’être nécessairement endogène, à la différence de la maintenance industrielle qui nécessite dans la vaste majorité des cas une action extérieure. Dans le cas des protéines, le vieillissement dépend de façon intrinsèque de leur séquence et de leur repliement. Deux acides aminés jouent un rôle central dans cet inévitable vieillissement. L’aspartate et l’asparagine se cyclisent spontanément en succinimide (avec désamidation dans le cas de l’asparagine), puis forment un résidu analogue, un iso-asparate, qui déforme localement le squelette polypeptidique [105]. Ce phénomène universel, connu depuis longtemps, mais curieusement occulté, impose que toutes les cellules contiennent simultanément un ensemble de protéines jeunes et de protéines vieillies, ces dernières voyant leur fonction altérée ou perdue [106]. Il doit donc exister en leur sein un ensemble d’agents capables de gérer cette information et de faire en sorte que, lorsque la cellule produit une descendance, ce soit une descendance seulement ou de préférence jeune [107]. Ces agents discriminateurs, qui font partie intégrante du programme génétique de la cellule, identifient des classes d’objets, par exemple ce qui est jeune et ce qui est vieux, et les conduisent à un destin différent selon leur classe d’âge. D’une façon générale, la réflexion sur le châssis nécessaire à la synthèse totale d’une cellule qu’on peut reconnaître comme vivante n’a pas encore pris en considération l’importance de ces agents classificateurs qui sont pourtant présents même au sein des cellules ayant le plus petit génome [108].
On souligne ici que l’information est un concept nécessaire à la compréhension de ce qu’est la vie. Elle doit être prise en compte dans les expériences ou les applications qui visent à synthétiser des cellules vivantes, en particulier via l’identification de ces agents discriminateurs et de leur rôle. Malheureusement, l’usage du terme est particulièrement flou, et n’est pas bien formalisé, sauf dans un domaine restreint de la physique statistique. Pourtant, l’idée que l’information, plus qu’une façon de dire, est une authentique catégorie de la nature est très ancienne. Cela a été longtemps illustré par l’image du vaisseau de Thésée, rapporté dans Les Vies des hommes illustres par Plutarque :
Le navire à trente rames sur lequel Thésée s’était embarqué avec les jeunes gens offerts au Minotaure, et qui le ramena victorieux à Athènes, fut conservé par les Athéniens jusqu’au temps de Démétrius de Phalère. Ils en changèrent les planches au fur et à mesure qu’elles vieillissaient, les remplaçant par des pièces neuves, plus solides. Aussi les philosophes, dans leurs discussions sur la nature du changement des choses choisirent-ils ce navire comme exemple, les uns soutenant qu’il reste le même, les autres qu’il est différent du navire de Thésée.
C’est que l’information en tant qu’objet fondamental de la physique n’a jusqu’à présent été vraiment discutée que pour rendre compte de la qualité de la transmission de messages constitués de séquences de symboles sans avoir à tenir compte de la signification du message. Shannon et Brillouin ont ainsi proposé une théorie de la communication qui est de façon abusive utilisée comme « théorie de l’information » [109]. Or l’utilisation du concept en biologie montre bien qu’il y a beaucoup plus que le modèle de Shannon dès qu’on se soucie non pas de l’intégrité du message, mais de sa signification : une molécule synthétique d’ADN ne conduit pas du tout au même résultat selon le contexte où elle est placée. C’est une observation cruciale dès qu’on imagine synthétiser la vie. Or, le rêve de l’ingénieur en biologie synthétique est que les cellules artificielles ne soient pas innovantes (qui volerait dans un avion qui innoverait au cours du vol ?) Il est bien sûr possible d’exclure les gènes qui permettent l’accumulation d’information. La conséquence est que, comme les usines, l’usine cellulaire vieillira et devra être systématiquement reconstruite. Cela pose problème pour l’utilisation à grande échelle de cellules synthétiques et l’incertitude sur leur devenir fait qu’il semble difficile de faire un usage malveillant de ce caractère original de toute forme de vie.
3.2. Biologie synthétique et contagion de l'information
Pour terminer avec l’importance de la prendre en compte, il existe encore un rôle tout à fait différent de l’information lorsqu’on explore ce que peuvent être de mauvais usages de la biologie synthétique. C’est que l’information elle-même est contagieuse, et alors facilement sujette à un double usage comme on peut facilement l’illustrer dans différents domaines. L’OMS tente d’éradiquer les maladies contagieuses spécifiques à l’homme, surtout virales. Cette éradication repose sur la vaccination et suppose que la majorité de la population mondiale se vaccine. Hélas, la propagation de la peur, utile pour limiter les contacts et la contagion de la maladie [110], va à l’encontre de cet objectif, en particulier par les réseaux sociaux lorsqu’elle propage la désinformation [111]. Un autre aspect négatif de l’information vient du caractère symbolique de la simple description explicite du génome d’un organisme pathogène, sous la forme d’un texte écrit dans un alphabet à quatre lettres. La connaissance du génome du virus de la variole a rendu son éradication inopérante. La séquence du virus elle-même n’étant pas pathogène, elle peut en effet se propager sans entraves par l’internet, par exemple [112], alors qu’on sait faire la synthèse complète des poxvirus, comme cela a été montré pour la variole du cheval [113].
Il existe d’autres formes plus raffinées d’aller et retour entre le texte d’une séquence génétique et ses applications concrètes malveillantes. Les ordinateurs peuvent être infectés par des logiciels hostiles traduits en séquences d’ADN, et les menaces biologiques peuvent être produites à partir de données accessibles au public, ce qui rend les bases de données d’organismes pathogènes particulièrement dangereuses [29, 114]. Ces bases de données peuvent ainsi contenir des séquences fictives qui cachent des programmes malveillants qui se mettent en marche à l’ouverture du fichier correspondant. Pour le comprendre, il est utile de se souvenir que les attaques informatiques reposent sur les défauts inévitables de l’implémentation d’un programme dans une machine [115]. En biologie, les failles sont particulièrement nombreuses dès qu’il s’agit d’échanger du matériel entre laboratoires. De même qu’ouvrir un fichier annexé à un courriel peut déclencher un processus malveillant, l’envoi de matériel fait d’acides nucléiques peut tromper. Les bonnes pratiques de laboratoire qui imposent la vérification de la séquence du matériel reçu sont cruciales en cas d’actions hostiles : en effet, un objet en apparence innocent peut en réalité être nuisible, ou tout simplement destiné, en étant autre chose que ce qui est attendu, à retarder ou à faire échouer des expériences coûteuses [116]. Mais le processus de vérification lui-même peut être cause de malveillance, si l’on envoie à séquencer un segment d’ADN dont la séquence encode des programmes hostiles destinés à contaminer l’équipement informatique lors du séquençage [117]. Comme les approches de biologie synthétique, industrielles en particulier, combinent tous les niveaux expérimentaux, de la conception in silico, à l’évolution in vivo et à la production in vitro, le tout piloté par ordinateur, les points d’entrée pour le piratage informatique sont très nombreux (Figure 3).
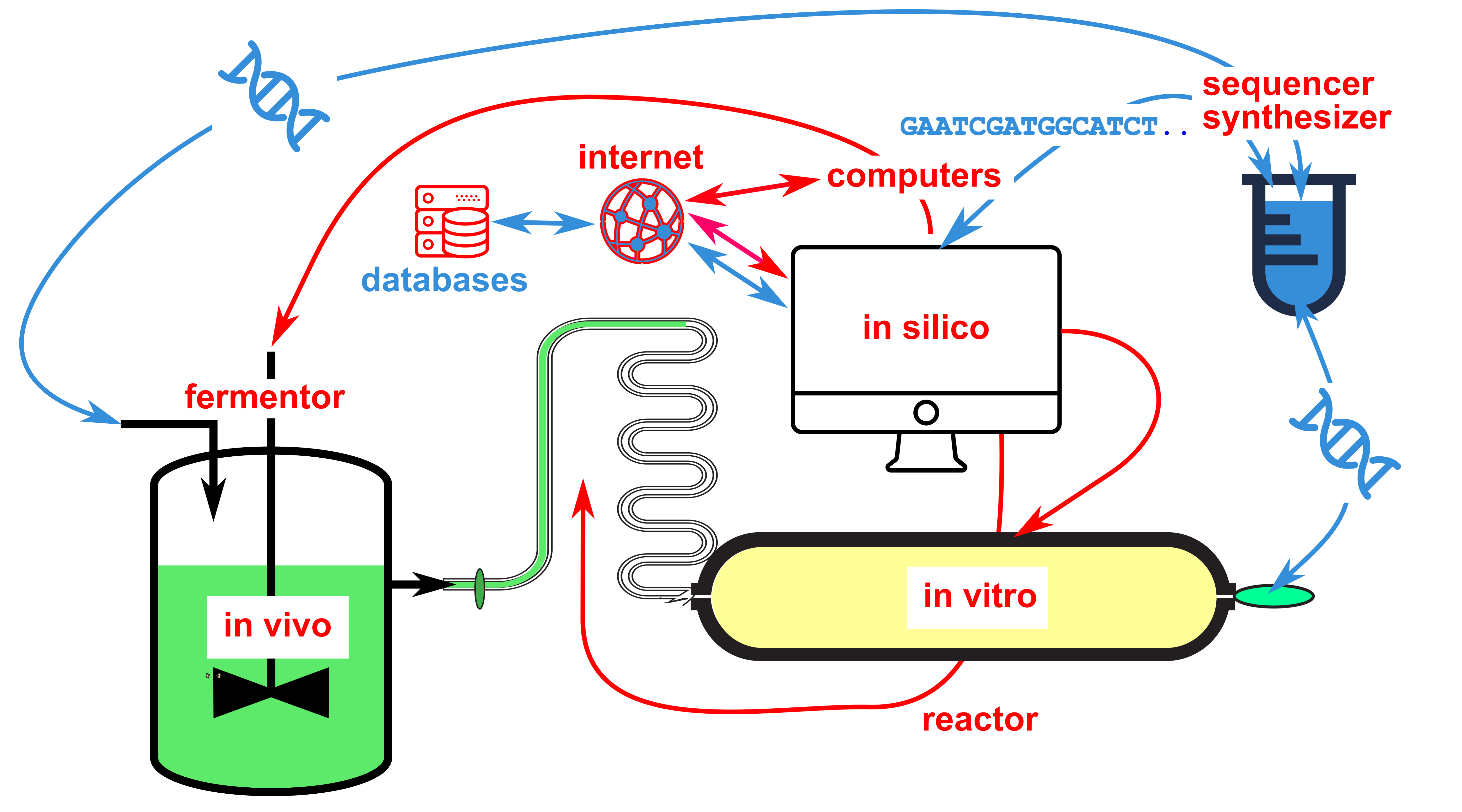
Aller et retour entre la conception et la synthèse d’acides nucléiques synthétiques et le pilotage d’un processus industriel intégré.
Dans un procédé industriel intégré, la biologie synthétique combine la synthèse des gènes choisis, produits à la suite d’une exploration in silico (à l’ordinateur), avec leur expression in vivo au sein de châssis qui sont multipliés en fermenteur, ou in vitro où ils servent à développer de nouvelles voies métaboliques ou à synthétiser des polymères nouveaux difficiles à obtenir avec un rendement suffisant. L’introduction de séquences corrompues peut permettre de dissimuler des programmes visant à asservir non seulement l’ordinateur de pilotage, mais aussi à infecter le réseau de télécommunications liant cet ordinateur à l’Internet, par exemple. Flèches bleues : flux de l’information associée aux séquences nucléiques, soit réelles soit sous forme de textes. Flèches rouges : flux de commandes. Masquer
Aller et retour entre la conception et la synthèse d’acides nucléiques synthétiques et le pilotage d’un processus industriel intégré.
Dans un procédé industriel intégré, la biologie synthétique combine la synthèse des gènes choisis, produits à la suite d’une exploration in silico ... Lire la suite
4. En guise de conclusion, une nécessaire prise de conscience
Lorsqu’émerge un nouveau domaine scientifique et technique, on ne peut anticiper son impact politique, économique, ou encore sociétal ou comportemental. Portée par la curiosité et l’activité inventive, la recherche scientifique est perçue comme une quête positive qui vise à faire progresser la connaissance et qui peut conduire à des applications au bénéfice de l’humanité et de son environnement. Hélas, l’histoire nous apprend que cet espoir a souvent été détourné par la quête du pouvoir. Pour éviter cet écueil et définir un objectif responsable pour la recherche scientifique, il faut tenter d’en écarter les mésusages et les prises de risque inacceptables. La raison d’être de la biologie synthétique est de chercher à comprendre ce qu’est la vie. En chemin, cela conduit à faire émerger des techniques nouvelles et des applications importantes pour des secteurs économiques légitimes comme la santé et la qualité de vie, qu’elle soit humaine, animale ou végétale. L’analyse du spectre des possibles, au travers des exemples cités ici, montre que le futur de ce domaine reste très ouvert et qu’il existe bien d’autres usages à connaître ou à inventer. La facilité d’accès croissante aux techniques de la biologie synthétique fait qu’il est de plus en plus difficile de contrôler efficacement les flux de matière et les moyens qui y sont associés. On ne doit pas ignorer pourtant qu’ils peuvent être détournés à des fins malveillantes ou que des accidents, en particulier liés à la nature évolutive du vivant, sont toujours possibles. Ainsi, du fait de son caractère très innovant, la biologie synthétique constitue un domaine « à double usage et à risque ». Or, et cela s’adresse à toute forme de « haute technologie », il faut que les personnes qui partagent l’innovation et l’acquisition de connaissances nouvelles prennent conscience des conséquences de leur divulgation. Cela implique notamment, pour le monde scientifique, de développer une culture de responsabilité qui tienne compte de l’acceptabilité des risques en recherche et innovation, et l’éthique que cela sous-entend. Trop souvent le danger ne vient pas d’acteurs hostiles, mais de personnes réticentes dès qu’il s’agit de mettre des limites à leur activité, qu’elles présentent comme d’intérêt général. C’est que la recherche de la notoriété est favorisée par le système actuel d’évaluation de l’activité d’un chercheur au travers de ses publications, fondé sur un modèle économique qui s’apparente à la publicité, participant à ce comportement condamnable qui se traduira, faute d’une prise de conscience collective, par la multiplication d’accidents évitables, d’épidémies et finalement par le développement d’un obscurantisme qui refuse l’exploration scientifique du monde. Le monde scientifique en a heureusement pris conscience avec la déclaration de San Francisco sur l’évaluation de la recherche (DORA https://sfdora.org/read/read-the-declaration-french/). Mais il reste essentiel d’introduire très tôt dans la formation des chercheurs et des ingénieurs la notion de « recherche à double usage et à risque » et savoir la détecter et en prévenir le développement, en particulier à partir d’une réflexion approfondie sur les modes d’évaluation de la recherche et des personnes qui la mettent en œuvre.
Déclaration d'intérêts
L’auteur ne travaille pas, ne conseille pas, ne possède pas de parts, ne reçoit pas de fonds d’une organisation qui pourrait tirer profit de cet article, et n’a déclaré dans ce contexte aucune autre affiliation que son affiliation académique.
Remerciements
Ces travaux, qui n'ont reçu aucun financement, ont bénéficié des discussions suscitées par le programme I2Cell de la Fondation Fourmentin-Guilbert, reconnu d'utilité publique.




 CC-BY 4.0
CC-BY 4.0