1 Introduction
La production d’huître creuse, Crassostrea gigas, est aujourd’hui la principale production française de coquillages. Le naissain mis en élevage dans les différents centres de production est obtenu par captage naturel ou par élevage en écloserie (actuellement de 10 à 15% du naissain mis en élevage)
En période estivale, à la faveur de températures élevées, de forts taux de mortalité de naissain d’huître creuse, C. gigas, (de 60 à 100%) sont rapportés régulièrement depuis 1991, en milieu naturel et, quelquefois, dans certaines écloseries nurseries. Au cours des dix dernières années, ces épisodes récurrents sont apparus comme un sujet préoccupant pour l’ensemble de la profession ostréicole 〚1〛.
Les études menées dans le but d’identifier les causes de ces épisodes de mortalité ont révélé l’existence d’un agent viral de type herpès chez les larves et le naissain d’huître creuse, ainsi que chez d’autres espèces de mollusques 〚2, 3〛. Si la pathogénicité de ce virus a pu être démontrée pour les stades larvaires 〚4〛, la reproduction expérimentale de la maladie sur naissain fait encore l’objet d’études. Par ailleurs, de nombreuses études ont établi le caractère pathogène de certaines souches de bactéries pour diverses espèces de bivalves, dont l’huître creuse 〚5–11〛.
Dans ce travail, des souches bactériennes ont été isolées lors d’un épisode de mortalité de naissain de C. gigas, survenu en site d’élevage. Ces souches ont été caractérisées phénotypiquement dans un premier temps. Parmi ces souches, des bactéries pathogènes du genre Vibrio ont été sélectionnées par reproduction expérimentale de la maladie sur naissain de C. gigas. L’identification moléculaire de la souche reconnue pathogène a été entreprise par séquençage du gène codant pour l’ARN 16S.
2 Matériel et méthodes
2.1 Prélèvement
Au cours de l’été 1997, un épisode de mortalité a été rapporté sur du naissain de captage naturel d’Arcachon placé en Charente Maritime pour prégrossissement sur filières en mer. La recherche de souches pathogènes a été effectuée sur un échantillon de naissain prélevé en septembre 1997, correspondant au taux maximal de mortalité (60%).
2.2 Bactériologie
Les souches bactériennes ont été obtenues à partir de broyats de chair de naissain dilués en eau de mer stérile et étalés sur deux types de milieux : milieu de Zobell, permettant d’isoler la flore hétérotrophe totale, et milieu TCBS, sélectif pour la flore Vibrionaceae. La caractérisation des souches isolées, en termes de genre et d’espèce, a été réalisée à partir de 35 tests biochimiques : Gram, mobilité, présence de flagelle, luminescence, oxydase, gélatinase, uréase, MEVAG, V.F., Citrate de Simmons, Kligler–Hajna, acidification de l’arabinose, du mannitol et du saccharose, ADH, LDC, ODC, indole, Voges–Proskauer, nitrate, ONPG, croissances à 0, 6, 8, 10% de NaCl, croissances à 4, 20, 35 et 40 °C, croissance sur α-kétoglutarate, D-glucosamine, L-arabinose, 〚12〛. Les tests nécessitant une croissance, excepté ceux à différentes salinités, ont été adaptés à l’étude de souches marines par addition de 2% de NaCl ou d’eau de mer artificielle dans les milieux et réactifs. Les résultats de ces tests sont utilisés dans des arbres dichotomiques d’identification, construits à partir de données bibliographiques 〚13, 14〛.
2.3 Pathologie expérimentale
Pour chaque souche testée, cinquante animaux de 5 mm à 1 cm ont été infectés par immersion dans une suspension bactérienne d’environ 107 bactéries ml–1, pendant 48 h à la température de 20 °C.
Dans la première série d’expériences, le naissain inoculé avec chacune des souches isolées a été rincé, puis placé en bac de 20 l pendant deux mois en structure d’élevage. Par la suite certains lots ont été maintenus dans un aquarium contenant 3 l d’eau de mer stérile et équipé d’un système d’oxygénation par bullage. Du phytoplancton (Chaetoceros calcitrans) a été distribué quotidiennement. La température a été maintenue à 20 °C pendant l’expérience.
Dans la seconde série d’expériences, le naissain a été inoculé, comme précédemment, avec certaines des souches sélectionnées. Le naissain a été rincé puis maintenu en aquarium contenant 3 l, comme précédemment. Le naissain a été observé régulièrement et a été considéré comme mort lorsqu’il y avait absence de fermeture des valves et de rétraction du manteau après stimulation par piqûre. Les animaux morts ont été prélevés afin de rechercher la souche bactérienne inoculée dans les tissus et de vérifier ainsi le postulat de Koch.
Plusieurs lots de naissain non inoculé fournissaient un contrôle, alors que deux contrôles positifs étaient utilisés : les souches de Vibrio tubiashi CRL10 et CRL22, pathogènes pour les larves et le naissain de mollusque 〚15–17〛.
2.4 Caractérisation moléculaire
L’ADN bactérien a été extrait par la technique classique de lyse en présence de protéinase K, extraction au phénol/chloroforme et précipitation à l’éthanol 〚18〛. Le gène codant pour l’ARN 16S a été amplifié par PCR à l’aide des amorces universelles dont les séquences suivent : 5’- CTCAGATTCAACGCTGGCGG-3’ et 5’-GGCGGTGTGTACAAGGCCCG-3’. La réaction d’amplification a été réalisée suivant les conditions standards définies pour la Taq DNA polymérase Silverstar (Eurogentec). Le produit de PCR a été cloné dans le vecteur pCRII (TA cloning kit, Invitrogen). Les mini-préparations d’ADN plasmidique obtenues par lyse alcaline 〚18〛 ont alors été utilisées pour le séquençage de l’insert (Appligène). La séquence obtenue a été alignée avec 32 autres séquences de Vibrionaceae (Tableau 1), tandis qu’une analyse phylogénétique a été réalisée en utilisant trois méthodes : neighbour-joining, maximum likelihood et maximum parsimony.
Numéros d’accession du gène codant pour l’ARN 16S des souches utilisées pour l’analyse phylogénétique.
| Souches | Numéro d’accession |
| Souche TNEMF6 | |
| V. splendidus (II) | AB038030 |
| V. splendidus (I) | A33125 |
| V. pelagius | AJ293802 |
| V. campellii | X74692 |
| V. parahaemolyticus | X74720 |
| V. carchariae | X7493 |
| V. proteolyticus | X74723 |
| V. halioticoli | AB000393 |
| V. natriegens | X74714 |
| V. fluvialis | X74703 |
| V. aestuarianus | AF172840 |
| V. lentus | AJ278881 |
| V. mediterranei | X74710 |
| V. orientalis | X74719 |
| V. vulnificus | X76334 |
| V. ordalii | X74727 |
| V. tapetis | Y08430 |
| V. scophthalmi | U46579 |
| V. pectenicida | Y13830 |
| V. shiloi | AF007115 |
| V. gazogenes | X74705 |
| V fischeri | X74702 |
| V. anguillarum | X71821 |
| V. salmonicida | X70643 |
| V. ordalii | X74718 |
| V. tubiashi | X74725 |
| V mytili | X99761 |
| V. diabolicus | X99762 |
| Photobacterium phosphoreum | X74687 |
| P. angustum | X74685 |
| P. leiognathi | X74686 |
| P. damsela | X74700 |
3 Résultats
Au cours de l’épisode de mortalité du naissain en mer, survenu en septembre 1997, des signes caractéristiques sont apparus sur les animaux moribonds échantillonnés: perception d’une forte odeur, faiblesse du muscle adducteur entraînant un défaut de fermeture des valves et présence d’une vase jaune-verdâtre au fond de la valve inférieure des animaux moribonds. Une recherche du virus de type herpès a été effectuée par PCR sur ces prélèvements et s’est révélée négative 〚19〛.
Les résultats de la caractérisation phénotypique des souches bactériennes isolées sont résumés dans le Tableau 2. Trois genres bactériens principaux ont été isolés à partir du naissain moribond : (1) souches apparentées au groupe Cytophaga/Flavobacterium, (2) souches apparentées aux Agrobacterium, (3) souches appartenant aux Vibrio. La souche MNEMF5, apparentée aux Cytophaga/ Flavobacterium est majoritaire sur milieu gélosé de Zobell, avec 105 bactéries par gramme de chair de naissain.
Identification phénotypique de souches bactériennes isolées sur boîte Zobell (MNEMF) et TCBS (TNEMF). Le dénombrement correspond au nombre de bactéries par gramme de chair de naissain broyé ; l’étude bactériologique définit la souche par genre, voire par espèce.
| Souche | Dénombrement | Genre | Espèce |
| MNEMF1 | 700 | Vibrio | anguillarum |
| MNEMF2 | 104 | Vibrio | splendidus II |
| MNEMF3 | 104 | Agrobacterium | sp. |
| MNEMF4 | 103 | Cytophaga | sp. |
| MNEMF5 | 105 | Cytophaga | sp. |
| MNEMF6 | 103 | Cytophaga | sp. |
| TNEMF1 | 180 | Vibrio | fluvialis |
| TNEMF2 | 103 | Vibrio | mediterranei |
| TNEMF3 | 360 | Vibrio | pelagius I |
| TNEMF4 | 103 | Vibrio | nigripulchritudo |
| TNEMF5 | 720 | Vibrio | mediterranei |
| TNEMF6 | 130 | Vibrio | splendidus II |
| TNEMF7 | 180 | Agrobacterium | sp. |
| TNEMF8 | 540 | Vibrio | splendidus II |
| TNEMF9 | 100 | Vibrio | splendidus II |
Ces différentes souches ont été testées en pathologie expérimentale sur naissain de C. gigas. Les six témoins négatifs font apparaître des taux de mortalité cumulée réduits (de 0 à 12%) à l’issue de l’expérience. Les deux souches de Vibrio tubiashi, CRL10 et CRL22, induisent respectivement des mortalités cumulées de 80 et 10% au bout de deux mois. Deux souches de Vibrio splendidus biovar II, MNEF2 et TNEMF6 entraînent une mortalité de 10 et 18%, alors que la souche TNEMF8 ne semble pas affecter les animaux. Enfin, la souche de Cytophaga MNEF5, majoritaire en dénombrement (Tableau 2) n’augmente pas le taux de mortalité cumulée.
Certains lots ont ensuite été maintenus en élevage pendant un mois supplémentaire en aquarium de 3 l. Les souches de Vibrio splendidus MNEF2 et TNEMF6 entraînent une mortalité cumulée respectivement de 12 et 80%, témoignant d’une pathogénicité différente, bien que ces deux souches appartiennent à la même espèce. Le contrôle CRL 22 induit une mortalité de 76%, alors que le naissain non inoculé subit une mortalité de 6%.
Les animaux morts au cours de l’expérience ont été analysés afin de quantifier les souches bactériennes présentes dans les tissus : les naissains inoculés avec la souche TNEMF6 présentent un dénombrement de 5 105 bactéries par gramme de chair ; cette souche apparaît comme majoritaire.
Dans une seconde série d’expériences d’infection expérimentale, le caractère pathogène de la souche TNEMF6 a été confirmé, avec des mortalités de 32 et 26% au bout de sept semaines, alors que les témoins donnent 12% pour CRL 10 et 2% pour CRL 22 (Tableau 3).
Seconde série de pathologie expérimentale. La souche Vibrio splendidus TNEMF6 (expérience dupliquée) et les souches contrôle Vibrio tubiashi CRL 10 et CRL 22 ont été inoculées par balnéation au naissain de Crassostrea gigas. La concentration de l’inoculum est déterminée par dénombrement sur boîte Zobell et donné en bactéries par ml. Après 48 h d’incubation et rinçage, le naissain est élevé en aquarium de 3 litres. Une surveillance régulière des animaux permet de définir le taux de mortalité cumulé au cours de 45 jours et sept semaines.
| Souches | Genre, espèce | Concentration | Mortalité à sept semaines |
| Néant | 6% | ||
| TNEMF6 | Vibrio splendidus II | 2,1 106 | 32% |
| 26% | |||
| CRL10 | Vibrio tubiashi | 7,8 105 | 12% |
| CRL22 | Vibrio tubiashi | 8,5 105 | 2% |
Le séquençage du gène codant pour l’ARN 16S de la souche TNEMF6 et l’alignement de cette séquence avec celle de 32 autres souches de Vibrio ont permis l’étude phylogénétique de cette souche (Fig. 1). Cette analyse moléculaire affilie sans ambiguïté cette souche à Vibrio splendidus II, confirmant ainsi la caractérisation phénotypique.
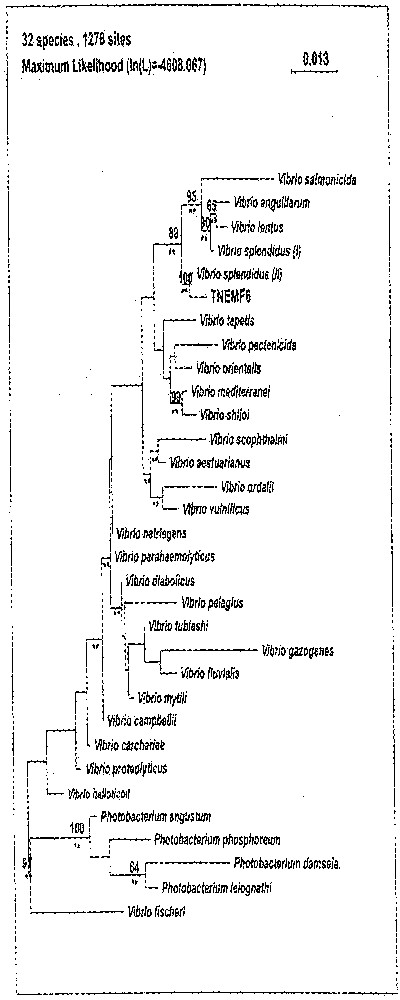
Position taxonomique de la souche TNEMF6 dans le genre Vibrio. La topologie de cet arbre non raciné a été obtenue par un algorithme d’analyse de maximum de vraisemblance (maximum likelihood). Les branches retrouvées par la méthode de parcimonie maximum sont notées par un double astérisque. Les valeurs supérieures à 50% marquent les branches obtenues par un algorithme d’analyse de matrice de distance (neighbour joining) et indiquent comment ces branches sont validées par l’analyse de réplication (analyse de bootstrap).
4 Discussion
Les épisodes récurrents de mortalité estivale de naissains d’huître inquiètent les professionnels, qui craignent qu’à l’instar de l’huître portugaise, C. angulata, C. gigas puisse disparaître à son tour, suite à une épizootie 〚20〛. Depuis 1995, notre laboratoire s’est attaché à décrire des souches bactériennes associées à des épisodes de mortalité. Dans ce travail, nous présentons l’isolement d’une souche bactérienne potentiellement pathogène pour le naissain d’huîtres creuses en France. Jusqu’à présent, la pathogénicité de souches bactériennes sur C. gigas a été démontrée, soit par infection d’animaux adultes, soit par infection de larves après immersion 〚5–11〛. Ici, pour la première fois, il a été possible d’induire expérimentalement des mortalités de naissain d’huîtres creuses C. gigas par balnéation.
Cette souche a été isolée à partir d’animaux de captage naturel et échantillonnés au cours d’un épisode de mortalité estivale (Tableau 4). D’après les études phénotypiques et génotypiques, cette souche (TNEMF6) appartient à l’espèce Vibrio splendidus biovar II. Les autres souches de Vibrio splendidus II isolées sont peu virulentes (exemple de MNEF2) ou non virulentes (exemple de TNEMF8). Les Vibrio splendidus sont très répandus dans le milieu marin et plusieurs études en font état 〚21–23〛. À l’intérieur de cette espèce ubiquiste, peu de souches sont virulentes. Par ailleurs, la classification dans une même espèce de toutes ces souches devrait être réexaminée à la lumière de la phylogénie moléculaire (Fig. 1) qui suggèrent l’existence de groupes distincts. Seules des hybridations ADN/ADN entre des souches représentatives de ces ensembles permettront de vérifier s’ils correspondent à une ou plusieurs espèces différentes.
Première série de pathologie expérimentale. Les différentes souches bactérienne isolées sur Zobell et TCBS ont été inoculées par balnéation au naissain de Crassostrea gigas. La concentration de l’inoculum est déterminée par dénombrement sur boîte Zobell et donné en bactéries par ml. Après 48 h d’incubation et rinçage, le naissain est élevé en bac de 20 l pendant deux mois. Une surveillance régulière des animaux permet d’obtenir le taux de mortalité cumulé au cours de cette période (taux de mortalité bac). Dans un second temps, certains lots sont sélectionnés et élevés en aquarium de 3 l. Le taux de mortalité donné correspond à trois semaines d’élevage.
| Souches | Genre, espèce | Concentration | Mortalité bac | Mortalité aquarium |
| Néant | 0% | 6% | ||
| Néant | 2% | |||
| Néant | 8% | |||
| Néant | 12% | |||
| Néant | 6% | |||
| Néant | 8% | |||
| MNEF1 | Vibrio anguillarum | 3 108 | 6% | |
| MNEF2 | Vibrio splendidus II | 106 | 10% | 12% |
| MNEF3 | Agrobacterium sp. | 4 107 | 6% | |
| MNEF5 | Cytophaga sp. | 108 | 2% | 5% |
| MNEF6 | Cytophaga sp. | 4 108 | 0% | |
| TNEMF1 | Vibrio fluvialis | 4 106 | 2% | |
| TNEMF2 | Vibrio mediterranei | 4 106 | 2% | |
| TNEMF3 | Vibrio pelagius I | 8 106 | 6% | |
| TNEMF5 | Vibrio mediterranei | 3 106 | 2% | |
| TNEMF6 | Vibrio splendidus II | 2 107 | 18% | 80% |
| TNEMF7 | Agrobacterium sp. | 3 107 | 2% | |
| TNEMF8 | Vibrio splendidus II | 4 107 | 4% | |
| CRL10 | Vibrio tubiashi | 4 106 | 80% | 76% |
| CRL22 | Vibrio tubiashi | 5 107 | 10% |
D’autres souches de Vibrio splendidus ont été associées à des mortalités de larves C. gigas 〚10〛, de coquilles Saint-Jacques Pecten maximus 〚24〛 ou de poissons 〚25〛. Pour ces différents modèles animaux, on observe une grande diversité de virulence entre souches appartenant à la même espèce. Actuellement le seul moyen d’établir la virulence d’une souche bactérienne est de la tester en pathologie expérimentale. Cette approche reste lourde et les résultats sont variables d’un essai à l’autre. Nous ne disposons pas de critères phénotypiques ou génotypiques, sur la base desquels il soit possible de détecter un Vibrio splendidus pathogène. Des études de typage moléculaire de différentes souches basées sur les profils de restriction des gènes ribosomiques (ribotypage) ou la RAPD n’ont pas permis de trouver un marqueur de la pathogénicité 〚Waechter, communication personnelle, p. 25〛. Nous désirons à présent développer des outils diagnostic spécifiques de facteurs de virulence. Notre démarche s’appuiera sur l’établissement d’une collection de souche de Vibrio splendidus, la soustraction génomique entre souche virulente et non virulente et clonage des gènes supports de virulence. En parallèle, des modèles in vitro seront développés pour tester la virulence de ces bactéries.
Un autre résultat intéressant de ce travail est la variabilité de virulence des souches pathogènes, en fonction des expériences d’épreuve. Dans notre étude, le naissain infecté par Vibrio tubiashi (CRL10) subit une mortalité de 80% en bac de 20 l dans la première expérience, alors que, dans la seconde expérience, il ne subit qu’une mortalité évaluée à 12%. Au cours de la première expérience, les naissains infectés par Vibrio tubiashi (CRL22) et Vibrio splendidus TNEMF6 présentent, après deux mois d’élevage en bac, des mortalités de l’ordre de 10%, puis, après un mois de culture en aquarium, une mortalité de 80%. En revanche, cet effet activateur du pouvoir pathogène par changement de la pratique d’élevage n’est pas observé pour la souche Vibrio splendidus MNEF2. Il semble donc que l’origine du naissain, le mode d’élevage ainsi que la souche de Vibrio soient des facteurs déterminants.
Ces résultats rejoignent ceux de l’étude des mortalités estivales sur le terrain, qui indiquent une étiologie multifactorielle de ce phénomène, effet concomitant de conditions environnementales, de paramètres physiologiques, génétiques et pathologiques. Afin de relier notre étude en bactériologie au phénomène des mortalités anormales, nous nous proposons, dans une étude ultérieure, de tester la virulence de nos souches en pathologie expérimentale avec des animaux de familles génétiquement différentes et en conditions environnementales différentes.
Remerciements
Les auteurs remercient la société Grainocéan pour son soutien technique et financier (bourse Cifre), ainsi que le Laboratoire communautaire de référence pour les maladies des mollusques. Nous remercions M. Tristan Renault et Melle Mélanie Gay d’avoir corrigé ce manuscrit.
Abridged version
1 Introduction
Today, the French shellfish production is mainly based on the Pacific cupped oysters, Crassostrea gigas. The spat used in oyster farms comes either from natural harnessing, or from hatcheries (nowadays between 10 and 15% of the total spat).
Since 1991, during the summertime, as the temperature increases, high C. gigas spat mortality rates (60 to 100%) occur in the field and also in some hatcheries. For ten years, oyster farmers have been very concerned by these outbreaks.
As these mortalities have been studied, a herpesvirus-like agent has been identified in cupped oysters larvae and spat as well as in other mollusc species. The pathogenicity of this agent has been demonstrated on larvae, but its effects on the spat are still being experimented. Furthermore, some strains of bacteria have been recognised as pathogenic for several bivalve species including the Pacific cupped oyster.
In the present study, some bacterial strains have been isolated from a mortality outbreak of C. gigas spat on a rearing site. These strains have been first phenotypically characterised. Among them, some pathogenic bacteria from the genus Vibrio have been identified by infection trials on C. gigas spat. The RNA 16S-coding gene has been sequenced so that molecular identification of the pathogenic strain could be achieved.
2 Experimental approach
During the summer 1997, pathogenic bacteria have been sought from natural harnessing spat during a mortality outbreak (mortality rate at sampling time: 60%).
Bacterial strains have been obtained from ground flesh of spat diluted in sterile sea water and spread out over two media: Zobell medium, on which grows the whole heterotrophic flora and TCBS, specific for the Vibrionaceae flora. The identification of the isolated strains has been realised with the results of 35 biochemical tests.
For each strain, fifty animals from 5 mm to 1 cm have been inoculated by bath in a bacterial suspension of about 107 bacteria ml–1 during 48 h at 20 °C. Animals have been rinsed and maintained in 20 l for two months at 20 °C with phytoplancton (Chaetoceros calcitrans) daily. Afterwards, some batches have been transferred to a 3-l aquarium. The spat has been regularly observed and considered as dead when the valves do not shut anymore and the mantle do not react after stimulation by prick. The dead animals have been sampled to seek for the inoculated bacterial strain and verify the Koch’s postulate.
Several batches of spat non-inoculated were used as negative control and two positive controls have been used: strains CRL10 and CRL22 of Vibrio tubiashi, pathogenic for molluscs’ larvae and spat.
After phenol/chloroform extraction, the bacterial 16S gene has been amplified by PCR with universal primers (5’-CTCAGATTCAACGCTGGCGG-3’ and 5’-GGCGGTGTGTACAAGGCCCG-3’). PCR product has been cloned and sequenced. The sequence obtained has been aligned with 32 other Vibrionaceae 16S sequences (Table 1) and phylogenic analysis has been conducted using three methods: neighbour-joining, maximum likelihood and maximum parsimony.
3 Results
During the mortality outbreak of September 1997, some characteristic signs were observed on the sampled dying animals: strong smell, weakness of the adductor muscle, which leads to a failure in the shutting of the valves. Samples were examined by PCR for the herpesvirus but the results were negative.
Three main bacterial genera have been isolated from the dying spat: (1) strains related to the Cytophaga/Flavobacterium group, (2) Strains related to Agrobacterium, (3) strains belonging to Vibrio. MNEMF5 strain related to Cytophaga/ Flavobacterium is the major strain on Zobell medium with 105 bacteria per gram of spat flesh.
The different isolated strains have been tested in infection trials with C. gigas spat. Six negative controls showed a low cumulative mortality rate (between 0 and 12%) at the end of the experiment. The two Vibrio tubiashi strains, CRL10 and CRL22, provoked cumulative mortality rates of 80% and 10% after two months. Two strains of Vibrio splendidus biovar II, MNEF2 and TNEMF6, caused a mortality of 10 and 18%, whereas TNEMF8 strain does not seem to have affected the animals. Finally, MNEF5 strain (Cytophaga) does not raise the cumulative mortality rate.
Some batches were then maintained in a 3-l aquarium for one more month. The V. splendidus strains MNEF2 and TNEMF6 caused a cumulative mortality of respectively 12 and 80%, showing a different pathogenicity, although they both belong to the same species. The positive control showed a mortality of 76%, whereas 6% of the non-inoculated spat died.
The animals that died during the experiment have been analysed to quantify the bacterial strains in the tissues: the animals infected with TNEMF6 showed 5 105 bacteria per gram of flesh and this strain was the major one.
In the second set of experiments, the pathogenic feature of TNEMF6 strain has been confirmed with mortalities of 32 and 26% after seven weeks, whereas the controls showed 12% for CRL 10 and 2% for CRL 22.
The molecular characterisation (16S gene sequencing and alignment with 32 other Vibrio strains) led to a phylogenetic study of this strain. Its result confirms the phenotypic identification by relating this strain to Vibrio splendidus biovar II.
4 Discussion
The frequently occurring outbreaks of summer mortality concerning the oyster spat are of great concern for the oysters’ farmers who fear the disappearance of C. gigas due an epizooty as the Portuguese oyster C. angulata. Since 1995, our laboratory described bacterial strains associated with mortality outbreaks. In this study, we present the isolation of the first bacterial strain potentially pathogenic for the cupped oyster’s spat in France. This strain (TNEMF6) has been isolated from natural harnessing spat sampled during a mortality outbreak. It has been phenotypically and genotypically identified as Vibrio splendidus biovar II. Other V. splendidus isolated strains have a low virulence (as MNEF2) or none (as TNEMF8). V. splendidus is widely spread in marine ecosystems. Few strains are virulent in this ubiquitous species. Furthermore, the classification within one species of all these strains should be re-examined with the actual knowledge of molecular phylogeny, which suggests the existence of distinct groups. The confirmation of the existence of several species could only be obtained by DNA-DNA hybridisations with strains representative of the different groups.
V. splendidus strains have been associated with mortalities of C. gigas larvae, of scallops Pecten maximus and of fish. A great diversity of virulence between strains of the same species has been observed with the different animal models. Up to now, the only way to determine the virulence of a strain is to realise experimental exposures. This approach is inconvenient and results may vary from one trial to another. We do not have any phenotypic or genotypic feature to distinguish a pathogenic V. splendidus strain. Studies on restriction profiles of ribosomal genes or RAPD did not lead to the identification of a pathogenicity marker. We are now working on the development of diagnostic tools specific of virulence factors. The steps will be: (1) establishment of a collection of V. splendidus strains, (2) genomic subtraction between virulent and non-virulent strains, (3) cloning of virulence genes. At the same time, in vitro models will be developed to test the virulence of the bacteria.
An interesting point of this work is the virulence variability of the pathogenic strains, depending on the experimental conditions. For example, the spat infected with V. tubiashi (CRL 10) shows a mortality rate of 80% in the 20-l tank and a mortality rate of only 12% in the second experiment. During the first set of experiments, the mortality rates of the spat infected with V. tubiashi (CRL 22) and V. splendidus (TNEMF6) were about 10% after two months in 20-l tanks, but after one more month in aquarium, the mortality rate reached 80%. On the other hand, although the batch infected with MNEF2 strain received the same experimental changes, the pathogenicity of this strain was not modified. Thus, the spat origin, the rearing methods and the Vibrio strain seem to be important factors.
These results are in agreement with the summer mortality field studies indicating a complex aetiology with several factors implicated (environmental conditions, physiological, genetic and pathological parameters). We propose to realise infection trials on the virulence of the isolated strains under variable environmental conditions and with distinct genetic families.


