1. Introduction
Most of life on Earth depends on the activity of photosynthetic organisms able to capture solar light energy to convert atmospheric CO2 to sugars, while releasing molecular oxygen from water [1]. Photosynthesis is vital not only for photosynthetic organisms, but also for heterotrophs like animals and humans that consume this organic matter and rely on the photosynthetically-produced oxygen for respiration. In the 20th century, plants, as major primary producers in terrestrial environments massively used in agriculture, have represented an active subject of research with the core aim of understanding the mechanisms underlying their growth and development [2]. Nevertheless, aquatic photosynthetic phytoplanktonic microorganisms, including eukaryotic microalgae and cyanobacteria, fix the same amount of carbon and evolve as much O2 as land plants [3]. Microalgae proliferate in marine and freshwater environments, but also in soils, rocks and ice, show a remarkable phenotypic diversity at the cellular, physiological and metabolic levels, and display a variety of life cycles and lifestyle traits [4, 5] (Figure 1). They greatly contributed to the spread of life in aquatic and terrestrial environments and play a critical role in sustainability of our planet by driving nutrient cycles and nourishing food webs [1].
(A) Schematic and simplified representation of the eukaryotic tree of life based on [6]. Groups that include microalgae are shown as colored ovals. SAR signifies the clade comprising Stramenopila, Alveolata, and Rhizaria. Plastid origins for each lineage are shown by colored circles as primary, secondary red or secondary green plastids, or plastid replacement (serial secondary or tertiary) based on [7]. Stramenopila and Alveolata also include non-photosynthetic species that have lost their plastids. (B) Transmission electron micrographs of a central longitudinal section of the green alga Chlamydomonas reinhardtii (y-1 yellow mutant) grown in the light (top panel: https://doi.org/doi:10.7295/W9CIL37252) and of the marine diatom Phaeodactylum tricornutum (oval morphotype) grown in the light (bottom panel, courtesy of Dr. Atsuko Tanaka, University of the Ryukyus, Okinawa, Japan). (C) Examples of different microalgae studied in the laboratory. Light microscope images of the diatoms (Stramenopila) Ditylum brightwellii (a), Thalassiosira pseudonana (b), P. tricornutum forming colonies (c) or as individual cells of the fusiform (d) or triradiate (e) morphotypes, and Cyclotella sp. (f); the green algae (Chloroplastida) C. reinhardtii (g) and Tetraselmis sp. (h); the dinoflagellate (Alveolata) Amphidinium carterae (i); Heterosigma akashiwo (Stramenopila) (j); the haptophytes Exanthemachrysis sp. (k) and coccolithophore Emiliania huxleyi (l); and a mixture of microalgae illustrating the morphology, size and color diversity (m). Images (a–m) were produced with the kind support of Gwenaëlle Gain. Marine microalgae were obtained from the Roscoff Culture Collection and Chlamydomonas (g) from the UMR7141 collection. Scanning electron micrograph of different marine species isolated from the bay of Naples, Italy. Image kindly provided by Diana Sarno and Marina Montresor (Stazione Zoologica Anton Dohrn, Napoli, Italy) (n).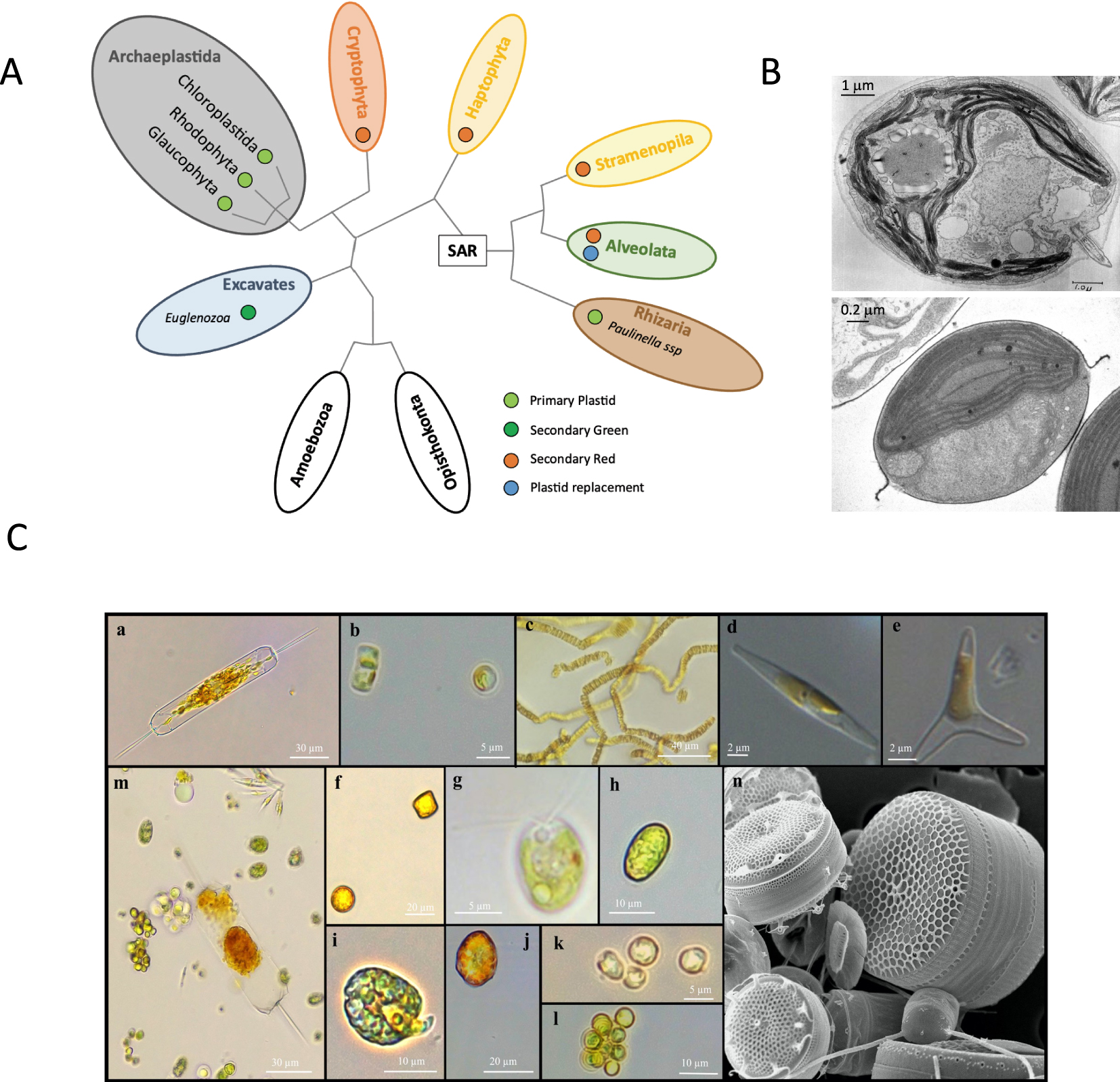
Eukaryotic microalgae result from a complex evolutionary history that still reveals unexpected detours. The plastid, the organelle hosting photosynthesis, is believed to derive from a free-living cyanobacterium, engulfed more than 900 million years ago by a heterotrophic host in a primary endosymbiosis [7]. This event gave rise to the clade of Archaeplastida, which includes the glaucophytes, the red algae and the green lineage, the latter comprising green algae and plants. However, most algae that dominate in marine environments originated from the subsequent engulfment and retention of a eukaryotic alga (red or green) through secondary, and even tertiary, endosymbiotic events. As a result, algae with secondary plastids are widely distributed throughout the eukaryotic tree of life [6] (Figure 1A). Among them, marine diatoms, which arose about 190 million years ago and comprise more than 100,000 species, represent the most diverse group of algae in contemporary oceans [8].
Functional biodiversity of photosynthetic organisms is an exciting new research frontier. How has photosynthesis evolved and diversified in such vastly different organisms? How do they handle the dialog between the nucleo-cytosolic and chloroplast compartments to ensure proper biogenesis and recycling of photosynthetic proteins? How do these phototrophs perceive their environment and modulate key physiological activities to ensure successful photosynthesis and growth in contrasting ecosystems? We need to address these questions practically, given the breadth of biodiversity, knowing that most microalgae have never been isolated from the environment, let alone cultivated in a laboratory.
The increasing availability of algal genomic data from different clades gathered over the last 20 years now enables a better exploration of microalgal biology [4]. With the increased accuracy of long-read sequencing, high-quality genome assemblies are now attainable for many microalgae. For example, the recent telomere-to-telomere assembly of the genome of the unicellular green alga Chlamydomonas reinhardtii [9] has allowed a precise description of its subtelomeric structures [10]. Comparative genome analysis and phylogenomics combine in a powerful strategy for predicting the function of genes based on their evolutionary history, in the framework of the overall evolution of chromosomal architecture. This strategic framework stands ready to analyze the avalanche of new data from environmental genomics and transcriptomics samples, through which the presence or differential expression of genes in different groups of microalgae can be studied in situ and related to the prevailing environmental conditions. We are about to start identifying the putative drivers of functional processes in global ecosystems [11] and unveiling regulatory gene networks involved in acclimation and adaptation to biotic or abiotic factors, even in species which have yet to be studied in laboratory conditions [12].
However, to integrate all these data, we need to better understand the molecular functions of individual genes and the properties of the regulatory networks they constitute. Function is mostly inferred from sequence conservation with homologues of known function, but inference rapidly becomes more hazardous as the phylogenetic distance between genes increases, as is often seen in environmental data. Added to this, a large proportion of genes in any newly-sequenced genome have no obvious homologue. They remain uncharacterized but may play a critical role in adaptation of microalgae. Today, the most common approach to infer algal gene function relates to acclimation processes in many marine algae, based on the analysis of transcriptomic data. It is however reasonable to expect that post-transcriptional and epigenetic modifications, as well as complex metabolic and cell signaling networks, also contribute to alga-environment relationships, as already demonstrated in land plants [13, 14] and currently under scrutiny in a few algal species [9, 13, 15, 16].
It is therefore essential to bring large-scale omics research closer to the study of characteristic regulatory processes in experimental model species. In the last decades, several model microalgae from different clades have emerged that lend themselves to molecular studies. In the green lineage, the freshwater alga C. reinhardtii is a model of choice for the study of photosynthesis, cell-cycle and cilia biology, and for the production of valuable bioproducts [17]. Its haploid nuclear genome facilitates the generation of loss-of-function mutations with observable phenotypes and ensuing genetic analyses (Table 1). Moreover, its nuclear, chloroplast and mitochondrial genomes are all readily transformable. C. reinhardtii is a facultative autotroph that can grow in the dark when provided with a source of reduced carbon, such as acetate, which is an advantage for the genetic dissection of photosynthesis. The systematic phenotyping of tens of thousands of mutant strains subjected to hundreds of different stresses has opened the way to large-scale functional studies of C. reinhardtii genes [18]. Taken together, C. reinhardtii thus stands as a reference phototrophic organism resulting from primary endosymbiosis in the green lineage. More recently, marine diatoms in the Stramenopile clade have been established as novel models for studying the biology and diversity of marine phytoplankton [8]. Due to its rapid growth rates, a small genome, and a wealth of associated genetic resources, Phaeodactylum tricornutum is currently the most established diatom model for research (Table 1) ranging from molecular and cell biology to ecophysiology and evolution, also proving to be a valuable new model for phototrophic organisms resulting from secondary endosymbiosis. Starting with genome sequencing, a significant number of other algae belonging to different clades are also becoming new models to answer specific biological and ecological questions or biotechnological challenges [4, 19].
Genomic features, currently available genome-enabled resources and biophysical methods for the green algae Chlamydomonas reinhardtii and the diatom Phaeodactylum tricornutum, based on [19] and [17]
| C. reinhardtii | P. tricornutum | |
|---|---|---|
| Nuclear genome size (Mbp) | 111.1 (strain CC503) | 27.4 (strain CCAP 1055/1) |
| Ploidy | haploid | diploid |
| Facultative phototrophy | Yes | No |
| Number of chromosomes | 17 | 34 pairs |
| Number of coding sequences (CDS) | 17,741 | 12,233 |
| Plastid genome size | 205.7 kbp | 117 kbp |
| Plastid number/ploidy | 1/80 | 1/130 |
| Plastid CDS | 75 | 128 |
| Mitochondria genome size | 15.7 kbp | 77.3 kbp |
| Mitochondrial CDS | 8 | 34 |
| 𝛼-solenoïd proteins | 14 PPRs, 140 OPRs | 42 PPRs, 4 OPRs |
| Genome portal (annotation version) | https://phytozome-next.jgi.doe.gov/info/CreinhardtiiCC_4532_v6_1 | https://genome.jgi.doe.gov/Phatr2/Phatr2.home.html (v2) |
| https://www.chlamycollection.org/chloro/genome.html | http://protists.ensembl.org/Phaeodactylum_tricornutum/Info/Index (v3) | |
| Centralized genome-enabled resources and databases | https://www.chlamylibrary.org/ | https://www.diatomicsbase.bio.ens.psl.eu/ |
| http://genomes.mcdb.ucla.edu/index.html | ASAFinder: https://rocaplab.ocean.washington.edu/tools/asafind/ | |
| https://www.chlamycollection.org/ | ||
| http://chlamystation.free.fr/ | HECTAR: http://www.sb-roscoff.fr/hectar/ | |
| Biophysical methods | Chlorophyll fluorescence (lifetime, low temperature) Absorption transient spectroscopy (redox changes, Electrochromic shift), Gas exchanges (O2 electrode, C14, Membrane Inlet Mass Spectrometry) |
Chlorophyll fluorescence (lifetime, low temperature) Absorption transient spectroscopy (redox changes, Electrochromic shift), Gas exchanges (O2 electrode, C14, Membrane Inlet Mass Spectrometry) |
| Nuclear transformation methods | Biolistic | Biolistic |
| Electroporation | Conjugation | |
| Glass beads | Electroporation | |
| Plastid transformation methods | Biolistic | Biolistic |
| Glass Beads | Electroporation | |
| Mitochondrial transformation | Biolistic | — |
| Modular cloning technology | Golden Gate (MoClo) | Gateway |
| Golden Gate | ||
| Gene silencing | Inverted repeat and antisense fragments | Inverted repeat and antisense fragments |
| Genome editing methods | CRISPR/Cas9, CRISPR/Cas9 delivered as RNP, ZFNs | TALEN, Meganucleases, CRISPR/Cas9, CRISPR/Cas9 delivered as RNP |
| Sexual reproduction | Heterothallic, isogamous | Not mastered in laboratory |
| Genetic analysis | Tetrad analysis |
Here, we focus on the intimately interconnected light-driven processes of photosynthesis and photoperception as well as on plastid biogenesis. We will provide concrete examples of how integrated approaches of ecophysiology, genomics and genetics offer novel opportunities to explore microalgal biology and diversity in phototrophs resulting from primary and secondary endosymbiosis.
2. Functional biodiversity of photosynthesis in microalgae
Oxygenic photosynthesis is the main process converting the energy of sunlight into chemical energy. The very first step of this conversion is the harvesting of photons by the pigment–binding protein complexes packed in the thylakoid membranes. In photosystems I and II (PSII and PSI) supercomplexes, light-harvesting complexes (LHCs) are associated with reaction centers where the photochemical reactions occur. Thanks to an electron transfer chain (ETC), PSI and PSII photochemistry work in series and allows both the oxidation of water and the supply of reducing power to the Calvin cycle, whereby carbon is fixed. The photosynthetic complexes, the soluble electron shuttles and the Calvin cycle enzymes are basically the same in microalgae and plants [1]. This strong conservation of elemental bricks contrasts with the huge diversity in light-harvesting strategies in terms of pigment composition, LHC proteins and photoprotection mechanisms.
Structural biology techniques, especially cryo-electron microscopy, have revealed a surprising variability among algae in the architecture of pigment–binding proteins and of their interactions to form supercomplexes. The number, nature, position and relative orientation of the pigments have been optimized for the typically low, but variable, light environment experienced in water. The light harvesting system allows a high efficiently energy transfer to the photochemical trap under low light but this efficiency is reversibly dampened under high light, through modifications in the nature, position or orientation of the pigments. These modifications favor non-radiative dissipation of the light energy as heat at the expense of photochemistry, which is visualized through the non-photochemical quenching (NPQ) of chlorophyll fluorescence. This widespread solution is of global ecological importance: a large survey of four ocean basins showed that 60% of the sunlight absorbed by marine microalgae is converted into heat [20].
We are just starting to unveil the biodiversity of microalgal photosynthesis. Photoprotection is a good example of how common observables and techniques (especially spectroscopy, genetics and structural biology), combined with progresses in genomics, provided precious insights into the biodiversity of this crucial aspect of photophysiology (reviewed in [21]). At first sight, studies in various microalgae (and photosynthetic organisms in general) revealed a common scheme: the joint action of a PSII subunit and the so-called xanthophyll cycle (the reversible deepoxidation of a carotenoid) creates the back-and-forth switch between a light-harvesting and a photoprotective (heat dissipation) mode. Depending on photosynthetic organisms, the molecular players and regulation processes, however, turned out to be widely different.
In higher plants, NPQ relies on pigment conversion, through the Violaxanthin–Antheraxanthin–Zeaxanthin (Vx/Ax/Zx) xanthophyll cycle (XC), and on conformational changes within PSII, mediated by the conditionally-expressed PSBS protein, which interacts with light-harvesting proteins and acts as the sensor for the luminal pH through its ability to bind protons [22]. In C. reinhardtii a different protein, LHCSR3, also plays a dual role: it is involved in pH sensing but also binds pigments (including Zeaxanthin), even though the direct role of Zeaxanthin is still not clear [23]. Considering microalgae arising from secondary endosymbiosis, NPQ is mostly studied in diatoms, especially P. tricornutum [24]. Contrary to plants and green algae, diatoms possess a diadinoxanthinin–diatoxanthin (Dd/Dt) xanthophyll cycle. The recent development of genetic techniques for diatoms has made it possible to dissect the NPQ mechanism. Using RNA interference and knock-out mutants, the LHCX proteins were shown to be involved in NPQ [25], and later to be strictly necessary [26]. The LHCX protein likely has a different role than PSBS and LHCSR, since it does not seem to sense lumenal pH [27, 28] while potentially binding Dt. Variability in LHCX expression contributes to phenotypic plasticity in diatoms, as evidenced by comparing several ecotypes of P. tricornutum that express NPQ at different levels [25]. The pioneering works on diatom LHCXs have been critical for our knowledge of the ecophysiological adaptation of aquatic microalgae to changing light conditions. Indeed, LHCX or LHCX-like genes have also been found in Haptophytes and Dinophytes [29]. These two groups use the Dd/Dt cycle as diatoms do. However, other organisms that arose from the secondary endosymbiosis possessing LHCX or LHCX-like proteins use the Vx/Ax/Zx cycle instead, for example, Chrysophytes, Eustigmatophytes, Raphidophytes and the alveolata Chromera velia. Initial analyses have indicated that the XC encoding genes in secondary-endosymbiosis algae derive from green algae, but the evolutionary history of LHCX is not fully understood.
The simple scheme presented in Figure 2, where microalgal NPQ is defined by the combination of a PSII subunit (PSBS, LHCSR or LHCX) and a XC (Vx/Ax/Zx or Dd/Dt), is already slated for updating. Cryptophytes do not possess XC nor PSBS, LHCSR or LHCX like proteins [30, 31] but display a significant NPQ, which indicates that novel NPQ actors await discovery. The photosynthetic amoeba Paulinella (whose plastid comes from an independent primary endosymbiosis) does not show any fast NPQ process and does not possess XC nor PSBS, LHCSR or LHCX-like proteins [32]. Moreover, the kinetics and mechanisms of the process are hugely diverse. Indeed, the dual roles of the xanthophyll pigment as direct quencher or indirect modulator of the energetic landscape in the light-harvesting system, combined with the dual role of PSII proteins in binding pigment and/or sensing pH, generate a high level of complexity that genomics data alone cannot reveal. From this point of view, there is a need for mechanistic studies of NPQ in model organisms representing Haptophytes, Dinophytes and non-diatom Stramenopiles possessing LHCX-like proteins and the Vx/Ax/Zx cycle.
Schematic representation of the photoprotection mechanism in the diatom P. tricornutum. When the light absorption by PSII-associated antenna is low (represented by the small yellow arrow), the proton concentration in the lumen remains low and the xanthophyll pigments are mostly in their epoxidized diadinoxanthin form (Dd). When the light absorption increases under bright light (represented by the large yellow arrow), the higher proton concentration (i.e. lower pH) in the lumen activates the diadinoxanthin de-epoxidase. The concentration of diatoxanthin (Dt) increases, which together with the presence of LHCX proteins, leads to enhanced heat dissipation (represented by the red arrow). (B) Schematic representation of the eukaryotic tree of life as in Figure 1. The circles next to each microalgal group represent the combinations of different NPQ regulators, PSII subunits (LHCX, PSBS or LHCSR) and xanthophyll cycles (Vx/Ax/Zx, green circle; Dd/Dt, orange circle) described in different lineages, based on [24].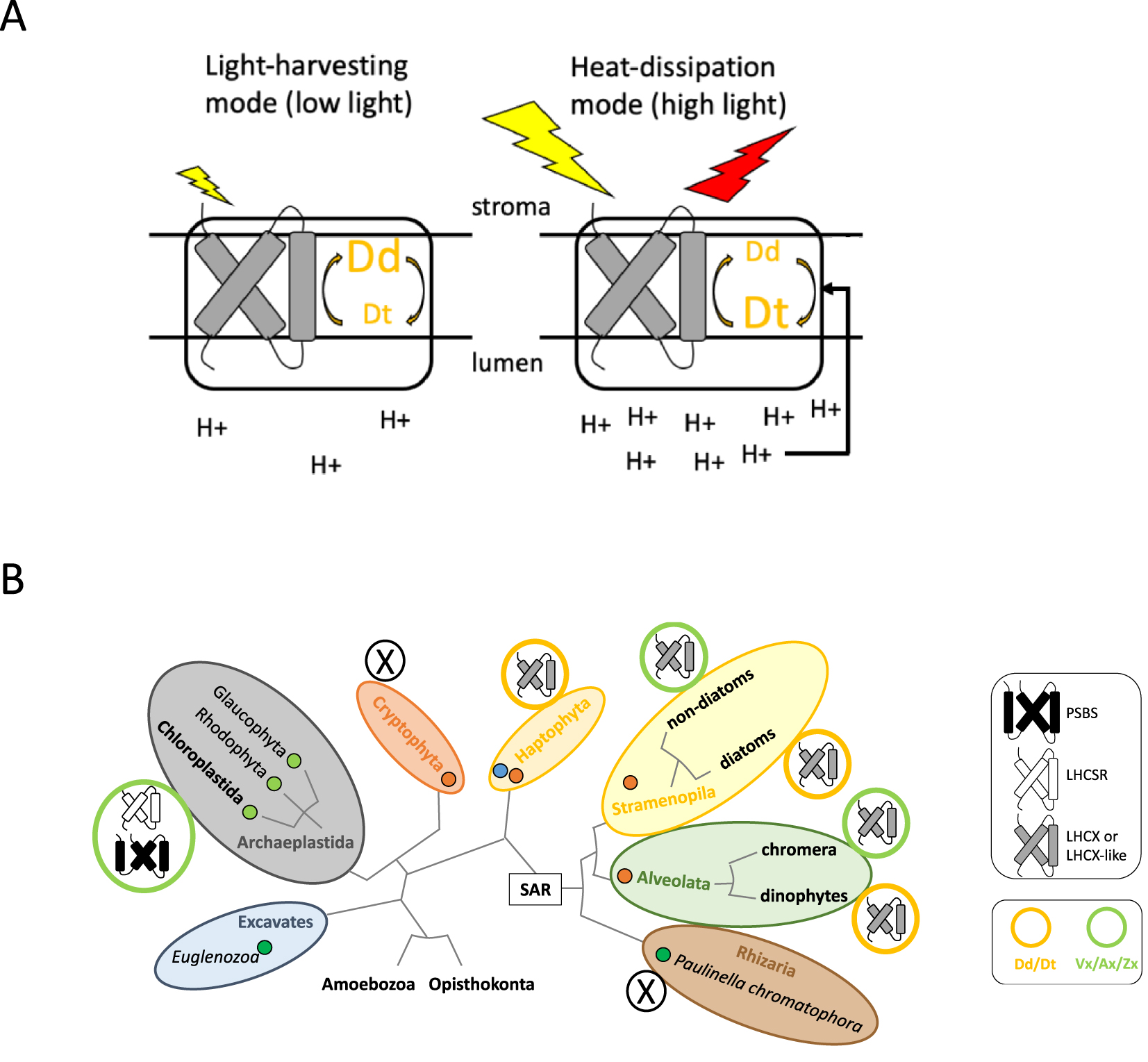
3. Sensing the light environment in microalgae
Light is a major source of information about the environment and influences pivotal aspects of development, physiology and metabolism in many organisms. Photoreceptors perceive light signals and activate downstream signaling pathways to regulate physiological responses. A plethora of photoreceptor-like sequences have been identified in algal genomes and, more recently, in metagenomic data [33, 34, 35]. Some algal photoreceptors are similar to those of terrestrial plants, but new species- or lineage-specific photoreceptors or spectral tuning variants are also found (Figure 3), likely to be specific adaptations to the underwater light field. Indeed, the light perceived by marine organisms varies significantly in intensity and spectral composition due to the absorption properties of water, which selects green and blue light with increasing depth [35]. Not surprisingly, diversified blue-light sensors are found in algal genomes. For instance, novel variants of the wide-spread blue-light cryptochrome photolyase family (CPF) have recently been described. This family encompasses DNA photolyases, blue-light activated enzymes that repair UV-induced DNA damages, and cryptochromes, known for their photoreceptor functions in terrestrial plants, and as transcriptional regulators of the circadian clock system in animals. In P. tricornutum, CPF1 is a novel protein with both DNA repair and photoreceptor activities [36] that may represent the missing link in the evolution of CPF catalytic activity from photolyase to photoreceptor. Since that discovery, CPF1 with DNA repair activity and diverse light sensing properties have been found in other microalgae, including Chlamydomonas and O. tauri [37], suggesting that a single protein with dual functions might be important in dynamic aquatic environments. Interesting novel CPFs have recently been identified by using a new computational method that extracts functional features from large protein sequence datasets [38]. The actual function of novel CPFs can now be tested experimentally in microalgal model species to confirm whether they are indeed light-responsive proteins with novel features. Diatoms also contain aureochrome photoreceptors, which are unique to stramenopiles [39]. Containing a light-oxygen-voltage (LOV) domain for blue light perception and a basic leucine zipper (bZIP) domain for DNA binding, these proteins act as light-regulated transcription factors and control several key biological processes such as gene expression, cell cycle and NPQ [39, 40]. Interestingly, green algae such as Chlamydomonas, have a different blue light photoreceptor with a LOV domain, called phototropin, which act as regulator of gene expression [41] and NPQ [42]. This suggests that different photoreceptors have independently evolved in different lineages to optimize photosynthesis, with blue light detection being a key driver, probably due to the abundance of this wavelength in the photic zones (Figure 3). By contrast, the existence of a diatom phytochrome (DPH) which absorbs red and far-red light and mediates far-red signaling is surprising, considering the strong attenuation of these wavelengths in deep water [43]. We are currently investigating whether DPH has a role as a depth detector or as a sensor of neighboring phototrophs, based on functional characterization of numerous DPH from diverse diatom species and the observed distribution of DPH genes in metagenomic data from the world’s oceans.
Main classes of photoreceptors encoded in the genomes of different microalgal groups. The schematic structure of these photoreceptors is indicated. The colors of the boxes indicate the classical light wavelengths the light-sensing domains perceive. White domains are those involved in signal transduction. PHR, photolyase related-domain; LOV, Light-Oxygen-Voltage domain; STK, serine/threonine kinase; OPM, output module differs according to the microalgae group; Rh, rhodopsin domain; PAS, Per-Arnt-Sim domain; GAF, cGMP phosphodiesterase/adenylyl cyclase/FhlA domain; PHY, phytochrome-specific domain. The functions indicated are those described for microalgae [34, 35].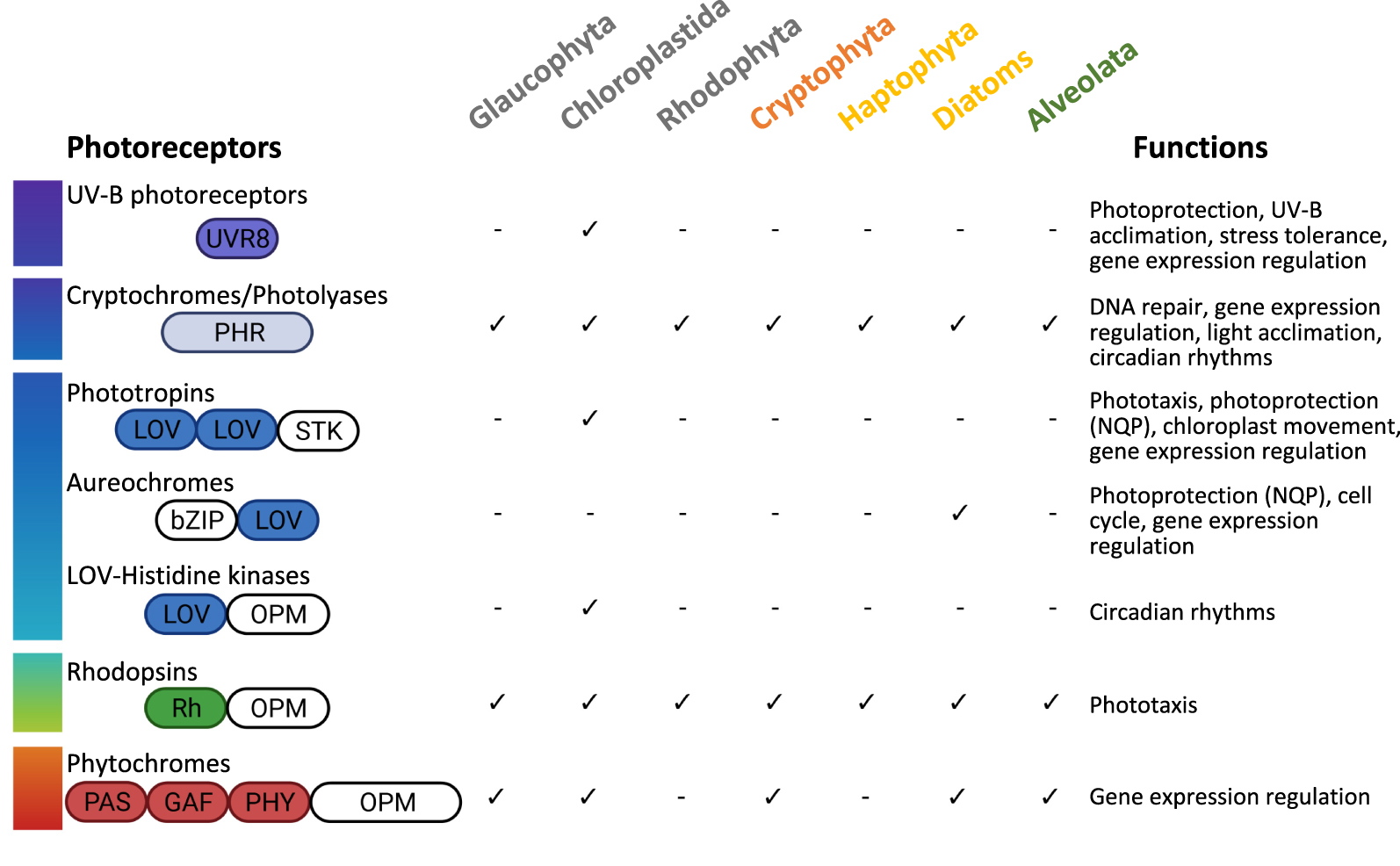
Photoreceptors also play a key role in the entrainment of the circadian clock, an endogenous regulator which governs the timing and synchronization of biological processes with periodic light-dark cycles in most organisms [44]. The molecular mechanisms of the clock have been extensively characterized in terrestrial organisms but only in a few algae of the green lineage, such as C. reinhardtii or O. tauri [45]. Circadian clock components are still largely unknown in many phytoplankton species resulting from secondary endosymbiosis, partly because the genome of these organisms lacks clear homologs of the clock components identified so far. By integrating functional genomic approaches with in vivo analyses of light-dependent physiological processes, we recently identified the existence of a functional circadian clock in P. tricornutum [46]. The bHLH-PAS transcription factor, named RITMO1, is the first known component of the diatom circadian clock system. Phylogenetic analysis reveals a wide distribution of RITMO1-like proteins in the genomes of diatoms and other marine algae, which may indicate they have a common function in these phototrophs. RITMO1 now represents a key molecular entry point for the identification of other clock components and for a deeper understanding of marine rhythms and of their evolutionary and ecological relevance.
4. Chloroplast diversification and biogenesis
Understanding eukaryotic photosynthesis requires a good knowledge of plastid biogenesis and repair, which have only been studied in the green lineage, but remain terra incognita in the other clades, including diatoms and their Rhodophyte ancestors.
A feature common to all plastids is the small number of genes, involved in photosynthesis or plastid gene expression, that remain encoded in their genome, as most genes of the ancestral endosymbiont were transferred to the nucleus during evolution. Indeed, the vast majority of the plastid proteome is encoded in the nucleus, translated in the cytosol and subsequently imported into the plastid. Thus, the biogenesis of the photosynthetic apparatus requires the crosstalk of plastid and nuclear genomes, as well as specific import machineries.
In the green lineage, most nucleus-encoded plastid proteins are routed to the organelle by amphipathic targeting peptides (TP), which interact with the outer membrane envelope, are then imported into the plastid by a TIC/TOC translocon, where they are removed by specific peptidases. TPs share their amphipathic properties with some antimicrobial peptides, which may be a legacy of early endosymbiotic events: TP would derive from antimicrobial peptides (AMP) [47, 48], while the TIC translocon would result from the cooptation of an endosymbiont defense mechanism against host innate immunity. Interestingly, in Chlamydomonas, antimicrobial sequences fused to a reporter gene allowed its import into organelles [49]. This AMP-resistance probably contributed to the retention of the cyanobacterial prey within the host by enabling the endosymbiotic transfer of genes, whose products can be imported back into plastid.
As a consequence, photosynthetic complexes and the plastoribosome are of dual genetic origin. How do microalgae coordinate the production of the different subunits in the stoichiometry required for functional complex assembly? In Viridiplantae, a first level of coordination relies on organellar trans-acting factors encoded in the nucleus that tightly control, in a gene-specific manner, all post-transcriptional steps of the expression of plastid mRNAs. Most of them belong to the 𝛼-solenoid superfamily: they are either pentatricopeptide repeat proteins (PPRs), defined by the tandem repetition of a degenerate motif of 35 residues and abundant in land plants, or octotricopeptide repeats proteins (OPR; 38 residues degenerate repeats) numerous in green algae but absent from red algae and scarce in land plants. The specificity of these two motifs for a given RNA results from the interaction of residues at defined positions within the repeat with a specific nucleotide of the transcript. A code governing these amino acid–nucleotide interactions has been established for PPR proteins [50], while our laboratory is establishing the “OPR code” and analyzing the evolutionary history of organelle-targeted 𝛼-solenoid proteins in phototrophs resulting from primary and secondary endosymbiosis.
In the green lineage, a second level of coordination lies in the concerted accumulation of subunits of a photosynthetic complex. In contrast to what is observed in cyanobacteria, mutants not expressing a core subunit of a complex pleiotropically lose all other subunits, thus avoiding the wasteful or even toxic accumulation of unassembled polypeptides (reviewed in [51]). This results from two complementary mechanisms: (i) some subunits are rapidly degraded by chloroplast proteases when they cannot assemble within a complex [52]. (ii) others, all encoded by the chloroplast genome, are controlled by epistasy of synthesis (so-called CES subunits): they are not degraded in absence of assembly, but their rate of synthesis is strongly reduced. This latter regulatory mechanism, first identified in Chlamydomonas by our laboratory, plays an important role in chloroplast biogenesis since all major photosynthetic complexes contain at least one, and often several, CES subunits [51]. By coupling the translation of some chloroplast genes to assembly of their products, CES allows rapid fine-tuning of the production of a subunit to its demand for the biogenesis of a photosynthetic complex.
Compared with green algae, microalgae resulting from secondary endosymbiosis face the same and also other challenges. As a eukaryotic alga was engulfed, the import system differs: proteins encoded in the nucleus but located in the plastids must cross four membranes to reach the stroma, and not two as in the archeplastidia. Bearing a bipartite pre-sequence comprised of a signal peptide followed by a plastid targeting peptide, they are first targeted to the endoplasmic reticulum (ER) and then transferred to the periplastidial compartment by a translocon derived from ER degradation machinery. Their further import into the chloroplast remains to be characterized.
In diatoms, the transfer of genes to the nucleus, largely begun in the red alga ancestor, continued after the secondary endosymbiosis, as their plastids contain fewer genes than those of red alga, but more than those of green algae. Still, diatom plastids encode several open reading frames inherited from red algal plastids but absent from the green lineage, whose role awaits study (Table 1). Diatoms must also coordinate the production of nucleus and plastid-encoded subunits. However, although their genomes encode organelle-targeted 𝛼-solenoid proteins, we still do not know, whether and how the nucleus controls the expression of plastid genes. Neither do we know if there is an assembly-controlled translation of plastid genes as is the case in the green lineage.
The plastid biogenesis features shared between diatoms, other secondary endosymbionts and Viridiplantae should tell us about the universal constraints for building a photosynthetic apparatus within an organelle. It should also provide insights into new regulatory mechanisms of photosynthesis explaining their ecological success in all aquatic environments.
5. Concluding remarks and future challenges
The integrated studies of photobiological processes highlighted here include exciting discoveries but also significant knowledge gaps that need to be bridged to truly understand microalgal biology. Studying the functional biodiversity of microalgae may seem a never-ending task, but it can now be confidently tackled, provided there is a continuous and iterative dialogue between laboratory studies of mechanisms in selected model species and of data from the natural environment. For example, comparative functional genomic investigations in diverse model species can reveal unknown regulators of relevant biological processes, still hidden in phytoplankton genomes, which can be projected in natural scenarios to predict their impact on population dynamics. New functions can also be hypothesized by exploring the genetic diversity through ecosystem biology approaches, e.g. by associating (meta)genes to species or genus levels and addressing their differential expression in different environments [11]. As shown above for photoprotection, phylogenomics can help to categorize hundreds of thousands of genes into a few functional groups, which in turn determine a minimum number of model organisms to study in the laboratory. Laboratory results should be validated in an ecological context, thus requiring new in situ explorations, and vice versa. To better link laboratory and environmental studies, and address the issue of heterogeneity in cell populations, monitoring the microalgae responses to multiple and carefully controlled environmental parameters will require a combination of cell imaging, flow cytometry, microfluidics, single cell reporter assays and omics technologies. Such tools will help identifying molecular fingerprints indicative of the physiological state of microalgae, as well as new biomarkers that, in turn, will facilitate the study of microalgal physiology in complex environments. These integrated studies will eventually feed robust ecosystem models, to complement hypothesis testing.
We will also need to develop new microalgal models from lineages still recalcitrant to genetic investigations, such as Dinophytes and Haptophytes. The identification of ecologically relevant species, based on estimations of the distribution and diversity of microalgae, should be considered alongside other practical criteria such as rapid growth, mastered life cycle in laboratory, suitability for genetic manipulations, and sufficient genomic data. Natural accession variants, sampled over a broad geographical scale, will make it possible to draw correlation between genotype, phenotype and ecological success [25], with pan-genomic studies focusing on this infra-specific diversity. The choice of a new model heavily depends on which specific processes will be studied. Indeed, studies of photosynthesis mutants, many of which were isolated in the facultative phototroph C. reinhardtii in our laboratory (http://chlamystation.free.fr/), have constantly driven photosynthesis research. Unfortunately, the most studied diatoms are obligate phototrophs, preventing isolation of such mutants. The diatom Cyclotella cryptica is a promising new model for the study of diatom photosynthesis because it grows both heterotrophically (in the presence of glucose) and autotrophically, while genomic and genetic resources are already available [53]. Our laboratory is currently working to establish this species as a fully-fledged genetic model for photosynthesis.
Human activities are continuing to alter oceans through pollution, acidification, deoxygenation and warming (Ref. [54], IPPC 6th Assessment Report) but to estimate the impact of these changes we need to understand their consequences on highly diverse primary producers like microalgae. Improving photosynthesis efficiency is also a challenge to tackle the ever-growing demands for food and energy and to provide potential strategies to mitigate the consequences of climate changes. Such ambitious objectives can only be attained by precisely studying how phototrophs have adapted to their environments and which acclimation strategies they use to fine-tune photosynthesis in highly variable conditions. Light-driven processes are useful entry points into key aspects of microalgal functional biodiversity because the classical methods used are applicable to different algal groups and light stimuli can be delivered in precisely controlled ways. Photosynthesis is one of the very few functions that can be measured in situ, thanks to the high sensitivity of spectroscopic and fluorescence techniques. Photoperception can also be approached at a variety of levels, from the molecular to the environmental level. Combining studies of these light-driven processes in the phytoplankton has enormous potential to advance our understanding of the mechanisms by which light supports life.
Conflicts of interest
Authors have no conflict of interest to declare.
Acknowledgements
We would like to thank Francis-André Wollman and Pierre Joliot for their constant support for our laboratory’s research, and for their enthusiasm and critical and constructive insights particularly as we enter this new phase of focusing on major ecological issues. We would like to thank all the non-permanent members of the laboratory, technicians, students and post-doctoral fellows who make the development of our research possible. We thank the Fondation Bettencourt-Schueller (Coups d’élan pour la recherche francaise-2018) and the “Initiative d’Excellence” program (Grant “DYNAMO,” ANR-11-LABX-0011-01) for supporting our research on the functional biodiversity of microalgae.
Version française
1. Introduction
La plupart des formes de vie sur Terre dépendent de l’activité d’organismes photosynthétiques capables de capter l’énergie lumineuse du soleil pour convertir le CO2 atmosphérique en sucres, tout en oxydant l’eau en oxygène moléculaire [1]. La photosynthèse est aussi vitale pour les hétérotrophes comme les animaux et les humains, qui dépendent de l’oxygène produit par les organismes photosynthétiques pour respirer et consomment la matière organique produite. Au cours du siècle, les plantes, producteurs primaires majeurs en milieu terrestre massivement utilisés dans l’agriculture, ont constitué un sujet de recherche extrêmement actif. Ces recherches visaient à comprendre les mécanismes sous-tendant la croissance, le développement et la résistance des plantes à différents pathogènes [2]. Cependant, les microalgues eucaryotes et les cyanobactéries constituant le phytoplancton aquatique fixent autant de carbone et libèrent autant d’oxygène que les plantes terrestres [3]. Ces microalgues prolifèrent dans les océans, mais aussi dans les eaux douces, les sols, la glace et sur les roches et présentent une diversité phénotypique remarquable aux niveaux cellulaire, physiologique et métabolique, ainsi qu’une immense variété de cycles et de modes de vie [4, 5] (Figure 1). Elles ont joué un rôle essentiel dans la propagation de la vie et sont cruciales pour la viabilité de notre planète en contribuant aux cycles des nutriments et en alimentant les réseaux trophiques [1].
(A) Représentation schématique et simplifiée de l’arbre de vie des eucaryotes d’après [6]. Les groupes comprenant des microalgues sont colorés. Le clade SAR, comprenant les Straménopiles, Alvéolés et Rhizariens, est mis en évidence. Le cercle coloré près des différentes lignées indique les plastes primaires, secondaires rouges ou secondaires verts, ou le remplacement des plastes (secondaires en série ou tertiaires), d’après [7]. Les Straménopiles et Alvéolés incluent également des espèces non-photosynthétiques, qui ont perdu leur plaste. (B) Haut, coupe longitudinale centrale de l’algue verte Chlamydomonas reinhardtii (mutant y-1 yellow) cultivée à la lumière en microscopie électronique à transmission (MET), https://doi.org/doi:10.7295/W9CIL37252; Bas, photographie de MET de la diatomée marine Phaeodactylum tricornutum (morphotype ovale) cultivée à la lumière, avec l’aimable autorisation du Dr. A. Tanaka, Ryukyus University, Okinawa, Japon. (C) Exemples des différentes microalgues étudiées à l’UMR7141 (IBPC). Photographies de microscopie optique des diatomées (Straménopiles) Ditylum brightwellii (a), Thalassiosira pseudonana (b), P. tricornutum formant des colonies (c) ou sous forme libre dans le morphotype fusiforme (d) ou triradié (e), Cyclotella sp (f); des algues vertes (Chloroplastidés) C. reinhardtii (g) et Tetraselmis sp (h); de la dinoflagellé (Alvéolés) Amphidinium carterae (i); de Heterosigma akashiwo (Straménopiles) (j); Exanthemachrysis sp (Haptophytes) (k); de la coccolithophore Emiliania huxleyi (Haptophytes) (l); et d’un mélange de microalgues illustrant leur diversité (m). Photographies réalisées par Gwenaëlle Gain (IBPC). Les microalgues marines proviennent de la Roscoff Culture Collection et les Chlamydomonas (g) de la collection de l’UMR7141. Photographie de microscopie électronique à balayage de différentes espèces marines isolées à Naples, Italie, réalisée par D. Sarno et M. Montresor (Stazione Zoologica Anton Dohrn, Naples, Italie) (n).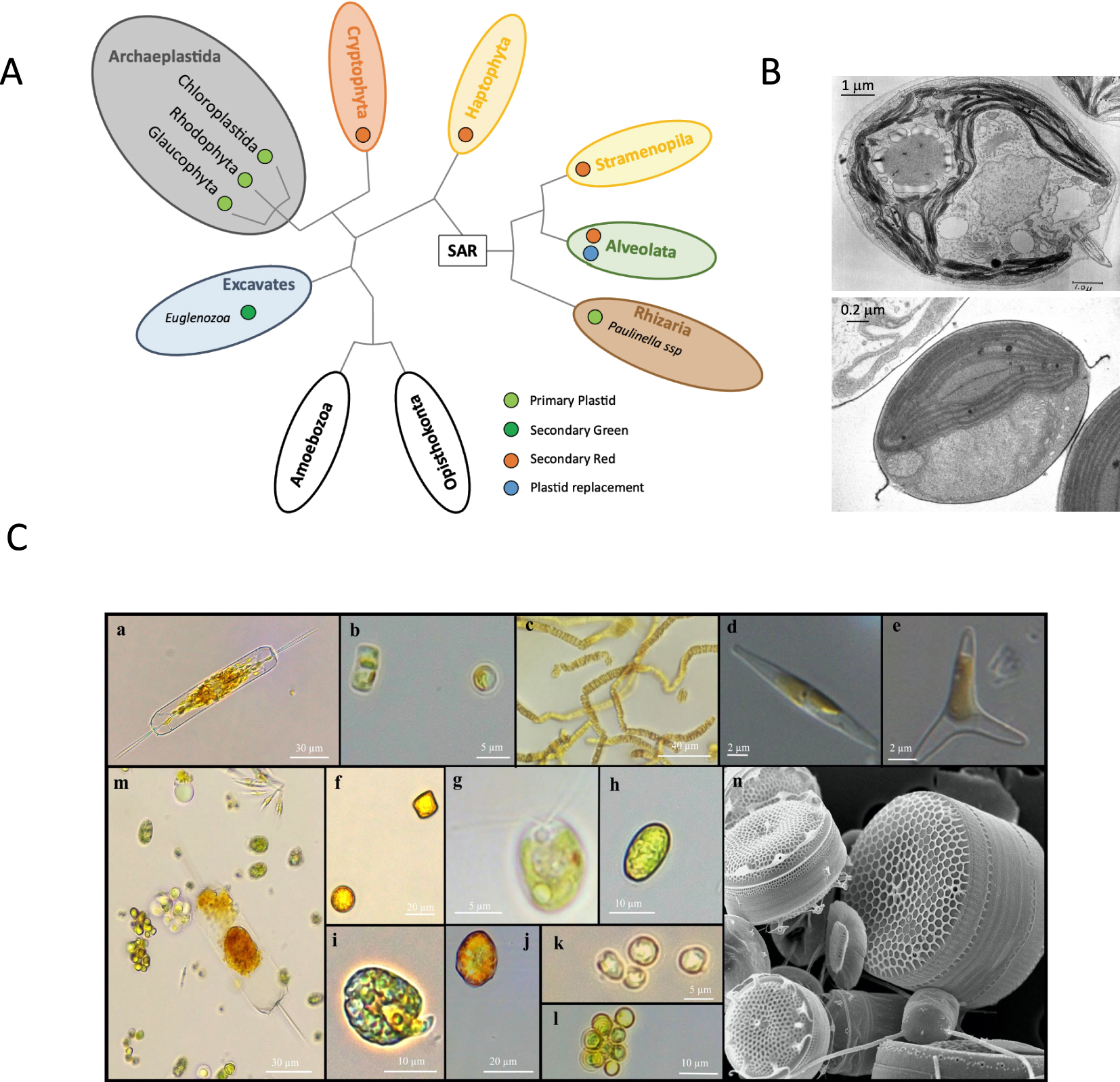
Les microalgues eucaryotes résultent d’une histoire évolutive complexe qui recèle péripéties et mystères. Le plaste, organite siège de la photosynthèse eucaryote, dérive d’une cyanobactérie libre, capturée il y a plus de 900 millions d’années par un hôte hétérotrophe au cours d’une endosymbiose primaire [7]. Cet événement a donné naissance au clade des Archaeplastida qui regroupe les glaucophytes, les algues rouges et la lignée verte (Viridiplantae), cette dernière comprenant les algues vertes (Chlorophytes) et les plantes. Cependant, la plupart des algues qui dominent dans les environnements marins résultent de la capture d’une algue eucaryote, rouge ou verte, lors d’événements d’endosymbiose secondaires, voire tertiaires. Ces algues dotées de plastes secondaires sont largement distribuées dans l’arbre de vie eucaryote (Figure 1A). Parmi elles, les diatomées, apparues il y a environ 190 millions d’années et comprenant au moins 100 000 espèces, représentent le groupe d’algues le plus diversifié dans les océans contemporains [8].
L’étude de la biodiversité fonctionnelle des organismes photosynthétiques est une nouvelle frontière passionnante de recherche. Comment la photosynthèse a-t-elle évolué et s’est-elle diversifiée dans des organismes aussi différents ? Comment les compartiments nucléo-cytosolique et chloroplastique dialoguent-ils pour assurer la biogenèse et le recyclage corrects des protéines photosynthétiques ? Comment ces phototrophes perçoivent-ils leur environnement et modulent-ils leurs activités physiologiques pour prospérer dans des écosystèmes très différents ? Comment aborder pratiquement ces questions étant donnée l’ampleur de la biodiversité des microalgues, dont la plupart n’ont jamais été isolées de l’environnement et encore moins cultivées en laboratoire ?
La disponibilité croissante de données génomiques de microalgues provenant de différents clades, recueillies au cours des 20 dernières années, permet de mieux explorer leur biologie [4]. Grâce aux progrès du séquençage de l’ADN et de l’ARN, des génomes de nombreuses microalgues de haute qualité sont désormais disponibles. Par exemple, l’assemblage récent « télomère à télomère » du génome de l’algue verte unicellulaire d’eau douce Chlamydomonas reinhardtii [9] a permis une description précise de ses structures sous-télomériques complexes [10]. L’analyse comparative des génomes combinée aux approches phylogénomiques offre une stratégie puissante pour prédire la fonction des gènes sur la base de leur histoire évolutive. Ce cadre permet désormais d’analyser l’abondance de données génomiques et transcriptomiques provenant d’échantillons prélevés dans différents milieux. L’existence ou l’expression différentielle de gènes dans différents groupes de microalgues peuvent ainsi être étudiées in situ et corrélées à des conditions environnementales précises. Ces avancées permettront d’identifier les facteurs contrôlant les principaux processus biologiques dans un écosystème global [11] ou de révéler les réseaux de régulation permettant l’acclimatation et l’adaptation aux facteurs biotiques ou abiotiques, y compris chez des espèces encore non étudiées en laboratoire [12].
Pour intégrer ces données, nous devons mieux comprendre les fonctions moléculaires des gènes individuels et également les réseaux fonctionnels qu’ils constituent. La fonction d’un gène est typiquement déduite de la conservation de sa séquence avec des gènes homologues de fonction connue. Mais cette prédiction devient rapidement aléatoire lorsque la distance phylogénétique entre les gènes augmente, comme souvent avec les données environnementales. En outre, le séquençage de tout nouveau génome révèle de nombreux gènes dépourvus d’homologues, dont la fonction reste inconnue. L’approche la plus courante pour identifier les gènes d’algues impliqués dans les phénomènes d’acclimatation est basée sur l’analyse de données transcriptomiques. Néanmoins, les mécanismes épigénétiques, ainsi que les réseaux métaboliques ou de signalisation cellulaire contribuent également aux interactions algues-environnement. Cela a été bien étudié chez les plantes terrestres [14], mais n’a encore été abordé que sur un faible nombre d’espèces de microalgues [9, 13, 15, 16, 55].
Il est par conséquent essentiel d’intégrer les approches « omiques » à grande échelle et l’étude des processus de régulation dans les espèces modèles. Au cours des dernières décennies, plusieurs microalgues modèles de différents clades et se prêtant aux études moléculaires ont émergé. Dans la lignée verte, C. reinhardtii est un modèle de choix pour l’étude de la photosynthèse, du cycle cellulaire et de la biologie des cils, ainsi que pour la production de bioproduits à haute valeur ajoutée [17]. Cette microalgue est un autotrophe « facultatif » et peut croître en absence de photosynthèse si on lui fournit une source de carbone réduit, comme l’acétate, ce qui permet la dissection génétique de la fonction photosynthétique. Son génome nucléaire haploïde facilite l’obtention et l’analyse génétique de mutants « perte de fonction » avec des phénotypes observables (Tableau 1). En outre, ses génomes nucléaires, chloroplastiques et mitochondriaux sont facilement transformables. Le phénotypage systématique de dizaines de milliers de souches mutantes soumises à des centaines de stress différents a récemment ouvert la voie à la génomique fonctionnelle à grande échelle chez C. reinhardtii [18].
Caractéristiques et ressources génomiques actuellement disponibles, et méthodes biophysiques d’étude pour l’algue verte C. reinhardtii et la diatomée P. tricornutum, d’après [17] et [19]
| C. reinhardtii | P. tricornutum | |
|---|---|---|
| Taille du génome nucléaire (Mpb) | 111.1 (souche CC503) | 27.4 (souche CCAP 1055/1) |
| Ploïdie | haploïde | diploïde |
| Phototrophie facultative | Oui | Non |
| Nombre de Chromosomes | 17 | 34 paires |
| Nombre de sequences codantes (CDS) | 17 741 | 12 233 |
| Taille du plastome | 205.7 kpb | 117 kpb |
| Nombre de plastes/Ploïdie | 1/80 | 1/130 |
| CDS plastidiales | 75 | 128 |
| Taille du génome mitochondrial | 15.7 kpb | 77.3 kpb |
| CDS mitochondriaux | 8 | 34 |
| Protéines 𝛼-solenoïdes | 14 PPRs, 140 OPRs | 42 PPRs, 4 OPRs |
| Accès génome (version d’annotation) | https://phytozome-next.jgi.doe.gov/info/CreinhardtiiCC_4532_v6_1 | https://genome.jgi.doe.gov/Phatr2/Phatr2.home.html (v2) |
| https://www.chlamycollection.org/chloro/genome.html | http://protists.ensembl.org/Phaeodactylum_tricornutum/Info/Index (v3) | |
| Ressources génomiques et bases de données centralisées | https://www.chlamylibrary.org/ | https://www.diatomicsbase.bio.ens.psl.eu/ |
| http://genomes.mcdb.ucla.edu/index.html | ASAFinder: https://rocaplab.ocean.washington.edu/tools/asafind/ | |
| https://www.chlamycollection.org/ | ||
| http://chlamystation.free.fr/ | HECTAR: http://www.sb-roscoff.fr/hectar/ | |
| Méthodes biophysiques | Fluorescence de la chlorophylle (temps de vie, basse température), Spectroscopie d’absorption (changements redox, Electrochromisme), échanges gazeux (C14, électrode O2, Spectrométrie de masse sur membrane) | Fluorescence de la chlorophylle (temps de vie, basse température), Spectroscopie d’absorption (changements redox, Electrochromisme), échanges gazeux (C14, électrode O2, Spectrométrie de masse sur membrane) |
| Méthodes de transformation nucléaire | Biolistique | Biolistique |
| Electroporation | Conjugaison | |
| Billes de verre | Electroporation | |
| Méthodes de transformation du plaste | Biolistique | Biolistique |
| Billes de verre | Electroporation | |
| Méthodes transf. mitochondriale | Biolistique | — |
| Technologie de clonage moléculaire | Golden Gate (MoClo) | Gateway |
| Golden Gate | ||
| Extinction de gènes | Répétition inversée et fragments antisens | Répétition inversée et fragments antisens |
| Méthodes d’édition génomique | CRISPR/Cas9, CRISPR/Cas9 fourni en RNP, ZFNs | TALEN, Meganucléases, CRISPR/Cas9, CRISPR/Cas9 fourni en RNP |
| Reproduction sexuée | Heterothallic, isogamous | Not mastered in laboratory |
| Analyse génétique | Tetrad analysis |
Plusieurs diatomées marines ont été récemment choisies comme nouveaux organismes modèles pour l’étude de la biologie et de la diversité du phytoplancton marin [8]. En raison de sa croissance rapide, de son petit génome et de la richesse des ressources génétiques associées, Phaeodactylum tricornutum est actuellement la diatomée la plus utilisée pour les recherches allant de la biologie moléculaire et cellulaire à l’écophysiologie et aux mécanismes évolutifs (Tableau 1). De nombreuses microalgues dont les génomes ont été séquencés, appartenant à différents clades, émergent comme des modèles complémentaires pour répondre à des questions biologiques et écologiques spécifiques ou relever des défis biotechnologiques [4, 19].
Nous nous concentrerons ici sur les processus gouvernés par la lumière (photosynthèse, photoperception et biogenèse des plastes) et illustrerons comment ces approches intégrées d’écophysiologie, génomique et génétique offrent de nouvelles possibilités pour explorer la biologie et la diversité des microalgues issues d’endosymbioses primaires et secondaires.
2. Biodiversité fonctionnelle de la photosynthèse chez les microalgues
La première étape de la photosynthèse oxygénique est la capture des photons par les complexes protéines–pigments (Light Harvesting Complexes ou LHC) insérés dans les thylakoïdes. Ces LHC s’associent avec les photosystèmes I et II (PSI et PSII), sièges des réactions photochimiques, pour former des supercomplexes. PSI et PSII fonctionnent en série au sein d’une chaîne photosynthétique de transfert d’électrons qui oxyde l’eau en O2 et fournit de l’ATP et du pouvoir réducteur au cycle de Calvin–Benson–Bassham de fixation du carbone. Les complexes photosynthétiques, les transporteurs solubles d’électrons et les enzymes du cycle de Calvin–Benson–Bassham sont pratiquement identiques chez les microalgues et les plantes [1], ce qui contraste avec la grande diversité des stratégies de collecte de la lumière en termes de pigments, de complexes protéines–pigments et de mécanismes de photoprotection. En effet, l’étude structurale, en particulier par cryomicroscopie électronique, des antennes collectrices des algues révèle une variabilité surprenante des protéines, de leurs liaisons avec les pigments et de leurs interactions pour former des supercomplexes. Le nombre, la nature, la position et l’orientation relative des chromophores ont été optimisés pour l’éclairement variable, mais généralement faible, en milieu aquatique. En faible luminosité, l’énergie collectée est très efficacement transmise aux centres photochimiques. En cas de forte illumination, des modifications de la nature, de la position ou de l’orientation des pigments diminuent réversiblement cette efficacité en favorisant, au détriment de la photochimie, la dissipation de l’énergie lumineuse sous forme de chaleur et l’extinction non photochimique (NPQ) de la fluorescence de la chlorophylle. Ce NPQ a une importance écologique critique: une vaste étude sur quatre bassins océaniques a montré que 60% de la lumière absorbée par les microalgues marines est convertie en chaleur [20].
Nous abordons tout juste la biodiversité photosynthétique des microalgues. La photoprotection (médiée par le NPQ) illustre parfaitement comment les techniques courantes (spectroscopie, génétique et biologie structurale), combinées aux progrès de la génomique, permettent d’étudier la biodiversité de ce mécanisme crucial [21]. Un schéma commun apparaît, impliquant une sous-unité du PSII et un cycle des xanthophylles (la dé-époxydation réversible d’un caroténoïde) qui, ensemble, permettent de basculer réversiblement d’un mode de collecte efficace de la lumière vers un mode de photoprotection par dissipation de chaleur.
Chez les plantes supérieures, ceci repose sur le cycle Violaxanthine–Antheraxanthine–Zéaxanthine (Vx/Ax/Zx) des xanthophylles, et sur des changements conformationnels de la sous-unité PSBS du PSII, un senseur du pH luménal, via ses résidus protonables [22]. Chez Chlamydomonas, LHCSR3, une protéine différente, répond également au pH et lie des pigments, dont la zéaxanthine, même si le rôle de cette dernière n’est toujours pas élucidé [23]. Parmi les microalgues issues d’endosymbiose secondaire, le NPQ est principalement étudié chez les diatomées, en particulier P. tricornutum [24]. À la différence des organismes de la lignée verte, leur cycle xanthophylle repose sur la Diadinoxanthine et la Diatoxanthine (Dd/Dt). Son mécanisme a été récemment disséqué grâce à des mutants obtenus par interférence ARN et par édition du génome : la protéine LHCX est impliquée dans le NPQ [25] et strictement nécessaire à ce dernier [26]. Son rôle est probablement différent de celui de PSBS ou LHCSR, car, si elle lie des xantophylles [56], elle semble insensible au pH [28, 56]. Les variations de l’expression de LHCX contribuent à la plasticité phénotypique des diatomées, comme en témoigne la comparaison de plusieurs écotypes de P. tricornutum montrant différents niveaux de NPQ [25]. Ces travaux pionniers sur LHCX ont été essentiels pour notre connaissance de l’adaptation des microalgues à des éclairements variables. En effet, des gènes de type LHCX sont présents chez les Haptophytes et Dinophytes [29], deux groupes qui utilisent le cycle Dd/Dt comme les diatomées. D’autres microalgues, les Chrysophytes, Eustigmatophytes, Raphidophytes et l’Alvéolaire Chromera velia utilisent des protéines de type LHCX mais le cycle Vx/Ax/Zx. Les analyses phylogénétiques montrent que les gènes codant les enzymes du cycle des xanthophylles comme ceux codant LHCSR ou LHCX dérivent d’algues vertes, y compris chez les algues dont le plaste dérive d’une algue rouge, mais l’histoire évolutive de ces dernières reste confuse.
La Figure 2, qui définit le NPQ des microalgues par la combinaison d’une sous-unité du PSII (PSBS, LHCSR ou LHCX) et d’un cycle des xanthophylles (Vx/Ax/Zx ou Dd/Dt), est déjà incomplète : les Cryptophytes ont un NPQ actif, mais ne possèdent ni cycle des xanthophylles, ni protéine PSBS, LHCSR ou LHCX [30, 31]. D’autres acteurs du NPQ restent donc à découvrir. De plus, la cinétique et les détails mécanistiques du NPQ sont très variés : le rôle dual des xanthophylles (extincteur direct ou modulateur indirect de l’énergétique des antennes collectrices) et de la protéine du PSII (liaison de pigment et/ou senseur de pH) génère un haut niveau de complexité que les données génomiques seules ne peuvent révéler. Il faut maintenant étudier en détail le NPQ dans des organismes modèles des clades Haptophytes, Dinophytes et Straménopiles non diatomées possédant des protéines LHCX et le cycle Vx/Ax/Zx.
Représentation schématique du mécanisme de photoprotection chez la diatomée P. tricornutum. À gauche : lorsque l’absorption de lumière par l’antenne associée au PSII est faible (flèche jaune), la concentration en protons dans le lumen reste faible et les pigments xanthophylles sont principalement sous leur forme époxydée (diadinoxanthine, Dd). À droite : lorsque l’absorption de la lumière augmente sous une forte lumière, la concentration plus élevée de protons (pH plus faible) dans le lumen active la diadinoxanthine déépoxidase. La concentration en diatoxanthine (Dt) augmente, ce qui, avec la présence des protéines LHCX, entraîne une meilleure dissipation de la chaleur (flèche rouge). (B) Représentation schématique de l’arbre de vie des eucaryotes comme dans la Figure 1. Les cercles attachés à chaque groupe de microalgues représentent la combinaison des différents régulateurs du NPQ, de la sous-unité PSII (LHCX, PSBS ou LHCSR) et du cycle des xanthophylles (Vx/Ax/Zx : cercle vert ; Dd/Dt : cercle orange), décrits dans différentes lignées, d’après [24].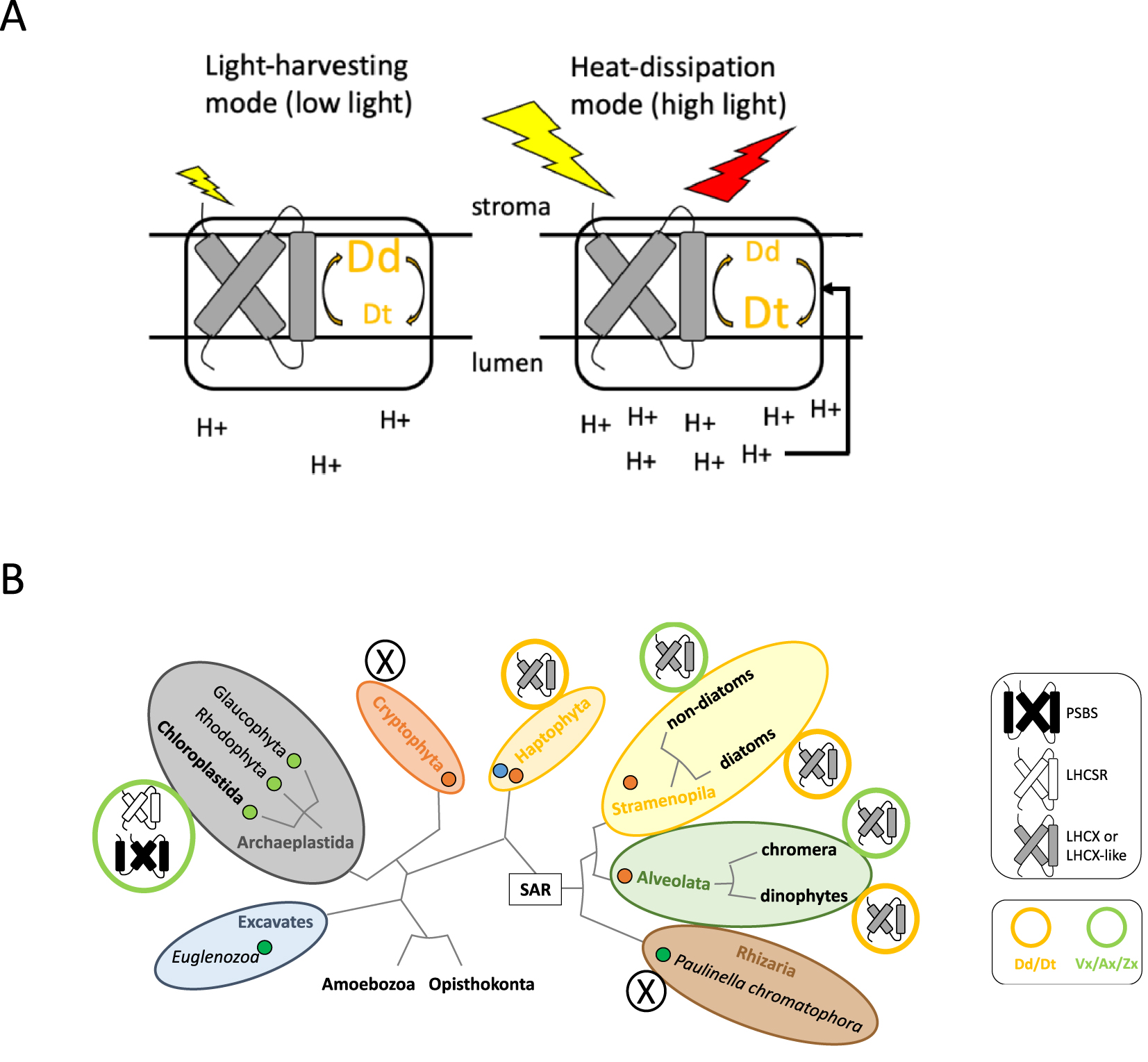
3. Perception de l’environnement lumineux chez les microalgues
La lumière est une source essentielle d’informations issues de l’environnement pour de nombreux organismes. Elle influence de nombreux aspects de leur développement, physiologie et métabolisme. Les signaux lumineux sont perçus par des photorécepteurs qui activent des voies de signalisation pour déclencher des réponses physiologiques [34]. De nombreuses séquences homologues à celles de photorécepteurs ont été identifiées dans les génomes d’algues [34, 35] et, plus récemment, dans des données de métagénomique océanique [33]. Certains de ces photorécepteurs sont similaires à ceux des plantes terrestres, mais d’autres, spécifiques de certaines espèces ou lignées, ou correspondant à des variants spectraux, ont également été découverts (Figure 3), et résultent probablement d’adaptations à la luminosité sous-marine. En effet, la lumière perçue par les organismes marins varie considérablement en intensité et en composition spectrale en raison des propriétés d’absorption de l’eau, qui sélectionne la lumière verte et bleue avec la profondeur [35]. Il n’est donc pas surprenant de trouver de nombreux récepteurs de lumière bleue dans les microalgues.
Principales classes de photorécepteurs identifiées dans les génomes de différents groupes de microalgues. La structure de ces photorécepteurs est représentée schématiquement. Les domaines colorés sont ceux impliqués dans la perception de la lumière selon les longueurs d’onde qu’ils absorbent classiquement; en blanc sont indiqués les domaines impliqués dans la transduction du signal. PHR : domaine apparenté aux photolyases; LOV: domaine Lumière-Oxygène-Voltage; STK: domaine Serine/Threonine kinase; OPM: module de transduction du signal, qui peut différer selon les groupes de microalgues, Rh: domaine rhodopsine; PAS : domaine Per-Arnt-Sim; GAF : domaine cGMP phosphodiesterase/adenylyl cyclase/FhlA; PHY : domaine phytochrome-spécifique. Les fonctions indiquées sont celles qui ont été décrites pour les microalgues [34, 35].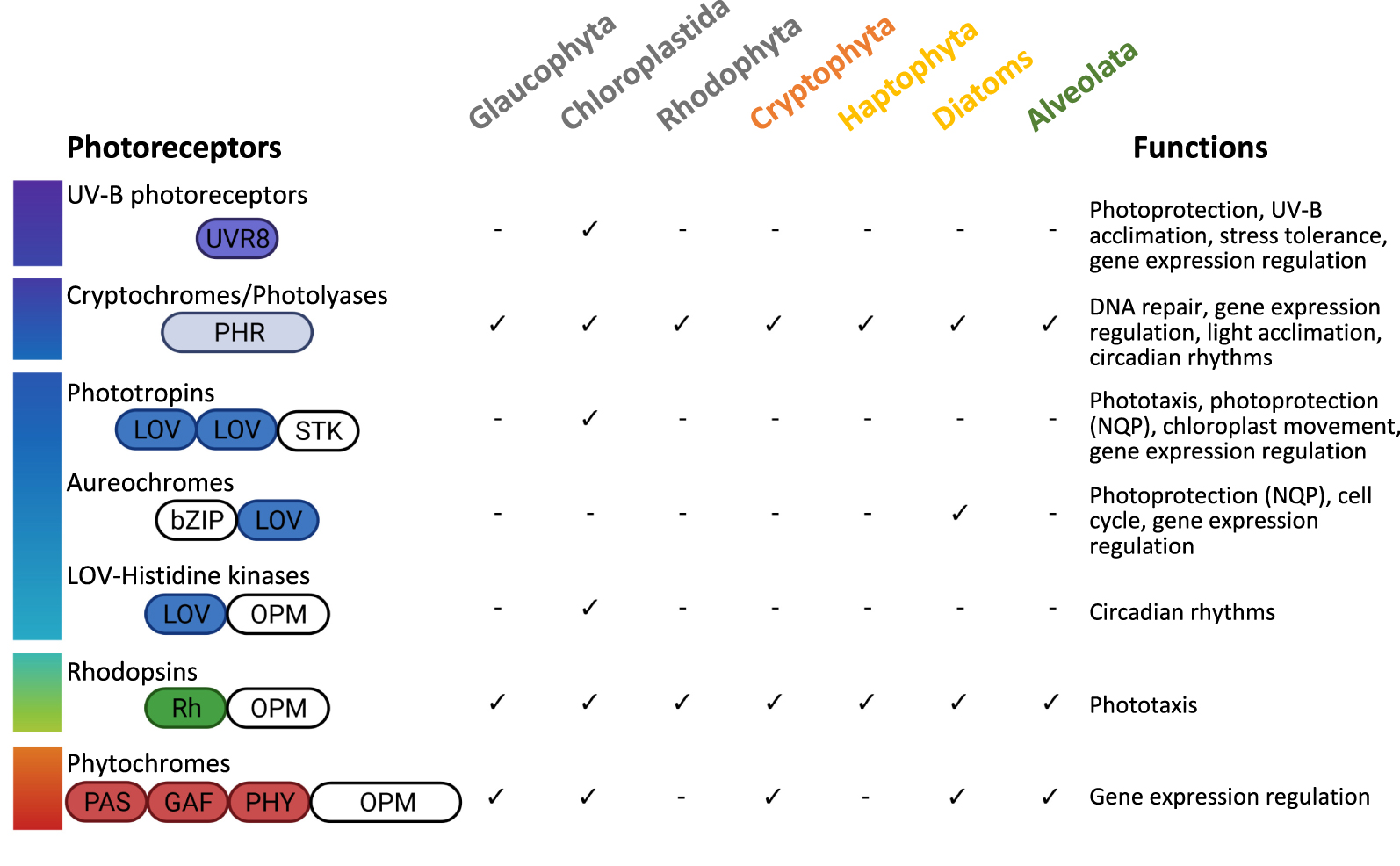
La famille des cryptochromes photolyases (CPF) comprend les ADN photolyases, des enzymes activées par la lumière bleue qui réparent les dommages à l’ADN induits par les UV, et les cryptochromes, fonctionnant comme photorécepteurs chez les plantes terrestres, et comme régulateurs transcriptionnels de l’horloge circadienne chez les animaux. Chez P. tricornutum, la protéine CPF1 présente les deux activités de réparation de l’ADN et de photoperception [36] et pourrait ainsi représenter un intermédiaire dans l’évolution de l’activité catalytique de photolyases à photorécepteurs. Depuis cette découverte, des CPF1 ayant une activité de réparation de l’ADN et diverses propriétés de détection de la lumière ont été trouvées dans d’autres microalgues, dont des algues vertes telles que Chlamydomonas et Ostreococcus tauri [37]. Ces protéines présentant une double fonction pourraient donc être particulièrement adaptées aux environnements aquatiques dynamiques. De nouvelles classes de CPF ont récemment été identifiées à l’aide d’une nouvelle méthode de calcul qui extrait les caractéristiques fonctionnelles de grands ensembles de données de séquence protéique [38]. La fonction exacte de ces nouveaux CPF pourra être testée expérimentalement chez des espèces modèles de microalgues pour confirmer qu’il s’agit bien de protéines photosensibles présentant de nouvelles caractéristiques.
Les diatomées contiennent également des auréochromes qui sont des photorécepteurs spécifiques des straménopiles [39]. Grâce à un domaine LOV (light-oxygen-voltage) permettant la perception de la lumière bleue et un domaine bZIP (basic leucine zipper) de liaison à l’ADN, les auréochromes agissent comme des facteurs de transcription régulés par la lumière et contrôlent des processus biologiques clés tels que l’expression génique, le cycle cellulaire et le NPQ [39, 40]. Il est intéressant de noter que les algues vertes telles que Chlamydomonas, utilisent un autre photorécepteur de lumière bleue avec des domaines LOV, la phototropine, pour réguler l’expression génique [41] et le NPQ [42]. Cela suggère que différents photorécepteurs pour la lumière bleue ont évolué indépendamment dans différentes lignées pour optimiser la photosynthèse, probablement en raison de l’abondance de cette longueur d’onde dans toutes les zones photiques (Figure 3). En revanche, la présence de phytochromes, photorécepteurs absorbant les lumières rouge et rouge lointain et induisant une signalisation par le rouge lointain chez les diatomées, est plus surprenante, étant donné la forte atténuation de ces longueurs d’onde dans la colonne d’eau [43]. Nous cherchons actuellement à savoir si ces phytochromes de diatomées (DPH) jouent un rôle de détecteur de profondeur ou de proximité d’autres organismes phototrophes, en réalisant la caractérisation fonctionnelle de DPH provenant de diverses espèces de diatomées et en étudiant la distribution des gènes DPH dans les données de métagénomiques collectées dans tous les océans du globe.
Les photorécepteurs jouent également un rôle clé dans l’entraînement de l’horloge circadienne, un système endogène qui contrôle dans la plupart des organismes le moment et la synchronisation des processus biologiques avec les cycles périodiques de lumière et d’obscurité [44]. Les mécanismes moléculaires de l’horloge ont été largement caractérisés chez les organismes terrestres mais seulement chez quelques algues de la lignée verte, comme C. reinhardtii ou O. tauri [45]. Les composants de l’horloge circadienne sont encore largement inconnus chez de nombreuses espèces du phytoplancton, en partie parce que les génomes de ces organismes manquent d’homologues clairs de ceux identifiés chez d’autres organismes. En intégrant des approches de génomique fonctionnelle et des analyses in vivo des processus physiologiques dépendant de la lumière, l’existence d’une horloge circadienne fonctionnelle a été récemment démontrée chez P. tricornutum [46]. Le facteur de transcription bHLH-PAS, nommé RITMO1, a été le premier composant de l’horloge circadienne identifié chez les diatomées. Des homologues de RITMO1 sont présents dans de nombreux génomes de diatomées et d’autres algues marines, ce qui pourrait suggérer une fonction similaire chez ces phototrophes. RITMO1 représente aujourd’hui un point d’entrée pour l’identification des autres composants de l’horloge circadienne et ouvre la voie à une meilleure compréhension des rythmes marins, de leur évolution et de leur importance écologique.
4. Diversification et biogenèse des plastes
Comprendre la fonction photosynthétique impose de connaitre les mécanismes de biogenèse et réparation des plastes, qui n’ont été étudiés que dans la lignée verte et restent une terra incognita dans les autres clades, y compris les diatomées ou leur ancêtre rhodophyte.
Une caractéristique commune à tous les plastes est le petit nombre de gènes retenus dans leur génome, principalement nécessaires à la photosynthèse ou à leur expression génétique. Cela résulte du transfert important, au cours de l’évolution, des gènes de l’endosymbionte ancestral vers le noyau de l’hôte. La grande majorité du protéome plastidial est maintenant codée dans le noyau, traduite dans le cytosol puis importée dans le plaste. Ainsi, la biogenèse de l’appareil photosynthétique nécessite le dialogue des génomes plastidial et nucléaire, ainsi qu’une machinerie d’import spécifique.
Dans la lignée verte, la plupart des protéines du plaste codées par le noyau sont importées grâce à un peptide d’adressage amphipathique (TP) qui interagit avec la membrane externe de l’enveloppe, et est ensuite importé par un translocon TIC/TOC dans le plaste, où il est éliminé par des peptidases spécifiques. Les TP partagent leurs propriétés amphipathiques avec certains peptides antimicrobiens, ce qui, plutôt que refléter une évolution convergente, serait l’héritage d’événements endosymbiotiques précoces : les TP dériveraient de peptides antimicrobiens (AMP), tandis que le translocon TIC résulterait de la cooptation d’un mécanisme de défense de l’endosymbionte contre l’immunité innée de l’hôte [47, 48]. De fait, chez Chlamydomonas, des séquences antimicrobiennes fusionnées à un gène rapporteur permettent d’acheminer son produit vers les organites [49]. Ce scénario a pu contribuer à la rétention de la proie cyanobactérienne par l’hôte en permettant le transfert endosymbiotique de gènes dont les produits pouvaient ensuite être réimportés dans le plaste.
En conséquence, les complexes photosynthétiques comme le plastoribosome ont une double origine génétique. Comment les microalgues coordonnent-elles la production des différentes sous-unités d’un complexe dans la stœchiométrie nécessaire à son assemblage fonctionnel ? Chez les Viridiplantae, un premier niveau de coordination repose sur des facteurs agissant en trans codés dans le noyau, qui contrôlent étroitement et d’une manière gène-spécifique, toutes les étapes post-transcriptionnelles de l’expression des ARNm plastidiaux. Pour la plupart, ils appartiennent à la superfamille des protéines 𝛼-solénoïdes : ce sont soit des protéines à répétitions pentatricopeptides (PPR), définies par la répétition en tandem d’un motif dégénéré de 35 résidus, abondantes dans les plantes terrestres, soit des protéines à répétitions octotricopeptides (OPR) avec des répétitions de 38 résidus, nombreuses chez les algues vertes mais absentes dans la plupart des algues rouges et rares chez les plantes supérieures. La spécificité de ces deux motifs pour un ARN donné résulte de l’interaction de résidus à des positions définies au sein de la répétition avec un nucléotide spécifique du transcrit. Un code régissant ces interactions acides aminés–nucléotides a été établi pour les protéines PPR [50], tandis que le « code OPR » est en court d’établissement dans notre laboratoire.
Dans la lignée verte, un deuxième niveau de coordination réside dans l’accumulation concertée des sous-unités d’un complexe photosynthétique. Contrairement à la situation chez les cyanobactéries, les mutants n’exprimant pas une sous-unité du cœur d’un complexe perdent pleïotropiquement toutes ses autres sous-unités, évitant ainsi l’accumulation potentiellement toxique de polypeptides non assemblés [51]. Ceci résulte de deux mécanismes complémentaires : (i) certaines sous-unités sont rapidement dégradées par les protéases chloroplastiques lorsqu’elles ne peuvent pas s’assembler [52], (ii) d’autres sous-unités, toutes codées par le génome chloroplastique, sont contrôlées par épistasie de synthèse (CES) : elles ne sont pas dégradées, mais leur vitesse de synthèse est fortement réduite en absence d’assemblage. Ce dernier mécanisme de régulation, identifié pour la première fois chez Chlamydomonas par notre laboratoire, joue un rôle important dans la biogenèse du chloroplaste puisque les principaux complexes photosynthétiques contiennent au moins une et souvent plusieurs sous-unités CES [51]. En couplant la traduction de certains gènes chloroplastiques à l’assemblage de leur produit, le CES permet d’ajuster finement la production d’un polypeptide à sa demande pour la biogenèse d’un complexe photosynthétique. Ce processus de régulation n’existe pas chez les cyanobactéries, où l’absence d’assemblage d’un complexe photosynthétique n’impacte pas l’expression de ses sous-unités constitutives.
Les microalgues issues d’endosymbioses secondaires sont confrontées aux mêmes défis ainsi qu’à d’autres. Parce qu’une algue eucaryote et non une cyanobactérie a été capturée, le système d’import diffère: les protéines codées dans le noyau mais localisées dans les plastes doivent traverser quatre membranes pour atteindre le stroma, et non deux comme chez les Archaeplastidia. Portant une préséquence bipartite comprenant un peptide signal suivi d’un peptide d’adressage au plaste, elles sont d’abord importées dans le réticulum endoplasmique (RE), avant d’être transférées dans le compartiment périplastidial par un translocon dérivé de la machinerie de dégradation du RE. Leur import ultérieur dans le plaste reste à caractériser.
Chez les diatomées, le transfert de gènes vers le noyau, largement commencé chez l’algue rouge ancestrale, s’est poursuivi après l’endosymbiose secondaire. Les plastes des diatomées contiennent en effet moins de gènes aujourd’hui que ceux des algues rouges, mais plus toutefois que ceux des algues vertes. Ainsi, les diatomées doivent également coordonner la production des sous-unités codées dans le noyau et le plaste. Leurs génomes codent pour des protéines PPR et OPR, dont certaines sont prédites adressées aux organites. Cependant, nous ignorons complètement, comme chez toutes les algues en dehors de la lignée verte, si et comment le noyau contrôle l’expression des gènes du plaste. De même, l’accumulation des sous-unités d’un complexe photosynthétique est-elle concertée chez les algues rouges et les diatomées et y a-t-il également un contrôle de la traduction de certains gènes plastidiaux par l’assemblage de leur produit ? En outre, les plastes de diatomées codent plusieurs cadres de lecture ouverts conservés soit dans les seules diatomées, soit dans les algues rouges et les endosymbiontes secondaires qui en dérivent, mais absents de la lignée verte, et dont le rôle dans le plaste reste à étudier. Les caractéristiques de la biogenèse des plastes communes aux diatomées (ou autres endosymbiontes secondaires) et aux Viridiplantae nous renseigneront sur les contraintes universelles de construction d’un appareil photosynthétique au sein d’un organite. Les nouveaux mécanismes régulant le processus photosynthétique que nous découvrirons lors de l’étude des plastes de diatomées nous aideront à mieux comprendre les raisons de leur succès écologique dans tous les milieux aquatiques.
5. Conclusions et perspectives
Les exemples d’études intégrées des processus photobiologiques décrits ici mettent en évidence des découvertes passionnantes mais aussi d’importantes lacunes dans nos connaissances, qui doivent être comblées pour véritablement comprendre la biologie de ces microalgues. L’étude de la biodiversité fonctionnelle des microalgues peut sembler une tâche sans fin, mais peut dorénavant être abordée avec confiance à condition d’un dialogue continu et itératif entre études mécanistiques chez des espèces modèles sélectionnées et études in situ. Les analyses comparatives de génomique fonctionnelle pour diverses espèces modèles faciliteront la découverte de régulateurs biologiques pertinents encore cachés dans les génomes du phytoplancton. De nouvelles fonctions pourront également être prédites en explorant la diversité génétique avec des approches de biologie des écosystèmes, par exemple en associant les séquences environnementales au niveau de l’espèce ou du genre et leur expression différentielle dans différents environnements [11]. La phylogénomique peut aider à simplifier un ensemble de centaines ou de milliers de gènes à quelques groupes fonctionnels, qui détermineront à leur tour les organismes modèles à étudier. Les résultats obtenus en laboratoire peuvent conduire à de nouvelles explorations in situ afin de valider ces résultats dans un contexte écologique et vice versa. Pour aborder la question de l’hétérogénéité des populations cellulaires, il parait important d’intégrer des approches de rapporteurs cellulaires étudiés par imagerie cellulaire, cytométrie en flux et microfluidique, ainsi que des technologies « omique » en cellule unique. Ces approches nous aideront à identifier des empreintes moléculaires caractéristiques de l’état physiologique des microalgues, ainsi que de nouveaux biomarqueurs qui, à leur tour, faciliteront l’étude de la physiologie des microalgues dans des environnements complexes, alimentant une modélisation plus robuste des écosystèmes.
Il est également essentiel de développer de nouvelles espèces modèles de microalgues appartenant à des lignées encore récalcitrantes aux investigations génétiques, comme les dinophytes et haptophytes. Le choix de ces nouveaux modèles expérimentaux devrait idéalement résulter d’un dialogue ouvert entre communautés scientifiques : de nouvelles estimations de la distribution et de la diversité des microalgues aideront à identifier des espèces écologiquement majeures. Celles-ci devraient également répondre à plusieurs critères pratiques tels qu’une croissance rapide, la maîtrise du cycle de vie en laboratoire, la possibilité de manipulations génétiques et la génération de données suffisantes sur leur génome et leur physiologie. L’analyse d’accessions naturelles échantillonnées sur une large échelle géographique nous aidera à mettre en évidence les corrélations entre génotype, phénotype, et succès écologique [25]. Les études pangénomiques se concentreront sur cette diversité infraspécifique.
Le choix de nouvelles espèces modèles sera également guidé par l’intérêt porté à des processus spécifiques. Les études de mutants, dont beaucoup ont été isolés chez C. reinhardtii dans notre laboratoire (http://chlamystation.free.fr/), ont constamment orienté les recherches sur la photosynthèse. Les diatomées les plus étudiées sont des phototrophes obligatoires, pour lesquels les mutants photosynthétiques sont à ce stade impossibles à générer. La diatomée Cyclotella cryptica apparaît comme un nouveau modèle prometteur pour l’étude de la photosynthèse des diatomées car elle se développe à la fois en hétérotrophie (en présence de glucose) et en autotrophie (sans autre source d’énergie que la lumière). Des ressources génomiques et génétiques pour cette microalgue sont déjà disponibles [53]. Notre laboratoire développe actuellement des techniques pour établir cette espèce comme modèle génétique de la photosynthèse.
Les activités humaines continuent de modifier les océans par la pollution, l’acidification, la désoxygénation et le réchauffement (Ref. [54], Sixième rapport d’évaluation de la CIPV), et nous ne pouvons pas estimer l’impact de ces changements sans comprendre leurs conséquences sur des producteurs primaires aussi diversifiés que les microalgues. L’amélioration de l’efficacité de la photosynthèse est également un défi majeur pour, d’une part, faire face à des besoins alimentaires et énergétiques toujours croissants, et d’autre part, développer des stratégies visant à atténuer les conséquences du changement climatique. Ces objectifs ambitieux ne peuvent être atteints qu’en étudiant précisément la manière dont les phototrophes se sont adaptés à leur environnement et les stratégies d’acclimatation qu’ils emploient dans des conditions variables. Les processus pilotés par la lumière sont au centre de ces recherches car les méthodes classiquement utilisées sont applicables à différents groupes d’algues, et la lumière peut être délivrée de manière précisément contrôlée pour déclencher les processus. La photosynthèse est ainsi l’une des très rares fonctions pouvant être mesurées in situ et de façon non-invasive grâce à la sensibilité des techniques de fluorescence et spectroscopie La photoperception peut également être abordée à différents niveaux, de la molécule à l’environnement. L’étude combinée de la photosynthèse et de la photoperception présente ainsi un énorme potentiel pour élargir notre compréhension des mécanismes par lesquels la lumière est exploitée pour soutenir la vie.
Conflit d’intérêt
Les auteurs n’ont aucun conflit d’intérêt à déclarer.
Remerciements
Nous tenons à remercier Francis-André Wollman et Pierre Joliot pour leur soutien constant aux recherches de notre laboratoire, et pour leur enthousiasme et leur regard critique et constructif notamment au moment où nous entrons dans cette nouvelle phase de focalisation sur des enjeux écologiques majeurs. Nous tenons à remercier tous les membres non-permanents du laboratoire, les techniciens, les étudiants et les post-doctorants qui rendent possible le développement de nos recherches. Nous remercions la Fondation Bettencourt-Schueller (Coups d’élan pour la recherche française-2018) et le programme « Initiative d’Excellence » (Bourse « DYNAMO », ANR-11-LABX-0011-01) pour le soutien de nos recherches sur la biodiversité fonctionnelle des microalgues.




 CC-BY 4.0
CC-BY 4.0