Abridged English version
Gene targeting permits precise and controlled modifications of a given locus. It is commonly used to achieve gene disruption, replacement or modification. This technique is based on the natural ability of eukaryotic cells to integrate an exogenous DNA molecule into their genome by homologous recombination. The efficiency of random integration and homologous recombination varies from species to species.
In this review, we will focus on the major applications of gene targeting, on the limitations of current strategies and the new strategies developed to overcome those limitations.
Gene targeting is a powerful tool with many applications in public-health research. It can be used to inactivate a gene encoding a protein of unknown function, resulting in the production of mutant cells lines for the functional characterisation of orphan genes. The use of gene targeting in embryonic stem (ES) cells results in the production of engineered ES cells that can be introduced into a blastocyst, in which they contribute to embryonic development, leading to the production of genetically modified animals. Such animals may be used for the characterisation of genes encoding proteins involved in processes that cannot be investigated in cell lines, typically genes displaying a spatio-temporal pattern of expression regulation during embryonic development. This technique can also be used to produce model animals bearing mutations similar to those involved in human genetic diseases. Model animals are useful not only for studies of diseases, but also for the development of therapeutic strategies. Beside the ethical questions that are still raised, the in-vitro differentiation of ES cells can also be used to produce genetically modified tissues for human gene therapy. It results in perfect correction of the mutation causing the disease, without disturbing the rest of the genome.
However, if they are to be widely used as a therapeutic strategy, and even as a laboratory tool, gene-targeting protocols must be greatly improved. The exogenous therapeutic nucleic acid fragment, also called the targeting vector, must be delivered very efficiently to the nucleus of targeted cells. Once this has been accomplished, the targeting vector must recombine with its target in a precise and efficient homologous reaction. This reaction requires the whole set of recombination proteins and the DNA mismatch repair system (MMR). Unfortunately, such reactions occur at a frequency of roughly one event per 105 to 108 treated cells. This reflects not only the low general capacity of mammalian cells to perform homologous recombination, but also the 1000 times higher frequency of random integration in such cells. It is still unclear what determines whether the targeting vector is directed to homologous recombination or to random integration.
The efficiency of a gene-targeting experiment depends on the properties of the target and vector and on the recombination activities of the targeted cells. The location of the genomic target and its transcriptional activity are not clearly correlated with the efficiency of the homologous recombination. However, the target primary sequence may affect the efficiency of the reaction. Indeed, some repeated sequences seem to disturb the process of homologous recombination by affecting the formation, stabilisation and resolution of intermediate joint molecules formed during the recombination reaction. Classical targeting vectors, such as replacement vectors, are several kilobases long DNA molecules that are homologous to the target on both sides and bear a selectable maker between the two homologous ‘arms’. It has been shown that the length and the level of sequence identity between the vector and the genomic target are of major importance for efficient gene replacement. As the region of sequence similarity and the quality of the match between the two sequences increases, so the efficiency of targeting is improved.
The low efficiency of current protocols for gene-targeting experiments makes it necessary to carry out selection steps to isolate the targeted cells. To bypass the long and costly selection steps and the design constraints (requirement for selectable markers) that they impose, ways to increase the frequency of homologous integration and/or to inhibit random integration of the targeting vector must be found. The new strategies designed to increase the efficiency of gene targeting are of three main types: alterations to the genomic target, the use of new kinds of targeting vector and modification of cellular recombination activities.
As double-strand breaks are known to initiate homologous recombination, strategies facilitating the specific cutting of the targeted locus provide a means of stimulating the efficiency of gene targeting. Inspired from natural rare-cutting endonucleases such as I-SceI from S. cerevisiae, chimeric artificial meganucleases are beginning to be developed. These bi-functional molecules consist of an endonuclease and a zinc-finger DNA-binding domain, able to bind only the targeted locus. Such systems have been shown to be efficient with an episomal substrate in Xenopus oocyte cells. Triplex-forming oligonucleotides (TFO), which bind polypurine–polypyrimine duplexes in a sequence-specific way, can also be used to target the genomic DNA. The TFO may be coupled to a DNA-damaging agent such as psoralen. The damage caused by UVA activation of psoralen, and even by the TFO itself, stimulates recombination in the vicinity of the genomic target of the TFO. The TFO can also be coupled to a targeting vector, leading to the rapid formation of the early intermediates of the recombination reaction and their stabilisation, thereby increasing the efficiency of the targeted recombination reaction.
The use of small synthetic oligonucleotide vectors for gene targeting has also been widely developed. The single-stranded nature of these molecules is known to have a recombinogenic effect. Moreover, the possibility of producing oligonucleotides from modified bases has raised hopes of promising new types of targeting vector such as chimeric RNA/DNA oligonucleotides (RDO) or modified DNA oligonucleotides (ODN). These molecules are much more stable in the cell than the classical single-stranded DNA molecule. However, experiments involving these new types of vector suffer from a lack of reproducibility, due to currently unknown limiting factors.
A large number of studies have focused on the effects on gene-targeting efficiency of the overproduction or inhibition of the production of proteins involved in homologous recombination. It has been shown in human cells that overproduction of the central recombination proteins, HsRad51 and its bacterial homologue RecA, stimulates homologous recombination between a targeting vector and the genomic target. Modulation of the level of production of several other genes, such as RAD54, and the RAD51 paralogues, BRCA1, BRCA2, p53 and BLM remains to be investigated. Inactivation of the proteins known to be active in the NHEJ pathway during recombinational repair has no effect on gene-targeting efficiency. This suggests that random integration events depend on activities that are currently unknown and require investigation.
1 Introduction
Les expériences de ciblage de gène, également nommées expériences de mutagenèse dirigée, permettent de modifier une séquence d'ADN spécifique à son locus génomique de manière précise et contrôlée. Ces manipulations peuvent induire une perte ou un gain de fonction. De manière plus précise, elles peuvent conduire à l'invalidation d'un gène par sa délétion complète ou par la création de mutations plus subtiles, au remplacement du gène par un autre gène ou par une séquence quelconque d'ADN, ou encore à la correction d'une mutation. Il a été mis en évidence que les levures et les cellules de mammifères ayant incorporé des molécules d'ADN exogènes sont capables d'intégrer ces fragments d'ADN au sein de leur génome par recombinaison homologue [1,2]. Ce mécanisme moléculaire permet l'échange d'information génétique entre une séquence chromosomique endogène, la cible à modifier, et une séquence exogène portant une région homologue à la cible. La séquence exogène est choisie et introduite dans la cellule par l'expérimentateur. Comparativement, les événements de recombinaison homologue sont plus rares dans les cellules mammifères que dans les levures, et l'intégration aléatoire de séquences exogènes dans le génome des cellules mammifères est beaucoup plus fréquente que l'événement de recombinaison homologue.
Le but de cette revue est triple, il est de montrer l'intérêt de réaliser des expériences de ciblage de gène, de montrer les limites de cette technologie, et de dresser un bilan des principales stratégies utilisées.
2 Les applications des techniques de ciblage de gène
Au cours des deux dernières décennies, les techniques visant à modifier les génomes de mammifères ont énormément progressé. Ces techniques présentent un intérêt majeur pour les industries pharmaceutiques et agroalimentaires, ainsi que pour de nombreux domaines de la recherche en biologie fondamentale et appliquée à la médecine. En matière de santé publique, la possibilité de modifier précisément un génome permet d'étudier la fonction et la régulation de gènes associés à certaines pathologies génétiques, de créer des organismes modèles et de mettre au point les thérapies géniques.
2.1 Production de lignées cellulaires mutantes et d'organismes modèles
Un très grand nombre de lignées cellulaires mutantes et d'organismes modèles ont été créés par génie génétique depuis une quinzaine d'années. La fin des programmes de séquençage des génomes humains et murins devrait être associée à un essor encore plus considérable de la génomique fonctionnelle. Le recours aux techniques de ciblage de gènes va permettre d'inactiver totalement ou partiellement la fonction de protéines inconnues, afin de déterminer leur rôle. Cependant, l'étude fonctionnelle d'un gène menée dans des lignées cellulaires en culture ne prend pas en compte la complexité des organismes vivants pluricellulaires. Typiquement, la fonction des gènes dont l'expression est régulée spatio-temporellement au cours du développement embryonnaire est impossible à étudier de manière physiologique dans des lignées de cellules en culture. En appliquant l'ensemble des techniques de génétique moléculaire modernes, il est possible de produire des mutants murins après avoir employé les techniques de mutagenèse dirigée sur des cellules embryonnaires souches (ES). Des lignées de cellules ES ont été établies à partir de rongeurs comme la souris ou le rat, et à partir de primates comme les singes Rhésus ou l'homme [3]. Les cellules de ces lignées maintiennent leur totipotence en culture. Les cellules ES murines peuvent être génétiquement modifiées in vitro, puis réintroduites dans un blastocyste où elles vont contribuer au développement de l'embryon. Ces expériences conduisent à la production d'animaux génétiquement modifiés. Elles permettent en particulier l'obtention d'animaux modèles présentant des mutations similaires à celles connues dans des pathologies génétiques humaines. Ces animaux sont d'une utilité majeure pour l'étude du développement des pathologies, mais aussi pour la mise au point de thérapies.
2.2 Thérapies géniques et cellulaires
Un grand nombre de syndromes et de pathologies humaines, dont de nombreux cancers, résultent de mutations génétiques héréditaires, acquises ou sporadiques. Ces mutations conduisent à la synthèse de protéines défectueuses ou à une dérégulation de leur expression. Elles peuvent provoquer des perturbations des mécanismes moléculaires et cellulaires fondamentaux. Les thérapies classiquement proposées visent à traiter les symptômes engendrés par les défauts génétiques et non leur cause. La thérapie génique cible l'origine de la pathologie. Elle consiste à apporter une copie fonctionnelle du gène muté ou d'un autre gène codant pour une activité thérapeutique, ou encore à réparer la mutation qui est à la source du désordre physiologique.
La complémentation de la mutation par l'apport d'un gène fonctionnel est la stratégie de thérapie génique la plus développée à l'heure actuelle. Elle consiste à introduire dans les cellules mutées un vecteur d'expression capable de conduire à la synthèse d'une « protéine-médicament » compensant le défaut génétique. L'expression du gène introduit est généralement transitoire, mais peut devenir continue si le vecteur d'expression s'intègre dans le génome de la cellule au niveau d'un locus favorisant sa transcription. Bien que cette stratégie présente des avantages de par sa simplicité apparente, elle présente un certain nombre d'inconvénients. Pour un bon contrôle de l'activité du gène thérapeutique, il est nécessaire d'introduire non seulement la partie codante du gène mais aussi les séquences régulant son expression, qui ne sont pas nécessairement caractérisées. Par ailleurs, en s'intégrant de manière aléatoire dans le génome, le vecteur peut perturber ou inactiver l'expression de gènes. Les conséquences peuvent être catastrophiques. Des rejets des cellules « soignées » peuvent également s'observer. Enfin, cette stratégie n'est envisageable que pour le traitement de défauts génétiques de nature récessive.
Une seconde stratégie, qui consiste à remplacer le gène muté par son allèle sauvage, ne présente aucun de ces inconvénients : en permettant la correction du défaut génétique, elle fournit une solution idéale pour la thérapie génique. Elle n'est ni vue, ni reconnue par l'hôte. Les techniques de ciblage de gène utilisées pour ce type d'application doivent permettre de délivrer des molécules d'acide nucléique correctrices jusqu'aux noyaux des cellules ciblées avec une grande efficacité. Une fois dans le noyau, les molécules délivrées doivent être capables de corriger la mutation de manière efficace et précise, sans engendrer d'autres réarrangements dans le génome.
3 Les techniques actuelles de ciblage : systèmes, mécanismes et facteurs limitants
Une expérience de ciblage de gène peut être découpée en trois étapes. La première étape consiste à délivrer au noyau de la cellule à modifier une molécule d'acide nucléique portant la modification à introduire, le vecteur de ciblage. Ce vecteur devra traverser la membrane cytoplasmique, le cytoplasme et la membrane nucléaire. Les différentes méthodes actuellement utilisées, ou en cours de développement, sont décrites plus en détail par Luo et Saltzman [4]. Suivant la lignée cellulaire et la nature de la molécule exogène, une même méthode aura une efficacité variable. Un protocole précis doit donc être mis au point pour chaque système. Cette mise au point expérimentale dépend du type cellulaire, du type de molécule à introduire, de la qualité des produits de culture cellulaire et de la dextérité de l'expérimentateur.
Au cours d'une deuxième étape, la molécule délivrée au noyau pourra être intégrée dans le génome après une réaction de recombinaison homologue avec sa cible. À ce jour, et ceci quel que soit le locus ou la lignée cellulaire étudiée, l'événement de recombinaison homologue a lieu à une fréquence faible (10−8 à 10−5). Cette faible fréquence est inhérente à l'activité de recombinaison des cellules de mammifères. Elle est certainement également associée aux techniques de ciblage de gène utilisées.
Des stratégies permettant la sélection des cellules recombinantes ont été développées [5–8]. Après cette étape de sélection et d'après la littérature, la plus haute fréquence de recombinaison homologue obtenue atteint 80 % [9]. Il est à noter que la moyenne est beaucoup plus basse, de l'ordre 0,3 à 1 % des cellules sélectionnées.
La sélection des cellules recombinantes est longue, coûteuse et traumatisante pour les cellules. Le développement de techniques permettant d'augmenter l'efficacité de la recombinaison homologue dans les cellules de mammifères permettrait de réduire, voire d'éviter, cette dernière étape. La fréquence d'un événement de recombinaison homologue ciblé dépend de trois facteurs : la nature la cible à modifier, la nature de la molécule exogène introduite et les activités de recombinaison de la cellule.
3.1 Influence de la nature de cible
Les expériences de ciblage de gène dans les cellules ES ne paraissent pas être réalisables avec une efficacité semblable à tous les loci génomiques. À la fin des années 1980 et au début des années 1990, les auteurs ont essayé de rechercher des hypothèses permettant d'expliquer ces différences. Une corrélation entre le niveau d'expression des gènes dans les cellules ES et l'efficacité de recombinaison homologue a par exemple été évoquée. Il a été montré que l'efficacité de ciblage du gène fortement exprimé hprt est 16 fois supérieure à celle du proto-oncogène int-2 faiblement exprimé [6]. Mais une autre étude a cependant montré une absence de corrélation entre le niveau d'expression d'un gène et sa fréquence de recombinaison avec un vecteur de ciblage [10]. La localisation de la cible dans le génome, indépendamment de son niveau d'expression, ne semble pas non plus être un facteur déterminant la fréquence des événements de recombinaison homologue [11]. Les variations de l'efficacité du ciblage observées d'un gène à l'autre pourraient être expliquées par une différence dans leur séquence primaire. Certaines séquences de l'ADN ont des propriétés particulières vis-à-vis des protéines de la recombinaison. Il a notamment été mis en évidence que la protéine RecA d'E. coli et son homologue chez les mammifères, Rad51, impliqués dans l'initiation des réactions de recombinaison, se fixent de manièrent préférentielle sur certaines séquences répétées de l'ADN puis inhibent la réaction d'échange de brin [12,13]. In vivo, les séquences répétées de type microsatellites inhibent la recombinaison chez la levure S. cerevisiae [14]. De plus, le ciblage de la E-cadhérine dans les cellules ES a été effectué en utilisant deux vecteurs de ciblage identique à l'exception du « bras gauche » d'homologie. Le « bras gauche », soit incluait, soit n'incluait pas les séquences répétées du promoteur de la E-cadhérine. Le taux de recombinaison était de 0,5 % avec les séquences répétées et de 10 % sans ces séquences ([15] et résultat non publié). Il est donc probable qu'une réaction de ciblage de gène impliquant une région comportant de telles séquences, extrêmement représentées dans les génomes, ne soit pas efficace.
Les réactions de ciblage de gène pourraient également être limitées par la dilution de la cible dans le génome. Une fois la molécule exogène introduite dans le noyau de la cellule à modifier, elle doit rechercher sa cible dans l'ensemble du matériel génétique. Cependant, ni l'augmentation de la quantité de vecteurs de ciblage introduits dans les cellules [11], ni celle du nombre de cibles dans le génome de la cellule [16] ne conduisent à l'amélioration de l'efficacité du ciblage de gène dans les cellules de mammifères. Une expérience de co-transfection transitoire de vecteurs entrant en compétition pour la même cible, un seul d'entre eux portant l'information nécessaire à la sélection des recombinants, a confirmé que la recherche d'homologie n'est pas une étape limitante dans les expériences de ciblage de gène [17]. La stabilité des intermédiaires précoces de recombinaison et/ou leur résolution seraient donc les principales barrières aux expériences de ciblage de gène.
3.2 Le vecteur du ciblage
La taille de la molécule d'acide nucléique portant la modification à introduire, le vecteur de ciblage, peut être de plusieurs kilobases ou plus réduite (de quelques dizaines à quelques centaine de bases ou de paires de bases). Les techniques classiques de ciblage de gène ont majoritairement recours à des vecteurs de ciblage contenant deux « bras » d'homologie, dont la taille totale est de 3 kb. L'utilisation de vecteurs de taille inférieure à 1 kilobase (kb) sera décrite plus loin.
3.2.1 Mécanismes de recombinaison impliqués dans les réactions utilisant des vecteurs de ciblage de grande taille
Initialement, deux types de vecteurs de recombinaison homologue pour les cellules de mammifères ont été décrits : les vecteurs d'insertion (dits ends-in ou de type O) ou les vecteurs de remplacement (dits ends-out ou de type ) [5].
Les vecteurs d'insertion sont des plasmides portant un gène codant pour une activité sélectionnable et une homologie à la région ciblée du génome (Fig. 1A). La linéarisation d'un tel vecteur au niveau de l'homologie qu'il partage avec le génome ciblé stimule son insertion dans ce génome [7,18,19]. La totalité du vecteur est intégrée dans le génome selon une réaction similaire à celle décrite par Szostak pour la réparation des cassures double brin par recombinaison homologue [20]. Les extrémités générées par la cassure sont dégradées pour donner des extrémités simple brin sortantes 3′-OH (Fig. 1, A1). Une de ces extrémités envahit le duplex homologue ciblé et s'hybride avec le brin qui lui est complémentaire pour former un ADN hétéroduplexe (ADNh). Le deuxième brin du complexe intact s'hybride réciproquement avec le brin complémentaire de la molécule portant la cassure (Fig. 1, A2). Les deux extrémités 3′-OH hybridées permettent d'initier une synthèse d'ADN pour restaurer les extrémités dégradées (Fig. 1, A3). Deux jonctions de Holliday sont ainsi formées. Leur migration va étendre les régions hétéroduplexes. Une résolution asymétrique des jonctions conduit à l'intégration du vecteur et à la duplication de la région ciblée (Fig. 1, A4). Les mésappariements éventuellement présents dans les régions d'ADNh sont réparés par le système de réparation des mésappariements (MMR) [21].
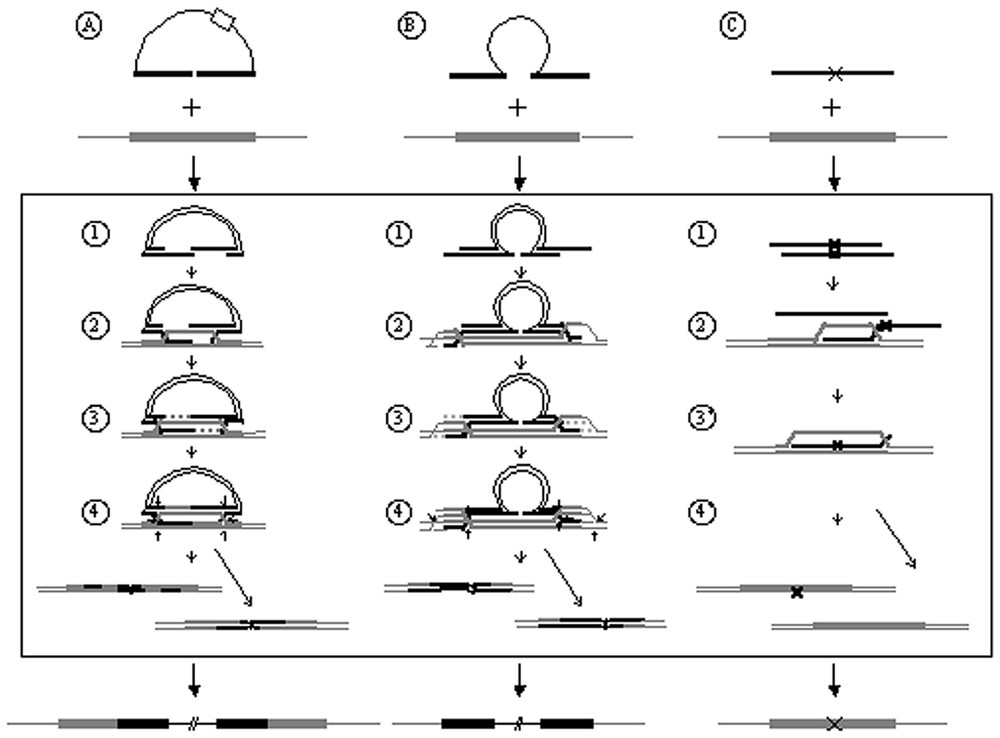
Mécanismes impliqués dans le ciblage de gène par un vecteur d'insertion (A), un vecteur de remplacement portant un large bloc d'hétérologie à la cible (B), un vecteur de remplacement portant une hétérologie à la cible de quelques bases seulement (C). Le vecteur est représenté en noir, la cible génomique en gris. Les zones d'homologie qu'ils partagent sont représentées par un trait épais, les régions hétérologues par un trait fin ou un X. Sur le vecteur de remplacement (A), la boı̂te blanche représente un gène codant pour une activité sélectionnable. Pour plus de simplicité, ce marqueur n'est pas représenté dans les figures décrivant les étapes conduisant à l'insertion du vecteur dans sa cible génomique. (1) : Dégradation des extrémités 5′ du vecteur de ciblage. (2) : Invasion du duplexe génomique homologue par les extrémités 3′-OH du vecteur de ciblage. (3) : Néosynthèse d'ADN initiée à partir des extrémités 3′-OH de la molécule lésée conduisant à la formation de deux jonctions de Holliday. (4) : Résolution des jonctions de Holliday. Le mode de résolution majoritairement observé est indiqué par des flèches plus épaisses. (3′) : Formation d'une longue région hétéroduplexe. (4′) : Réparation du mésappariement présent sur l'hétéroduplexe. Le sens de réparation majoritairement observé est indiqué par la flèche plus épaisse.
À la différence des vecteurs d'insertion, les vecteurs de remplacement conduisent à un échange non réciproque de l'information génétique : à la fin de la réaction de recombinaison, l'information initialement portée par la cible est perdue au profit de celle présente sur le vecteur (Fig. 1B). Les événements de recombinaison impliquant un vecteur de remplacement sont le plus fréquemment expliqués par un double événement de crossing-over se produisant à chaque extrémité du vecteur [22]. Leung et al. suggèrent l'existence d'un autre mécanisme pour le ciblage de gène par un vecteur ends-out [23]. Ces auteurs ont montré, chez la levure S. cerevisiae, l'existence d'un intermédiaire de recombinaison constitué par un long hétéroduplexe, de 2 à 4 kb, créé par l'assimilation d'un brin du vecteur par le duplex chromosomique homologue (Fig. 1, C2). Les mésappariements contenus dans ce long ADNh seraient corrigés par le MMR pour aboutir au produit final de la recombinaison (Fig. 1, B4). Dans le système de Leung et al., le long hétéroduplexe potentiellement formé comprend un mésappariement de seulement cinq paires de bases. La formation du long ADNh pourrait être inhibée par la présence d'un bloc d'hétérologie de taille plus importante sur le vecteur de ciblage.
La recombinaison des vecteurs d'insertion et de remplacement avec leur cible génomique utilise donc vraisemblablement des voies similaires, impliquant non seulement la machinerie de la recombinaison homologue, mais aussi la voie de la réparation des mésappariements.
3.2.2 Influence de la séquence du vecteur
Quelle que soit la nature du vecteur, la taille et le degré d'homologie qu'il partage avec sa cible sont des facteurs déterminants pour la réaction de ciblage de gène [24]. Un vecteur portant 9 kb d'homologie va conduire à une efficacité de ciblage 20 fois supérieure à celle observée avec le même vecteur portant seulement 4 kb d'homologie avec la cible génomique [5]. L'homologie peut être répartie sur les deux bras du vecteur, mais la fréquence des événements de recombinaison infidèle augmente lorsqu'un des deux bras est d'une longueur inférieure à 1 kb [25]. Ceci est très probablement lié aux événements de dégradation susceptibles de se produire naturellement aux extrémités du vecteur. L'existence de petites divergences de séquence entre la cible génomique et la région homologue portée par le vecteur de ciblage contribue également à la faible fréquence des événements de recombinaison homologue [24]. Une divergence de 1,2 % entre un vecteur d'insertion et sa cible entraı̂ne une diminution d'un facteur 6 de l'efficacité d'intégration à la cible [26]. L'utilisation de séquences isogéniques à la lignée cellulaire à modifier paraı̂t donc préférable [27]. Par ailleurs, la taille des blocs d'hétérologie portés par un vecteur de ciblage modifie certainement l'efficacité de la réaction de ciblage. En utilisant des vecteurs portant, comme seule hétérologie, la modification à introduire dans le génome, la fréquence des événements de ciblage de gène devrait être améliorée.
3.3 Limitations inhérentes à l'activité cellulaire de recombinaison non homologue
L'efficacité des expériences de ciblage de gène dans les cellules de mammifères n'est pas seulement limitée par la faible efficacité de la voie de recombinaison homologue. La fréquence élevée des événements de recombinaison non homologue est également un handicap majeur à l'obtention de cellules modifiées de manière ciblée. Au cours des expériences de ciblage de gène, les recombinants obtenus résultent majoritairement d'événements de recombinaison non homologue se produisant à des loci non ciblés du génome. Dans les cellules de mammifères, la recombinaison illégitime est en effet de l'ordre de mille fois plus efficace que la recombinaison homologue [2,11]. Le mode de recombinaison illégitime le mieux caractérisé est le non-homologous end-joining (NHEJ). Le NHEJ permet la jonction de deux extrémités d'ADN possédant très peu ou aucune homologie de séquence. Lorsqu'une cassure double brin est formée dans la cellule, elle peut être réparée par recombinaison homologue ou par NHEJ et la manière dont se produit le choix entre les deux voix de réparation reste inconnue. Le stade du cycle cellulaire semble être un facteur modulant ce choix dans les lignées de cellules leucémiques de poulet induites par un virus aviaire (cellules DT40) [28]. Cependant, il a été montré que la recombinaison homologue et le NHEJ peuvent être couplés dans la réparation d'une même cassure dans des cellules de mammifères [29]. Ces deux modes de recombinaison pourraient agir de manière séquentielle au cours du processus de réparation de la cassure [30,31].
4 De nouvelles stratégies pour une meilleure efficacité de ciblage de gène
Afin d'améliorer les techniques actuelles de ciblage de gène, de nouvelles stratégies permettant d'augmenter l'efficacité de la recombinaison homologue et/ou de diminuer l'efficacité des mécanismes de recombinaison illégitime sont développées. Ces stratégies peuvent être divisées selon trois axes : un premier visant à stimuler la recombinaison homologue en touchant à la cible génomique, un deuxième visant à créer de nouveaux vecteurs de ciblage plus efficaces et un dernier visant à modifier les activités cellulaires de manière à favoriser les événements de recombinaison ciblée.
4.1 Introduction de dommage dans la cible
Il a été montré que la présence d'un dommage dans l'ADN, en particulier une cassure double brin, augmente la fréquence des événements de recombinaison homologue. La création de cassure à l'aide d'enzymes de restriction à site de coupure rare comme I-SceI stimule la recombinaison intra-chromosomique et le ciblage de gène de 10 à 10 000 fois [32,33]. Cependant, la création d'une cassure au niveau du gène ciblé par le biais d'une méganucléase classique nécessite l'introduction préalable d'un site de reconnaissance pour cette enzyme. Cette étape consistant elle-même en un ciblage de gène, cette stratégie n'est pas avantageuse. Le récent développement de méganucléases chimériques permet, en théorie, de cliver de manière spécifique un gène ciblé non modifié. Ces méganucluéases chimériques sont constituées d'un domaine endonucléase et d'un domaine de fixation à l'ADN à séquence spécifique en doigt de zinc. Leur efficacité a été démontrée sur un substrat de recombinaison épisomal dans des ovocytes de xénope [34]. Le développement de protéines en doigt de zinc possédant une forte affinité et une spécificité pour une cible unique dans un génome donnera la possibilité de stimuler le ciblage de gène par introduction d'une cassure double brin au niveau de n'importe quel locus du génome [35,36].
Les oligonucléotides capables de former des triples hélices d'ADN (TFO pour triplex-forming oligonucleotides) couplés à des agents endommageant l'ADN, sont d'autres outils permettant de diriger des dommages à des sites spécifiques du génome. Les TFO forment des triples hélices, au niveau de séquences particulières du génome comprenant uniquement des bases puriques sur un brin et des bases pyrimidiques sur l'autre brin [37]. Ces séquences sont retrouvées en moyenne tous les 1 000 pb dans les génomes de vertébrés, ce qui permet en théorie de pouvoir cibler n'importe quel gène [38]. Le couplage de psoralènes aux TFO est largement utilisé (pour une revue, voir [39]). L'activation des psoralènes par les UVA induit la formation de lésions (monoadduits et pontages inter-brins) au niveau de la région ciblée par le TFO. Ces lésions stimulent la recombinaison extra-chromosomique et intra-chromosomique [40,41]. La formation de la structure en triple hélice paraı̂t permettre à elle seule une stimulation locale de la recombinaison [42]. Pour tirer partie de cette propriété, des systèmes bi-fonctionnels, incluant, d'une part, un TFO stimulant le ciblage de gène et, d'autre part, une molécule portant la modification à introduire dans la cible, ont été développés (Fig. 2) [43–46]. Dans ces systèmes, l'affinité du TFO pour sa cible permet en outre d'accélérer la formation des intermédiaires précoces de la réaction de recombinaison et de les stabiliser.

Utilisation de molécules bi-fonctionnelles comportant un TFO pour le ciblage de gène. La molécule bi-fonctionnelle est constituée d'un oligonucléotide (TFO, en blanc), capable de former une triple hélice avec une cible génomique polypurine/polypyrimidine (en blanc à points noirs) et d'un ADN (en noir à points blancs) portant la modification à introduire de manière ciblée (X) dans le génome. Ces deux éléments sont reliés par un adaptateur, dont la nature diffère selon les systèmes. Le TFO interagit avec sa cible génomique par des liaisons de Hoogsteen (losanges), alors que l'ADN vecteur du ciblage interagit avec le duplexe homologue par l'intermédiaire de liaison Watson–Crick (traits verticaux).
4.2 Utilisation de petits vecteurs simple brin
Les vecteurs classiquement utilisés sont de longues molécules d'ADN double brin. L'utilisation de vecteurs simple brin paraı̂t intéressante, car les événements de recombinaison sont naturellement initiés grâce à des extrémités d'ADN simple brin. Cependant, les difficultés inhérentes à la production de longs ADN simple brin et leur faible stabilité dans les cellules rendent l'utilisation de ces molécules assez complexe.
L'utilisation pour le ciblage de gène de courts fragments d'ADN simple brin d'environ 500 nucléotides (SFHR pour Small Fragment Homologous Replacement) a été testée. Ils ont permis de corriger avec une efficacité importante (dans 1 % des cellules transfectées par le vecteur) une petite délétion de 3 pb dans le gène CFTR des lignées de cellules humaines CF transformées ou primaires [47,48]. La nature des mécanismes moléculaires impliqués dans l'utilisation de ce type de vecteur n'a pas été étudiée, mais elle est certainement rattachée au modèle proposé par Leung et al. ([23] décrit plus haut). Il est à noter que l'utilisation de fragments d'ADN double brin de tailles similaires a permis d'obtenir la correction d'une mutation non-sens dans des myoblastes de souris en culture avec une efficacité de 15 à 20 % [49]. Ceci suggère que l'état simple brin du vecteur n'est pas requis pour obtenir une fréquence de ciblage de gène élevée.
L'utilisation de courtes molécules oligonucléotidiques comme vecteurs pour le ciblage de gène est de plus en plus développée. Ces molécules sont synthétisées chimiquement, ce qui permet d'y introduire facilement des nucléotides modifiés qui augmentent leur stabilité dans les cellules. Les RDO (RNA/DNA Oligonucleotides) sont des oligonucléotides chimériques d'environ 70 nucléotides, composés de bases ribonucléiques modifiées (2′-O-méthyl) et de bases désoxyribonucléiques (Fig. 3). Leur séquence primaire leur permet de former une structure en double épingle à cheveux. Ils ont été utilisés pour corriger ou introduire des mutations ponctuelles dans des cellules de mammifère en culture et, in vivo, dans des animaux et des plantes (pour une revue, voir [50]). Lors d'une étude menée in vitro dans des extraits cellulaires de cellules humaines, il a été montré que la partie 2′-O-méthyl ARN de ces oligonucléotides facilite et stabilise la formation des intermédiaires précoces de recombinaison [51]. Les mécanismes précis sur lesquels reposent le ciblage de gène par les RDO ne sont pas connus, mais ils impliqueraient des voies de recombinaison homologue et de réparation de l'ADN [52]. Dérivant des RDO, des oligonucléotides chimères de structure plus simple ont été utilisés avec succès pour le ciblage de gène [53]. Ces molécules, nommées ODN, sont composées de quelques dizaines de bases désoxyribonucléiques. Elles comprennent en plus, à chaque extrémité, quatre bases ribonucléiques modifiées (2′-O-méthyl), qui ont pour fonction de protéger l'oligonucléotide contre les exonucléases. Dans un même système, l'efficacité de correction d'une mutation ponctuelle par un RDO ou un ODN peut aller de plusieurs cellules corrigées sur 100 cellules transfectées à aucune cellule corrigée pour 106 cellules transfectées ([54] et nos propres résultats non publiés). Des efforts permettant de comprendre quels sont les facteurs limitant la reproductibilité de ces expériences doivent être fournis, afin de permettre une utilisation effective de ces molécules prometteuses.
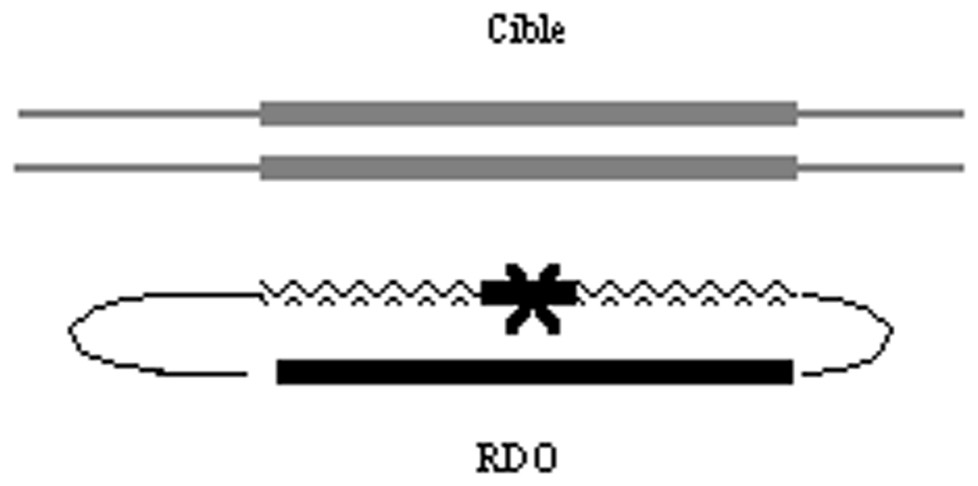
Utilisation de petits oligonucléotides chimères de type RDO pour le ciblage de gène. La molécule RDO est un oligonucléotide comportant 50 à 80 nucléotides de type ribonucléique (zig-zag) et désoxyribonucléique. Les traits noirs épais correspondent aux séquences composées de bases désoxyribonucléiques homologues à la séquence ciblée. La séquence ces oligonucléotides leur permet d'adopter une structure en double épingle à cheveux. Des régions hétérologues à la cible (traits noirs fins) facilitent la formation de cette structure secondaire. La modification à introduire dans le génome ciblé (X) est portée par des bases désoxyribonucléiques entourées de ribonucléotides.
4.3 Modulation des activités cellulaires
Une troisième stratégie visant à améliorer les techniques actuelles de ciblage de gène consiste à modifier le niveau d'expression des gènes codants pour les protéines participant aux processus de recombinaison, de manière à stimuler la fréquence des événements de recombinaison homologue ou à diminuer celle des événements illégitimes.
Des études génétiques et biochimiques ont permis de mettre en évidence chez la levure un groupe de gènes impliqués dans la réparation des cassures double brin de l'ADN par recombinaison homologue : le groupe d'épistasie ScRAD52 [55]. Des homologues structuraux de ces différents gènes ont pu être identifiés chez les vertébrés et leur similarité de fonction, démontrée in vitro, ainsi que la capacité de certains d'entre eux à complémenter des mutants de levure suggèrent une conservation dans la fonction de ces gènes de la levure à l'homme. La sur-expression de certaines de ces protéines, en particulier les homologues de Rad51, Rad52, Rad54, Rad55 et Rad57, qui participent aux étapes d'appariements et d'échange de brins, pourrait permettre d'augmenter la fréquence des événements conduisant au ciblage de gène.
Il a été montré que la sur-expression de RAD51 dans des cellules humaines augmente la fréquence de ciblage de gènes à deux loci différents [56]. L'analyse des recombinants ainsi obtenus a montré que la fréquence des événements d'intégration illégitime était identique dans des cellules sauvages et dans des cellules sur-exprimant hsRAD51, alors que les événements de recombinaison homologue ciblés étaient de deux à trois fois plus fréquents dans les cellules sur-exprimant hsRAD51. Ce résultat suggère que la protéine Rad51 est impliquée dans les réactions de recombinaison ciblée et que son niveau d'expression est limitant. La sur-expression de l'homologue bactérien de RAD51, RecA, dans des cellules murines, permet également la stimulation des réactions de ciblage de gène [57]. En revanche, la sur-expression de RAD52, qu'elle soit associée ou non à celle de RAD51, provoque une réduction de l'efficacité de ciblage de gène [58]. La protéine Rad52 est impliquée dans différentes voies de recombinaison [59]. Il est possible que sa surproduction stimule une voie alternative à celle utilisé par le ciblage de gène. La surproduction des autres protéines accessoires de Rad51 n'a pas été testée, mais l'inactivation de chacune de ces protéines (Rad54, Rad51B, Rad51C, Rad51D, XRCC1, XRCC3 [60–62]) provoque une diminution de l'efficacité de recombinaison entre un vecteur et une cible génomique homologue. Il est donc possible que leur surproduction stimule le ciblage de gène.
La modification de l'expression de certains gènes extérieurs aux groupes d'épistasie RAD52 mais reliés au mécanisme de la recombinaison homologue, tels que BRCA1, BRCA2, p53 ou BLM, paraı̂t également intéressante à tester (pour une revue, voir [63]). Cependant, les effets délétères engendrés par la dérégulation de l'expression de suppresseurs de tumeur ne favorisent pas le développement de ces stratégies. De même, une sur-expression de hsRad51 ayant été décrite dans différentes lignées de cellules cancéreuses, il n'est pas exclu que la modification de l'expression de ce gène entraı̂ne, parallèlement à une amélioration de l'efficacité du ciblage de gène, la transformation des cellules ainsi manipulées [64,65].
Il est également possible d'améliorer les techniques de ciblage de gène en diminuant l'activité cellulaire de recombinaison illégitime. Des expériences de ciblage de gène ont été menées dans des lignées déficientes pour des protéines du NHEJ [66]. La fréquence de ciblage n'est augmentée dans aucune des lignées mutantes testées (cellules ES de souris Ku70−/−, XRCC4−/− et DNA-PKcs−/−), alors que la réparation par recombinaison homologue de cassures induites par l'endonucléase I-SceI est fortement augmentée dans la lignée Ku70−/−. Cette observation, ainsi que d'autres études menées dans des lignées déficientes dans la voie de NHEJ [67,68], laissent penser à l'existence d'une voie alternative de recombinaison illégitime. La caractérisation de cette voie ainsi que celle des systèmes régulant l'utilisation des différentes voies de recombinaison sont des axes de recherche qui bénéficieront certainement aux techniques de ciblage de gène.
Il a cependant été mis en évidence une stratégie permettant de diminuer l'activité de recombinaison illégitime dans des cellules de mammifères. Elle consiste à inhiber une protéine nucléaire capable d'ajouter des polymères d'ADP-ribose sur des protéines, la poly-(ADP-ribose)-polymérase (PARP), qui est activée en présence de cassures double brin de l'ADN [69]. L'inhibition de la PARP par le 3-méthoxybenzamide (3-MB) permet de diminuer la fréquence des événements de recombinaison illégitime et par ce biais d'augmenter la proportion de recombinants issus d'événement de recombinaison homologue lors d'expériences de ciblage de gène [70]. Il est à noter que l'observation de cet effet dépend du mode de transfection utilisé pour délivrer le vecteur de ciblage au noyau.
5 Discussion
La production des animaux modèles porteurs de mutations génétiques, similaires à celles retrouvées dans des pathologies humaines d'origine génétique, ainsi que la mise au point de thérapies géniques efficaces constituent deux voies de recherche d'enjeu majeur. Ces deux applications requièrent la mise au point de techniques permettant de modifier les génomes de manière fine, contrôlée et avec une grande efficacité. Les techniques classiques de ciblages de gène ne conduisent à l'obtention de recombinants modifiés de manière ciblée qu'avec une fréquence très faible. La majorité des recombinants obtenus est issue d'événements de recombinaison illégitime se produisant en un locus aléatoire du génome. De ce fait, ces approches classiques excluent la possibilité de générer des recombinants ne présentant pas un phénotype sélectionnable. Diverses stratégies permettant d'obtenir une efficacité de ciblage suffisante pour se dispenser de l'étape de sélection et de criblage ont été récemment développées. Cependant, il faut prêter attention aux effets délétères que ces nouvelles approches sont susceptibles d'avoir sur le génome, en particulier si elles doivent être appliquées à la thérapie génique. L'utilisation de molécules capables d'endommager la cible, notamment celle de psoralènes, est problématique, de part, leur action mutagène. La modification de l'expression de gènes impliqués dans la recombinaison génétique, même si elle n'est envisagée que sous une forme transitoire, est également problématique. La plupart de ces gènes jouent un rôle très important dans le métabolisme de l'ADN et une dérégulation de leur expression pourrait entraı̂ner des dommages et réarrangements chromosomiques létaux ou cancérigènes. L'approche offrant un maximum de sûreté est celle qui consiste à utiliser des vecteurs plus recombinogènes que les vecteurs classiques. Les petites molécules simple brin, naturelles ou synthétiques, semblent posséder cette propriété. Le développement de leur utilisation et des méthodes permettant de les délivrer efficacement au noyau des cellules à modifier pourrait être la solution idéale pour le ciblage de gène.
Remerciements
Ce travail a pu être réalisé grâce aux contrats PIC IGRT de l'Institut Curie et CNRS-RP n°RL/PT/971691322. EB a été financé par l'ARC.



