Abridged English version
Instead of vibrational phenomena, which are implicated in vision and hearing, biochemical interactions involving structural complementarity between molecules are required to elicit the olfactory signals. All living organisms, including human beings, are able to discriminate between myriads of odours, which are airborne, therefore usually hydrophobic, molecules. The olfactory repertoire is remarkably diverse, composed of aromatic and aliphatic molecules with varied functional groups and diverse carbon backbones. The first step of olfactory detection consists of the interaction of odorants with protein receptors embedded within the plasma membrane of olfactory sensory neurone dendritic cilia. In humans, these sensory neurones are found in the olfactory epithelium, situated in the olfactory cleft at the top of the nasal cavities just under the skull. Axons of these neurones project to glomeruli in the two olfactory bulbs of the brain through the cribriform plate of the ethmoid bone, which forms the skull base.
Linda Buck and Richard Axel, which were rewarded with the Physiology and Medicine Nobel Prize in 2004, demonstrated that these receptors, endowed with seven transmembrane domains, belong to the vast family of G-protein-coupled receptors (GPCR), whose role in cellular communication is largely documented. When activated by odorant binding, the olfactory receptors (ORs) interact with a trimeric G protein, which then dissociates, liberating its subunit. This subunit in turn binds a membrane effector, adenylate cyclase III, which transforms ATP into a secondary messenger, cyclic AMP. The latter molecule provokes the opening of a nucleotide-gated ion channel. Then positive ions, entering the neurone cytoplasm, depolarize the cell, eliciting an action potential, and further the nervous influx. Odour elaboration therefore relies on odorant–receptor interaction, based on size, form and chemical function recognition, at the initiation of sensory coding. Although the number of odorant molecules is only limited by their volatility at usual temperature and atmospheric pressure, human ORs are only about 350, which is nevertheless the largest multigenic family in our genome. Electrophysiology experiments run on isolated olfactory neurones and functional expression of ORs, followed by calcium imaging, permitted to understand this paradoxical situation. Surprisingly, a broad specificity was found for all ORs so far studied. Their molecular receptive ranges were found to be highly tolerant in some aspects, but highly specific in other areas, therefore establishing a complex combination of ORs. Finally, the olfactory system rests on fuzzy combinatory coding, opposite to the immune system, which involves the very specific binding of antigens on a single specific antibody among millions. A given OR can be activated by numerous more or less structurally related odorants, and, reciprocally, an odorant can serve as an agonist for many receptors, or even as an antagonist, which complicates this combinatorial code. Even with a mere 350 receptors and a system in which 10 receptors are on average required to make discrimination, there are more than receptor combinations, far more than the number of possible chemical compounds. So that the system allows detection of molecules that never existed before in Nature, which is the case of new flavours resulting from human cooking and processing, and also of odorants produced by chemical synthesis.
In order to understand how odorants bind onto their receptors, the knowledge of their 3D structure is a prerequisite. As GPCRs are very hydrophobic transmembrane proteins, chances of obtaining an OR 3D-structure by X-ray crystallography is unlikely by now. Nevertheless, protein modelling can be helpful in studying the odorant–receptor interactions at atomic scale, based on the crystal structure of the only known prototypical model GPCR, bovine rhodopsin. The prediction of functional sites provided concrete suggestions of site-directed mutagenesis experiments for altering odorant specificity, which were used to validate the models. This approach shows that odorants bind through hydrophobic and van der Waals interactions into a binding pocket, opened to the exterior of the cell, and made by four transmembrane domains, which form a α-helix bundle. This view helps to define the odotope as the region of an odorant molecule that is specifically recognized by an OR binding site, so that a single molecule can have several odotopes, depending on how it interacts with the OR.
In vertebrates, only one OR gene is expressed in any given OR neurone, i.e. “one neurone, one receptor”. Even very homologous ORs are expressed in different neurones, so that OR gene expression was shown to be derived exclusively from only one of two alleles, which are mutually excluding the expression of one another. Not only the expression of ORs is clonal, but also all neurones expressing the same receptor converge to the same olfactory glomerulus. In the olfactory bulb, their axons form synapses with mitral cells and tufted relay neurones, resulting in a stereotyped spatial map in which inputs from different ORs are targeted to different bulb glomeruli. Consistent with patterns of odour-induced activity, an odorant receptor code is represented by a dispersed ensemble of neurones and, in the olfactory bulbs, by a specific combination of activated glomeruli. A stereotyped sensory map is further created in the olfactory cortex in which signals from a particular OR are targeted to specific clusters of neurones. In addition, signals from the same OR are targeted to multiple olfactory cortical areas, permitting the parallel processing of inputs from a single OR before delivery to the neocortex and limbic system. So odour coding results from the combinatory activation of a set of ORs, which bind different odotopes (or different odorants in a mixture), giving rise to an activated topographical neuronal map in the cortex.
In vertebrates, the study of OR evolution shows a period of increasing abundance of functional genes with appearance of aerial life (from 100 OR genes in Fishes to 400 in Amphibians); then the OR number exceeded 1000 in dog and rodents (up to 1500 in rat). However, when nostrils went far from the soil (the principal odour source), this number significantly decreased. This is the case of Primate standing (∼350 ORs in humans) and bird takeoff (only 80 ORs in chicken). Nonetheless, comparison of the OR gene repertoire of human with its closest living evolutionary relative, the chimpanzee, reveals three human-specific OR subfamily expansions, suggesting that a subset of OR genes are under positive selection, by contrast to recurrent pseudogenisation (loss of gene function due to deleterious mutations). In humans, the OR genes are found scattered in nearly all chromosomes except 20 and Y, but only six chromosomes contain about three ORs out of four. These genes are classified in 17 families, which are grouped in two major classes, class I corresponding to the ancestral fish ORs, and class II to ORs that have appeared with aerial life. Surprisingly, 10% of human ORs belong to class I, while 90% belong to class II. At least one member of the former class was shown to be functional.
In our species, it is well known that differences in olfaction may obey a genetic determinism, well known in the case of androstenone smell. Detailed genetic studies, based on single-nucleotide polymorphism, showed both a great individual diversity and a significant interethnic segregation of OR genes, resulting from genetic drift due to rapid random pseudogenisation. This unprecedented prevalence of segregating pseudogenes revealed one of the most pronounced cases of functional population diversity in the human genome, suggesting that different evolutionary pressures may have shaped the chemosensory repertoire in different human populations. Absence of uniformity in the human olfactory genome (statistically no one can share the same set of functional OR genes) could explain, at least partially, that there is no semantic reference for olfaction, which remains a subjective sense, unable to classify odours unambiguously.
As regards humans, one can ask these questions: Are we microsmatic animals (i.e. endowed with poor sense of smell)? Did evolution result in loss of certain peculiar receptors, which would imply accumulation of specific anosmias? The comparison of the olfactory genomes of human and mouse shows that pseudogenisation only affects genes whose sequences are very close to each other, so the human odour space is not considerably restricted compared to that of the mouse. In addition, it is worth emphasizing that the absolute size of human olfactory bulbs is as large as those of rodents, which comprise 3 times less glomeruli. At last, humans have a far larger brain with a huge cortex for signal processing. Therefore we are not microsmatic, but have developed vision and hearing to such an extent that smell does not appear as useful.
In conclusion, it is worth noticing that the OR role is not restricted to olfaction: these receptors are involved in axonal guidance of olfactory neurones (necessary to rigorously maintain proper connections with glomeruli during neurone renewal) or in spermatozoa chemotaxis. Is it plausible that new functions will be discovered in cellular migration occurring during embryogenesis, for instance? Although only at its beginning, the understanding of the molecular bases of olfaction is now well established on sound convergent data from diverse biological approaches, dismissing specious theories such as the vibrational theory. The combinatorial performance of the peripheral detection explains that smell is not an analytical sensory system, but rather a synthetic one referring to odotopic maps, globally acknowledged like recognition of faces (gnosis).
1 Introduction
En matière d'olfaction, les questions suivantes se posent de façon récurrente : comment se forme la représentation des odeurs ? Pourquoi ne peut-on les nommer ? Pourquoi le sens olfactif n'est-il pas universel ? La connaissance des mécanismes moléculaires qui sous-tendent la capture des molécules odorantes permet d'apporter des éléments de réponse à ces questions. Bien que l'olfaction soit essentielle pour la survie de tout organisme, notamment pour déterminer si l'aliment est comestible ou non, ou pour fuir des dangers potentiels, l'étude neurophysiologique de l'odorat ne fut longtemps qu'effleurée en raison de la complexité de son approche. Cependant, les essais de corrélations entre structure chimique des molécules odorantes et réponse sensorielle sont aussi vieux que la chimie de synthèse [1]. Au-delà des études des relations structure–activité des odorants [2,3], des théories sans fondement biologique furent bâties pour établir des déterminismes, reprenant la théorie vibrationnelle introduite dans les années 1930 pour expliquer les interactions entre molécules chimiques [4–6]. Les études modernes sur les mécanismes biologiques de l'olfaction n'ont pris leur essor que dans les années 1980 et ont entamé une ère nouvelle, sanctionnée par le prix Nobel de médecine et de physiologie, attribué à Linda Buck et à Richard Axel en 2004 pour la découverte des récepteurs olfactifs [7], marquant ainsi clairement les progrès décisifs accomplis. Il a ainsi été formellement démontré que la perception des molécules odorantes implique des interactions biochimiques reposant sur la complémentarité structurale de molécules, contrairement à la vision et à l'audition, qui procèdent de phénomènes de nature vibratoire.
1.1 Les systèmes chimiosensoriels et la multimodalité
La compréhension du traitement de l'information sensorielle chimique demeure cependant un défi, car il implique tous les étages d'intégration, du niveau périphérique jusqu'au cortex, et fait appel à la mémoire et à la sémantique, tout en reposant sur la multimodalité, qui rend formidablement complexe l'étude de la perception des molécules odorantes [8]. En effet, le cerveau combine simultanément olfaction, gustation, somesthésie (toucher, thermoception, nociception), et même vision et audition, pour donner une perception unique, alors que ces sens procèdent de voies nerveuses totalement différentes. Par exemple, la sensation de menthe, due au L-menthol, résulte de la fixation indépendante de ce composé sur des récepteurs qui activent des neurones olfactifs, produisant la sensation odorante, et, parallèlement, sur des récepteurs très différents, activés normalement par le froid, et portés par des neurones trigéminaux. Odeur et fraîcheur forment ainsi une seule perception combinée de nature multimodale.
1.2 Les molécules odorantes et les odotopes
Pour les animaux aériens, théoriquement tout composé chimique suffisamment volatil pour atteindre l'épithélium olfactif peut stimuler un neurone olfactif et provoquer une sensation olfactive, c'est-à-dire toute molécule suffisamment volatile pour atteindre le fond des cavités nasales, donc de petite taille (<300 Da) et douée d'un certain caractère hydrophobe. Ce caractère hydrophobe explique pourquoi parfums et arômes sont quasi nécessairement associés aux lipides et aux alcools. Le répertoire olfactif est donc remarquablement varié, comprenant des molécules aromatiques et aliphatiques avec divers squelettes carbonés et dotés de groupes fonctionnels tels que les aldéhydes, esters, alcools, acides carboxyliques, cétones, alkènes, amines, imines, thiols, dérivés halogénés, nitriles, sulfures et autres éthers. Il convient ici d'introduire le concept d'odotope, qui est un des motifs stéréochimiques d'une molécule odorante capable d'interagir avec au moins un récepteur olfactif (RO). Un odorant peut avoir plusieurs sites d'interactions différents, lui permettant de se fixer indépendamment sur plusieurs RO différents et avec des affinités différentes.
1.3 Anatomie du système olfactif
Chez les Vertébrés, et notamment l'être humain, les neurones olfactifs sont les capteurs sensoriels primaires. Situés au sein du neuroépithélium olfactif qui tapisse le fond de la cavité nasale dans les fentes olfactives, sous la boîte crânienne, ils projettent leurs axones à travers la lame criblée de l'os ethmoïde (le plancher de la boîte crânienne) vers des structures glomérulaires dans les deux bulbes olfactifs à la base du cerveau [9]. Les neurones sensoriels olfactifs sont bipolaires, avec une seule dendrite se terminant par des cils olfactifs baignant dans le mucus nasal au rôle protecteur, et un axone qui, via les cribles de l'os ethmoïde, se projette dans les glomérules, structures situées dans les deux bulbes olfactifs, localisés à la base du cerveau. Chez l'Homme, les très étroites fentes olfactives occupent moins de 10% de la surface des cavités nasales [10], et, dans chaque cavité nasale, quelque six millions de neurones se projettent dans les 8000 glomérules de l'un des deux bulbes olfactifs. Il convient de noter que les neurones olfactifs sont les seuls neurones à être en contact direct avec l'extérieur de l'organisme, raison vraisemblable pour laquelle ils se renouvellent tous les deux mois environ.
1.4 La compréhension du codage périphérique de l'olfaction
La question est donc de comprendre comment l'odorant, qui est une molécule chimique pénétrant dans la cavité nasale, induit une sensation, qui est l'odeur. Avant que l'information soit traitée par le système nerveux central, ce qui implique un passage par les bulbes olfactifs et différentes aires corticales, la première étape concerne le codage dit périphérique, qui a lieu au niveau des cils des neurones sensoriels de l'épithélium olfactif. Cette première étape de la détection olfactive réside dans l'interaction des odorants avec des protéines enchâssées dans la membrane de ces cils, les récepteurs olfactifs. Comme les molécules odorantes sont généralement hydrophobes, des protéines de transport ou odorant-binding proteins aideraient au franchissement de la couche de mucus hydrophile [11,12]. Le neurone olfactif transforme le signal chimique en un signal biochimique, puis, finalement, en signal électrique (influx nerveux).
2 Les récepteurs olfactifs
Linda Buck et Richard Axel ont été à l'origine de la démonstration que les RO appartiennent à la très vaste famille des récepteurs couplés aux protéines G (RCPG), protéines connues pour leur rôle dans la communication cellulaire [7]. Leur découverte a eu comme point de départ l'observation du fait que l'activation des neurones olfactifs par des molécules odorantes impliquait des messagers secondaires connus pour être ceux que l'on observe dans la cascade de transduction en aval des RCPG. La surprise de leur découverte fut moins la nature que le nombre de gènes de récepteurs olfactifs, un millier chez les Rongeurs, qui constituent la plus grande famille multigénique, représentant de 2 à 4% du génome. Ce nombre d'environ 350 seulement chez l'être humain est cependant bien inférieur à celui des molécules odorantes [13].
2.1 Les récepteurs couplés aux protéines G (RCPG)
Ces récepteurs forment une très vaste famille de protéines membranaires dotées de propriétés communes. Elles sont le capteur d'un processus à trois étapes, impliquant aussi une protéine G comme transducteur (par exemple la transducine dans la rétine) et une enzyme effectrice (phospholipase C, adénylate cyclase...). Les RCPG sont très variés et peuvent capter des messages aussi différents que des photons lumineux, des petites molécules organiques, des sucres, des nucléotides, des peptides, des lipides et aussi des macromolécules. La structure générale des RO est comparable à celle de la rhodopsine bovine, seul élément de cette famille dont la structure cristallographique a pu être déterminée [14]. Ils sont dotés de sept domaines transmembranaires réunis en faisceau, constitués d'acides aminés très hydrophobes et reliés par trois boucles externes et trois boucles cytoplasmiques, avec un domaine extracellulaire (ectodomaine N-terminal) et un domaine intracellulaire (endodomaine C-terminal). Les récepteurs à sept domaines transmembranaires sont impliqués dans de très nombreux processus de communication cellulaire (régulation hormonale, neurotransmission...) et sont très étudiés, car ce sont notamment les cibles de plus de la moitié des médicaments actuellement vendus en pharmacie. La plupart des RCPG sont cependant orphelins, c'est-à-dire que les agonistes (les molécules qui les activent) n'ont pas encore été identifiés ; c'est notamment le cas des récepteurs olfactifs.
2.2 Caractéristiques propres aux récepteurs olfactifs
Ces récepteurs sont de petite taille (300 à 350 acides aminés), du fait d'un très court ectodomaine, et leurs gènes dépourvus d'introns. Ils se distinguent par leur très grande seconde boucle extracellulaire, une troisième boucle courte et quelques critères de séquence [13,15].
2.3 Voies de signalisation des récepteurs olfactifs
Lorsqu'ils sont activés par une molécule odorante, à l'instar d'autres RCPG, les RO induisent une cascade de transduction intracellulaire impliquant des protéines trimériques appelées protéines G. Par analogie avec les autres RCPG, on pense que la fixation de l'odorant induit un changement de conformation des domaines transmembranaires 3 et 6 [16], ce qui permet l'interaction avec la protéine G trimérique et induit sa dissociation en une sous-unité α et un dimère βγ, ainsi que l'hydrolyse du GTP en GDP. Celle-ci conduit à la réassociation du trimère et arrête le processus d'activation [17]. Une sous-unité particulière, , qui active une adénylate cyclase, est le plus souvent rapportée intervenir dans les processus olfactifs [18,19]. Cette enzyme convertit l'ATP intracellulaire en AMP cyclique (AMPc), lequel à son tour induit l'ouverture d'un canal ionique de la membrane plasmique du neurone olfactif, permettant l'entrée de cations tels que Na+ et Ca2+ [20]. Il s'ensuit une dépolarisation du neurone, puis la génération d'un potentiel d'action aboutissant à l'influx nerveux. Ce processus fonctionne comme un amplificateur qui permettrait à une seule molécule d'odorant de générer un potentiel d'action par neurone [21,22]. Des voies alternatives pourraient être utilisées en olfaction, notamment celle de l'inositol triphosphate (IP3), qui implique une phospholipase C, via d'autres types de protéines G [23].
2.4 Les répertoires d'odorants des récepteurs olfactifs
Sachant que les protéines membranaires perdent leur fonction dès lors qu'on les extrait de leur membrane, comment identifier les ligands des RO ? Comme il n'y a qu'un seul gène de RO exprimé dans un neurone donné (cf. infra) [24], une approche consiste à étudier la réponse électrophysiologique de neurones olfactifs isolés par patch-clamp stimulés avec des odorants [25], puis à identifier le récepteur par RT–PCR. L'expression clonale des RO a aussi été utilisée pour pratiquer des études in vivo chez le rat [26].
Une autre approche plus générale, fondée sur les méthodes pharmacologiques, l'imagerie calcique couplée à l'expression hétérologue, consiste à transformer une cellule qui n'exprime pas naturellement de RO avec le gène d'un RO préalablement cloné. En plaçant dans la cellule un colorant fluorescent qui a la propriété de ne fluorescer qu'en présence d'ions calcium, on peut visualiser l'activation d'un RO qui lie un odorant, puisque ce phénomène induit la libération de calcium dans le cytoplasme, donc l'émission de lumière par le fluorophore. On peut ainsi avoir accès à l'affinité des odorants pour leurs récepteurs [27,28].
Bien que seulement quelques RO aient été étudiés [25–34], quelles que soient les approches méthodologiques utilisées, les résultats permettent de généraliser les travaux originaux de Malnic et al. [25], qui ont formellement éclairé le domaine : un récepteur donné reconnaît de nombreux odorants et tout odorant peut servir de ligand pour de nombreux récepteurs. Il existe cependant des critères communs de forme et de fonction pour les ligands d'un récepteur donné. Par exemple (Fig. 1), dans le cas du récepteur humain hOR1G1, parmi un centaine d'odorants testés, cinq s'avèrent être des activateurs forts qui ont en commun d'avoir neuf ou dix carbones et une extrémité polaire portant diverses fonctions (aldéhyde, alcool, cétone...), alors qu'une vingtaine d'autres molécules de taille un peu différente activent plus faiblement ce récepteur [27]. Araneda et al. [28] ont montré que, pour un récepteur donné, il y a simultanément une grande spécificité pour certaines caractéristiques moléculaires et une grande tolérance pour d'autres. Cette capacité des RO à interagir avec plusieurs odorants est ainsi bien adaptée au codage d'un vaste espace olfactif : avec seulement 350 récepteurs différents et un système dans lequel 10 récepteurs en moyenne seraient requis pour discriminer des odorants, il y a plus de combinaisons de récepteurs, plus que le nombre de composés chimiques possibles. Le système olfactif repose donc sur une spécificité de liaison de nature floue.
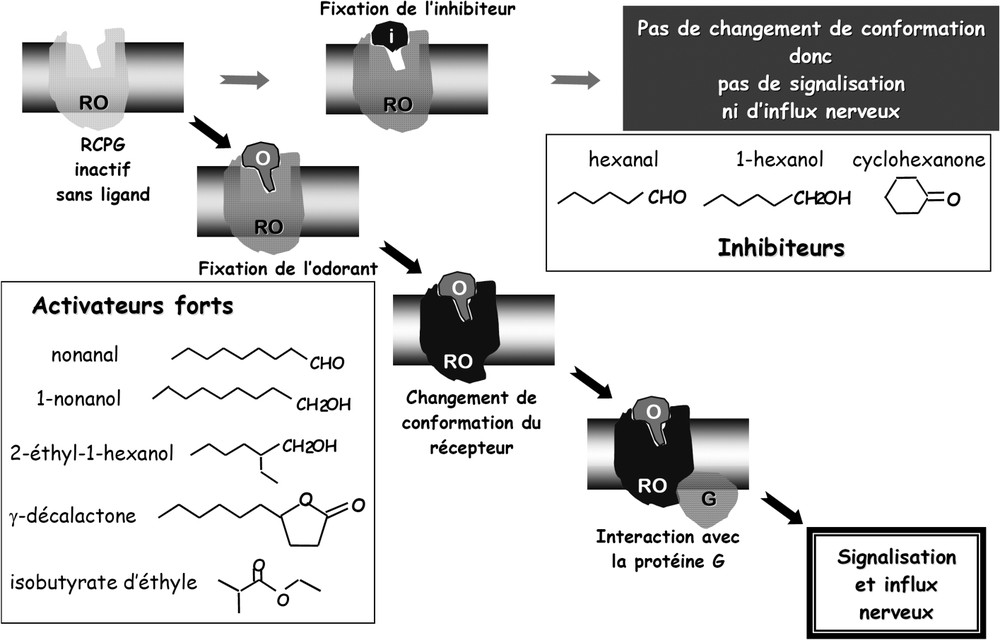
Représentation schématique de l'activation d'un récepteur olfactif par un odorant (O), agoniste entraînant un changement de conformation permettant l'interaction avec la protéine G, et de son inhibition par un antagoniste. Les odorants qui activent le récepteur OR1G1 (RO) et ses inhibiteurs (i) [27] illustrent, à titre d'exemple, la parenté structurale entre ces deux ensembles de molécules.
2.5 Mise en évidence d'antagonistes
De fait, il s'avère que le codage olfactif est encore plus complexe, car l'activation par les agonistes peut être perturbée par des odorants, qui se comportent comme des inhibiteurs. En étudiant les ligands du récepteur hOR1G1 (Fig. 1), on peut constater que des molécules dotées des mêmes fonctions chimiques que les activateurs forts, mais comportant six carbones au lieu de neuf, inhibent les réponses du récepteur : tout se passe comme si ces molécules se fixaient à l'entrée d'un site actif sans être suffisamment volumineuses pour provoquer le changement de conformation nécessaire à la transduction du signal [27]. Un tel résultat, observé par d'autres équipes [33,34], illustre la complexité du codage olfactif, qui intègre l'action d'agonistes et d'antagonistes, certaines molécules pouvant avoir un rôle activateur sur un récepteur et inhibiteur sur un autre, sans occulter en outre le rôle de la concentration des odorants.
2.6 Relations entre l'activation des récepteurs et la perception sensorielle
La question est de savoir si ce phénomène d'antagonisme périphérique observé in vitro a des répercussions effectives sur la perception des odeurs in vivo, donc s'il induit une inhibition sensorielle. Dans un premier temps, l'équipe de Hans Hatt étudia le répertoire du récepteur hOR17-4, qui présente la particularité d'être exprimé dans les spermatozoïdes [33]. Parmi les agonistes les plus actifs, le bourgeonal se révéla être aussi un excellent attracteur qui induit le chimiotactisme des spermatozoïdes. En revanche, le chimiotactisme, comme la liaison au récepteur, sont inhibés par une autre molécule, l'undécanal. Or le récepteur hOR17-4 est aussi exprimé dans l'épithélium olfactif. En faisant des mesures psychophysiques sur des patients (analyse sensorielle), ainsi que des électro-olfactogrammes pour suivre la réponse électrophysiologique de l'épithélium olfactif, ces auteurs ont montré qu'en présence simultanée de bourgeonal et d'undécanal, il y a perte de perception de l'odeur de muguet due au bourgeonal chez l'être humain [35].
2.7 Les sites actifs des récepteurs olfactifs
Comment peut-on expliquer, au niveau moléculaire, voire atomique, la faible spécificité d'interaction des RO ? Comme les RO sont de nature membranaire, il est impossible d'identifier les sites actifs par l'observation de la structure tridimensionnelle (diffraction aux rayons X). En effet, en dépit de l'intérêt considérable de l'industrie pharmaceutique pour ces molécules, un seul récepteur à sept segments transmembranaires parmi des milliers, la rhodopsine, qui se trouve dans un état quasi cristallin dans la membrane des cellules de la rétine, a pu faire l'objet d'une telle étude [14], mais il est très illusoire d'espérer une telle approche pour les RO. Dans l'état actuel des moyens disponibles, le site actif ne peut être déduit que de la comparaison des nombreuses séquences en acides aminés des différents récepteurs : orienté vers la face extracellulaire, ce site de liaison correspond en toute logique aux régions hypervariables des séquences situées dans le faisceau des hélices transmembranaires 3 à 6 [36,37]. En prenant la structure de la rhodopsine comme modèle, il est possible de prédire la structure tridimensionnelle de RO à l'aide d'algorithmes de modélisation moléculaire [38,39]. Parmi de nombreuses tentatives, les résultats récents de Katada et al. [34] sont les plus convaincants, car leur modèle a été validé expérimentalement. Après avoir prédit le site actif, ces auteurs ont positionné les ligands connus du récepteur de souris mOR-EG par calcul (amarrage moléculaire), identifié les acides aminés impliqués dans la liaison avec les odorants et prédit des modifications de spécificité en envisageant le changement de ces acides aminés. Ils ont réalisé ces modifications par mutagenèse dirigée et vérifié expérimentalement leurs prédictions, validant ainsi le modèle structural. Ils ont ainsi pu suggérer que, bien que la grande majorité des autres récepteurs couplés aux protéines G reconnaissent leurs ligands principalement par des interactions ioniques et des liaisons hydrogènes, les RO lient les odorants par des liaisons hydrophobes et des interactions de van der Waals, en accord avec leur nature hydrophobe. Ce type d'interactions permet d'expliquer la sélectivité floue de la fixation, de même que son affinité plutôt faible, d'environ 106 à [29,40].
2.8 Conclusions sur l'activation des récepteurs par un système combinatoire flou
L'élaboration de l'odeur repose donc sur l'interaction odorant–récepteur, fondée sur une reconnaissance de taille, de forme et de fonction chimique, point de départ du codage de l'information sensorielle. Le système repose ainsi nécessairement sur un codage combinatoire, puisqu'il n'y a que 350 RO pour des myriades d'odorants : un récepteur donné reconnaît de nombreux odorants et tout odorant peut servir de ligand pour de nombreux récepteurs, sans oublier les antagonistes qui compliquent ce code combinatoire. Ce système permet la détection de molécules qui n'ont jamais encore existé dans la Nature, ce qui est notamment le cas des nouvelles flaveurs résultant des préparations culinaires et des molécules produites par la chimie de synthèse.
3 Le codage olfactif
3.1 La connectique neuronale
La simple description de l'interaction d'une molécule odorante avec un jeu de RO au niveau périphérique ne permet pas d'expliquer comment s'élabore le codage neuronal. Ce codage repose sur deux phénomènes : le premier réside dans le fait qu'un neurone olfactif n'exprime qu'un seul allèle de gène de RO [15,24,41–45], le second dans le fait qu'un glomérule olfactif ne reçoit les projections que de neurones qui expriment le même récepteur [46]. C'est l'organisation anatomique du bulbe olfactif qui transforme la détection stéréochimique des odorants en une carte d'activation neuronale, dite « odotopique », à la base du codage périphérique. En effet, en dépit d'un renouvellement permanent des neurones olfactifs et grâce à la stabilité de l'expression monoallélique des récepteurs [45], la projection des neurones dans les glomérules est ciblée : tous les neurones qui expriment le même récepteur convergent vers un même glomérule, ce qui confère une dimension spatiale à la reconnaissance stéréochimique dans le bulbe olfactif, car l'activation d'un neurone olfactif n'entraîne que celle des glomérules qui sont connectés aux neurones qui expriment le même récepteur. Comme leurs axones forment des synapses avec les cellules mitrales, qui à leur tour forment des synapses avec des assemblées de neurones à des sites spécifiques dans de multiples aires du cortex olfactif, se crée une autre carte sensorielle stéréotypée dans le cortex olfactif [46]. Le codage de l'odeur résulte de l'activation combinatoire d'un ensemble de récepteurs fixant différents odorants, aboutissant à une topographie d'activation dans le cortex. Sachant que les neurones olfactifs, dont la durée de vie est de deux mois environ, se renouvellent sans cesse, la constance des connexions est cruciale. Ce sont là encore des RO, exprimés cette fois dans les axones, qui guident le câblage axonal [47].
3.2 Effet de la concentration sur le codage de l'odeur
La notion d'odotope et celle de carte odotopique permettent d'expliquer pourquoi une même molécule pure peut présenter des notes olfactives variées à des concentrations différentes. C'est le cas de l'indole, qui exhale une odeur de jasmin à faible concentration et une odeur fécale à forte concentration, ou encore celui du décanal qui, à faible concentration, a une odeur plaisante, fraîche, d'orange ou de citron, et, à forte concentration, une odeur désagréable et oppressive de bougie et de graisse. Comme les différents récepteurs n'ont pas la même affinité pour les différents odotopes d'un odorant, l'ensemble des récepteurs activés dépend de la concentration de l'odorant [48]. En conséquence, la carte odotopique résultant de l'activation des récepteurs change en fonction de la concentration, et la note de l'odeur évoquée varie en conséquence.
4 Génomique et génétique des récepteurs olfactifs
4.1 Classification et localisation chromosomique des gènes de récepteurs olfactifs
La comparaison des séquences des gènes des RO en permet une classification [7]. En prenant certains critères d'homologie de séquence, on aboutit à 17 sous-familles [49,50] ; certains récepteurs d'une même sous-famille n'ont toutefois que 20% d'identité de séquence d'acides aminés [51], ce qui illustre l'étendue de leur divergence, alors que d'autres ne diffèrent que par un très faible nombre d'acides aminés. Il convient de retenir deux grands sous-ensembles de gènes, les uns apparentés à ceux des poissons (classe I), bien qu'aussi exprimés chez les mammifères, et les autres uniquement présents chez les animaux aériens (classe II), observés à partir des amphibiens [52,53]. Chez un ancien vertébré, la lamproie, on observe une troisième classe distincte de RO [54].
Chez l'être humain, il y a environ 350 gènes potentiellement fonctionnels et 450 pseudogènes, c'est-à-dire des gènes qui ont subi des mutations invalidantes, soit rendant leur expression impossible, soit altérant la fonction des protéines exprimées [13]. Comme chez les autres mammifères, on observe, dans notre espèce, des gènes de classe I, dont au moins un a été démontré être fonctionnel [27], et, de manière générale, ces gènes comprennent moins de pseudogènes (52%) que ceux de la classe II (77%) [55]. La classification systématique de ces gènes est difficile, puisque l'homologie moyenne n'est que de 27% [13]. Une classification fondée sur la proximité de séquence [55] est consultable sur le site Web du Human Olfactory Receptor Data Exploratorium (http://bioinformatics.weizmann.ac.il/HORDE) [56], cependant que toutes les séquences disponibles de gènes de RO sont accessibles sur le site de l'Olfactory Receptor Database (http://senselab.med.yale.edu/senselab/ORDB/default.asp) [57]. Les gènes des RO se trouvent localisés sur tous les chromosomes, sauf les chromosomes 20 et Y [58]. Il n'y a donc pas de dimorphisme sexuel au niveau de ces récepteurs. Cependant, la répartition des gènes sur les chromosomes est loin d'être uniforme : six chromosomes portent les trois quarts des gènes de RO.
4.2 Évolution des récepteurs olfactifs
Pour mieux appréhender l'olfaction humaine, il est intéressant de comparer les génomes olfactifs au cours de l'évolution. La comparaison du nombre de gènes et de pseudogènes est permise par la disponibilité des génomes actuellement séquencés [53]. Si l'on se réfère aux gènes potentiellement fonctionnels, les Poissons possédaient déjà une bonne centaine de RO et, lorsque les Vertébrés ont quitté le milieu aquatique, le nombre de gènes fonctionnels s'est considérablement accru, avec 400 gènes chez un Amphibien, le Xénope, pour culminer chez les Mammifères, chez lesquels il semble atteindre un optimum avec la Souris, dotée de 1040 gènes, le Chien de 1100 gènes, et surtout le Rat de 1500 gènes [59]. Il est frappant de constater que, quand leurs narines se sont éloignées du sol, c'est-à-dire quand les primates se sont redressés et quand les oiseaux se sont envolés, nombre de gènes olfactifs ont progressivement perdu leur fonction, vraisemblablement parce que l'éloignement de la source principale d'odeurs qu'est le sol a fait perdre de l'importance à l'odorat, en contrepartie du développement de la vision. Rouquier et al. [58,60] ont montré qu'une réduction significative des gènes de RO s'observe chez les primates prosimiens (les « singes de l'ancien monde », les plus évolués), alors que les primates inférieurs (ou « singes du nouveau monde ») possèdent moins de pseudogènes, ce qui a été confirmé par Gilad et al. [61]. Le nombre de gène de RO potentiellement fonctionnels n'est plus que de 350 chez l'Homme et de 80 chez le Poulet. Sans pression de sélection, une dérive génétique par perte de fonctionnalité de nombreux gènes de RO serait responsable d'une évolution très rapide. On considère maintenant que ce sont les gènes des RO qui évoluent le plus vite dans l'espèce humaine. Cependant, la récente comparaison des génomes de l'Homme et du Chimpanzé a montré qu'à côté de cette pseudogénisation, un sous-ensemble de RO a évolué sous une pression de sélection positive, que ce soit chez le Chimpanzé comme chez l'Homme, depuis la séparation de ces deux espèces [62], ce qui soutient l'hypothèse selon laquelle l'olfaction joue toujours un rôle biologique important dans notre espèce.
4.3 Variabilité génétique des récepteurs olfactifs
Chez l'être humain, la perception sensorielle de l'androsténone, un androgène à 19 carbones doté d'une odeur particulière, est sujette à une importante variation individuelle. Parmi les adultes, approximativement 50% ne perçoivent aucune odeur, même à forte concentration. En revanche, 15% détectent une odeur subtile, non gênante voire éventuellement plaisante. Les 35% restants sont extrêmement sensibles à l'androsténone, détectant moins que 200 parties par trillion dans l'air ; ils attribuent une odeur pestilentielle à ce stéroïde. Cette variation individuelle a un déterminisme génétique [63]. L'étude des allèles des RO, bien que difficile en raison du nombre de gènes et de leur ressemblance, a été abordée par Menashe et al. [64], qui ont étudié le polymorphisme nucléotidique de RO humains d'individus d'origines ethniques différentes. En génotypant 51 gènes de RO chez près de 200 personnes, ces auteurs ont démontré, d'une part, que les individus étaient tous différents (aucun ne possédant la même distribution de gènes fonctionnels) et, de l'autre, que certains gènes étaient plus fréquemment invalidés dans une population que dans une autre [65]. Les gènes de RO sont apparus comme le cas le plus prononcé de diversité fonctionnelle du génome humain, suggérant que des pressions de sélection différentes ont modelé les répertoires chimiosensoriels dans des populations distinctes. Cette étude a révélé une ségrégation significative, qui peut expliquer les différences de sensibilité olfactive entre populations.
5 L'être humain est-il microsmique ?
Bien que cette dérive génétique ne soit pas uniquement négative [62], on peut s'interroger sur les conséquences de cette pseudogénisation des gènes de RO sur l'odorat de l'espèce humaine. En particulier, comme il est convenu de dire que l'être humain est un piètre opérateur olfactif, il est légitime de se demander s'il est réellement microsmique, terme désignant un être doté d'un odorat de piètre efficacité. Cette hypothèse peut être clairement invalidée [66]. Tout d'abord, on peut souligner que, dans certains cas, l'Homme a un meilleur odorat que le Chien. Par exemple, si on compare les seuils olfactifs d'une série d'aldéhydes aliphatiques, les Chiens ne sont meilleurs que l'être humain que pour les courtes chaînes, alors que l'Homme est aussi performant, sinon meilleur, pour les aldéhydes à longues chaînes ; dans ce cas, il s'avère aussi meilleur que le Rat [67]. La comparaison des séquences de gènes de RO humains avec celles des autres espèces permet d'identifier des gènes orthologues dont les séquences diffèrent peu, et indique qu'ils ont vraisemblablement la même fonction [58,68], comme de récentes études in vitro portant sur un récepteur de classe I l'attestent [27]. La comparaison des gènes de l'Homme et de la Souris [50] montre que, bien que moins nombreux, les gènes humains potentiellement fonctionnels ont des orthologues murins répartis de manière régulière dans presque toutes les sous-familles. Ce qui indique que les êtres humains ont un espace olfactif peu différent de celui de la Souris et que notre système olfactif a conservé la capacité à identifier un vaste spectre de molécules chimiques, même si plus de la moitié de ses gènes ont été invalidés. En outre, il convient de souligner que les bulbes olfactifs humains ont la même taille, en valeur absolue, que ceux du Rat, et, de plus, qu'il y a 8000 glomérules chez l'Homme, pour seulement 2500 chez le Rat. Enfin, l'Homme a un cortex autrement développé pour traiter l'information. L'être humain n'est donc pas microsmique, mais la vision et l'audition sont devenues des sens majeurs, reléguant l'olfaction qui, faute d'entraînement suffisant, s'avère sous-employée.
6 Conclusions
Bien qu'actuellement la très grande majorité des RO soient encore orphelins, la convergence des résultats des disciplines moléculaires, cellulaires et neurophysiologiques permet de valider définitivement le modèle de perception des molécules odorantes au niveau périphérique et d'exclure d'anciennes théories spécieuses, comme la théorie vibrationnelle soutenue par Turin [69]. Cette théorie, par ailleurs soumise à des tests expérimentaux soigneusement conduits, a été définitivement invalidée expérimentalement [70]. Ce sont donc les aspects stéréochimiques des interactions odorant–RO qui président à l'élaboration des odeurs. Ces résultats montrent aussi que l'odorat n'est pas un sens analytique, mais synthétique, qui fonctionne par rapport à un référentiel complexe fondé sur la mémorisation de cartes odotopiques résultant de l'activation peu spécifique d'ensembles de RO.
Les mécanismes mis en jeu dans la reconnaissance des odorants sont cependant différents de ceux du système immunitaire : il s'agit d'une reconnaissance combinatoire floue fondée sur un relativement faible nombre de protéines, par opposition à une reconnaissance extrêmement spécifique reposant sur des millions de protéines différentes, les anticorps. L'intégration sensorielle dès le niveau périphérique empêche de distinguer un mélange d'odorants d'un mélange d'odotopes ; c'est vraisemblablement pourquoi même les nez les plus entraînés ont la plus grande difficulté à distinguer et à identifier les composants d'un mélange de plus de trois composants purs, malgré un entraînement intensif. En revanche, grâce à sa nature combinatoire, ce système possède un avantage considérable, car il autorise une adaptation à la détection de toute nouvelle molécule.
La nature synthétique de l'olfaction repose aussi sur une capacité à mémoriser des cartes odotopiques correspondant à des mélanges très complexes et à les reconnaître, vraisemblablement par un système analogue à la gnosie des visages, processus de reconnaissance globale. Elle pose cependant des difficultés pour classer les odeurs [2], conséquences de la variabilité génétique considérable des RO. Cette diversité génétique est vraisemblablement à l'origine de l'incapacité de nommer une odeur et explique l'absence de sémantique commune pour l'odorat, par incapacité à verbaliser des sensations olfactives qui diffèrent d'une personne à l'autre, aboutissant à une incontournable subjectivité pour décrire des perceptions olfactives [64,65]. C'est pourquoi la mise en place de panels de dégustation fiables et efficaces reste un souci incontournable. De même, la variabilité des populations due à la ségrégation des gènes des RO explique pourquoi l'industrie agroalimentaire est contrainte de développer des produits ciblés. Cette variabilité génétique est en outre compliquée par une variabilité culturelle, qui prend son origine dès le plus jeune âge.
Avant de conclure, il est important de rappeler que le rôle des RO n'est pas cantonné à l'olfaction : comme on l'a vu, ces protéines participent au guidage axonal [71] et au chimiotactisme des spermatozoïdes. Il est probable qu'on leur découvrira des rôles nouveaux, notamment dans les migrations cellulaires impliquées dans le développement embryonnaire.
L'ensemble de ces fondements scientifiques nouvellement acquis en matière d'olfaction constitue désormais un tremplin pour la recherche et le développement de nombreuses industries, de l'agro-alimentaire à la cosmétique en passant par la parfumerie, ou encore le développement de biocapteurs [72]. Cependant, l'étude des RO n'en est en fait qu'à ses débuts et bien des questions restent en suspens, en particulier pour comprendre les phénomènes de cinétique d'activation très rapide, et surtout de désensibilisation, qui, chez les RCPG, peuvent être de nature très variée [73].


