Abridged English version
Salinity and drought represent the major environmental constraints that limit crop production and yield in Tunisia. Since grapevine production and planting area are greatly affected by soil salinity and drought, it is of agricultural importance to analyze and improve the tolerance of grapevine to these stresses. So far, attempts for grape productivity improvement are mainly focalized on the processes of berry development and ripening, pathogens and diseases resistance. However, little or poor achievements have been accomplished regarding the improvement of the tolerance of grapevine to abiotic stresses. Among strategies leading to the genetic improvement and adaptation of plants to these stresses, there is the identification and the transfer of genes involved in the tolerance to drought and salinity. Although many genes are induced by drought and salinity, only a few of them seem to be efficient for this stress tolerance. In this respect, we developed a candidate gene approach to identify such genes using a berry cDNA library (var. Cabernet Sauvignon) at the ‘véraison’ stage. The conditions for PCR amplification were 94 °C for 2 min, followed by 30 cycles at 94 °C for 1 min, 54 °C for 30 s, 72 °C for 2 min, and 72 °C for 10 min for the final extension step. On the basis of the sequences of the amplified fragments and in order to get both ends of the cDNA, additional nested primers and universal SP6 and T7 primers were used for asymmetric PCRs on the cDNA library. The assembly of contiguous sequences provided a full-length cDNA of 1.358 kb. Thus, we have identified and cloned ‘Vvrd22’, a gene encoding a response to dehydration protein from var. Cabernet Sauvignon. The predicted ORF encodes a protein of 364 amino acids with a calculated molecular mass of 39.2 kDa that shared similarities with rd22 proteins from other plants (i.e. 73% with Bruguiera gymnorrhiza). The physicochemical and structural properties of the putative protein deriving from this cDNA have been characterized using bioinformatics tools. The structural parameters, the conserved domains and the post-translational modification sites identified are of great importance in understanding the function of this protein. Similarly to other rd22 proteins in higher plant, the Vvrd22 protein contained the conserved BURP domain. RT–PCR was used to examine Vvrd22 expression during berry development as well as in the organs and tissues of the plant. In this respect, var. Cabernet Sauvignon berries were harvested from a vineyard in Bordeaux (France) between June and September in order to obtain samples corresponding to the five phenological stages of the berry development. Roots and leaves were sampled from plants grown under hydroponic conditions. For first-strand cDNA synthesis, RT was run with reverse transcriptase on 2 μg of total RNA (denatured at 75 °C for 10 min) at 42 °C for 60 min. After completion of first-strand cDNA synthesis, 5-μl aliquots were taken for PCR. These RT–PCR amplifications allowed concluding that Vvrd22 is constitutively expressed among all tissues and organs of grape, but at a very low level. Rooted cuttings were submitted to salt and drought stress. Northern analysis showed that Vvrd22 mRNA is induced by salt stress as well as by water deficit. Indeed, salt treatment (100 mM NaCl) was able to induce early and late expression of Vvrd22 in a tolerant variety (Razegui), but not in the sensitive one (Syrah), suggesting that this gene is involved in the response to salt stress. Moreover, under control condition, Vvrd22 displayed constitutively a higher level of expression in the tolerant variety than in the sensitive one. Regarding drought, Vvrd22 is induced in both tolerant and sensitive varieties. Since the plant hormone abscisic acid (ABA) is produced under water-deficit conditions and is instrumental in the development of tolerance against drought, we studied the expression of Vvrd22 in berry cell suspensions under both ABA and sucrose treatments. The grape berry cell suspension derived from Cabernet Sauvignon berries was maintained at 25 °C in an orbital shaker (100 rpm) by weekly subculture. At the exponential growth phase, ABA (10 μM) and/or sucrose (58 mM) were supplemented to the medium. Northern blots showed that ABA could poorly induce Vvrd22. However, combined with sucrose, ABA could enhance the expression of Vvrd22, even if it is still at a low level. Molecular cloning, preliminary characterization and further overexpression of this protein through genetic transformation will allow the validation of its function and consequently would promote the improvement of the tolerance of grapevine to drought and salinity.
1 Introduction
L'amélioration génétique classique des cultures s'est donnée, pendant longtemps, pour objectif primordial l'augmentation de la productivité. Toutefois, le succès de cette stratégie d'amélioration est étroitement lié à l'existence de conditions pédoclimatiques favorables. Or, le territoire tunisien est en grande partie marqué par des contraintes de déficit hydrique et de salinité [1,2], qui entravent le bon développement des cultures en général et celui de la vigne (Vitis vinifera L.) en particulier. Outre la dégradation de la structure des sols et les baisses substantielles de la productivité des vignobles, ces conditions stressantes engendrent des effets dépréciatifs sur la qualité du raisin, et par conséquent des problèmes quant à sa commercialisation [3,4]. Les efforts déployés pour améliorer la tolérance des plantes à la sécheresse et à la salinité à travers les travaux de sélection classique n'ont eu jusqu'à aujourd'hui qu'un succès très limité, en raison de la complexité génétique des espèces et de la lourdeur de la technique [5,6]. Des progrès sont actuellement initiés par le biais d'études de génomique comparative au sein d'organismes modèles, et à travers l'utilisation de techniques nouvelles telles que l'analyse d'EST (étiquette de gènes exprimés), l'analyse parallèle de l'expression d'une large gamme de gènes (microarray), de mutagenèse ciblée ou aléatoire… [5,7]. L'environnement dans lequel les plantes sont cultivées n'offre généralement pas des conditions optimales de croissance [8]. De ce fait, et en guise de réponse et d'adaptation à ces conditions stressantes, les plantes ont élaboré des mécanismes et adopté des stratégies de tolérance et de survie complexes, qui, au niveau moléculaire, impliquent le fonctionnement coordonné d'un grand nombre de gènes [9]. Plusieurs gènes ont déjà été identifiés chez les céréales, les cultures maraîchères, et particulièrement chez la plante modèle Arabidopsis thaliana. Cependant, chez la vigne, les recherches ont été plutôt axées sur les aspects qualitatifs de la baie de raisin et phytosanitaires de la plante, délaissant quelque peu les conditions environnementales de culture. Pourtant, ce dernier volet représente en Tunisie une des contraintes les plus dépressives sur le développement de la vigne, sur les rendements et la qualité des raisins. Au niveau du laboratoire de physiologie moléculaire de la vigne (centre de biotechnologies de Borj-Cédria, Tunisie), nous avons axé nos recherches sur l'identification, l'isolement et la caractérisation de gènes impliqués dans la tolérance aux stress salin et hydrique chez la vigne. L'identification et la validation de la fonction de tels gènes représentent un préalable pour les exploiter dans un programme d'amélioration génétique. À cet égard, nous avons développé une approche gène candidat qui consiste d'abord en une exploration bibliographique de gènes pertinents dont l'efficacité au niveau de la tolérance aux stress abiotiques a été déjà prouvée chez diverses espèces végétales ; le principe est ensuite de se baser sur l'homologie des séquences et d'essayer de les identifier chez la vigne en les amplifiant par PCR, et enfin de les isoler par clonage dans des vecteurs adéquats pour des études de génomique fonctionnelle. La découverte de nouveaux gènes, la détermination de leur modalité d'expression ainsi qu'une meilleure compréhension de leurs rôles dans l'adaptation aux stress pourraient conduire à l'établissement des bases effectives d'une stratégie conférant aux plantes une meilleure tolérance au stress [5,10]. En effet, la disposition d'une base moléculaire et d'un pool génétique varié constitue une étape indispensable avant d'entamer des travaux de génomique fonctionnelle et de transformation génétique visant à améliorer la tolérance des plantes aux contraintes environnementales. Dans ce cadre, nous avons identifié et isolé un gène de réponse à la déshydratation rd22, dont nous avons réalisé une caractérisation moléculaire et étudié l'expression sous différentes conditions, afin de pouvoir l'exploiter efficacement dans un programme d'amélioration génétique de la tolérance aux stress abiotiques chez la vigne.
2 Matériel et méthodes
2.1 Matériel végétal
2.1.1 Cultures en plein champ et hydroponique de la vigne
Les baies de raisin de Vitis Vinifera var. cabernet sauvignon, utilisées pour les études d'expression du gène Vvrd22 au cours du développement, ont été prélevées dans le vignoble du domaine Inra du Grand Parc de Bordeaux. Les prélèvements ont eu lieu entre les mois de juin et septembre 2000, afin de pouvoir disposer de cinq stades représentatifs du développement de la baie de raisin. Les stades 1, 2, 3, 4 et 5 correspondent respectivement à 3, 6, 8, 10 et 12 semaines après la floraison. Les baies sont débarrassées de leur pinceau, puis sont immédiatement congelées dans de l'azote liquide et stockées à , jusqu'à leur utilisation [11]. Ultérieurement, les baies ont été pesées au laboratoire pour homogénéiser les différents poids à chaque stade. Les études d'expression sur la pellicule et sur la pulpe ont nécessité de peler la pellicule des baies et de séparer la pulpe des pépins sur le lieu de récolte. En ce qui concerne les racines et les feuilles, les prélèvements d'organes ont été effectués sur des boutures fructifères de la même variété cabernet sauvignon, cultivées en conditions hydroponiques [11].
2.1.2 Bouturage ligneux
L'étude de l'expression des gènes par « Northern Blot » a été effectuée sur deux variétés de vigne à comportement contrasté, l'une, Razegui, tolérante, et l'autre, Syrah, sensible au sel. Les plantes issues de bouturage ligneux effectué en décembre sont enracinées et repiquées en pot individuel pour subir graduellement le stress salin (100 mM NaCl) [12]. Des rameaux lignifiés ont été prélevés sur des ceps de vigne dont les bourgeons sont dormants (novembre–décembre). Ensuite, ceux-ci sont débités en fragments ligneux portant deux bourgeons. Après éborgnage, les boutures sont trempées à leur base dans de l'exubérone (solution d'AIB commerciale) afin de favoriser la rhizogenèse, puis paraffinées et repiquées dans des cagettes remplies de sable. Les cagettes sont ensuite mises dans une chambre climatisée (température : 25 °C, humidité relative : 70%, photopériode : 16 h). Après deux semaines, les boutures commencent à débourrer et à développer le système racinaire [12]. Après enracinement des boutures, les jeunes plants ont été repiqués dans des pots individuels en plastique de 2 l contenant du sable inerte lavé avec de l'acide sulfurique concentré et rincé plusieurs fois avec de l'eau distillée. Après une période d'acclimatation de deux semaines les boutures enracinées sont arrosées avec la solution nutritive de Long Ashton [13], additionnée de 100 mM de NaCl. Le sel est apporté progressivement dans le milieu nutritif à raison de 25 mM de plus de NaCl tous les 15 jours, jusqu'à atteindre 100 mM [12]. Les échantillons foliaires sont prélevés 6 et 24 h après le début d'application de 100 mM de NaCl. Le stress hydrique a été appliqué sur les deux variétés Kahli (tolérante) et Galb Serdouk (sensible) de façon progressive, par cycles de déshydratation suivis par un arrêt total de l'irrigation pendant un mois, stade auquel les échantillons foliaires ont été prélevés [14].
2.1.3 Suspension cellulaire de vigne
Les cultures de suspensions cellulaires ont été obtenues à partir de cals de tissus fructifères de la variété cabernet sauvignon, puis stabilisées à l'obscurité dans une chambre thermostatée à 25 °C sous agitation (160 rpm) dans le milieu MS [15] additionné de 920 μl ANA (0,5 mg/l), de 1,2 ml BAP (0,1 mg/l), de 20 g/l saccharose et de 0,25 g/l hydrolysat de caséine enzymatique (Sigma, C0626) [16]. Pour les études d'expression du gène, les apports de saccharose (58 mM) et d'ABA (10 μM) ont été réalisés quatre jours après le repiquage des suspensions, afin que les cellules se trouvent en phase de croissance exponentielle [11].
2.2 Identification et clonage du gène Vvrd22
Pour identifier des gènes de vigne associés à la tolérance à la sécheresse et/ou à la salinité, une approche gène candidat a été adoptée. Celle-ci consiste en premier lieu à identifier des régions conservées à partir de l'alignement de différentes séquences de gènes d'intérêt en vue de désigner des oligonucléotides en guise d'amorces. Par la suite, en utilisant ces amorces, des réactions PCR sont réalisées sur une banque d'ADNc de baie de raisin (var. cabernet sauvignon) au stade de la véraison, en vue d'amplifier le gène recherché. Les conditions d'amplification sont les suivantes : une phase d'initiation à 94 °C pendant 2 min suivie d'une phase d'élongation de 30 cycles (94 °C : 1 min, 54 °C : 30 s et 72 °C : 2 min) et une phase de terminaison à 72 °C pendant 10 min. Les fragments amplifiés sont purifiés à partir des gels d'agarose puis clonés dans le vecteur pGEMT-easy (Promega) et enfin séquencés (Génome Express). L'obtention de l'ADNc complet est effectuée par des PCR asymétriques additionnelles en utilisant des amorces universelles SP6 et T7 et des amorces internes désignées selon la séquence de ces fragments préliminaires. L'assemblage final des séquences contiguës permet l'obtention de l'ADNc complet. C'est ainsi qu'un ADNc dénommé Vvrd22 a été identifié et isolé. Sa séquence finale a été enregistrée sous le numéro d'accession AY634282 au niveau de la base de données Genbank (NCBI).
2.3 Étude de l'expression spatio-temporelle du gène Vvrd22
L'analyse par RT–PCR semi-quantitative a été utilisée pour décrire l'expression spatiotemporelle du gène Vvrd22, mais également sa réponse à certains éliciteurs, tels que le saccharose et l'ABA.
2.3.1 Extraction de l'ARN à partir des différents organes de la vigne
L'extraction de l'ARN des feuilles et des racines a été effectuée sur des plants (var. cabernet sauvignon) provenant de cultures hydroponiques, en utilisant le protocole au bromure de cétyltriméthylammonium et la précipitation au chlorure de lithium, tel qu'ils ont été décrits par Chang et al. [17]. Les baies (var. cabernet sauvignon) destinées à l'extraction de l'ARN sont prélevées dans le vignoble et au cours des différents stades phénologiques précédemment cités. La baie de raisin est un matériel récalcitrant à l'extraction de l'ARN total, du fait de l'accumulation au cours du développement de polyphénols et de sucres. Nous avons utilisé le protocole mis au point par Boss et al. [18], qui a permis d'obtenir de bons rendements pour les stades compris entre le stade de la véraison et le stade de la récolte. Toutefois, les rendements pour les extractions des stades plus précoces (avant véraison) se sont avérés faibles. Nous avons donc opté pour un autre protocole utilisé chez la banane [19], qui nous a permis d'obtenir de meilleurs rendements pour ces stades. Afin d'éliminer les contaminations par l'ADN génomique, l'ARN total a été traité par la DNAase I (Promega) et extrait de nouveau par la méthode utilisant le phénol/chloroforme/alcool iso-amylique et précipité par l'acétate de sodium et finalement resuspendu dans de l'eau distillée stérile traitée par du diéthylpyrocarbonate. L'ARN des suspensions cellulaires de la variété cabernet sauvignon a été extrait à l'aide du kit d'extraction d'ARN, selon les recommandations du fournisseur (Qiagen).
2.3.2 Réactions de RT-PCR
Pour la synthèse du premier brin d'ADNc, la réaction de transcription inverse a été effectuée à l'aide d'une reverse transcriptase (100 unités de MMLV RNase H, Promega) sur 2 μg d'ARN total (dénaturé à 75 °C pendant 10 min) durant 60 min à 42 °C, en présence de 1,2 μM d'oligo(dT), tampon RT 5X, 4 mM DTT, 800 μM dNTPs, et 20 unités de RNAasin. Par la suite, 5 μl d'aliquots sont prélevés de cette réaction pour l'étape de PCR. La détection des transcrits Vvrd22 (200 pb) a été effectuée en utilisant les amorces sens 5′-GGACTTGAGATTTGTTAG-3′ et antisens 5′-TACGGAATAATATGACTC-3′. La PCR est constituée d'une phase d'initiation à 94 °C pendant 2 min, suivie d'une phase d'élongation de 24 cycles (94 °C : 1 min, 42 °C : 30 s, et 72 °C : 1 min) et une phase de terminaison à 72 °C pendant 10 min. Le facteur d'élongation EF1γ de la vigne (numéro d'accession : AF176496), amplifié (200 pb) à l'aide des amorces sens 5′-TCAATCTGTCTAGGAAAGGAAG-3′ et antisens 5′-GCGGGCAAGAGATACCTCAA-3′, a servi de contrôle pour ces réactions PCR (avec une température d'hybridation de 54 °C). Les produits d'amplification sont analysés par électrophorèse sur gel d'agarose (1%).
2.4 Étude de l'expression de Vvrd22 suite à l'application de stress salin et hydrique
Les feuilles ont été récoltées après l'application du stress et conservées à avant de subir l'extraction d'ARN et les analyses par Northern Blot. Dix microgrammes d'ARN total sont séparés par électrophorèse sur gel d'agarose (1%) formaldéhyde (2,3 M) dans un tampon MOPS (1 X) durant 3 h à 100 V [20]. L'ARN total est transféré sur une membrane de nitrocellulose Hybond N+ (Amersham Pharmacia biotech) dans un tampon SSC (10 X, NaCl 1,5 M, citrate trisodique 0,15 M, pH 7,0). Les étapes d'hybridation et de lavage de la membrane sont effectuées selon la procédure décrite par Höfer et al. [21]. L'hybridation est effectuée avec un fragment d'ADNc de 349 pb synthétisé par PCR. Cette sonde a été marquée au [P] selon les recommandations du Random Primed Labeling Kit (MBI-Fermentas). La révélation et la détection ont été faites par exposition de la membrane sur écran phosphor-imager (Molecular Imager FX, Bio-Rad).
2.5 Analyse bioinformatique
L'alignement des séquences et l'établissement des relations phylogénétiques ont été effectués à l'aide du programme CLUSTALX [22]. Les prédictions topologiques de structure ont été réalisées avec les logiciels TopPred2 (http://bioweb.pasteur.fr/seqanal/interfaces/toppred.html), DAS (http://www.sbc.su.se/~miklos/DAS/), TMpred (http://www.isrec.isbsib.ch/software/TMPRED_form.html) et ProtParam (http://www.expasy.org/tools/protparam.html). Les séquences ont été analysées à l'aide du logiciel DNASTAR (Expert Sequence Analysis Software, DNASTAR Inc.), et la recherche de séquences homologues a été effectuée au niveau de la base de données NCBI [23].
3 Résultats et discussion
3.1 Caractérisation de Vvrd22
L'ADNc Vvrd22 (var. cabernet sauvignon) que nous avons identifié et isolé est constitué de 1358 nucléotides et possède une région 5′ non traduite de 81 nucléotides, une région 3′ non traduite de 182 nucléotides et une région codante de 1095 nucléotides. Le cadre de lecture identifié code une protéine putative de 364 acides aminés, dont la masse moléculaire a été estimée à 39,2 kDa. La Fig. 1 montre l'organisation génomique du gène Vvrd22. Ce dernier est localisé au niveau du chromosome 4 du génome de la vigne ; il possède 10 exons et mesure 1,8 kb (the first genome sequence of an elite grapevine cultivar Pinot noir Vitis vinifera L. clone ENTAV 115). Notons qu'une autre isoforme du gène Vvrd22 a été identifiée au niveau du même chromosome, avec seulement trois exons et une longueur de 1,7 kb. La recherche dans la base de données NCBI a permis d'identifier une région, située en amont du site de départ de la transcription (SDT) du gène Vvrd22, contenant des éléments putatifs de régulation de la transcription. Cette partie génomique, adjacente à Vvrd22 et mesurant environ 0,5 kb, a fait l'objet d'une recherche d'éléments régulateurs potentiels au niveau des deux brins. Ainsi, différents éléments de régulation ont été identifiés en amont du SDT du gène Vvrd22. Les premières boîtes TATA et CCAAT sont situées 50 pb en amont du SDT. Un groupe d'éléments de réponse aux facteurs de transcription Myb/Myc est localisé directement en amont du SDT. Par ailleurs, six boîtes Myb/Myc ont été recensées sur ce fragment et sont toujours présentes conjointement sur les deux brins. Enfin, deux éléments ABRE, potentiellement requis pour la réponse à l'ABA, ont été localisés 500 pb en amont du SDT. De plus, des motifs impliqués dans l'expression au niveau de tissus spécifiques (tels que le pollen et le mésophylle) ont également été identifiés au niveau de ce fragment. La présence de tous ces éléments régulateurs suggère qu'il s'agit très probablement d'une région régulatrice et promotrice du gène Vvrd22, et que ce dernier pourrait jouer un rôle important dans la réponse aux stress hydrique et salin et que les facteurs de transcription Myb/Myc seraient impliqués dans la régulation de sa transcription ainsi que dans la signalisation du stress (via l'ABA). Cette dernière hypothèse a été effectivement démontrée chez A. thaliana [24,25]. L'étude de la séquence protéique a révélé que Vvrd22 présente un niveau de similitude élevé avec les autres protéines rd22 végétales (Fig. 2). Par ailleurs, un domaine BURP a été identifié au niveau de la séquence peptidique. La présence de ce domaine, conservé au niveau de la partie C terminale de diverses protéines végétales, laisse supposer que ce domaine joue un rôle important [26]. Ce dernier est constitué de 210 à 230 résidus d'acides aminés et est caractérisé par l'enchaînement suivant : deux aa phénylalanine au niveau de la partie N terminale, puis deux aa cystéine, et quatre motifs répétés cystéine–histidine arrangés de la manière suivante CH–X(10)–CH–X(25–27)–CH–X(25–26)–CH, où X peut être n'importe quel aa. Le domaine BURP a été identifié pour la première fois chez Brassica napus, et sa fonction biologique demeure encore inconnue [26]. La protéine possède une multitude de sites putatifs de régulation et de modification post-traductionnelles, dont une quinzaine de sites de phosphorylation (Tableau 1). La présence d'un site d'attachement du glycosaminoglycan à la position 74 indique un probable mécanisme de glycosylation de la protéine, qui deviendrait une glycoprotéine à la suite de cette modification. Les aa lysine, glycine et valine représentent ensemble 30% de la protéine totale. Les espaces entre les différentes cystéines présentes dans la protéine sont disposés de la sorte : H2N–220–C–11–C–51–C–11–C–26–C–26–C–13–COOH, permettant ainsi plusieurs possibilités pour l'édification des ponts disulfures nécessaires à l'établissement de la structure tertiaire. Par ailleurs, le motif en tandem simple VGVGKGTGVNVHAGKGKPGGGTT est répété trois fois au niveau des positions 58–79, 80–102 et 103–119. Cette répétition a été également identifiée trois à six fois chez les protéines rd22 de Prunus, Medicago, Brassica, Avena et Arabidopsis. La structure de Vvrd22 est organisée à 16,5% en hélice α, 31,3% en feuillet β et 52,2% en simple coude ou en chaîne linéaire. Bien qu'aucun peptide signal d'adressage de la protéine n'ait été identifié chez Vvrd22, celle-ci possède une région hélicoïdale transmembranaire tout au début de la séquence (aa : 2–24) pouvant jouer le rôle d'ancrage au niveau des membranes des organites et structures cellulaires, notamment le réticulum endoplasmique [27].

Organisation génomique du gène Vvrd22 (1,8 kb) au niveau du chromosome 4 du génome de la vigne (cultivar Pinot noir Vitis vinifera L. clone ENTAV 115). Boîte noire : exon ; boîte blanche : région non traduite (RNT) ; trait noir : intron.
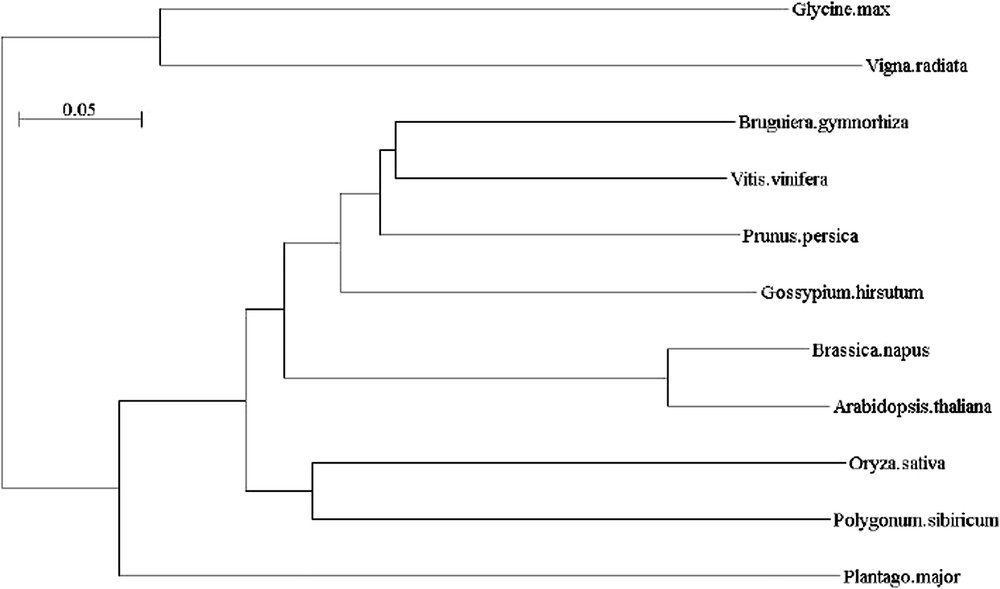
Arbre phylogénique de Vvrd22 et ses homologues (Glycine max : AAM03361, Vigna radiata : BAC22500, Bruguiera gymnorhiza : BAB60848 ; Prunus persica : AAL26909, Vitis vinifera : AAV36561, Gossypium hirsutum : AAQ22345, Brassica napus : AAC15700, Arabidopsis thaliana : BAA01546, Orysa sativa : BAF06075, Polygonum sibiricum : ABH09493, Plantago major : CAH59196).
Inventaire des sites putatifs de modification post-traductionnelle chez Vvrd22
| SITE | Attachement du glycosaminoglycan | Phosphorylation par tyrosine kinase | Phosphorylation par protéine kinase C | Phosphorylation par caséine kinase II | N-Myristoylation |
| Position | 74 | 131–261 | 191–203–213–217–245–302 | 132–164–217–235-239–257 | 55–75–81–98–104–124–312 |
3.2 Étude de l'expression de Vvrd22 chez la vigne
Vvrd22 s'exprime de manière constitutive dans tous les tissus analysés, mais à un niveau réduit (Fig. 3). Il est transcrit de manière basale dans les feuilles (jeune et adultes), racines et baies à tous les stades, mais, cependant, à un moindre degré au niveau des racines. Il ne semble pas être spécifiquement lié à un stade de développement particulier de la plante, mais son expression serait probablement induite sous des conditions de stress hydrique et/ou osmotique. Le profil d'expression des gènes rd22 est variable tant sur le plan quantitatif que spatial [28]. Chez le soja (Glycine max), les gènes rd22 Gm3 et Gm4 sont principalement exprimés au niveau des feuilles sénescentes et quasiment absents des autres organes de la plante, alors que Gm5 est plutôt exprimé au niveau des fleurs, bourgeons végétatifs et graines [28]. Chez A. thaliana, le gène rd22 est spécifiquement exprimé au niveau de la fleur et de la graine [29,30]. Chez la pomme de terre (Solanum tuberosum), celui-ci semble être exprimé au niveau des méristèmes du tubercule dormant, mais réprimé au cours de la levée de dormance [31].
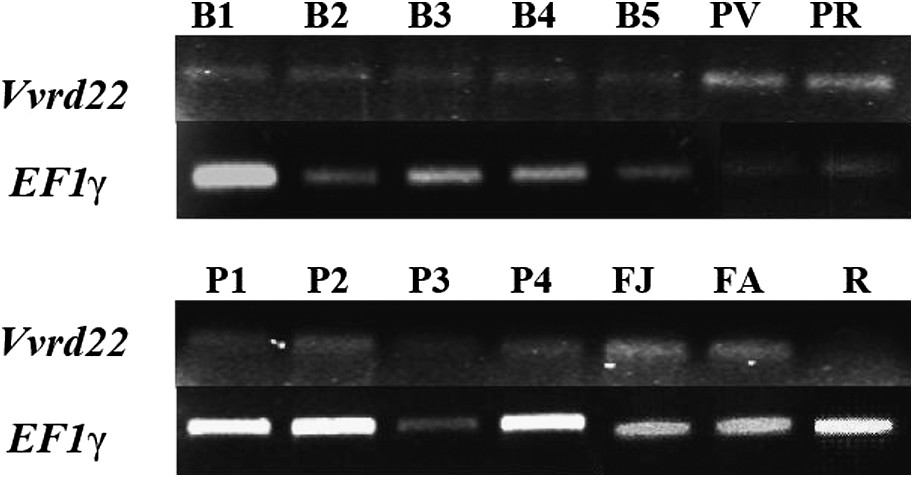
Étude par RT–PCR du niveau d'expression de Vvrd22 au cours des différents stades du développement de la baie et dans différents organes de la vigne (Bi : baie au stade i, Pi : pépin au stade i, PV : pellicule verte, PR : pellicule rouge, FJ : feuille jeune, FA : feuille adulte, R : racine). Le facteur d'élongation EF1γ est utilisé en guise de contrôle interne.
3.3 Étude de l'expression de Vvrd22 en conditions de stress salin et hydrique
Les analyses par Northern Blot (Figs. 4 et 5) montrent que Vvrd22 est induit par les stress salin et hydrique. Le stress hydrique induit l'expression du gène Vvrd22 à la fois chez les variétés tolérante et sensible, avec toutefois un niveau d'expression plus intense chez la variété tolérante. En revanche, le stress salin induit l'expression de Vvrd22 uniquement et spécifiquement chez la variété tolérante. Cette réponse est rapide, elle est visible après 6 h de stress et se poursuit en s'intensifiant après 24 h. On constate, par ailleurs, que chez la plante contrôle de la variété tolérante Razegui, Vvrd22 affiche un niveau d'expression constitutif supérieur à celui de la plante contrôle de la variété sensible Syrah. Ainsi, Vvrd22 semble être différemment exprimé au niveau des variétés tolérante et sensible aussi bien en condition standard qu'en conditions de stress hydrique ou salin. Il serait par conséquent impliqué dans la réponse et la tolérance aux stress hydrique et salin chez la vigne. La régulation du niveau d'expression serait un facteur déterminant pour la tolérance au stress. L'expression de rd22 en réponse aux stress hydrique et salin a été également démontrée chez A. thaliana [25,30]. Chez Bruguiera gymnorrhiza, Banzai et al. [32] ont constaté, consécutivement à l'application d'un stress salin (500 mM), une surexpression de gènes homologues à rd22 au niveau des feuilles apicales, contrairement aux feuilles basales.
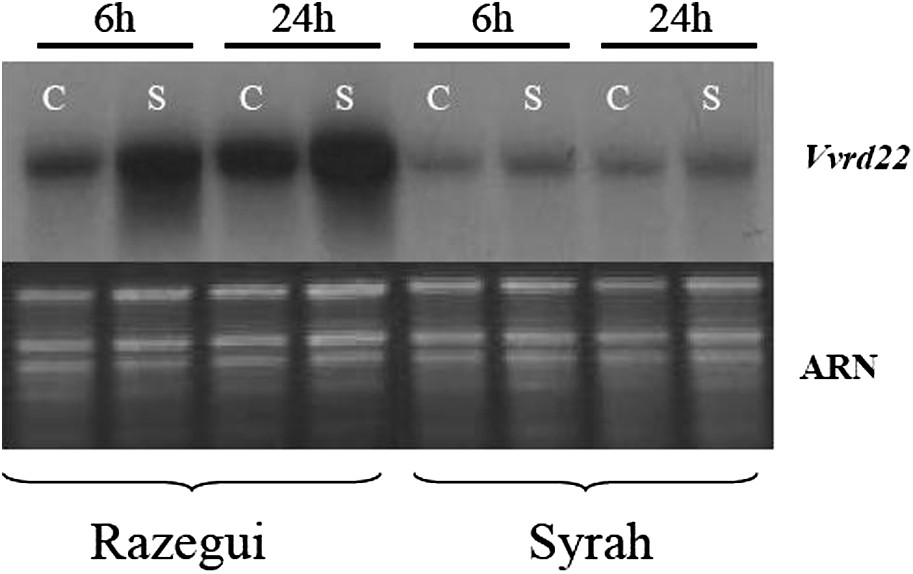
Analyse par Northern Blot de l'expression du gène Vvrd22 consécutive à l'application d'un stress salin chez les variétés Razegui (tolérante) et Syrah (sensible). C : Plante contrôle, S : plante stressée. Les analyses ont été effectuées 6 et 24 h après l'application du stress.
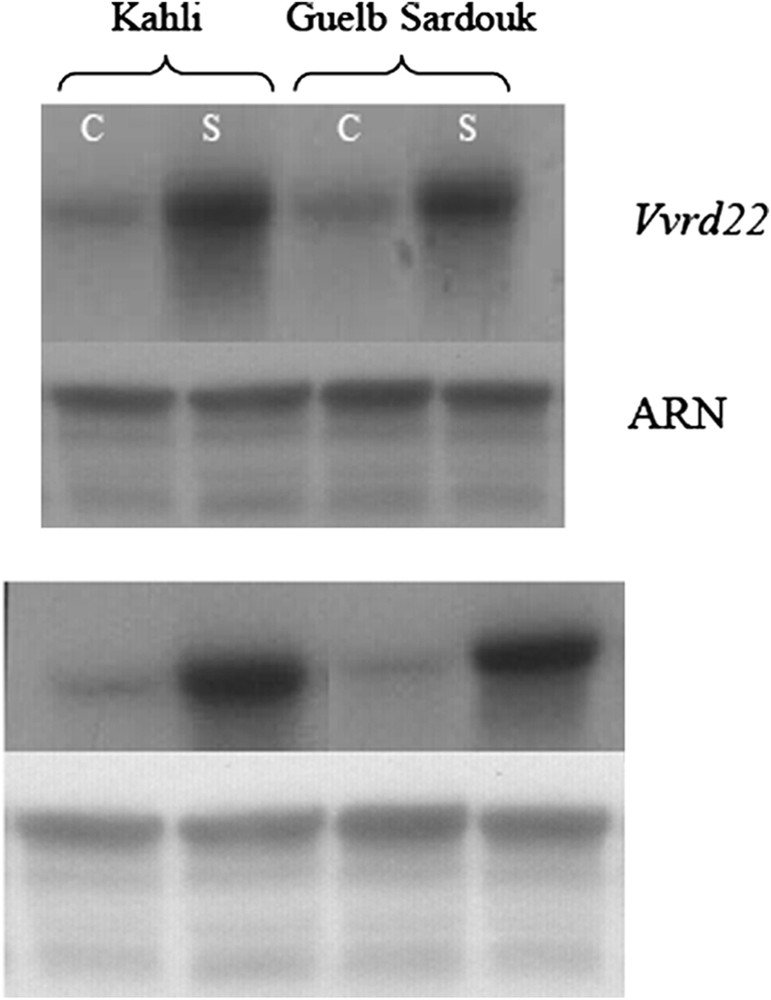
Analyse par Northern Blot de l'expression du gène Vvrd22 suite à l'application d'un stress hydrique chez les variétés Kahli (tolérante) et Guelb Serdouk (sensible). C : Plante contrôle, S : plante stressée.
3.4 Étude de l'expression de Vvrd22 en réponse à l'ABA et/ou au saccharose
Ayant démontré que Vvrd22 est induit par les stress salin et hydrique, nous avons réalisé des essais préliminaires pour rechercher les voies de signalisation de ces stress. Sachant qu'une des voies de signalisation des stress hydrique et salin pouvait suivre celle de l'ABA [33–35], nous avons, d'une part, étudié l'effet de cette hormone sur l'expression du gène Vvrd22 et, d'autre part, comme il est également connu que le saccharose induit une tolérance à la déshydratation des tissus et cellules végétales cultivés in vitro [33,36], nous l'avons combiné avec l'ABA dans nos essais.
3.4.1 Utilisation de l'ABA
Nos résultats ont montré que le gène Vvrd22 est faiblement induit par 10 μM d'ABA (Fig. 6). Chez A. thaliana, une forte induction du gène rd22 a été rapportée à partir de 40 μM d'ABA [25,37,38], ce qui expliquerait la faible induction de Vvrd22, avec seulement 10 μM d'ABA dans notre cas. Pourtant cette dernière concentration est suffisante pour induire l'expression de gènes de réponse à la déshydratation et de tolérance au stress salin chez la vigne [39,40]. Dans le cas de la mangrove, l'application d'un traitement ABA a inhibé l'expression de gènes homologues à rd22 [32]. Par ailleurs, différentes études ont mis en évidence l'expression de gènes suite à l'application de stress hydrique mais sans pour autant que ces derniers soient induits par l'ABA [29,30]. Chez A. thaliana, l'expression du gène rd22 pouvait être induite par l'ABA dans tous les organes de la plante, sauf dans la graine où elle ne semblait pas être régulée par l'ABA [30].
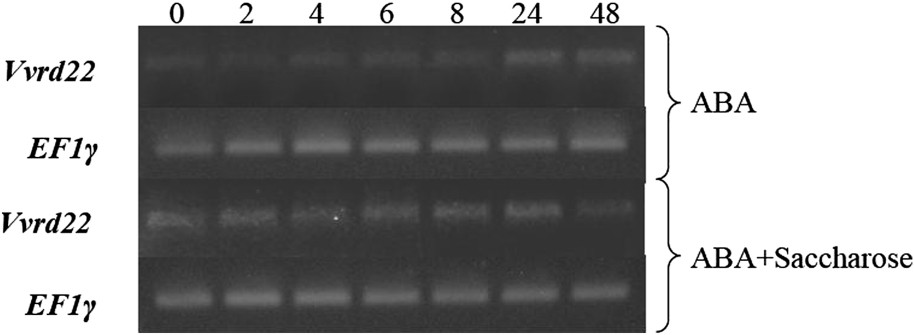
Étude par RT–PCR du niveau d'expression du gène Vvrd22 en réponse à l'application d'ABA (10 μM) et de saccharose (58 mM) sur des suspensions cellulaires de baie de raisin. Les numéros des pistes (0, 2, 4, 6, 8, 24 et 48) indiquent le temps écoulé (en h) depuis l'application du traitement.
3.4.2 Utilisation du saccharose
L'application de saccharose seul n'engendre aucune induction d'expression du gène Vvrd22.
3.4.3 Utilisation de la combinaison ABA/saccharose
L'expression du gène suit le même profil que celui du traitement contenant l'ABA uniquement, mais gagne légèrement en intensité et en rapidité de réponse lors du traitement ABA + saccharose. En effet, la Fig. 6 décrit une légère amplification du signal lorsque les deux composés sont additionnés au milieu de culture des suspensions cellulaires. Des résultats similaires ont été obtenus pour les gènes VvNHX1 [39], VvMyb [40] et VvDhn (non publié) de la vigne. Le saccharose semble interagir positivement avec l'ABA au niveau de la signalisation du stress.
4 Conclusions et perspectives
En utilisant une approche gène candidat, nous avons identifié un gène codant une protéine de type rd22 impliquée dans la réponse à la déshydratation, mais aussi au stress salin chez la vigne. Au niveau structural, celle-ci est composée de 365 aa et possède un domaine conservé BURP dont la fonction demeure inconnue, bien qu'il ait été identifié chez plusieurs protéines végétales. L'analyse bioinformatique effectuée a révélé la complexité de la régulation de cette protéine, notamment par la présence putative de multiples sites de régulation ainsi que de motifs probables de modifications post-traductionnelles. Sur le plan fonctionnel, ce gène affiche un faible niveau d'expression constitutif au niveau des différents tissus et organes de la vigne. Son expression semble plutôt être induite par les stress hydrique et salin, particulièrement chez la variété tolérante à ces stress. De plus, des molécules de signalisation de stress comme l'ABA (appliqué seul ou en combinaison avec le saccharose) induisent faiblement son expression dans des cellules de baie de raisin cultivées en suspension. Ainsi, malgré la présence d'un domaine conservé BURP chez les gènes rd22, ceux-ci affichent un profil d'expression variable et seraient par conséquent impliqués dans diverses fonctions cellulaires, notamment la réponse aux stress salin et hydrique. De ce fait, nos travaux d'identification et de caractérisation du gène Vvrd22 chez la vigne ainsi que la détermination spatiotemporelle de son niveau d'expression dans les différents organes de la plante constituent une étape primordiale avant d'entamer des travaux d'analyse d'activité et de caractérisation fonctionnelle. À cet égard, l'ADNc Vvrd22 a été cloné dans le vecteur d'expression pGREEN muni d'un promoteur constitutif P35S afin de pouvoir réaliser sa surexpression dans des plantes modèles (tabac, Arabidopsis) afin de valider sa fonction. L'ensemble de ces démarches constitue un pré-requis indispensable avant d'entamer un programme d'amélioration génétique de la vigne ou de tout autre plante d'intérêt agronomique dans le but d'obtenir des plants capables de mieux tolérer le stress hydrique et/ou salin.


