Abridged English version
The Gilthead sea bream Sparus aurata is a coastal fish species common in the Mediterranean. The genetic structure of its natural populations has been so far relatively little documented, several studies [6,7] reporting little or no differentiation, with signals difficult to interpret geographically and rarely more than one sample in the western Mediterranean (MedW). However, for several species of similar ecology, this water basin seemed to be often genetically homogeneous [9,10]. Before proceeding to comparative biology studies in S. aurata, we aimed at ascertaining this homogeneity between the southern and northern banks of the MedW. Four samples of 48 individuals each were captured in France (Sète and Thau lagoon) and Algeria (Annaba bay and Mellah lagoon) (Fig. 1). They were typed for three microsatellites loci, two of them close to the Somatotropine and the Prolactine gene loci (termed hereafter 5′PRL and 5′GH respectively) and one anonymous (TG)18. Two RAPDs primers were also scored as a supplement.
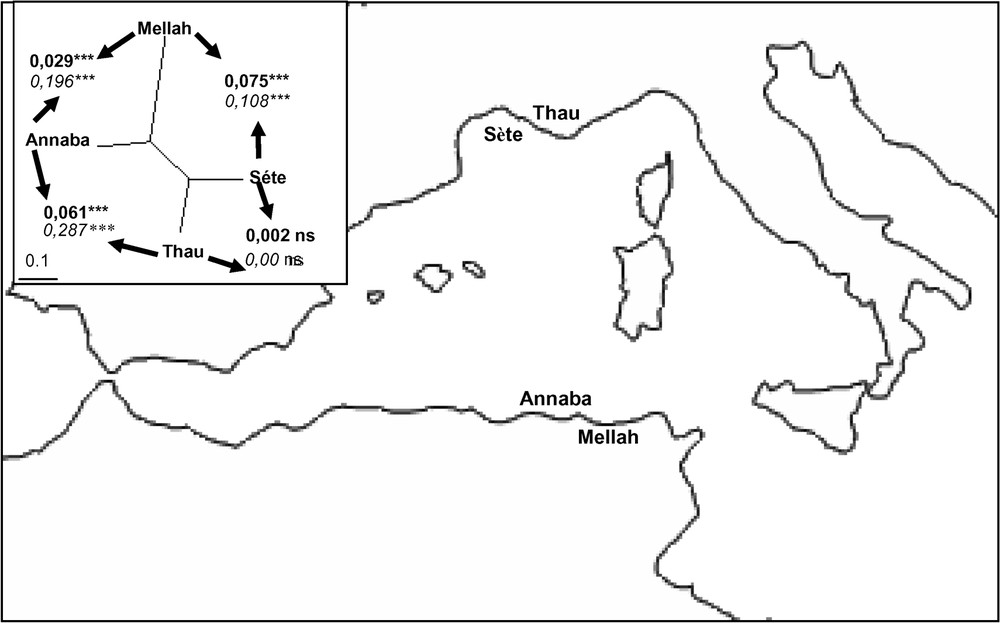
Lieux d'échantillonnage de la daurade Sparus aurata en Méditerranée occidentale. Encadré : arbre NJ sur quatre échantillons de Sparus aurata, basé sur la distance génétique de Reynolds à trois locus microsatellites (5′PRL, 5′GH et (TG)18). Les flèches indiquent les estimations du par paire d'échantillons pour les trois locus microsatellites (en gras) et les deux systèmes RAPD (en italiques).
Table 1 gives the values of total number of alleles, heterozygosity and for each microsatellite locus in each sample. Table 2 provides with the estimates of Wright's pairwise differentiation for each microsatellite locus (Weir & Cockerham Theta estimator, Genetix 4.02 software) and for the RAPDs (Lynch & Milligan estimator, AFLP-SURV 1.0 software). The North and the South of the MedW appear as quite differentiated, with a North/South average microsatellite , and a corresponding RAPDs value of , while at the same time the two geographically close sample of Sète and Thau appeared undifferentiated. In the southern bank, the Mellah and Annaba samples showed levels of differentiation varying from nil to significant according to the marker considered.
Nombre total d'allèles (), hétérozygoties attendues non biaisées () et écart à Hardy–Weinberg () pour chaque locus microsatellite dans chacun des échantillons.
| Annaba | Mellah | Sète | Thau | |
| 5′PRL | ||||
| N | 31 | 24 | 42 | 36 |
| 13 | 13 | 12 | 11 | |
| 0,908 | 0,926 | 0,887 | 0,870 | |
| 0,114⁎⁎ | 0,057 ns | 0,251⁎⁎⁎ | 0,075 ns | |
| 5′GH | ||||
| N | 18 | 10 | 35 | 36 |
| 12 | 9 | 15 | 8 | |
| 0,855 | 0,8211 | 0,902 | 0,866 | |
| 0,357⁎⁎⁎ | 0,404⁎⁎⁎ | 0,083 ns | 0,233⁎⁎⁎ | |
| (TG)18 | ||||
| N | 31 | 33 | 39 | 36 |
| 8 | 7 | 8 | 6 | |
| 0,804 | 0,773 | 0,773 | 0,744 | |
| 0,038 ns | 0,101 ns | 0,140⁎⁎ | 0,181⁎⁎ |
⁎⁎ ;
⁎⁎⁎ .
Valeurs de l'estimateur de selon [22] par paire d'échantillons aux locus microsatellites confondus et pris séparément.
| Microsatellites | ||||||||||||
| Mellah | Sète | Thau | ||||||||||
| Locus confondus | 5′PRL | GH | (TG)18 | Locus confondus | 5′PRL | 5'GH | (TG)18 | Locus confondus | 5′PRL | 5′GH | (TG)18 | |
| Annaba | 0,029 ⁎⁎ | −0,009 ns | 0,081⁎ | 0,014 ns | 0,051 ⁎⁎⁎ | 0,052 ⁎⁎⁎ | 0,068 ⁎⁎⁎ | 0,029 ⁎⁎⁎ | 0,061 ⁎⁎⁎ | 0,074⁎⁎⁎ | 0,044 ⁎⁎⁎ | 0,066 ⁎⁎⁎ |
| Mellah | 0,075 ⁎⁎⁎ | 0,035 ⁎⁎⁎ | 0,116 ⁎⁎⁎ | 0,071 ⁎⁎⁎ | 0,087 ⁎⁎⁎ | 0,054⁎⁎⁎ | 0,100 ⁎⁎⁎ | 0,106 ⁎⁎⁎ | ||||
| Sète | 0,002 ns | −0,007 ns | 0,003 ns | 0,013 ns | ||||||||
| RAPD | ||||||||||||
| Mellah | Sète | Thau | ||||||||||
| A01&A10 | A-01 | A-10 | A01&A10 | A-01 | A-10 | A01&A10 | A-01 | A-10 | ||||
| Annaba | 0,196 ⁎⁎⁎ | 0,351 ⁎⁎⁎ | 0,000 ns | 0,204 ⁎⁎⁎ | 0,310 ⁎⁎⁎ | 0,064 ⁎⁎⁎ | 0,287 ⁎⁎⁎ | 0,312 ⁎⁎⁎ | 0,177 ⁎⁎⁎ | |||
| Mellah | 0,108 ⁎⁎⁎ | 0,183 ⁎⁎⁎ | 0,064⁎⁎ | 0,178 ⁎⁎⁎ | 0,187 ⁎⁎⁎ | 0,173 ⁎⁎⁎ | ||||||
| Sète | 0,000 ns | 0,000 ns | 0,043 ns |
⁎ .
⁎⁎ .
⁎⁎⁎ ; les valeurs significatives après l'ajustement de Bonferroni sont en gras et valeurs de par paire d'échantillons aux locus RAPD A1 et A10, confondus et pris séparément, obtenues selon [20].
This differentiation between the two banks of the MedW seems to be unprecedented for similar species for which information exists. Two mutually non-exclusive hypotheses could be advanced to explain this, either S. aurata is a species which migrates and disperses much less than others both at larval and adult stages, for instance because they would be more infeodated to very coastal environments, or the loci used would be subjected to selection by differential adaptation to North/South environmental gradients. This last possibility is nevertheless difficult to admit for such locus as (TG)18 locus and the RAPDs that are in essence anonymous. Identifying which genes are implicated in the genetic differentiation between the two banks of the MedW will be an interesting challenge to evidence eventual differential selective pressures. Whatever the hypothesis retained, our study demonstrates that there does not exist a single stock at the scale of the occidental Mediterranean and that this should be taken into account for the studies undertaken at this scale in this species.
1 Introduction
La daurade Sparus aurata (L. 1758) est une espèce côtière commune en Méditerranée occidentale (MedOuest), moins fréquente à l'est et au sud-est de cette mer et très rare en Mer Noire [1].
Hormis les travaux qui concernent les populations d'élevage [2–4], la structure génétique des populations naturelles de daurade S. aurata a été jusqu'à présent relativement peu abordée. Ben Slimen et al. [5], utilisant des marqueurs allozymiques (21 locus), ont mis en évidence une différenciation géographique de cette espèce sur les côtes tunisiennes, avec des valeurs du paramètre de différenciation génétique comprises entre et entre les localités du nord et de l'est de la Tunisie, autour du détroit siculo-tunisien. Cependant, les valeurs de au sein de la Méditerranée occidentale n'étaient pas significatives (). Alarcon et al. [6], utilisant plusieurs types de marqueurs, ont montré un faible degré de différenciation globale pour sept échantillons méditerranéens et atlantiques (microsatellites : , 6 locus ; allozymes : , 16 locus ; région de contrôle de l'ADN mitochondrial : aucune variabilité), sans qu'il ne soit possible d'en donner une signification géographique claire. La présence d'un seul échantillon de Méditerranée occidentale ne permet pas, néanmoins, d'estimer l'hétérogénéité génétique à l'intérieur de ce bassin. De Innocentiis et al. [7], à l'aide de quatre locus microsatellites, ont étudié cinq échantillons de Méditerranée occidentale, un échantillon adriatique et un échantillon atlantique. Ces auteurs mettent en évidence une différenciation globale faible, mais significative, (), avec des valeurs de par paire significatives essentiellement entre l'Atlantique, l'Adriatique (représentant la Méditerranée orientale) et le reste. Au sein de la Méditerranée occidentale, seul l'échantillon situé le plus au sud (sud de la Sicile) montrait une différenciation avec les quatre autres situés plus au nord (). Cette faible différenciation entre MedOuest, détroit siculo-tunisien et Adriatique semble être confirmée par l'analyse de 26 locus enzymatiques par Rossi et al. [8] avec des valeurs de comparables (0,017).
La structuration génétique de cette espèce est donc encore, à l'heure actuelle, assez mal connue, avec peu ou pas de signal clairement interprétable du point de vue de la phylogéographie, et pour autant qu'on puisse en juger sur un échantillonnage suffisant, il semble pour le moins que les populations du bassin occidental de la Méditerranée étudiées jusque-là soient peu différenciées avec les marqueurs étudiés.
Chez d'autres poissons démersaux ayant des biologies similaires à celle de S. aurata, des informations relativement fiables existent quant à une éventuelle hétérogénéité génétique de leurs peuplements en Méditerranée. Ainsi, chez les sparidés Diplodus vulgaris et Lithognathus mormyrus provenant de huit localités Est et Ouest méditerranéennes, Arculeo et al. [9] mettent en évidence, sur une vingtaine de locus allozymiques, une différenciation génétique faible () chez D. vulgaris, alors que chez L. mormyrus ces auteurs trouvent une homogénéité génétique quasi-totale à travers toute la Méditerranée (). Par ailleurs, Lenfant et Planes [10] ont montré que les stocks de Diplodus sargus étaient génétiquement homogènes sur des distances supérieures à 1000 km en Méditerranée nord-occidentale. Bargelloni et al. [11] ont examiné la variabilité de séquence d'une partie de la région de contrôle mitochondriale et de 21 locus allozymiques, de la Méditerranée orientale aux côtes portugaises, chez D. sargus et D. puntazzo. Pour cette dernière espèce, il existe une différentiation génétique marquée entre l'Atlantique et la Méditerranée, avec un fort global ( et respectivement pour les allozymes et l'ADNmt). Un significatif est aussi observé entre MedOuest et MedEst ( pour les allozymes et pour l'ADNmt). En revanche, la présence d'un seul échantillon de MedOuest ne permet pas, là non plus, d'évaluer l'hétérogénéité dans ce bassin. Chez D. sargus, par contre, aucune différenciation globale n'est observée. Cette homogénéité est confirmée par l'étude de Domingues et al. [12], basée sur la région de contrôle mitochondriale et le premier intron du gène de la protéine ribosomale S7, qui n'a révélé aucun signe de différentiation génétique entre la Méditerranée Est, Ouest et l'Atlantique chez cette espèce. Chez un autre poisson exploitant des biotopes similaires à la daurade, le loup (ou bar) Dicentrarchus labrax (Moronidé), plusieurs études basées sur le polymorphisme de différents fragments de l'ADN mitochondrial [13–15], ne trouvent aucune différenciation entre les échantillons de MedOuest. Naciri et al. [16] et Bahri-Sfar et al. [17] confirment ce résultat en utilisant des marqueurs microsatellites. Selon ces auteurs, ces résultats soutiennent un mode de fonctionnement en métapopulation quasi-panmictique à l'échelle de la Méditerranée occidentale, causé par des flux de gènes fréquents et importants.
On voit donc que l'homogénéité génétique semble être la règle en Méditerranée occidentale, voire au-delà, pour plusieurs espèces de poissons d'écologie similaire. Ceci serait en relation avec leur capacité de déplacement à l'âge adulte, ainsi qu'à la présence d'œufs et de larves planctoniques permettant des flux géniques importants entre les populations.
Le but du présent travail est de vérifier si S. aurata, espèce pour laquelle nous ne disposons que de données fragmentaires, se conforme à ce modèle. Ceci donnerait une base robuste aux études de biologie comparée chez cette espèce. Nous avons pour cela analysé quatre échantillons en provenance du golfe d'Annaba (baie d'Annaba et la lagune de Mellah – Algérie) au sud et du golfe du Lion (Sète et étang de Thau – France) au nord, qui ont été typés pour trois locus microsatellites. Nos résultats ont été complémentés par l'analyse de 19 locus RAPD.
2 Matériel et méthodes
2.1 Echantillonnage
Quarante-huit individus ont été échantillonnés dans chaque site (Fig. 1). Sur chaque individu frais, un fragment de nageoire caudale d'environ 1 cm2 a été prélevé et conservé dans de l'alcool à 95°, afin de procéder ultérieurement à l'extraction de l'ADN.
2.2 Typage des locus microsatellites
L'ADN a été extrait avec le kit d'extraction QIAGEN (France), selon le protocole du fournisseur. L'ADN extrait est dosé et sa concentration est ajustée à 50 ng/μl.
Trois locus microsatellites ont été analysés dans les conditions PCR ci-après : S. aurata (TG)18 (poly GT) défini par [18] (SauANINRA (TG)18 R 5′-GAGCTGTAAACCCAGG-3′ et SauA (TG)18 F 5′-TGTTGGAGCTTGGCAC-3′, le volume réactionnel total = 25 μl : 1,2 mM de MgCl2 et la T° d'hybridation = 60–50 °C), le locus 5′GH avec 14 répétitions du motif de base « CA » défini par Almuly et al. [19] dans la région 5′ du gène de l'hormone de croissance et pour lequel nous avons redéfini des amorces (GHm1 5′-CCT-TAA-ATC-AGT-TCA-GCC-G-3′ et GHm2 5′-GGT-TCA-AGT-TCT-GGT-GAC-TAG-3′, le volume réactionnel total = 25 μl avec 2,25 mM de MgCl2 et une T° d'hybridation = 60–50 °C) et un locus équivalent défini par [20] dans la région 5′ du gène codant pour la Prolactine, 5′PRL, microsatellite imparfait avec le motif « CA » répété 12 fois (PRL R5′-GAAGAGTTTGCTTCCATTGG-3′ et PRL F5′-CATCCATCCAATTAAGATGG-3′, le volume réactionnel total = 25 μl : 1,2 mM de MgCl2 et une T° d'hybridation = 56 °C). Les amorces reverse des trois locus étaient rendues fluorescentes par ajout du fluorochrome 6-Fam. Les produits de PCR ont migré sur un gel d'acrylamide à 6% et ont été visualisés à l'aide d'un scanner Hitachi FMBIO2.
Par ailleurs, deux amorces RAPD : A-01 et A-10 (Operon Technologies Inc.) ont permis l'amplification de 29 systèmes de bandes présence/absence dont 19 étaient polymorphes, soit six pour l'amorce A-01 (5′-AGGGGTCTTG-3′) et treize pour l'amorce A-10 (5′GTGATCGCAG-3′). Les PCR ont été effectuées dans un volume réactionnel de 25 μl, contenant 5 μl d'extrait d'ADN, 2 mM de MgCl2, avec 44 cycles de dénaturation à 94 °C (45 sec), une hybridation à 37 °C (1 mn). Ces bandes sont visualisées par coloration au bromure d'ethidium (BET), après migration sur gel d'agarose à 2%. La durée de migration est de 3 h à 100 W.
La technique RAPD [21] est basée sur l'existence de polymorphismes dans le site d'amorçage d'un oligonucléotide décamère. Ces marqueurs sont donc dominants, car seuls deux phénotypes sont détectables : présent ou absent. Un grand nombre de fragments est généré en une seule réaction d'amplification étant donné la courte taille des oligonucléotides. Cette technique est réputée peu reproductible, cependant nous avons procédé à un grand nombre de réplicats en ne conservant que les bandes stables et répétables. Plusieurs amplifications et migrations ont été faites, en intercalant les échantillons des différentes populations.
2.3 Analyse des données
Les indices de diversité (hétérozygotie attendue non biaisée de Nei , nombre d'allèles ) et de structuration génétique ( de Wright estimé par le θ de Weir et Cockerham) ont été calculés à l'aide du logiciel Genetix 4.02 [22] à partir des données génotypiques microsatellitaires individuelles. Pour les RAPD, chaque bande est considérée comme un locus. Une matrice de présence-absence de bande (individus*locus) est ainsi obtenue, où seules les bandes polymorphes ont été prises en compte. Les individus présentant des profils anormaux, tels qu'un trop grand nombre de bandes privées, sont retirés de l'analyse. Comme les marqueurs RAPD sont dominants pour la présence, la fréquence de l'allèle récessif peut être estimée par la racine carrée de la fréquence du phénotype « absent ». Pour ces locus, nous avons estimé le par paires d'échantillons grâce au logiciel AFLP-SURV 1.0 [23] en utilisant la méthode de Lynch and Milligan [24].
3 Résultats
3.1 Diversité génétique
Le Tableau 1 donne les valeurs de diversité génique (), ainsi que le nombre total d'allèles par échantillon (). Nous constatons que le locus (TG)18 est moins riche en allèles que les deux autres locus 5′GH et 5′PRL. Le paramètre indique quant à lui la valeur des déficits en hétérozygotes qui sont relativement élevés et significatifs pour tous les échantillons et tous les locus.
Les amorces RAPD utilisées ont produit des fragments ayant des tailles comprises entre 165 et 650 pb. Parmi les 29 bandes obtenues, 19 sont polymorphes, soient six pour l'amorce A-01 et treize pour l'amorce A-10.
3.2 Structure génétique
Le Tableau 2 montre les valeurs de l'estimateur de de Weir et Cockerham [25] par paire d'échantillons aux trois locus. Une nette différenciation nord/sud est visible entre les échantillons d'Annaba et ceux de Sète et Thau d'une part ( et ) et de Mellah par rapport à ces deux mêmes sites d'autre part ( et ). Chaque locus pris séparément montre la même tendance. En revanche, cette valeur est très faible et non significative entre Sète et Thau, alors qu'elle ne montre pas de tendance claire entre Annaba et Mellah (5′PRL : ; 5′ GH : ; TG18 : ). Des résultats tout à fait comparables sont obtenus avec les locus RAPD : Les valeurs de estimées selon la méthode de Lynch and Milligan [24] montrent une différenciation hautement significative entre les échantillons d'Annaba par rapport à ceux de Sète et Thau ( et respectivement) d'une part et ceux de Mellah par rapport à ces deux sites pris séparément ( et respectivement) ; les deux systèmes d'amorces pris séparément montrent la même tendance. Les deux localités Sète et Thau s'avèrent similaires avec une valeur nulle, tandis que Annaba et Mellah montrent une différenciation soit nulle soit significative selon le système d'amorce considéré (A01 : ; A10 : ).
La Fig. 1 représente la proximité relative des quatre échantillons, basée sur la distance de Reynolds et al. [26] pour les locus microsatellites. Les valeurs de sont reportées en gras. Qualitativement, le même profil est obtenu pour les locus RAPD (valeurs en italique), même si les valeurs absolues sont plus fortes que pour les trois locus microsatellites.
4 Discussion et conclusion
La revue de la littérature disponible sur S. aurata montre des données très fragmentaires et difficiles à interpréter, avec rarement plus d'un point en MedOuest (voir Introduction). Néanmoins, contrairement à ce qu'on aurait pu attendre, nos échantillons de S. aurata des rives nord et sud de la Méditerranée occidentale apparaissent comme génétiquement très divergents ( Nord/Sud moyen = 0,069 pour les microsatellites), puisque aucune valeur obtenue à une échelle géographique équivalente ne dépasse ce chiffre pour les quelques espèces pour lesquelles il existe des données comparables (ceci est encore plus marqué pour les RAPD avec un Nord/Sud estimé à 0,194 ; il est cependant difficile de donner une signification absolue à ce chiffre). Par ailleurs, à l'échelle des quelques kilomètres qui séparent la lagune de Thau de la pleine mer au large de Sète, nous ne notons aucune divergence génétique significative, ce qui est conforme à ce que l'on pouvait attendre et pourrait signifier des flux géniques importants. En revanche entre Annaba et Mellah, l'existence ou non d'une différenciation significative dépend du marqueur considéré dans l'analyse (le locus 5′GH et le système RAPD A01 montrent une différenciation significative, les autres marqueurs étant indifférenciés).
Chez les espèces génétiquement homogènes sur de grandes distances, comme Dicentrarchus labrax [16,17] ou Diplodus sargus [10,12], c'est en général la dispersion par la phase larvaire pélagique qui est invoquée pour expliquer l'homogénéisation de proche en proche des populations. La durée de cette phase chez la daurade est estimée à 45 jours [27], ce qui est comparable à celle des autres espèces citées. Deux hypothèses non-exclusives pourraient alors a priori expliquer les résultats obtenus : soit la biologie de S. aurata est telle que les individus, à tous les stades de leur cycle de vie, migreraient beaucoup moins que les autres espèces et resteraient inféodés à des milieux très côtiers, sans beaucoup se disperser. Si cette hypothèse est vérifiée, on devrait alors également observer des valeurs de du même ordre dans l'étude microsatellitaire de De Innocentis et al. [7], ce qui n'est pas le cas. Une alternative serait que les locus utilisés ne sont pas strictement neutres et reflèteraient une adaptation différentielle aux conditions du milieu entre le nord et le sud (température, niveau trophique, etc...). Cette dernière hypothèse est cependant difficile à admettre telle quelle, étant donné que globalement le même signal est obtenu sur des microsatellites situés à proximité de gènes (Somatotropine et Prolactine) ou sur des marqueurs réputés anonymes (locus TG18 et systèmes RAPD). Ces locus anonymes peuvent représenter n'importe quelle portion du génome codant ou pas. Savoir quels gènes sont impliqués dans la différenciation génétique de la daurade Sparus aurata entre les deux rives de la Méditerranée, peut permettre de mieux appréhender la nature d'éventuelles pressions de sélection différentes.
Quelque soit l'hypothèse retenue, notre étude montre qu'il n'existe pas un stock génétiquement uniforme chez S. aurata à l'échelle de la Méditerranée occidentale. Cette information doit être prise en compte dans les études biologiques et halieutiques menées chez cette espèce. A partir de nos observations, l'étude comparée de la biologie larvaire et de la physiologie comparée de ces animaux de part et d'autre de la Méditerranée, devient dès lors un objectif intéressant.
Remerciements
Cette recherche est réalisée dans le cadre d'un projet de coopération franco-algérien (Accord-programme de coopération CMEP No 04 MDU 628), intitulé « Ichtyofaune côtière du golfe d'Annaba : diversité génétique et biologie halieutique ».


