1 Introduction
Les premières observations de structures enveloppées d’une double membrane qu’on appela autophagosomes furent réalisées grâce à la microscopie électronique sur les cellules hépatiques de souris et de rats dans les années 1960. L’observation dans ces cellules de vésicules denses emplies d’enzymes lysosomales et accolées aux mitochondries [1,2] amena quelques années après Christian de Duve à introduire le terme « autophagie ». Lors du « CIBA Foundation Symposium » dédié au lysosome en 1963, il décrivit ces vésicules à simple ou double membrane contenant des portions de cytoplasme et d’organelles à des stades variés de désorganisation sous le terme de vésicule d’autophagie (voir [3,4] pour une revue). L’implication du glucagon et de l’insuline dans l’apparition des vésicules d’autophagie fut par la suite largement documentée (voir [5–7] pour revue). Il fut aussi montré que l’autophagie était induite en situation de carence en acides aminés et que la levée de cette carence par la production d’acides aminés produits par la voie de l’autophagie exerçait un rétrocontrôle sur la voie [8,9]. À partir des observations en microscopie, les différentes structures (phagophore) impliquées dans la formation de l’autophagosome et dans sa digestion (autolysosome) furent décrites. L’autophagie décrite dans les années 1960 par de Duve et considérée au départ comme un mécanisme de dégradation non sélective de portions cytoplasmiques fut ensuite revisitée. En 1973, Bolender et al. [10] montrèrent que des fragments de réticulum endoplasmique (RE) pouvaient être séquestrés dans des autophagosomes et suggérèrent une sélectivité pour le RE. Le rôle de l’autophagie dans la dégradation spécifique de mitochondries lors de la métamorphose des insectes fut ensuite montré [11].
Ce fut l’obtention de données moléculaires chez la levure par le groupe du professeur Ohsumi qui permit d’affiner la description du processus d’autophagie. L’intervention des acteurs protéiques essentiels au processus, les protéines ATG (AuTophaGie), fut alors décrite ([12,13] pour revue). Le criblage de mutants incapables de réaliser le recyclage de leurs protéines et de leurs organites permit en 1997 d’identifier le premier gène d’autophagie ATG1.
2 Les grandes étapes de l’autophagie
Quatre types d’autophagie furent décrits :
- • la macro-autophagie, considérée comme non sélective ;
- • la cytoplasm to vacuole targeting pathway (CVT) ayant une sélectivité pour certaines protéines cibles ;
- • la mitophagie ciblant la mitochondrie ;
- • et la pexophagie ciblant les peroxisomes ([14] pour revue).
Ces quatre types d’autophagie bien que différents par leurs fonctions partagent et utilisent un ensemble de gènes ATG communs qui forment le cœur de la machinerie moléculaire. Ces gènes participent tous à la formation de l’autophagosome. L’induction du processus d’autophagie par les signaux de l’environnement impliquent des gènes de régulation qui permettent la formation de phagophores (Fig. 1). Le phagophore est la structure de nucléation de l’autophagosome composée d’une bicouche lipidique et de la structure pré-autophagosomale (« Pre-Autophagosomal Structure » [PAS]). Le PAS se compose d’un assemblage de protéines qui permettent l’élongation de la membrane du phagophore en participant au recrutement de protéines et de lipides. L’origine de la source lipidique est encore soumise à polémique. Il se pourrait que la mitochondrie, le RE, le Golgi ou encore la membrane plasmique puissent contribuer à la fourniture des lipides [15–19]. Quoi qu’il en soit, l’apport incessant de lipides permet au phagophore de s’étendre et son incurvation d’englober des portions de cytoplasmes et des organelles. La fusion des membranes entraîne la formation de l’autophagosome, [20] qui est une vacuole de petite taille délimitée par une double membrane. L’autophagosome ainsi formé voyage dans la cellule jusqu’à d’autres vésicules lytiques, lysosomes ou jusqu’à la vacuole centrale chez la levure et les végétaux. Lorsque la membrane externe de l’autophagosome fusionne avec celle de la vacuole ou du lysosome, le corps autophagique, constitué de la membrane interne de l’autophagosome et de son contenu, est libéré dans la lumière où il est dégradé. Les acides aminés et les squelettes carbonés libérés par les lipases et hydrolases sont vraisemblablement exportés vers le cytosol et recyclés.
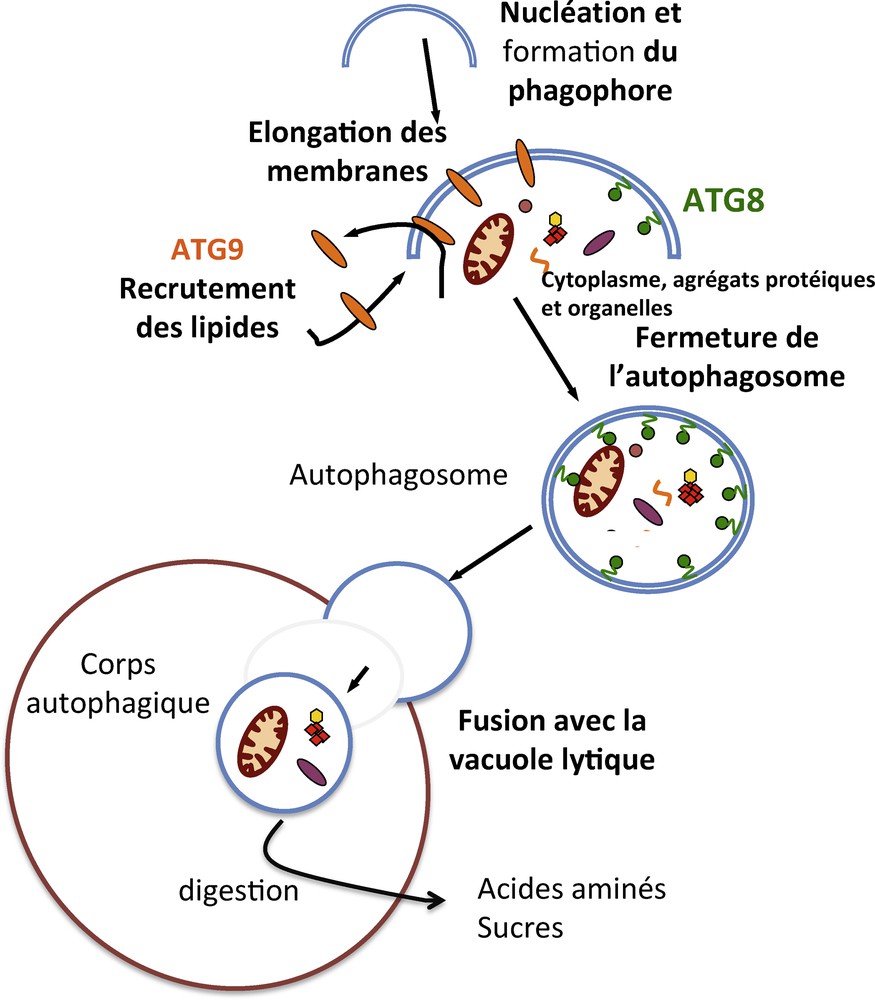
Schéma présentant les bases morphologiques de l’autophagie. L’autophagie débute par la formation du phagophore. L’origine de la membrane reste à ce jour inconnue, mais son expansion dépend de la protéine ATG9. Peu à peu des portions de cytoplasme sont séquestrées par la vésicule cytosolique à double membrane que constitue l’autophagosome. La fusion de la membrane externe de l’autophagosome à la vacuole lytique permet l’expulsion du corps autophagique dans la lumière de la vacuole. L’hydrolyse de la membrane interne de l’autophagosome libère son contenu qui est alors hydrolysé et les molécules qui en résultent sont exportées vers le cytosol via des perméases membranaires.
Adapté d’après Thompson et Vierstra [23].
3 Une machinerie moléculaire conservée chez tous les organismes eucaryotes
Les gènes de l’autophagie découverts chez la levure Saccharomyces cerevisiae au milieu des années 1990 lors d’un crible génétique visant à trouver des mutants incapables de s’adapter et de survivre à la carence nutritionnelle [21,22] ont été également identifiés chez les eucaryotes animaux et végétaux. Le nombre de gènes ATG a atteint 33 chez la levure. Parmi ces 33 gènes, certains sont dédiés aux voies spécifiques (CVT, pexophagie et mitophagie), les autres composent le cœur de la machinerie moléculaire qui participe aussi bien à la macrophagie qu’aux voies spécifiques. Ce cœur de la machinerie autophagique compte quatre groupes fonctionnels (Fig. 2). Le complexe de régulation ATG1/13 et le complexe PI3K sont importants dans l’étape d’initiation de formation de la membrane. Les systèmes de conjugaison ATG5/12 et ATG8 interviennent dans l’élongation de cette membrane et son ornementation ([23] ; Tableau 1).
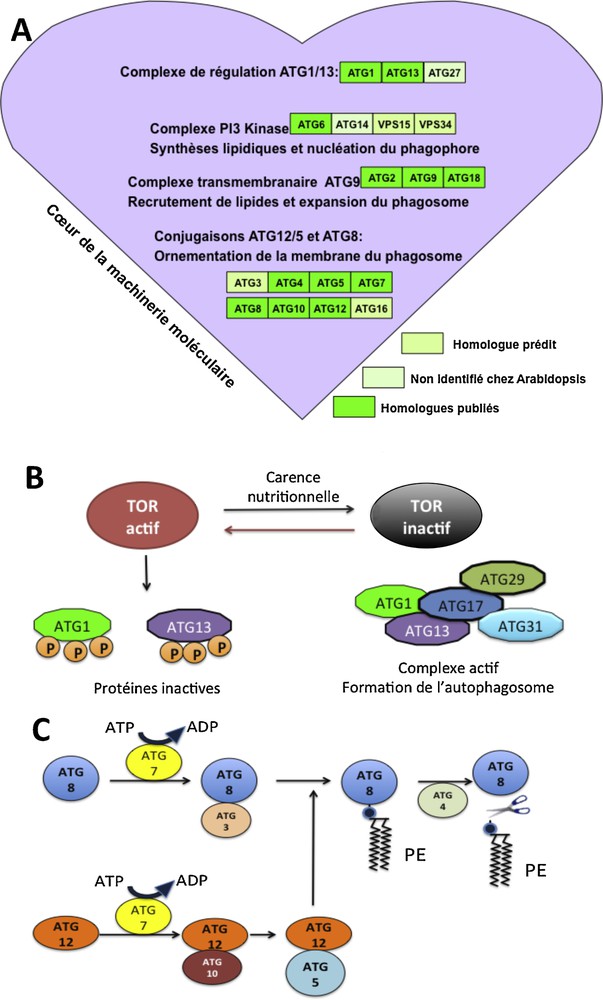
A. Illustration des différentes protéines autophagie (ATG) participant au cœur de la machinerie moléculaire de l’autophagie. Sont représentées en vert foncé les protéines homologues de la levure dont des mutants sont publiés chez Arabidopsis thaliana, en vert clair les protéines homologues prédites et enfin en vert pâle les protéines non identifiées à ce jour chez A. thaliana (D’après [23,112,130]). B. Le complexe de régulation ATG1/13. Lorsque le complexe TOR (Target Of Rapamycin) est inhibé à la suite d’une limitation en nutriments ou un traitement à la rapamycine, les protéines ATG13 et ATG1 sont déphosphorylées. Ceci permet l’association d’ATG1 avec ATG13, suivie de l’activation de l’activité kinase d’ATG1 et le recrutement des autres protéines sur la structure pré-autophagosomale (PAS) pour initier la formation de l’autophagosome. Ces évènements sont immédiatement réversés par la fourniture de nutriments (D’après [24]). C. Les systèmes de conjugaison. ATG8 est activée par ATG7 (une enzyme E1), transférée sur ATG3 (une enzyme E2) et éventuellement conjuguée à la phosphatidyléthanolamine (PE) grâce à l’action du complexe ATG5/12. En clivant le pont entre ATG8 et PE, ATG4 libère la protéine ATG8 de la membrane de l’autophagosome et permet son recyclage. ATG12 est fixée sur le résidu lysine (K) d’ATG5 de façon similaire à la réaction de conjugaison d’ATG8, excepté le fait qu’ATG10 fonctionne comme une enzyme E2 dans ce système à la place d’ATG3. Le complexe ATG12-ATG5 interagit avec ATG16 et forme un oligomère. Le complexe ATG12-ATG5-ATG16 exerce la fonction d’une enzyme E3 sur la réaction de conjugaison d’ATG8 à la PE ; la réaction de transfert d’ATG8 depuis ATG3 sur la phosphatidyléthanolamine (PE) est stimulée par ce complexe (D’après [24]).
Liste des homologues des gènes ATG chez Arabidopsis identifiés et décrits à ce jour.
| Nom du gène | AGI | Références |
| Complexe de régulation ATG1 | ||
| ATG1a | At2g37840 | [25] |
| ATG1b | At3g53930 | |
| ATG1c | At3g61960 | |
| ATG1t | ||
| ATG13a | At3g49590 | |
| ATG13b | At3g18770 | |
| Complexe PI3K | ||
| ATG6 | At3g61710 | [28] |
| VPS34 | ||
| Complexe ATG9 | ||
| ATG2 | At3g19190 | [33] |
| ATG9 | At2g31260 | [131] |
| ATG18a | At3g62770 | [32] |
| ATG18b | At4g30510 | |
| ATG18c | At2g40810 | |
| ATG18d | At3g56440 | |
| ATG18e | At5g05150 | |
| ATG18f | At5g54730 | |
| ATG18g | At1g03380 | |
| ATG18h | At1g54710 | |
| Systèmes de conjugaison | ||
| ATG3 | At5g61500 | |
| ATG4a | At2g44140 | [34] |
| ATG4b | At3g59950 | |
| ATG7 | At5g45900 | [130] |
| ATG8a | At4g21980 | [130] |
| ATG8b | At4g04620 | |
| ATG8c | At1g62040 | |
| ATG8d | At2g05630 | |
| ATG8e | At2g45170 | |
| ATG8f | At4g16520 | |
| ATG8g | At3g60640 | |
| ATG8h | At3g06420 | |
| ATG8i | At3g15580 | |
| ATG5 | At5g17290 | [23,33] |
| ATG10 | At3g07525 | [133] |
| ATG12a | At1g54210 | [130] |
| ATG12b | At3g13970 |
3.1 Le complexe de régulation ATG1/ATG13
La protéine kinase ATG1 et les protéines ATG13, ATG17, ATG29 et ATG31 fonctionnent ensemble dans le même complexe [24], (Fig. 2A, B). La protéine kinase ATG1 joue un rôle prépondérant dans l’activation de l’autophagie en agissant en aval du complexe « Target of Rapamycin » (TOR) décrit plus loin dans cette revue. La déphosphorylation d’ATG1 et d’ATG13, suite à l’inactivation de TOR (Fig. 2B) permet au complexe ATG13, ATG1 et ATG17 de s’assembler, ce qui induit le processus d’autophagie en permettant l’augmentation de l’activité kinase d’ATG1 qui est essentielle pour l’induction de la machinerie autophagique et est fortement induite en réponse à la carence nutritionnelle chez la levure. La cible de l’activité kinase ATG1 reste toutefois inconnue à ce jour. Chez Arabidopsis thaliana, quatre gènes ATG1 et deux gènes ATG13 ont été identifiés [25]. Une étude récente décrit la formation du complexe ATG1/13 dans des protoplastes d’Arabidopsis et montre que ce complexe est à la fois régulateur et cible de la machinerie autophagique. Les doubles mutants d’Arabidopsis ne présentant plus les protéines ATG13 sont hypersensibles à la carence nutritionnelle et présentent une sénescence précoce typique des mutants d’autophagie [25].
3.2 Le complexe PI3K
« Vacuolar Protein Sorting » (VPS34) est la seule phosphatidylinositol 3-kinase décrite chez la levure. Cette protéine peut former deux complexes. Le complexe I est composé de VPS34, VPS15, VPS30 (aussi appelé ATG6 chez les végétaux et Beclin 1 chez les animaux) et ATG14. Le complexe II comporte VPS38 à la place d’ATG14. Les complexes I et II sont localisés sur le PAS et sur la membrane des endosomes suivant la présence spécifique d’ATG14 ou de VPS38 [26,27]. Les complexes I et II ont des rôles différents dans l’autophagie ou dans la voie « Vacuolar Protein Sorting » respectivement. L’activité kinase de VPS34 est nécessaire à la génération de phosphatidylinositol 3-phosphate, lipide participant à la formation du PAS [13]. Seules les protéines ATG6 et VPS34 ont été identifiées jusqu’à présent chez les plantes. Chez Arabidopsis, les mutants ATG6 et VPS34 sont embryon létaux [28,29].
3.3 Formation de la structure autophagosomale : le complexe ATG9
ATG9 est la seule protéine transmembranaire essentielle à l’autophagie connue à ce jour. Cette protéine transite entre le PAS et la couche lipidique du phagophore mais n’a jamais été retrouvée dans l’autophagosome une fois celui-ci formé. Comme les autres protéines du processus d’autophagie, ATG9 est libérée et recyclée avant même que l’autophagosome ne soit clos [30]. Comme ATG9 est une protéine transmembranaire, son retrait de la membrane ne se fait pas par simple dissociation, mais nécessite l’intervention d’autres protéines situées à la périphérie de la membrane : ATG2 et ATG18. ATG18 se lie au phosphatidylinositol-3-phosphate de la membrane. La kinase ATG1 facilite l’association de ATG2 et ATG18 avec ATG9. La formation de ce complexe permet le recyclage continuel d’ATG9 à partir du phagophore en croissance et avant qu’il ne devienne autophagosome [31]. Chez Arabidopsis, contrairement à ATG2 et ATG9, et contrairement à la levure qui ne contient qu’une copie du gène ATG18, ATG18 est codée par une famille multigénique comprenant huit membres dont l’expression varie en fonction des tissus de la plante, de son stade de développement et des stress auxquels la plante est soumise. La présence de plusieurs ATG18 chez les mammifères et les plantes suggère des rôles spécifiques mais encore inconnus pour chacune des isoformes ATG18a-h [23]. Les mutants ATG2 et ATG9 d’Arabidopsis ont été décrits et montrent le même phénotype de sénescence foliaire et d’hypersensibilité à la carence nutritionnelle que les plantes RNAi 18a [32–34].
3.4 Les systèmes de conjugaison ATG5/12 et ATG8
Les deux systèmes de conjugaison font intervenir la protéine ATG7 (Fig. 2C). ATG7 active ATG12 suivant un processus similaire à celui de l’ubiquitinylation. L’ubiquitinylation est une modification post-traductionnelle nécessitant l’intervention successive de trois grandes classes d’enzymes appelées E1-, E2- et E3-ligases [35]. ATG7 est une enzyme comparable à une E1-ligase. En activant ATG12 elle transfère cette protéine sur le résidu cystéine de l’enzyme ATG10. ATG10, qui est comparable à une E2-ligase, permet alors de conjuguer ATG12 à ATG5 via la formation d’un pont isopeptide entre le résidu glycine d’ATG12 situé en position C-terminale et le résidu lysine d’ATG5 [24,36]. L’analyse des mutants ATG5 et ATG12 a permis de montrer que la formation de ce conjugué est essentiel à la formation de l’autophagosome [37,38]. Le complexe ATG5/12 permet d’assurer l’élongation de la membrane de l’autophagosome et de l’ornementer en favorisant le couplage d’ATG8 à la phosphatidyléthanolamine PE. La conjugaison de la protéine ATG8 à la PE est comparable à un processus d’ubiquitinylation. La protéine ATG8 nouvellement synthétisée est clivée par la protéase à cystéine ATG4 au niveau de sa partie C-terminale [39]. La protéine ATG8 ainsi modifiée est ensuite activée par ATG7. Cette réaction qui nécessite de l’ATP permet le transfert d’ATG8 sur le résidu cystéine d’ATG3. ATG3, qui pourrait correspondre à une E2-ligase d’un système d’ubiquitinylation, permet, avec la participation du complexe ATG5/12 la conjugaison d’ATG8 à la phosphatidyléthanolamine (PE), un lipide de la membrane de l’autophagosome. Un pont amide se forme liant le résidu glycine d’ATG8 et le groupe amine de la PE [34,40]. ATG8 est ainsi ponté à la membrane interne et externe du phagophore. Lors de la fermeture du phagophore en autophagosome les ATG8-PE situés sur la membrane externe des autophagosomes sont libérés par ATG4. La conjugaison ATG8-PE est donc un processus réversible puisque ATG4 est capable de libérer ATG8 de sa cible lipidique. Les protéines ATG8 recyclées permettent une progression efficace de l’autophagie en favorisant l’attachement et l’hémifusion des membranes des autophagosomes (Fig. 2C et 3), [24,41]. Tout comme ATG18, ATG8 est codée par plusieurs gènes chez les animaux et les plantes et par un seul chez la levure [23]. La diversification des gènes ATG8 chez les eucaryotes supérieurs (neuf isoformes chez Arabidopsis) suggère une spécialisation. ATG8, en plus de son rôle dans l’expansion des autophagosomes participerait à la sélectivité de dégradation [42]. Une interaction protéique spécifique entre les ATG8 et des récepteurs autophagiques permettrait de recruter dans les autophagosomes des cibles spécifiques. Des motifs d’interaction entre les récepteurs et ATG8 (AIM, ATG8-Interacting-Motif) commencent à être identifiés sur plusieurs protéines chez les animaux et les végétaux. Chez les animaux les protéines p62 et NBR1 font référence pour leur AIM. Chez Arabidopsis, la première protéine contenant un AIM identifiée est AtNBR1 par homologie aux NBR1 des mammifères [43]. Le second récepteur décrit est TSPO (Tryptophan Rich Sensory Protein) localisé dans la membrane plasmique. TSPO contient un AIM et se fixe aux hèmes dont l’action dans la cellule est potentiellement toxique puisque ces éléments sont susceptibles de générer des ROS. La fixation des hèmes à TPSO permet de dégrader sélectivement les hèmes par la voie de l’autophagie sélective [44]. Joka2 est une protéine de tabac présentant le même AIM typique de NBR1 et p62. Joka2 est localisée à la fois dans le noyau et dans des taches cytosoliques. L’expression de son gène est spécifiquement induite en situation de carence en soufre, tout comme ATG8f. Joka2 forme des homodimères qui interagissent avec un membre de la famille ATG8, présumé être ATG8f [45].
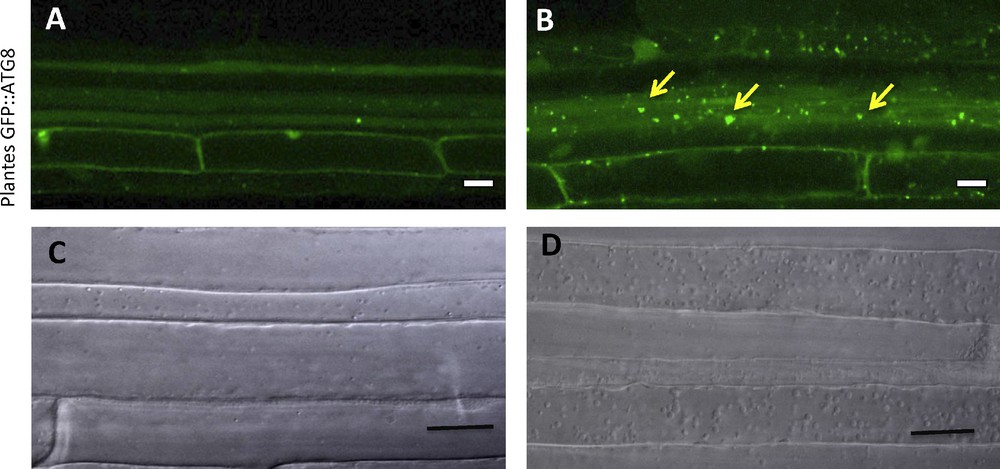
Illustration de la détection des autophagosomes (A, B) et des corps autophagiques (C, D) dans des racines d’Arabidopsis traitées (B, D) ou non (A, C) à la concanamycine A. Les fusions GFP : ATG8 permettent de localiser les autophagosomes en microscopie confocale sous la forme de petits points verts mobiles (B, quelques uns désignés par une flèche). L’utilisation de la microscopie photonique permet de visualiser les corps autophagiques. La concanamycine A bloque la dégradation des corps autophagiques dans les vacuoles et permet de visualiser l’accumulation des autophagosomes (B) et des corps autophagiques sous la forme de globules plus denses et statiques (D).
4 Méthodes cytologiques et moléculaires pour l’étude du flux autophagique
Afin d’étudier les mécanismes d’autophagie, leur régulation, leur dynamique, plusieurs méthodes complémentaires ont été développées. Certaines permettent de quantifier l’accumulation des autophagosomes en système contraint, d’autres d’observer le flux autophagique de façon dynamique [32,46].
La méthode traditionnelle utilisée depuis les origines de la découverte des autophagosomes est la microscopie électronique (voir paragraphes précédents). Elle permet de reconnaître les autophagosomes à leur double membrane. Cette technique qui requiert une grande expertise et l’accès à un microscope électronique n’est pas utilisée en routine. Des méthodes de microscopie confocale plus faciles d’accès l’ont peu à peu remplacée et permettent d’évaluer l’activité autophagique grâce à des fusions de protéines fluorescentes avec les ATG8 ([47–49] ; et références incluses).
L’utilisation de fusions GFP:ATG8 nécessitant le recours à la transgenèse, des essais ont été effectués afin d’observer les autophagosomes par marquage avec des colorants fluorescents spécifiques des compartiments acides comme la monodansylcadaverine ou encore le LysoTracker. Toutefois, parce que les autophagosomes ne sont pas les seuls compartiments acides de la cellule, l’utilisation de ces colorants reste une méthode approximative du flux autophagique. Bien que les fusions protéiques entre ATG8 et la GFP (Green Fluorescent Protein) permettent d’obtenir d’excellents marqueurs des autophagosomes, la perte de fluorescence de la GFP en condition acide a été évoquée et d’autres marqueurs fluorescents comme la RFP sont également utilisés pour tracer les corps autophagiques jusque dans les vacuoles [50] (Fig. 3).
Chez les plantes, l’observation des corps autophagiques en microscopie photonique est une méthode aisément réalisable sur des cellules de racine. Cette observation est facilitée par l’utilisation de drogues capables de bloquer la dégradation des corps autophagiques dans la vacuole comme la concanamycine A, le E-64d et la 3-MéthylAdénine (3-MA). Le E-64d est un inhibiteur des protéases à cystéine. La concanamycine A, quant à elle, inhibe les ATPases vacuolaires conduisant à une augmentation du pH vacuolaire. Les protéases ne sont alors plus actives et les corps autophagiques n’étant plus dégradés s’accumulent dans ce compartiment et deviennent de ce fait plus facilement observables ([34] ; Fig. 3).
Outre les méthodes de microscopie, des méthodes biochimiques permettant de quantifier les protéines ATG8 et ATG8-PE sur gel d’urée SDS-PAGE ont été proposées pour estimer le flux autophagique, la proportion de forme lipidée ATG8-PE étant corrélée au nombre d’autophagosomes et au taux de renouvellement d’ATG8 [34].
Toutes ces méthodes ont permis de caractériser les mutants d’autophagie chez la levure, les animaux et les plantes et ainsi de révéler le rôle des gènes ATG.
5 Sélectivité de l’autophagie chez la levure, les animaux et les plantes
À l’origine, l’autophagie a été décrite par de Duve comme un mécanisme de dégradation non sélectif. Par la suite, la découverte de protéines ATG requises pour la dégradation spécifique d’organites tels que les mitochondries et peroxysomes, les termes de mitophagie et pexophagie sont apparus dans la littérature.
5.1 La voie sélective du cytoplasme à la vacuole CVT
Le CVT pathway signifie « Cytoplasm to Vacuole Targeting pathway ». Cette forme d’autophagie n’a été décrite que chez la levure. Cette voie cible deux enzymes qu’elle véhicule spécifiquement à la vacuole : une aminopeptidase 1 appelée prApe1 et une alpha-mannosidase appelée Ams1 [51]. Les précurseurs de ces enzymes forment des oligomères puis s’assemblent en complexes protéiques reconnus par la protéine ATG19 qui alors interagit avec ATG11. Ces complexes sont alors délivrés au niveau du PAS où l’interaction entre ATG19 et ATG8 se fait grâce au motif LIR-like WXXL d’ATG19. Les complexes sont alors séquestrés dans une vésicule à double membrane appelée vésicule CVT. Cette vésicule est transportée vers la vacuole où sa membrane externe fusionne avec celle de la vacuole, libérant ainsi dans la lumière de la vacuole le corps protéique qu’elle contient. La vésicule CVT possède des similarités morphologiques avec l’autophagosome puisqu’elle possède une double membrane mais sa taille est bien inférieure à celui-ci (150 nm versus 400–900 nm). La formation des vésicules CVT requiert en plus d’ATG19 et ATG11 les acteurs du cœur de la machinerie moléculaire de l’autophagie.
Aucun homologue des protéines ATG19 et ATG11 n’a été identifié chez les végétaux et chez les animaux, laissant supposer que cette voie est spécifique de la levure.
Toutefois les études récentes montent que tous ces eucaryotes exploitent les motifs de type LIR pour sélectionner le contenu des vésicules autophagiques (voir plus loin).
5.2 Sélectivité pour des protéines cytosoliques
La sélectivité pour des cibles cytosoliques est restée méconnue jusqu’à la découverte chez les mammifères de la protéine p62. Cette protéine, impliquée dans plusieurs fonctions biologiques contient plusieurs domaines d’interaction protéique dont un domaine d’interaction avec l’ubiquitine appelé UBA, un domaine PB1 responsable de son oligomérisation et un domaine de type LIR [52]. Ainsi p62 fonctionne comme un acteur essentiel de l’autophagie capable de recruter des protéines ubiquitinées et d’interagir avec l’homologue animal d’ATG8 (LC3) afin de servir d’adaptateur et de récepteur pour les protéines ubiquitinées destinées à être digérées. Des cibles de p62 ont été identifiées, il s’agit d’enzymes ou facteurs de transcription impliqués dans la régulation du stress oxydatif [53,54] mais aussi de bactéries ubiquitinées et éliminées grâce à p62 via l’autophagie [55]. Par la suite Nbr1 et Alfy, deux autres adaptateurs impliqués dans l’autophagie et se liant à des protéines ubiquitinées, furent décrites. Nbr1 contient deux motifs LIR et interagit avec LC3. Alfy interagit avec ATG5 et les PI3P à la surface du phagophore. Des publications récentes montrent que p62 serait aussi impliquée dans l’autophagie sélective d’organelles comme les mitochondries et les peroxisomes ([14,56–60] pour revue).
Jusqu’à présent aucun homologue de p62 n’a été décrit chez la levure et chez les végétaux. L’homologue de Nbr1 a été récemment identifié chez A. thaliana [43]. Pour l’instant ses cibles ne sont pas connues.
5.2.1 Pexophagie
La dégradation des peroxysomes en excès ou altérés chez la levure est réalisée par la voie d’autophagie sélective appelée pexophagie [17,61]. Cette voie implique le cœur de la machinerie autophagique et comprend également des protéines qui lui sont spécifiques comme ATG30, ATG26 et ATG28. ATG30 est la phosphoprotéine réceptrice des peroxysomes identifiée chez la levure pour la pexophagie [62]. ATG30 s’associe aux peroxysomes via une interaction avec les protéines PEX3 et PEX14 situées dans la membrane des peroxysomes [62]. La phosphorylation des protéines ATG30 lorsqu’elles sont associées aux peroxysomes leur permet de s’attacher aux protéines adaptatrices ATG11 et ATG17 de la machinerie autophagique [17]. ATG24 intervient plus tard dans le processus de pexophagie, sans doute lors de la libération du contenu vésiculaire au niveau de la vacuole. L’activité enzymatique stérol glucosyltransférase d’ATG26 est cruciale pour déterminer la taille du « pexophagosome ».
Jusqu’à ce jour, les homologues des gènes ATG30 et PEX n’ont pas été identifiés chez les plantes.
5.2.2 Mitophagie
La mitophagie observée en microscopie dans les cellules de foie de rongeur dans les années 1960 fut ensuite caractérisée chez la levure en condition d’anaérobie et de carence en lactate [63–66]. Kanki et Klionsky [67] recherchèrent parmi les gènes ATG lesquels étaient nécessaires à la mitophagie. Ils utilisèrent chez la levure des fusions de protéines mitochondriales avec la GFP et un protocole visant à détecter la GFP libérée par clivage des protéines fusionnées après passage par la vacuole. Dans ces conditions, il fut montré que les délétions des gènes ATG11, ATG20 et ATG24 n’altéraient pas la macro-autophagie mais inhibaient la mitophagie, la pexophagie et également la voie CVT. En revanche, les délétions dans les gènes ATG17, ATG29 et ATG31 bloquent la mitophagie, la pexophagie et la macro-autophagie sans altérer la CVT chez la levure. Le criblage de mutants de levure incapables de mitophagie leur permit ensuite d’isoler les gènes ATG32 et ATG33 [68,69]. Ces deux gènes sont spécifiques de la mitophagie. La protéine ATG32 située sur la membrane externe de la mitochondrie se lie à ATG11 et à ATG8, ce qui permet le recrutement spécifique de mitochondries vers la structure pré-autophagosomale. ATG32 possède, comme la protéine de levure ATG19 et la protéine de mammifère p62, un motif de type LIR « WQAI » dans sa partie N terminale qui lui permet de se lier à ATG8 [17,70]. ATG33 est également située sur la membrane externe de la mitochondrie. L’action d’une délétion de ATG33 se fait ressentir essentiellement lorsque les cultures de levure sont à l’état stationnaire ce qui suggère que ATG33 a un rôle particulier dans le recrutement des mitochondries âgées vers la mitophagie. À ce jour, aucun homologue d’ATG32 et ATG33 n’a été identifié chez les plantes.
5.2.3 Ribophagie
Les ribosomes sont des complexes nucléo-protéiques qui représentent à peu près la moitié des protéines totales dans les cellules jeunes où la synthèse protéique est très active. La dégradation de leurs ARN et de leurs protéines en situation de carence nutritionnelle peut représenter une source non négligeable d’acides aminés [71]. De plus l’intégrité des ribosomes étant essentielle au bon fonctionnement de la machinerie traductionnelle et à la reprogrammation cellulaire face aux changements environnementaux, il est essentiel que leur turnover soit assuré et régulé. Depuis la découverte de l’autophagie, l’observation de ribosomes dans des autophagosomes en microscopie électronique, était attribuée à leur forte probabilité de présence dans des structures autophagosomales non sélectives, du fait de leur très grand nombre dans la cellule. En montrant que les ribosomes sont dégradés plus rapidement que les autres protéines cytosoliques, Kraft et Peter suggérèrent que leur dégradation était sélective [72,73]. Ayant été montré chez la levure que lors d’une carence, la dégradation de la sous-unité ribosomale 60S dépendait de l’activité de la protéase Ubp3 et de son co-facteur Bre5, l’interaction de ces deux protéines avec la protéine réceptrice ATG19 renforça l’idée d’une autophagie sélective des ribosomes. Ossareh-Nazari et al. [74] identifièrent deux protéines nécessaires à la ribophagie Cdc48, une protéine chaperon, et Ufd3, un adaptateur de Cdc48 se fixant à l’ubiquitine.
Si les homologues des protéines impliquées dans la ribophagie chez la levure n’ont pas été retrouvés chez les plantes, l’implication de RNS2, une ribonucléase de la famille RNase T2 pourrait participer à un mécanisme assimilé à de la ribophagie. Les mutants dans RNS2 présentent une accumulation d’ARN dans la vacuole et une autophagie constitutive. Ce phénotype associé à la localisation subcellulaire de la protéine RNS2 sur le RE et dans la vacuole sont aussi des arguments en faveur d’une ribophagie chez les végétaux [75,76].
5.2.4 Chlorophagie : autophagie sélective du chloroplaste chez les plantes
Les plastes sont les organites spécifiques des eucaryotes photosynthétiques. Ils se distinguent des autres organites par leur plus grande taille (5 à 10 μm). Les chloroplastes ont un rôle prépondérant dans la photosynthèse et se caractérisent par leur forte production de formes réactives de l’oxygène [77]. La particularité de ces organites végétaux est qu’ils renferment une quantité considérable de protéines, environ 50 % des protéines foliaires [78,79]. Les chloroplastes vieillissants aussi appelés gérontoplastes se dégradent, perdent en volume et deviennent une source d’azote majeure pour le recyclage de l’azote lors de carences et quand la feuille vieillit [78]. Les mécanismes impliqués dans la protéolyse des protéines chloroplastiques et dans la dégradation des chloroplastes sont peu connus. Le fait que le nombre de chloroplastes reste constant jusqu’à une phase de sénescence foliaire tardive, alors que les protéines du stroma sont dégradées dès la maturité de la feuille [78] indique que le contenu protéique des chloroplastes peut être dégradé sans que la structure entière du chloroplaste ne disparaisse. Leur taille décroissante indique que les gérontoplastes seraient vidés de leur contenu avant de disparaître. Des études menées sur les plantes supérieures et chez Chlamydomonas ont permis de montrer que la rubisco, protéine majeure du chloroplaste, pouvait être transférée du chloroplaste dans des vésicules puis transportée jusqu’à la vacuole [80–82]. Ces vésicules, identifiées par l’utilisation de lignées transgéniques exprimant des protéines du stroma ou la rubisco marquées avec la protéine fluorescente GFP, sont appelées les « Rubisco Containing Bodies » (RCB). Wada et Ishida [83,84] ont montré que le transport des RCB vers la vacuole dépend de la voie de l’autophagie. Dans les mutants d’autophagie, ces vésicules ne sont plus observées dans la vacuole. Leur modèle établit qu’en condition de carence nutritionnelle, un stromule bourgeonnerait du chloroplaste. Le bourgeon terminal à l’extrémité du stromule serait pris en charge par une membrane qui en fusionnant formerait une vésicule à double membrane : le RCB. Le transport de ce RCB vers la vacuole serait dépendant de la voie de l’autophagie. Enfin, ce corps serait dégradé à l’intérieur de la vacuole par les protéases y résidant. Le chloroplaste est un gros organite dont la taille varie entre 5 et 10 μm. Wada et al. [83] proposent que la formation des RCB participe au rétrécissement du chloroplaste et à sa conversion en gérontoplaste de taille inférieure qui pourrait être pris en charge par les autophagosomes dont la taille avoisine 1,5 μm [85].
Pour l’instant les mécanismes de prise en charge des structures chloroplastiques ne sont pas connus, aucune protéine adaptateur n’a été mise en évidence.
5.3 Les protéines à motif LIR et la sélectivité autophagique chez les plantes
La question de la sélectivité de l’autophagie chez les plantes n’a été abordée que très récemment. La mise en évidence d’un homologue de Nbr1 mobilisé vers la vacuole centrale chez A. thaliana a permis de détecter pour la première fois un adaptateur de l’autophagie sélective chez les plantes [43]. Par la suite l’homologue de Nbr1, la protéine Joka2, fut identifié chez le tabac suite à un crible double hybride visant à identifier les partenaires protéiques de la protéine UPC9 de fonction inconnue et induite en situation de carence en soufre [45]. L’expression du gène codant Joka2 est induite en carence en soufre et en carence en azote. Joka2 est localisée sous forme de spots cytosoliques et nucléaires et forme des homodimères. Joka2 interagissant avec ATG8f, la fusion de ces protéines avec des rapporteurs fluorescents a permis de confirmer leur colocalisation in situ au niveau de structures autophagosomales.
La découverte que la dégradation de la protéine régulatrice TSPO, capable de se lier aux tétrapyrroles, est inhibée dans le mutant ATG5 d’A. thaliana et est sensible aux inhibiteurs de type III phosphoinositide 3-kinases qui régulent l’autophagie dans les cellules eucaryotes, a désigné TSPO comme une cible potentielle impliquée dans la régulation de la dégradation des produits de dégradation de la chlorophylle via l’autophagie sélective chez les plantes [44]. L’interaction de TSPO avec AtATG8e et la colocalisation partielle de TSPO et AtATG8e in vivo renforce cette hypothèse.
Récemment le groupe de Gad Galili a recherché les protéines ciblées par la voie de l’autophagie et capables d’interagir avec ATG8f par la méthode du double hybride [86]. Cette approche leur a permis d’identifier deux protéines ATI1 et ATI2 spécifiques des plantes. En situation de suffisance en carbone, ces protéines sont associées à la membrane du RE alors qu’en situation de carence elles sont associées à des structures sphériques mobiles qui se déplacent en surface du réticulum. Ces structures sont différentes des péroxisomes, mitochondries, Golgi et autophagosomes et ne contiennent aucun marqueur protéique spécifique du réticulum. La destination finale de ces protéines est la vacuole. Les niveaux d’expression de leurs gènes sont plus élevés au cours de la phase finale de maturation de la graine et lors de sa dessication. Un rôle d’ATI1 et ATI2 dans la germination des graines a pu être montré par l’analyse de leurs mutants.
6 Rôles et régulations de l’autophagie chez les plantes
Active à un niveau basal, il est admis que l’autophagie a un rôle de contrôle-qualité de la cellule chez la levure, les animaux et aussi chez les plantes. Par l’élimination des agrégats protéiques ou des protéines altérées et des organelles défectueux, elle détoxifie et désencombre la cellule et permet ainsi de maintenir une homéostasie cellulaire [87]. C’est pourquoi l’autophagie est induite en réponse aux stress susceptibles d’altérer le fonctionnement de la cellule comme le stress du RE qui provoque l’accumulation de protéines mal repliées, les stress oxydants d’origines diverses ou encore les altérations mitochondriales [87,88]. L’autophagie est également un processus cellulaire qui permet de mobiliser des ressources nutritionnelles intracellulaires notamment en réponse aux carences, qu’elles soient azotées ou carbonées [89]. Même s’ils contribuent tous à stimuler l’autophagie pour aider à maintenir les cellules en bon fonctionnement, ces différents signaux font intervenir des voies de signalisation différentes [90]. Dans cette partie nous présenterons essentiellement les rôles de l’autophagie chez les plantes. Seules les données obtenues chez l’animal et utiles à la compréhension de ces rôles seront exposées ici.
6.1 Autophagie et gestion des carences nutritionnelles
La limitation nutritionnelle est le facteur physiologique induisant l’autophagie le plus décrit chez l’ensemble des eucaryotes. Pour la majorité des cultures cellulaires de mammifères, la carence en acides aminés induit l’autophagie dans les minutes qui suivent l’initiation du stress [91]. La dégradation des protéines qui en résulte permet à la cellule d’adapter son métabolisme et de répondre à ses besoins énergétiques. Dans le cas des nouveau-nés des mammifères, la rupture du lien ombilical crée une carence nutritionnelle sans précédent. La stimulation de l’autophagie est alors essentielle à la survie du nouveau-né. C’est grâce à l’autophagie que le muscle cardiaque trouve l’énergie nécessaire à son fonctionnement dans les instants qui suivent la naissance et avant que la digestion du lait maternel ne puisse remplir ses besoins énergétiques [88]. Chez les plantes il a été montré que les gènes d’autophagie sont induits par la carence en azote [92] et par la carence en carbone [93], ce qui suggère que cette voie est également impliquée dans un mécanisme de sauvegarde nutritionnelle chez les plantes.
Le mécanisme moléculaire par lequel les signaux de carence nutritionnelle sont perçus a d’abord été décrit chez la levure [94] puis chez le rat [95]. La protéine kinase TOR a été identifiée comme un élément essentiel à la régulation de l’autophagie [96]. TOR (Target Of Rapamycin) est une sérine/thréonine kinase très conservée qui appartient à la famille des PIKK (PhosphatidylInositol Kinase-related Kinase). Cette protéine décrite pour son rôle dans la sensibilité des cellules à la rapamycine intègre des signaux hormonaux, métaboliques et nutritionnels [97]. Activée par la présence de nutriments, TOR a un effet positif sur la croissance cellulaire et un effet négatif sur l’autophagie qui est alors à son niveau le plus faible. En situation de carence nutritionnelle, l’activité kinase de TOR est inhibée, ce qui réduit la croissance cellulaire et met la cellule sur un mode de recyclage nutritif assurant sa survie. Les cibles directes de TOR sont ATG13 et ATG1, qui sous forme déphosphorylées forment le complexe d’initiation de l’autophagie. La protéine kinase, AMPK (AMP-activated protein Kinase) est aussi un acteur important dans la régulation de l’autophagie en réponse au stress énergétique chez les animaux. AMPK, activée lorsque l’énergie de la cellule est faible, active le couple TSC1/TSC2 (Tuberous Sclerosis Complex), qui en retour inhibe TOR. AMPK peut également phosphoryler directement RAPTOR, un partenaire protéique essentiel à l’activité de TOR ([96] pour revue). En carence énergétique, AMPK contribue ainsi à induire l’autophagie via l’inhibition du complexe TOR/RAPTOR.
Les homologues des régulateurs AMPK et TOR ont été isolés et caractérisés chez Arabidopsis sous le nom de SnRK1 et AtTOR. Le génome d’Arabidopsis ne contient qu’un seul gène TOR contrairement à la levure qui en possède deux [98,99]. AtTOR fonctionne en complexe multimérique et les partenaires identifiés à ce jour sont LST8 et RAPTOR [100,101]. La mutation de AtTOR est embryon létale. L’utilisation de plantes AtTOR RNAi et de plantes qui surexpriment AtTOR a permis de montrer que ce gène est impliqué dans la croissance et la sénescence des plantes. L’utilisation de lignées AtTOR RNAi a permis de montrer récemment son rôle régulateur de l’activité autophagique [102]. Contrairement aux plantes, l’algue verte Chlamydomonas reinhardtii est sensible à la rapamycine [103,104]. L’augmentation de la vacuolisation dans les cellules de C. reinhardtii traitées à la rapamycine et l’augmentation simultanée du niveau de protéine ATG8 montrent que la régulation de l’autophagie passe par la voie de TOR chez cette algue [105,106].
Les protéines de la famille des SnRK1 (Snf1 [Sucrose non fermenting 1] Related protein Kinase) sont des homologues de la protéine SNF1 de levure et de la protéine AMPK chez les mammifères. Les SnRK1 jouent, chez les plantes, un rôle essentiel dans la réponse aux stress énergétiques, aux carences en sucres et à l’ABA [107,108]. Récemment, des analyses transcriptomiques ont révélé que la protéine KIN10, appartenant à la famille des SnRK1, est activée lors d’une carence en sucre et inhibée en présence de sucre [107]. KIN10 stimule la transcription de gènes du catabolisme de la paroi cellulaire, des lipides, du saccharose, des acides aminés, de l’amidon et des protéines, ce qui pourrait permettre de fournir à la cellule une source alternative d’énergie et de métabolites lors d’une carence. De manière intéressante, KIN10 active également l’expression de gènes d’autophagie comme ATG8e ainsi que des gènes impliqués dans la remobilisation de l’azote comme la Glutamate DésHydrogénase [109]. Le rôle possible des SnRK1 dans la régulation de l’autophagie reste pour l’instant à démontrer.
6.2 Autophagie et résistance aux stress de l’environnement
Chez les plantes, plusieurs études montrent que l’autophagie a un rôle important dans la gestion du stress oxydatif. L’exposition de plantules d’Arabidopsis à un stress oxydatif par traitement au H2O2 ou au méthylviologène conduit à une rapide et forte induction de l’activité autophagique [110] et les mutants d’Arabidopsis (AtATG18a-RNAi) et de riz (Osatg10b) sont hypersensibles au stress oxydatif. La forte accumulation de protéines oxydées observée chez ces deux plantes montre que l’autophagie est un processus qui permet de les dégrader et les éliminer [110,111]. Les lignées transgéniques AtATG18a-RNAi d’Arabidopsis s’avèrent également plus sensibles au stress salin et à la sécheresse [112,113].
6.3 Autophagie et immunité chez les plantes
La mort cellulaire programmée chez les végétaux est un mécanisme immunitaire de défense spécifique contre les agents pathogènes connu sous le nom de réponse hypersensible (HR). Lorsqu’une plante est infectée par une forme avirulente de l’agent pathogène, les cellules infectées déclenchent des mécanismes de mort cellulaire programmée associés à la réaction hypersensible autour du site de l’infection créant ainsi un environnement défavorable à la dispersion de l’agent pathogène. La HR est provoquée par la reconnaissance spécifique de l’agent pathogène par la plante via des interactions gènes d’Avirulence (Avr)-gènes de résistance (R). L’activation des voies de signalisation de défense au site de perception du parasite engendre la production de messagers secondaires, hormonaux tels que l’acide salicylique, ou encore de ROS qui provoquent la mort des cellules attaquées et sont également véhiculés à distance dans les tissus adjacents. L’acide salicylique (SA) et les ROS constituent des signaux de la résistance systémique acquise [114]. Les espèces réactives de l’oxygène (ROS) générées naturellement au cours du développement des plantes le sont de manière plus importante, plus rapide et plus localisée lors de l’attaque parasitaire. Ce phénomène est appelé « bouffée oxydante » et fait intervenir le peroxyde d’hydrogène (H2O2), son précurseur l’anion superoxide (O2.−) et le radical hydroxyle (ȮH) [115]. L’excès de ROS au site d’infection est sans doute responsable de la mort des cellules. Les résultats obtenus lors de l’infection de mutants d’autophagie par divers agents pathogènes montrent que la mort cellulaire liée à la HR ne se limite pas au site de l’infection et s’étend sans limite dans les tissus sains des mutants d’autophagie [116,117]. Les signaux de mort cellulaire générés dans les cellules infectées migrent de proche en proche vers des tissus sains sans être atténués. L’autophagie pourrait intervenir à différents niveaux dans l’inhibition de la propagation de la zone de mort cellulaire. Elle pourrait dégrader directement les ROS et le SA et éliminer les protéines qui traduisent et amplifient ces signaux. L’autophagie, en ciblant et dégradant au fur et à mesure les protéines oxydées, permettrait le retour à la normale [118]. En croisant les mutants d’autophagie avec des mutants déficients dans la synthèse ou l’accumulation d’acide salicylique (sid2 et NahG), Yoshimoto et al. [117] sont parvenus à supprimer l’extension de la HR chez les mutants d’autophagie, montrant ainsi le rôle de l’autophagie dans la gestion de la HR par un contrôle de la voie de signalisation du SA.
6.4 Autophagie, vieillissement et remobilisation de l’azote chez les plantes
Contrairement aux animaux, le vieillissement, aussi appelé sénescence, a une fonction biologique chez les plantes [119,120]. Cette fonction de la sénescence foliaire est de permettre le recyclage des nutriments et leur export des feuilles en sénescence dont la plante va bientôt se séparer, vers les organes en formation et demandeurs en énergie et nutriments. Ce recyclage nutritionnel au niveau de la plante entière est appelé remobilisation et les études menées jusqu’à présent sur le sujet concernent essentiellement la remobilisation de l’azote, qui avec le carbone est l’une des sources d’autotrophie chez les plantes. Le processus de remobilisation de l’azote est un mécanisme d’économie nutritionnelle qui est essentiel à l’adaptation des plantes à leur environnement [121,122]. Les protéines chloroplastiques sont une source majeure pour la remobilisation de l’azote. En effet le chloroplaste contient près de 70 % des protéines foliaires et celles ci sont représentées en majorité par une seule protéine, la rubisco (50 % des protéines foliaires chez les plantes en C3) qui en plus d’être l’enzyme responsable de la fixation photosynthétique du carbone est une forme de stockage de l’azote foliaire [124,125].
Les nombreuses études sur la sénescence foliaire et les mécanismes de remobilisation de l’azote révèlent que de nombreuses protéases sont induites à la sénescence [109,122]. Le transfert de la rubisco et des protéines du stroma chloroplastique dans des RCB vers la vacuole permettrait leur protéolyse. Il a été récemment montré que les RBC ne sont plus observés dans les cellules des feuilles sénescentes des mutants d’autophagie [83,123]. Ces observations suggèrent que le processus d’autophagie est nécessaire à la formation des RCB et à leur transport vers la vacuole lytique. Le modèle (Fig. 4) propose un mécanisme de dégradation des protéines du stroma. Les protéines solubles du chloroplaste sont sensibles à l’oxydation et l’action des formes réactives de l’oxygène est capable de les fragmenter en peptides [124–126]. Il a été montré que les peptides oxydés de la rubisco s’accolent à la membrane du chloroplaste [127,128]. Il est connu que les chloroplastes sont capables d’émettre des stromules qui sont des bras évaginés se terminant par un bourgeon [129]. Nous supposons que les stromules bourgeonnant du chloroplaste contiendraient dans leurs bourgeons les peptides oxydés destinés à être hydrolysés dans la vacuole. Ces bourgeons seraient pris en charge et englobés par une membrane autophagique conduisant à la formation d’un RCB. Le RCB serait alors véhiculé jusqu’à la vacuole par une voie qui dépend de l’autophagie pour y être dégradé.
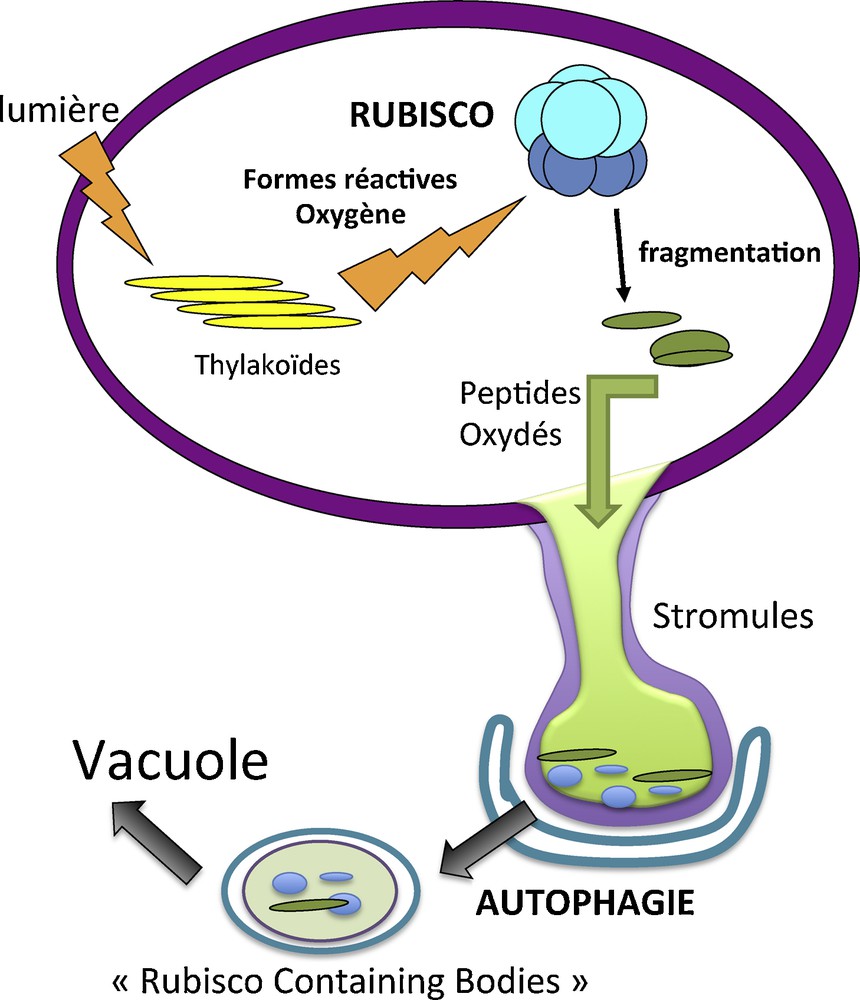
Modèle hypothétique pour la dégradation de la rubisco et probablement d’autres protéines du stroma par autophagie via les « Rubisco Containing Bodies » (RCB). En condition de carence nutritionnelle ou lors de la sénescence foliaire naturelle, les formes réactives de l’oxygène sont plus abondantes dans le chloroplaste du fait d’un mauvais fonctionnement des chaînes de transfert d’électrons des antennes lumineuses. Il en résulte une oxydation des protéines du stroma qui sont fragmentées. Les stromules bourgeonnant du chloroplaste contiennent alors les protéines oxydées destinées à la protéolyse. Le bourgeon du stromule ainsi chargé est séquestré par une membrane d’isolation. Le RCB, un autophagosome contenant des peptides du stroma est formé et transporté vers la vacuole où il sera dégradé par les protéases vacuolaires.
D’après [84].
Il a été constaté que tous les mutants d’autophagie présentent dans leurs cellules des stromules surabondants [84] et qu’ils ont tous une sénescence foliaire plus précoce que les plantes sauvages et ceci d’autant plus en situation de limitation en nutriments [32,34,117,130–133]. Ceci suggère que la gestion de leurs nutriments foliaires est inefficace et délétaire pour l’organe foliaire. L’engorgement des cellules végétales en formes oxydées et en organelles déviants pourrait être la cause de leur intoxication et de leur sénescence accélérée.
Dans ce contexte nous avons récemment exploré de façon globale le rôle de l’autophagie dans les processus de recyclage nutritionnel et de remobilisation de l’azote dans les feuilles d’Arabidopsis lorsque la plante est limitée ou non en azote. Nos travaux nous ont permis de montrer que l’intégrité de la machinerie autophagique est essentielle chez les plantes au remplissage du grain et à la remobilisation de l’azote des parties végétatives vers la graine [134]. L’autophagie induite au cours du vieillissement foliaire serait un des mécanismes responsables de la remobilisation des nutriments au niveau plante entière.
7 Conclusion
Les rôles de l’autophagie sont étudiés depuis longtemps dans le monde animal. Les données dont nous disposons dans le règne végétal restent plus modestes, la découverte de ce mécanisme chez les plantes ayant débuté plus tard que chez les animaux et la levure, dans les années 2000. Bien que bénéficiant de l’avancée des connaissances obtenues sur les animaux et ayant pu constater une forte homologie des processus moléculaires de l’autophagie et de ses régulations entre cellules animales et végétales, le rôle de l’autophagie dans le contrôle nutritionnel et dans l’adaptation des plantes à leur environnement reste à explorer. Le rôle de l’autophagie dans la physiologie devrait permettre de mettre en évidence certaines spécificités de fonction liées au fait que les végétaux sont des organismes autotrophes pour l’azote et le carbone. On peut en effet s’attendre à ce que les cibles et la régulation du système soient différentes.
Déclaration d’intérêts
Les auteurs n’ont pas transmis de déclaration de conflits d’intérêts.


