1 Introduction
Ces dernières années, les méthodes de biologie moléculaire ont permis de mettre en évidence de nouvelles formes d’acides ribonucléiques (RNA) complétant la famille des RNA non codants (tRNA, rRNA, snRNA…). Parmi ceux-ci figurent des RNA non codants de très petite taille, appelés « microRNA » ou encore « miRNA ». Les microRNA sont des RNA d’une vingtaine de nucléotides, dont les précurseurs sont transcrits à partir du génome, puis soumis à des processus de maturation qui régulent l’expression de protéines cibles en s’appariant de manière spécifique à leurs RNA messagers (mRNA), inhibant ainsi leur traduction ou entraînant leur dégradation. On dénombre actuellement plus de 2000 microRNA [1], qui seraient impliqués dans la régulation d’environ un tiers du génome [2]. En raison, d’une part, de la dérégulation de l’expression cellulaire des microRNA observée dans de nombreuses pathologies, se caractérisant par une augmentation ou une diminution de leur biogenèse et, d’autre part, de la capacité des cellules à sécréter ou libérer certains d’entre eux dans les milieux extracellulaires, expliquant ainsi leur présence au niveau des liquides biologiques, il a été suggéré que les microRNA circulants pourraient avoir un intérêt comme biomarqueurs non invasifs. Cette revue de synthèse a pour objectif de faire le point sur l’utilisation actuelle des microRNA en tant que biomarqueurs, essentiellement au niveau des liquides biologiques, et d’aborder les perspectives qui ressortent du fait que leur formes vésiculaires circulantes pourraient permettre d’évaluer l’état des cellules et des tissus qui les produisent.
2 Les microRNA
Le premier microRNA, dénommé lin-4, fut découvert en 1993 chez Caenorhabditis elegans (C. elegans) par Ambros et al. [3] grâce à l’identification d’une mutation de type « perte de fonction » qui entraînait des anomalies du développement chez le nématode. La découverte de lin-4 a été un temps considérée comme une « singularité » dans la génétique des nématodes jusqu’à la mise en évidence par Ruvkun et al. [4], en 2000, soit sept ans plus tard, d’un deuxième microRNA, appelé let-7, jouant à nouveau un rôle régulateur dans le développement de C. elegans. Plusieurs études ont ensuite montré que let-7 se retrouvait non seulement chez C. elegans mais aussi chez d’autres espèces telles que Drosophila melanogaster ainsi que chez les mammifères (l’homme inclus), avec une structure quasiment identique dans la plupart des cas. Et c’est alors que, dès 2002, paraît la première étude rapportant une relation entre la dérégulation des microRNA et une pathologie humaine, en l’occurrence un cancer, avec la découverte par Calin et al. [5] de l’implication de la délétion de deux gènes de microRNA dans de nombreux cas de leucémies lymphoïdes chroniques. Les microRNA s’avèrent donc dotés de structures évolutivement très conservées, qui leur permettent de réguler l’ensemble des processus physiologiques, allant bien au-delà des aspects développementaux à l’origine de leur découverte, et d’intervenir dans de nombreuses pathologies, soit indirectement comme facteur intermédiaire d’une dérégulation, soit directement comme facteur responsable de celle-ci [6].
2.1 Biogenèse et dégradation des microRNA
Les gènes des microRNA sont localisés dans tous les chromosomes sauf, semble-t-il, le chromosome Y (Fig. 1). La plupart des microRNA proviennent, soit de gènes spécifiques non codants localisés dans les régions intergéniques du génome, soit de régions introniques ou plus rarement exoniques de gènes codants que l’on qualifie aussi de gènes hôtes. Dans le cas des gènes codants, le promoteur utilisé peut correspondre à celui du gène ou, au contraire, à un promoteur additionnel intronique [7]. Les gènes sont transcrits par l’enzyme RNA polymérase II en de longs transcrits primaires, appelés « pri-microRNA » ou encore « pri-miRNA », qui peuvent atteindre plusieurs milliers de nucléotides. Ils possèdent, comme pour les pré-mRNA, une « coiffe » en 5′ (7-méthylguanosine triphosphate [m7G]), qui est mise en place dès le début de la transcription et une « queue » polyadénylée à son extrémité 3′. Les pri-microRNA ont une structure tridimensionnelle comportant des régions en épingle à cheveux (constituées par une tige et une boucle), parmi lesquelles se trouve celle comprenant les séquences du futur microRNA [8]. Ces structures particulières en épingle à cheveux sont clivées à la base de leur tige par une endoribonucléase nucléaire de type III, donnant ainsi naissance à un précurseur de microRNA appelé « pré-microRNA » ou encore « pré-miRNA », comportant entre 60 et 100 nucléotides. La reconnaissance et le clivage des structures relatives aux futurs microRNA est plus précisément le fait d’un complexe protéique dénommé « microprocesseur ». Il est formé par la protéine Drosha, qui est l’endoribonucléase, et le facteur DGCR8 (DiGeorge syndrome Critical Region Gene 8), qui est une protéine de liaison des jonctions RNA simple brin–RNA double brin dans la structure en épingle à cheveux. Le mécanisme canonique implique DGCR8, qui reconnaît la structure caractéristique du pri-microRNA (une trentaine de paires de bases plus ou moins bien appariées) où se fera le clivage. DGCR8 se charge aussi de stabiliser l’association de Drosha avec le pri-microRNA et favorise le clivage à une distance de 11 paires de bases de la jonction RNA simple brin–RNA double brin. Les gènes de microRNA sont assez fréquemment polycistroniques, de sorte qu’à partir d’un même pri-microRNA peuvent être générés plusieurs pré-microRNA différents. Les pré-microRNA sont ensuite transportés du noyau vers le cytoplasme. Ce transport s’effectue grâce à la formation d’un complexe avec l’exportine-5, en association avec son cofacteur RanGTP (guanosine triphosphate) [9]. Ce complexe transportera spécifiquement les pré-microRNA à l’extérieur du noyau. L’hydrolyse par la protéine GAP (GTPase activating protein) de la molécule RanGTP en RanGDP (guanosine diphosphate) dans le cytoplasme va modifier la structure de l’exportine-5, qui va libérer le pré-microRNA [10]. Dans le cytoplasme, le pré-microRNA, dont la structure est réduite à une tige formée par deux brins plus ou moins bien appariés reliés par une boucle, va être pris en charge par un complexe protéique constitué à nouveau par une endoribonucléase de type III nommée Dicer et par le facteur protéique TARBP2 (Trans-Activation Responsive [TAR] RNA-Binding Protein 2) encore appelé TRBP2 (TAR RNA-Binding Protein 2). Dicer est comparable à une « règle moléculaire » et va permettre la réalisation de deux coupures de part et d’autre des brins à la base de la boucle, générant ainsi un RNA double brin d’environ 22 nucléotides chacun. Le microRNA duplex est ensuite pris en charge par les protéines de la famille Argonaute (AGO) pour former avec d’autres facteurs protéiques le complexe pré-RISC (pre-RNA-Induced Silencing Complex), dont le rôle primordial est de choisir, entre les deux brins, lequel sera conservé et lequel sera éliminé. Les deux brins peuvent être différenciés en prenant en compte leur orientation d’origine dans le pré-microRNA, de sorte que l’on distingue un brin « 5p » et un brin « 3p », qui sont imparfaitement complémentaires. Un des deux brins est sélectionné selon des principes complexes et constituera de ce fait le brin « miRNA » ou brin « guide » ou brin « majeur », le brin complémentaire constituant alors le brin « miRNA* » ou brin « passager » ou brin « mineur », qui est généralement soumis à dégradation. En réalité, la génomique nous apprend que les situations peuvent être très diverses : dans certains cas, le brin 5p est quasiment le seul à être conservé, dans d’autres cas c’est le brin 3p, et souvent les deux brins coexistent dans des pourcentages variables selon les microRNA, voire les types cellulaires, reflétant ainsi la complexité des mécanismes de sélection. Le microRNA correspondant au RNA simple brin sélectionné dans le complexe multienzymatique donnera naissance au complexe RISC (RNA-Induced Silencing Complex), constitué par un microRNA et une protéine Argonaute auxquels viennent s’associer d’autres facteurs tels que la protéine GW182, qui joue un rôle majeur dans la répression traductionnelle et la déstabilisation du mRNA. C’est donc sous forme de ce complexe RISC que les microRNA sont capables de reconnaître leurs mRNA cibles et d’induire l’inhibition de leur expression [9].
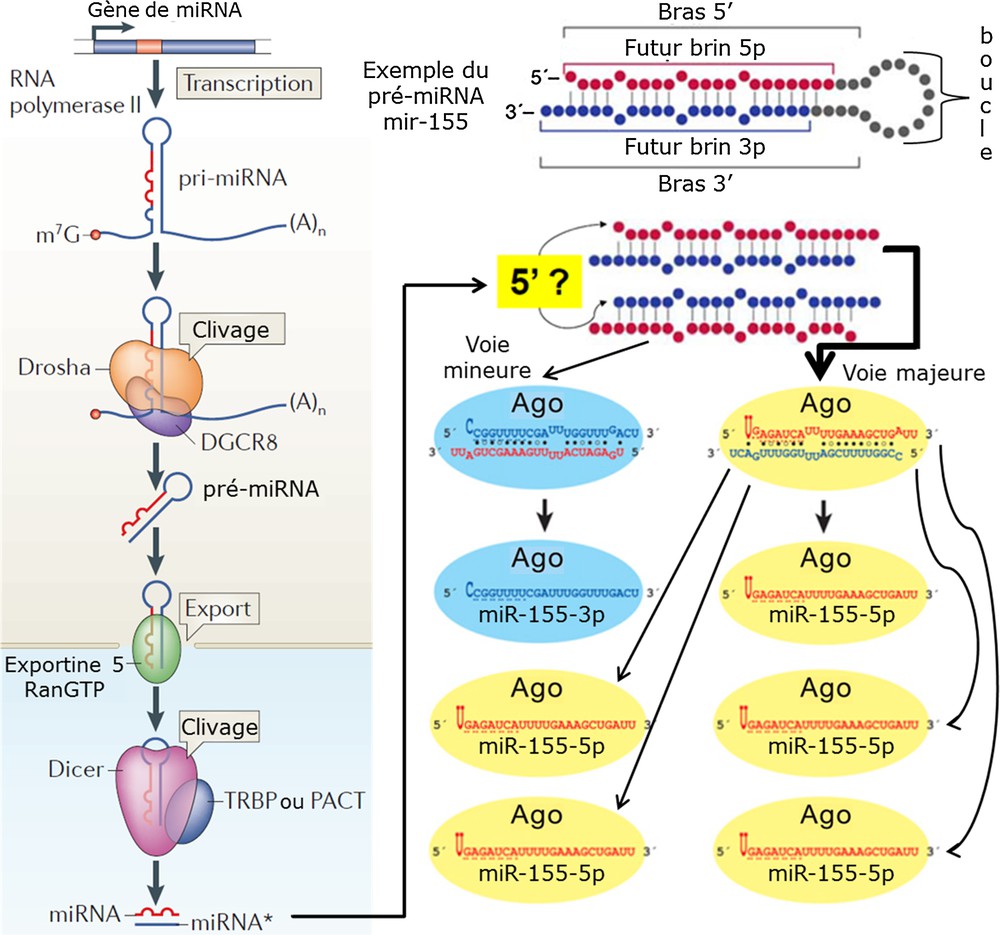
Biogenèse des microRNA. Un gène de microRNA (miRNA) est transcrit par l’RNA polymérase II en un long transcrit primaire (pri-miRNA) qui possède une « coiffe » m7G (7-méthylguanosine triphosphate) à son extrémité 5′ et une « queue » (A)n (polyadénylée) à son extrémité 3′. Le pri-miRNA comporte une région en épingle à cheveux comprenant la/les séquence(s) du/des futur(s) miRNA. Cette structure est clivée à la base de sa tige par le complexe Drosha/DGCR8, ce qui donne naissance au précurseur de miRNA (pré-miRNA). Le pré-miRNA est ensuite transporté du noyau vers le cytoplasme grâce au complexe Exportine-5/RanGTP. Libéré dans le cytoplasme, le pré-miRNA est clivé à la base de sa boucle par le complexe Dicer/TRBP/PACT, générant ainsi un RNA double brin (miRNA/miRNA*) d’environ 22 nucléotides chacun. Ce miRNA duplex, imparfait et asymétrique, est ensuite pris en charge par les protéines de la famille Argonaute (Ago) pour former un complexe dont le rôle primordial est de choisir, entre les deux brins du duplex, lequel sera conservé et lequel sera éliminé. On distingue un brin « 5-p » et un brin « 3-p » par référence à leur orientation d’origine dans le pré-miRNA (mir-155). Selon l’orientation du duplex, le brin 5-p est conservé et le brin 3-p est éliminé, ou au contraire le brin 3-p est conservé et le brin 5-p est éliminé. La sélection repose sur des principes complexes, mais les deux mécanismes peuvent coexister, avec généralement une voie majeure et une voie mineure. On obtient au final un complexe ribonucléoprotéique stable entre un brin d’RNA (miR-155-5p ou miR-155-3p) et une protéine Argonaute (Ago). Masquer
Biogenèse des microRNA. Un gène de microRNA (miRNA) est transcrit par l’RNA polymérase II en un long transcrit primaire (pri-miRNA) qui possède une « coiffe » m7G (7-méthylguanosine triphosphate) à son extrémité 5′ et une « queue » (A)n (polyadénylée) à son extrémité 3′. ... Lire la suite
Les mécanismes intervenant dans la dégradation des microRNA sont, pour leur part, encore très imparfaitement connus. Le temps de demi-vie des microRNA dépend à la fois de l’activité transcriptionnelle de leurs gènes et des processus de maturation, et pour la composante dégradation, de leur séquence (notamment en relation avec des modifications post-transcriptionnelles dans le cas des isoformes), de leur association avec différents facteurs protéiques (tels que les protéines Argonautes), de leur état ou non d’association avec leurs mRNA cibles, de leur localisation dans des structures granulaires cytoplasmiques particulières impliquées dans le stockage et la dégradation des mRNA (telles que les mRNA-processing bodies ou P bodies et les granules de stress) et, in fine, de l’activité d’enzymes encore mal caractérisées [11]. Si les microRNA ont pour leur majorité des temps de demi-vie longs (> 24 heures), certains ont dans des situations physiologiques ou pathologiques particulières des temps de demi-vie beaucoup plus courts en relation avec une vitesse accrue de leur dégradation. Sur le plan de la stabilité on observe donc une certaine hétérogénéité qui dépend à la fois de la nature même du microRNA et du type cellulaire dans lequel il est exprimé [12,13].
Il est communément considéré que la transcription des microRNA s’effectue au niveau du noyau et que les produits générés sont ensuite transportés dans le cytoplasme qui constitue leur lieu d’action principal. Toutefois, la présence de microRNA matures a été démontrée dans le noyau, à la fois dans le nucléoplasme et dans les nucléoles [14], ainsi que dans d’autres compartiments subcellulaires tels que la mitochondrie (« MitomiR » [15]), le réticulum endoplasmique rugueux [11] et le système endosomique [16]. En plus des aspects fonctionnels particuliers en relation avec la présence de microRNA spécifiques dans des organites, ceci peut avoir un impact important sur la durée de vie des microRNA et, par la même occasion, sur la durée de leur action sur les mRNA.
2.2 Mécanisme d’action des microRNA
Pour schématiser, on peut considérer que la spécificité de reconnaissance d’un microRNA est principalement déterminée par la séquence de ses nucléotides 2 à 7 ou 2 à 8, correspondant à la région dénommée seed située du côté 5′ (Fig. 2). Cette séquence hexamérique ou heptamérique s’apparie avec celle du mRNA cible. Quatre nucléotides forment, en aval de celle-ci, la partie centrale du microRNA, mais leur caractéristique étant d’être généralement sans complémentarité avec la cible, cela crée une boucle au sein du duplex constitué par l’association microRNA–mRNA. Quant à la région située du côté 3′, elle montre une complémentarité qui est assez variable selon les cas. Ce qui caractérise donc les microRNA, c’est qu’ils forment le plus souvent un hétéroduplex avec leur cible. Cette propriété les distingue des siRNA (small- ou short-interfering RNA) pour lesquels la complémentarité avec leur cible est stricte tout le long de leur séquence avec pour résultat la formation d’un homoduplex. La formation d’un homoduplex ou d’un hétéroduplex explique en grande partie le fait que le mécanisme d’action au niveau du mRNA cible est généralement différent entre microRNA et siRNA. Au plan physiologique, les microRNA se différencient d’ailleurs des siRNA par d’autres aspects, tels que leur biogenèse et leur finalité. Des RNA double brin ou en épingle à cheveux peuvent être des précurseurs pour la synthèse de siRNA endogènes. Les pseudogènes transcrits en orientation antisens produisent des RNA capables de s’apparier au mRNA du gène correspondant ou aux séquences répétitives intergéniques, à l’instar des transposons. Une fois arrivés au cytosol, ces siRNA sont pris en charge par Dicer et ses protéines associées comme TARBP2 (TRBP2) et PACT (Protein ACTivator of the interferon-induced protein kinase R [RNA-activated]). Les siRNA matures sont ensuite chargés sur les protéines Argonautes 2 (AGO2). Les siRNA exogènes sont issus de RNA doubles brins exogènes (typiquement d’origine virale, mais également synthétiques dans le cas d’expérimentations) par l’action de Dicer et de ses protéines associées (TARBP2 et PACT), puis sont chargés sur AGO1, AGO2, AGO3 ou AGO4 [8]. Les microRNA régulent plutôt le niveau de traduction et la stabilité des mRNA, alors que les siRNA endogènes, en clivant systématiquement leurs cibles, exercent une défense face aux agents pathogènes viraux. Les siRNA participent aussi à l’extinction de l’expression des transposons, et d’une façon plus générale à celle de l’expression de certains RNA responsables d’un bruit de fond nuisible au plan génomique. Les protéines de la famille Argonaute qui interagissent directement avec les microRNA sont des facteurs clés pour l’assemblage et le fonctionnement du complexe RISC. On en retrouve plusieurs classes ; chacune d’entre elles prendra typiquement en charge un modèle d’RNA interférence et agira par un mécanisme pouvant être différent. Par exemple, AGO2 est la seule, parmi les isoformes connues (AGO1 et AGO2 chez la drosophile ; AGO1, AGO2, AGO3 et AGO4 chez les mammifères), qui possède une activité endonucléolytique et qui puisse faire un clivage direct de la séquence cible. Mais, pour cela, il faut que le microRNA et le mRNA cible forment un homoduplex, car le site de clivage d’AGO2 se trouve dans la partie centrale de l’hybride. Par conséquent, les très rares microRNA ayant une complémentarité parfaite avec leurs cibles permettront à AGO2 d’exercer son activité endonucléolytique (ils se comportent là, en fait, comme des siRNA). En réalité, les microRNA forment dans la majorité des cas des hétéroduplex avec leurs mRNA cibles, caractérisés par l’existence d’une boucle dans la partie centrale, ce qui empêche AGO2 d’exercer son activité de clivage. Finalement, comme dans le cas d’une prise en charge des microRNA par les isoformes n’ayant pas d’activité endonucléolytique (AGO1, AGO3 et AGO4), il n’y a pas de clivage direct du mRNA cible, mais un blocage de la traduction. D’une façon générale, les séquences cibles reconnues par les complexes RISC se situent dans la partie 3′ non traduite (3′UTR) des mRNA. Il semble que le blocage de la traduction par les microRNA soit l’élément déterminant dans l’engagement consécutif des mRNA dans un processus de dégradation. Cependant, la vitesse avec laquelle la dégradation des mRNA succède au blocage de leur traduction dépend de nombreux facteurs [17]. Le niveau traductionnel revêt de toute évidence une grande criticité parmi les différents mécanismes de régulation incriminés, et des exemples récents montrent que la localisation subcellulaire des microRNA peut en influencer le déroulement. C’est ainsi que parmi les microRNA mitochondriaux, tous très vraisemblablement d’expression majoritairement nucléaire, certains sont capables d’exercer une action d’activation de la traduction alors que celle-ci serait inhibitrice dans le cytoplasme, ceci posant entre autres questions celle de la nature et de la composition du complexe RISC dans les mitochondries [18]. Il convient de noter aussi que les microRNA semblent pouvoir exercer d’autres fonctions sur le plan de l’expression génomique, lesquelles sont sous-tendues par des mécanismes différents de ceux, post-transcriptionnels, abordés précédemment. Certains, comme hsa-miR-122, sont connus en tant que facteurs majeurs dans la réplication du virus de l’hépatite C, et d’autres contribuent à la répression ou à l’activation transcriptionnelle de certains gènes des cellules eucaryotes.
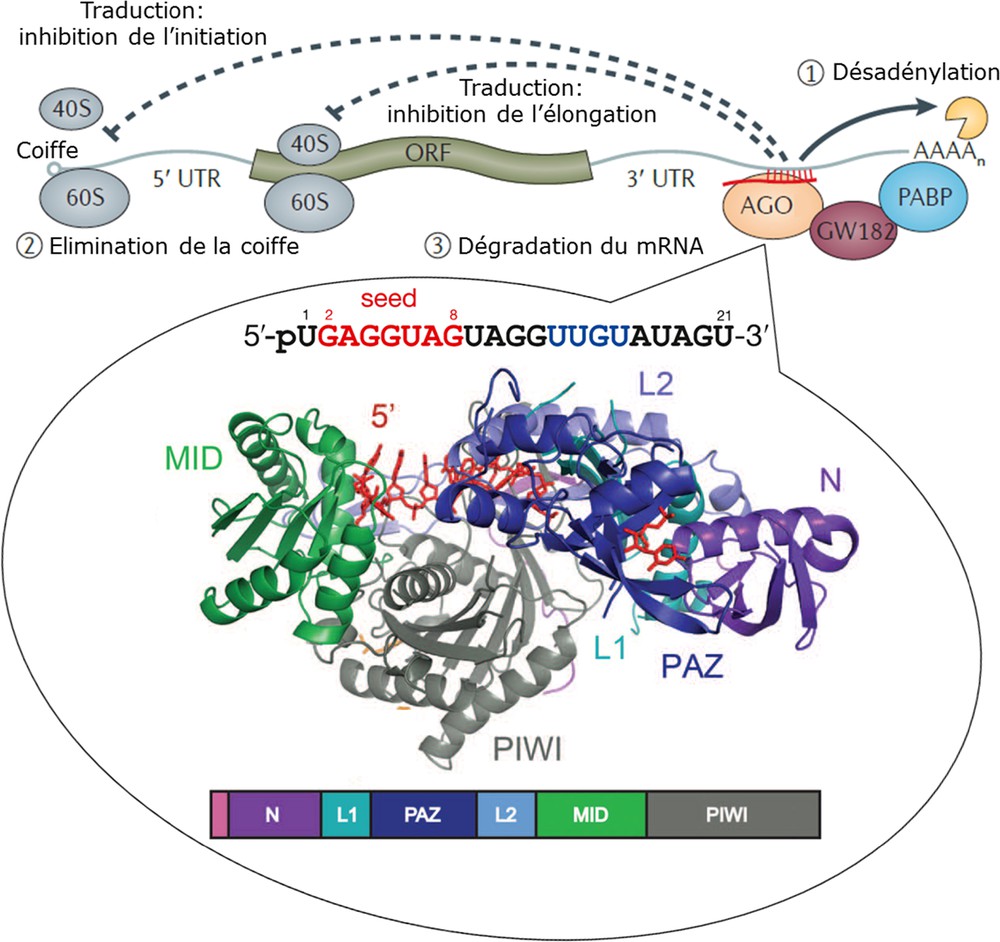
Mécanisme d’action des microRNA. Le complexe ribonucléoprotéique microRNA/Argonaute (AGO) reconnaît, via la séquence seed du microRNA, une séquence complémentaire dans la région 3′UTR (3′ non traduite) d’un RNA messager (mRNA), et est associé via la protéine AGO au facteur protéique GW182, lequel, en interagissant avec la protéine PABP (protéine de liaison de la queue polyadénylée [AAAAn]) joue un rôle essentiel dans la répression traductionnelle et la déstabilisation du mRNA. Il semble que le blocage de la traduction soit un élément déterminant dans l’engagement consécutif des mRNA dans un processus de dégradation. L’inhibition de la traduction peut concerner l’initiation (assemblage des sous-unités ribosomiques 40S et 60S) et l’élongation (progression du ribosome dans la région codante [ORF]). Après désadénylation de sa queue polyadénylée (AAAAn) et élimination de sa coiffe, le mRNA devient sensible à l’action des ribonucléases cytoplasmiques. L’encart montre la structure schématique d’un microRNA et des domaines constitutifs d’une protéine Argonaute, ainsi que la structure 3D du complexe ribonucléoprotéique tirée de la Protein Data Bank (Crystal Structure of Human Argonaute2 [MMDB ID : 117351–PDB ID : 4OLA]). Masquer
Mécanisme d’action des microRNA. Le complexe ribonucléoprotéique microRNA/Argonaute (AGO) reconnaît, via la séquence seed du microRNA, une séquence complémentaire dans la région 3′UTR (3′ non traduite) d’un RNA messager (mRNA), et est associé via la protéine AGO au facteur ... Lire la suite
2.3 Nomenclature des microRNA
Certains microRNA ont gardé leurs noms historiques comme « lin-4 » et « let-7 » [19,20]. Cependant, les microRNA matures, c’est-à-dire ceux inclus dans un complexe RISC, doivent désormais être caractérisés en prenant en compte leurs origines génomiques, leurs homologies de séquences et leurs modes de maturation. Un microRNA est généralement identifié sous la forme suivante : hsa-miR-155. Les trois premières lettres désignent l’espèce (ici Homo sapiens), alors que le nombre (dans ce cas 155) désigne l’ordre de découverte du microRNA chez cette espèce et celui de son incorporation dans la base de données miRBase (http://www.mirbase.org). Historiquement, quand des études mettaient en évidence deux microRNA originaires d’un même précurseur, le plus abondant était nommé miR-155 et le moins abondant miR-155*, ce qui ne donnait aucune indication sur leur orientation d’origine dans le pré-microRNA. Ce mode de notation est désormais abandonné. Quelle que soit l’abondance des deux formes, on distinguera les microRNA sur la base de leur position dans le pré-microRNA de la manière suivante : miR-155-5p (miR-155) et miR-155-3p (miR-155*) par exemple (Fig. 1). Le gène et le précurseur (pré-microRNA) d’un microRNA sont notés « mir », alors que le produit mature correspondant est noté « miR ». Lorsque des microRNA identiques sont obtenus à partir de deux gènes différents, on écrira hsa-mir-24-1 et hsa-mir-24-2 pour désigner les gènes, et par exemple hsa-miR-24-3p pour désigner l’une des formes du microRNA mature. Pour deux microRNA similaires (produits par deux gènes différents), c’est-à-dire ceux qui ont une même séquence de reconnaissance de leurs cibles mRNA (séquence seed comprenant les nucléotides en 5′ des positions 2 à 7 ou 8), mais qui ne différent que par un, deux ou trois nucléotides dans les parties 3′, on désignera les deux gènes en leur conservant un même nombre, mais on les distinguera en ajoutant une lettre : ainsi hsa-mir-10a et hsa-mir-10b seront respectivement à l’origine des microRNA hsa-miR-10a-5p et hsa-miR-10b-5p. On peut trouver toutes sortes de combinaisons comme avec hsa-mir-29 : hsa-mir-29a, hsa-mir-29b-1, hsa-mir-29b-2 et hsa-mir-29c.
2.4 Dérégulations des microRNA
Le processus de biogenèse peut se déréguler à plusieurs niveaux et aboutir à une diminution ou à une augmentation de la quantité de microRNA dans la cellule. Cette dérégulation peut être d’ordre génétique. Ainsi, les microRNA peuvent être dérégulés par une perte chromosomique, une amplification, ou une translocation ou encore par des mutations ponctuelles de leurs gènes. Les facteurs épigénétiques peuvent aussi influencer l’expression des microRNA. La méthylation de résidus cytosine au niveau du ADN (acide désoxyribonucléique) avec l’hyperméthylation ou l’hypométhylation des îlots CpG ainsi que des modifications post-traductionnelles des histones sont fréquemment retrouvées dans de nombreux cancers. Les dérégulations des facteurs de transcription comme p53 et c-Myc peuvent elles aussi jouer un rôle dans la dérégulation des microRNA au niveau de leur promoteur. On peut également trouver des anomalies touchant des protéines intervenant à différentes étapes de la maturation des microRNA et, de ce fait, perturbant leur expression. Celles-ci peuvent être dues à des mutations ou à des dérégulations au niveau du microprocesseur Drosha, de l’exportine-5, ou encore de Dicer ou de son cofacteur TARBP2 (TRBP2). Des études récentes suggèrent que les RNA non codants longs (lncRNA), d’une longueur d’environ 200 nucléotides (ce qui dans la catégorie des RNA non codants et par rapport aux microRNA est considéré comme long), et les RNA circulaires (circRNA) faisant également partie de la famille des RNA non codants, pourraient interagir non seulement avec les autres classes d’RNA mais aussi avec les microRNA. Certains possèderaient des séquences complémentaires de celles des microRNA, créant ainsi une compétition avec leurs mRNA cibles. Pour cette raison, ils sont surnommés « les éponges à microRNA ». En effet, la dérégulation des lncRNA ou des circRNA induit une dérégulation au niveau des microRNA, qui a été retrouvée dans certains cancers [21].
2.5 Fonctions des microRNA
Les microRNA sont des régulateurs principalement post-transcriptionnels entraînant l’inhibition de la production de protéines spécifiques. Ils régulent l’expression d’environ un tiers du génome et provoquent des modifications au niveau des grandes voies de signalisation et du métabolisme. Les microRNA ont donc un rôle essentiel dans les différents processus cellulaires comme la différenciation, la prolifération, ou encore l’apoptose. Si leurs voies de biogenèses sont dérégulées, ils peuvent devenir des acteurs majeurs dans de nombreuses pathologies.
2.6 Sécrétion et libération des microRNA dans le milieu extracellulaire
Les microRNA sont présents non seulement dans les cellules des tissus, mais aussi dans tous les liquides biologiques (Fig. 3). En effet, les microRNA sont retrouvés dans des liquides biologiques aussi divers que le sérum, le plasma, les urines, le liquide céphalorachidien (LCR) et la salive. Plusieurs des microRNA les plus abondants se retrouvent en commun entre plusieurs types de fluides, alors que d’autres ne sont présents que dans des liquides bien spécifiques [22]. La présence des microRNA dans les liquides biologiques a été mise en évidence pour la première fois en 2008 par Chim et al. [23], qui ont démontré que des microRNA d’origine placentaire sont retrouvés au niveau du plasma maternel. La même année, Gilad et al. [24] ont également rapporté la stabilité des microRNA circulants, et les auteurs ont compris dès ce moment-là l’intérêt que pourraient avoir les microRNA comme biomarqueurs. Le passage des microRNA dans le milieu extracellulaire s’effectue par différents mécanismes et sous différentes formes. Ils peuvent être sécrétés par la cellule par l’intermédiaire de vésicules extracellulaires telles que les exosomes et les microvésicules, ou libérés sous forme de corps apoptotiques [25]. On peut également les retrouver, pour certains, associés aux lipoprotéines HDL (lipoprotéine de haute densité) et pour la plus grande partie sous forme de complexes ribonucléoprotéiques avec AGO2 ou NPM1 (nucleophosmin 1) du fait de leur libération par nécrose cellulaire. Les microRNA, vésiculaires ou non, passent ensuite des milieux extracellulaires vers la circulation générale. Environ 90 % des microRNA circulants se trouvent sous une forme non vésiculaire, associés aux protéines Argonautes et plus particulièrement aux AGO2 [26]. Les exosomes constituent une nouvelle forme de communication intercellulaire. Ce sont des petites vésicules membranaires d’origine endosomique de 30 à 100 nm de diamètre. Elles sont sécrétées par différents types de cellules, normales ou pathologiques, et leur production a été observée dans des cellules telles que les cellules hématopoïétiques (cellules dendritiques, lymphocytes B et T, plaquettes), les cellules neuronales et gliales, les cellules épithéliales intestinales ou encore les cellules embryonnaires. Les cellules tumorales ont une activité particulièrement importante de production d’exosomes [27]. En plus des microRNA, les exosomes renferment un ensemble de molécules d’une grande diversité comme des facteurs de traduction, des enzymes du métabolisme, des protéines jouant un rôle dans l’apoptose (Alix), des protéines chaperonnes (protéines de choc thermique : Hsp70 et Hsp90). Ils renferment également d’autres types d’acides nucléiques comme les mRNA. Enfin, ils peuvent transporter des virus et des prions provenant d’une cellule infectée. Un certain nombre d’éléments constitutifs des exosomes pourraient donc être des indicateurs de l’origine de la cellule productrice ainsi que des changements physiologiques ou pathologiques qu’elle subit. Certaines protéines intravésiculaires comme les protéines de choc thermique (Hsp70 et Hsp90) et des protéines transmembranaires comme les tétraspanines (CD9, CD63, CD81, CD82) sont caractéristiques des exosomes, quel que soit le type cellulaire qui les produit. Inversement, certaines protéines, notamment membranaires, comme des protéines d’adhérence cellulaire et des protéoglycanes, sont plutôt spécifiques de leur cellule d’origine. Ceci permet d’identifier l’origine cellulaire de sous-populations d’exosomes dans un liquide biologique parmi l’ensemble de la population des exosomes. La sélectivité relative à la présence de certaines molécules semble répondre à des mécanismes complexes. Les microRNA représentent près de 50 % des RNA totaux vésiculaires, et il a été montré récemment que leur présence impliquait des mécanismes de tri faisant que certains microRNA étaient préférentiellement adressés vers les exosomes [28]. Les exosomes ont été considérés un temps comme un moyen pour la cellule de se débarrasser d’éléments inutiles, comme des protéines obsolètes. Cependant, il est désormais prouvé que ces vésicules sont bien plus que de simples « poubelles cellulaires » et qu’elles jouent un rôle important dans les communications intercellulaires. En effet, après transfert des exosomes de souris à des mastocytes humains, des protéines de souris ont été retrouvées dans les cellules receveuses [29]. Les exosomes servent donc au transfert d’informations (mRNA, virus) et d’autres matériels (protéines, microRNA) d’une cellule à une autre (cellule receveuse). D’une part, il a été montré que les exosomes produits par des cellules soumises à un stress apportent une information visant à induire la résistance des cellules environnantes par effet paracrine. Par exemple, les microRNA régulent l’activité des cellules bêta du pancréas par transfert des exosomes [30]. D’autre part, il semble que les exosomes de cellules tumorales soient capables de participer à distance à la formation des niches prémétastatiques. Les exosomes jouent également un rôle significatif dans l’immunité cellulaire (présentation d’antigène et réponse immunitaire) [31].
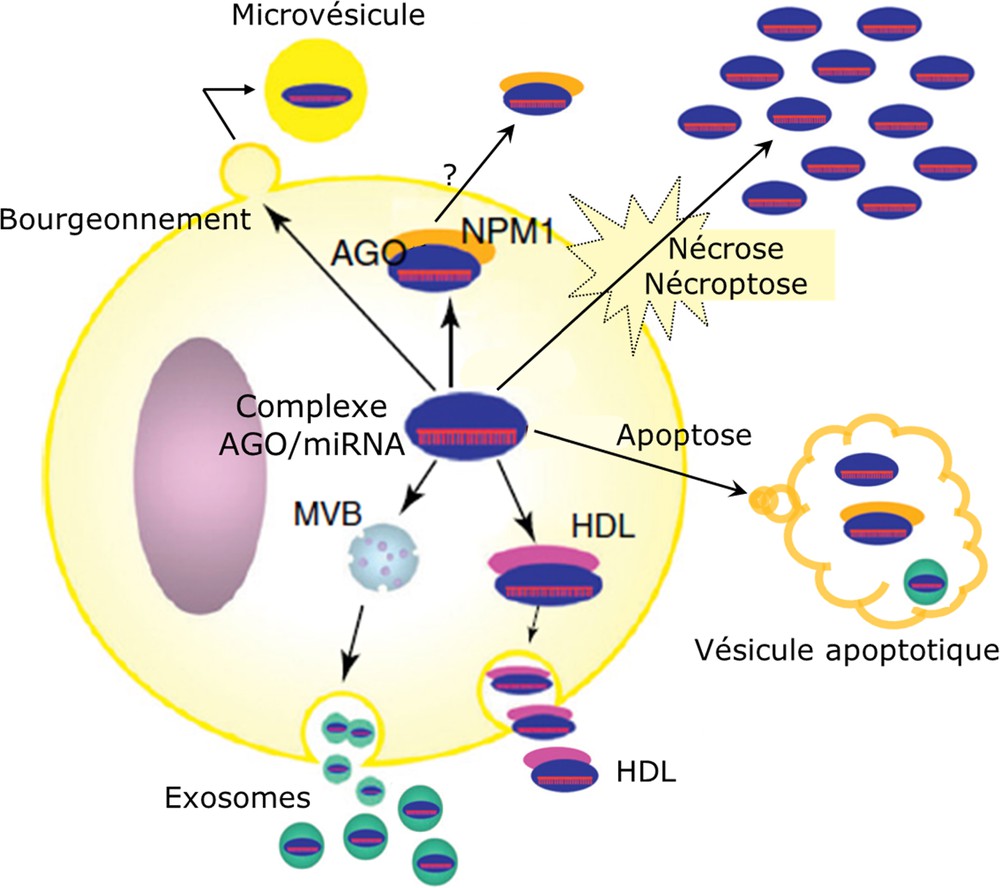
Sécrétion et libération des microRNA dans le milieu extracellulaire. Les cellules tumorales ainsi que les cellules non tumorales libèrent des microRNA dans les milieux extracellulaires, et cette production peut se faire sous plusieurs formes : par sécrétion sous forme d’exosomes via les corps multivésiculaires (MVB) du système endosomique ou sous forme de microvésicules par bourgeonnement de la membrane cytoplasmique, par apoptose sous forme de vésicules apoptotiques, par nécrose ou nécroptose sous forme de complexes libres (non vésiculaires), et par d’autres mécanismes également (complexe ribonucléoprotéique associé à une lipoprotéine HDL ou à la protéine nucléophosmine [NPM1]). Les miRNA passent ensuite des milieux extracellulaires vers la circulation générale. Masquer
Sécrétion et libération des microRNA dans le milieu extracellulaire. Les cellules tumorales ainsi que les cellules non tumorales libèrent des microRNA dans les milieux extracellulaires, et cette production peut se faire sous plusieurs formes : par sécrétion sous forme d’exosomes via ... Lire la suite
3 En quoi les microRNA constituent-ils des biomarqueurs intéressants ?
L’étude des microRNA peut s’inscrire dans une approche complémentaire de celle du ADN (génome), des mRNA (transcriptome) et des protéines (protéome). D’ailleurs, on parle actuellement de miRnome. Malgré une moindre diversité lorsque l’on considère les mRNA ou les protéines, les microRNA sont vraisemblablement par rapport à eux des biomarqueurs plus directement représentatifs de l’activité génomique cellulaire. Ils sont notamment moins profondément affectés que les mRNA par certaines modifications post-transcriptionnelles telles que l’épissage alternatif, par exemple. De plus, sur un plan quantitatif, si l’on compare les microRNA et les protéines, le niveau de sensibilité atteint pour les microRNA du fait de l’amplification inhérente à la PCR (Polymerase Chain Reaction) est assez supérieur à ce dont on dispose actuellement pour les protéines. En outre, dans les liquides biologiques, les microRNA présentent une stabilité bien meilleure que celle des mRNA. En effet, des microRNA ont pu être isolés d’échantillons cliniques archivés, provenant y compris de l’urine et de la salive [32]. Les microRNA « échappent » à la dégradation par les RNAses contenues dans les liquides biologiques grâce à leurs formes de transport, dont font partie les exosomes et les complexes avec les protéines Argonautes. Selon leur forme de transport, vésiculaire ou non vésiculaire, la signification des microRNA en tant que biomarqueurs sera bien différente. Les formes non vésiculaires peuvent refléter un phénomène de souffrance et de mort cellulaire par nécrose ou nécroptose, caractéristique d’un tissu particulier si l’on étudie les microRNA les plus exprimés par ces tissus. En effet, certains microRNA ont une assez forte spécificité cellulaire, tels que miR-122 pour les hépatocytes ou miR-1 et miR-133 pour les cardiomyocytes. Les formes vésiculaires reflètent quant à elles d’autres processus. Différentes études ont montré que les exosomes sont des moyens de communication intercellulaires permettant aux microRNA d’agir sur des voies de signalisation d’autres cellules que celles qui les ont synthétisés. Le profil des microRNA contenus dans les exosomes est en partie spécifique de la cellule qui l’a sécrété et est modifié selon l’état physiologique ou pathologique dans lequel elle se trouve. Ainsi, une cellule tumorale ne sécrétera pas le même profil de microRNA qu’une cellule normale. Le dosage des microRNA dans les liquides biologiques peut donc refléter les changements moléculaires qui se produisent dans les cellules dont ils dérivent, et apporter ainsi des informations d’aide au diagnostic et à la prise des décisions thérapeutiques [33]. Un même microRNA n’est cependant pas toujours spécifique d’une cellule ou d’un tissu, c’est pourquoi la meilleure approche consiste à doser un panel de microRNA, pouvant constituer une sorte de « signature » caractéristique d’une pathologie donnée.
4 Aspects méthodologiques de l’analyse des microRNA utilisés comme biomarqueurs
4.1 Méthodes utilisées
Les méthodes de dosage des microRNA sont essentiellement représentées par les microarrays (microréseaux ou puces), la RT-PCR (Reverse Transcription-Polymerase Chain Reaction) quantitative en temps réel ou qRT-PCR et le deep sequencing (séquençage profond).
4.1.1 Les microarrays
Ce sont des systèmes d’hybridation miniaturisés qui permettent l’analyse simultanée à haut débit de plusieurs centaines de microRNA. Les microarrays ont l’avantage de coûter moins cher que la qRT-PCR et le deep sequencing. En outre, cette méthode offre la possibilité d’effectuer une analyse combinatoire entre les microRNA et l’expression des gènes sur un même échantillon, ce qui permet d’étudier les fonctions des microRNA en connaissant leurs gènes cibles. Le principe de cette méthode repose sur l’hybridation inverse en phase solide. Les sondes sont liées de manière covalente au support solide, alors que les microRNA sont marqués et présents sous phase liquide. Les microRNA sont courts (une vingtaine de nucléotides) ; il est donc difficile de normaliser les températures de fusion (Tm). Ce problème a été résolu dans certaines plates-formes (Exiqon®) en utilisant des nucléotides modifiés ou « LNA » (Locked Nucleic Acid) qui sont incorporés dans les sondes d’hybridation. Avec ces nucléotides, la création d’un pont méthylène entre l’hydroxyle en position 2′ et l’atome de carbone en position 4′ de l’hétérocycle du ribose entraîne une stabilisation conformationnelle de la sonde, ce qui augmente la stabilité des hybrides. Le choix approprié des LNA atténue la différence de Tm entre chaque sonde et leur apporte une excellente spécificité [34]. De plus, grâce à l’incorporation de LNA dans les sondes, il est possible de différencier des microRNA ayant des séquences très proches. L’apparition récente du Nanostring nCounter® constitue une autre innovation dans les méthodes d’hybridation servant à la détection des microRNA. Cette technologie nécessite deux sondes spécifiques pour chaque microRNA d’intérêt. L’une d’elles contient de la biotine et se lie à l’extrémité 3′ pour la fixer à un support solide, alors que la seconde sonde se lie à l’extrémité 5′ et possède une séquence de couleurs particulières qui permet sa détection. Les avantages de cette méthode sont la mesure directe des microRNA sans nécessiter d’amplification ou de clonage, une détection multiplex de plusieurs microRNA dans une réaction unique et la capacité de différencier des microRNA ayant des séquences très proches avec une grande précision [34]. Cependant, le rendement d’extraction des microRNA à partir des liquides biologiques est bien plus faible que celui obtenu à partir des cellules ou des tissus, et les microarrays ne représentent pas vraiment une technique d’analyse quantitative. C’est pourquoi, il convient d’utiliser les microarrays pour effectuer un screening initial qui doit être suivi par une validation individuelle des microRNA d’intérêts par qRT-PCR.
4.1.2 La qRT-PCR
La qRT-PCR est utilisée depuis plusieurs années pour doser les microRNA circulants et est considérée comme la méthode de référence [35]. Elle présente l’avantage d’être facilement utilisable en pratique quotidienne. En outre, elle est sensible, spécifique et offre un large domaine de mesure. Avec cette technologie, les qRT-PCR peuvent être réalisées soit individuellement pour un microRNA donné, soit désormais sous forme de panels de plusieurs centaines de microRNA, ce qui nécessite l’utilisation de microplaques dans lesquelles chacun des puits contient les amorces spécifiques de microRNA différents. Certaines propriétés propres aux microRNA rendent leur détection particulièrement délicate. En effet, la petite taille de ces oligonucléotides et l’absence de séquence commune comme la queue poly (A) pour les mRNA, ne permettent pas l’utilisation d’amorces classiques. C’est pourquoi deux catégories de méthodes sont utilisées selon les plates-formes de qRT-PCR. Elles se distinguent par les amorces et les chimies de détection qui ont été développées. La première repose sur l’utilisation d’une amorce de type « tige-boucle » pour réaliser la transcription inverse du microRNA. Le cADN formé est ensuite amplifié avec des amorces classiques et quantifié en temps réel grâce à des sondes marquées par un fluorophore qui sont ajoutées au milieu réactionnel. Cette méthode est utilisée dans les plates-formes telles que TaqMan Cards® (Life Technologies®). La seconde implique la polyadénylation de l’extrémité 3′ du microRNA en utilisant une enzyme : la polyA polymérase d’Escherichia coli. Ensuite, une amorce antisens contenant une séquence poly (T) et de part et d’autre une séquence universelle en 5′ et une séquence dégénérée en 3′ permet d’effectuer la réaction de transcription inverse, puis l’amplification, de concert avec l’amorce sens. Les plates-formes telles que Qiagen® et Stratagene® utilisent des amorces classiques, tandis qu’Exiqon® utilise des amorces contenant des nucléotides LNA, ce qui leur confère une grande spécificité. Dans tous les cas, les produits d’amplification sont détectés grâce à un agent intercalant, le SYBR® Green, dont la fluorescence augmente d’environ cent fois lorsqu’il est incorporé dans une structure à double brin. Comme cette liaison n’est pas spécifique du ADN double brin, il est importance d’analyser la courbe de fusion afin de détecter la présence d’éventuels produits indésirables. L’introduction récente de la PCR digitale (dPCR) pourrait améliorer la quantification des microRNA. C’est une méthode dérivée de la qRT-PCR, qui se caractérise par une très bonne sensibilité, une précision accrue et une relative insensibilité aux inhibiteurs de la PCR. La dPCR repose sur la distribution de l’échantillon dans des milliers de compartiments individuels ou partitions de volume bien défini. La répartition des microRNA recherchés dans ces partitions s’effectue de manière aléatoire. Après amplification, la mesure de la fluorescence permet de savoir, pour chaque partition, si elle contient ou non les microRNA d’intérêt et d’estimer le nombre de ces microRNA sous l’hypothèse d’une distribution de Poisson. Ainsi, il est possible de réaliser une quantification absolue des microRNA sans avoir recours à des étalons ou à des microRNA de références pour la normalisation [36].
4.1.3 Le deep sequencing
C’est une méthode basée sur le séquençage de nouvelle génération qui connaît actuellement un essor important. Il s’agit d’une technique robuste qui détermine le profil d’expression des microRNA grâce au séquençage rapide de millions de séquences simultanément ; on parle de « miR-séquençage » [37]. Les instruments actuels sont capables de réaliser 200 millions (ou plus) de lectures par librairie et peuvent multiplexer 48 (ou plus) librairies en une seule fois [38]. Par rapport aux puces, le deep sequencing analyse une plage plus large. Il n’est pas sujet aux problèmes de bruit de fond et d’hybridation croisée et peut permettre l’identification de nouveaux microRNA. Le deep sequencing a également permis de caractériser plusieurs « isomiR », qui sont des microRNA présentant des variations dans leurs séquences (à leur extrémité 3′) par rapport à la référence ; ceci est d’autant plus important que les isomiR sont impliqués dans plusieurs pathologies humaines, notamment les cancers. Malgré tout son intérêt, le deep sequencing reste encore peu utilisé à cause de son coût élevé et de la nécessité d’avoir des bio-informaticiens entraînés pour l’analyse d’un nombre important de données qui sont fournis par cette méthode [39].
4.1.4 Avantages et inconvénients des différentes méthodes
Mestdagh et al. [40] ont comparé 12 plateformes disponibles dans le commerce, en utilisant 20 contrôles positifs et négatifs normalisés (Tableau 1). Cette étude a montré des différences significatives entre les performances analytiques de ces plateformes en termes de reproductibilité, sensibilité, spécificité, précision, ainsi que de concordance de l’expression différentielle. Ces différences concernent non seulement les plateformes basées sur des méthodes d’analyses différentes, mais aussi celles qui font appel au même principe de dosage. Dans une méta-analyse récente, les données brutes de 1539 et 705 miRnomes obtenus respectivement par les microarrays et le deep sequencing ont également montré l’impact du choix de la plate-forme sur les microRNA. Ainsi, le deep sequencing reconnaît essentiellement les microRNA riches en uracile, tandis que les microarrays détectent surtout les microRNA riches en guanine [41]. Chaque méthode possède ses avantages et ses limites. Le choix d’une plate-forme doit donc être guidé par l’objectif de l’analyse, et il est vivement recommandé pour les études rapportant une nouvelle signature de valider celle-ci par une autre méthode.
Comparaison entre les différentes méthodes de dosage des microRNA circulants.
| Méthode | Microarrays | qRT-PCR | Deep sequencing |
| Sensibilité | Moyenne | Excellente | Bonne |
| Domaine de mesure | Moyen | Très large | Large |
| Dosage de nouveaux microRNA | Non | Non | Oui |
| Praticabilité | Utilisation facile | Utilisation facile | Difficile |
| Coût | Relativement bas | Élevé | Très élevé |
| Exemples de plateformes | Microarray (Affymetrix) Microarray (Agilent) |
miRCury® (Exiqon) miScript® (Qiagen) TaqMan® Cards (Life technologies) |
TruSeq® (Illumina) Ion Torrent® (Life Technologies) |
4.2 Difficultés méthodologiques liées à l’analyse des microRNA circulants
Il y a peu de chevauchements des résultats obtenus dans la littérature : ce peut être en partie expliqué par les variables pré-analytiques et analytiques, à savoir :
- • le choix du type d’échantillons utilisés pour l’analyse et les conditions pré-analytiques ;
- • la contamination par les cellules sanguines ;
- • et surtout le manque de consensus dans la normalisation des résultats.
4.2.1 Variables pré-analytiques
Le choix de l’anticoagulant contenu dans les tubes de prélèvement influence considérablement la nature et la quantité des microRNA dosés. Le sérum semble avoir un contenu un peu plus important en microRNA que le plasma [42]. Le volume de liquide utilisé pour réaliser l’extraction peut également influencer les résultats ; il est donc important que ce volume soit clairement décrit dans le protocole [43]. L’hémolyse augmente certains microRNA, comme miR-16 et miR-451. Ceci est d’autant plus important à prendre en considération que miR-16 est largement utilisé pour la normalisation des résultats. Les conditions du recueil des liquides biologiques autres que le sang n’ont pratiquement pas été étudiées. Le choix de la vitesse et du temps de la centrifugation dépend, d’une part, du microRNA recherché et, d’autre part, des contaminants que l’on souhaite éliminer. En effet, les débris cellulaires, les corps apoptotiques et les plaquettes sanguines ont des profils d’expression qui leurs sont propres. Lorsque le plasma est utilisé comme matériel biologique, il est recommandé de préparer un plasma appauvri en plaquettes, en réalisant une double centrifugation avec une première centrifugation du sang vers 800 g, puis une seconde centrifugation du plasma vers 12 000 g pour éliminer les plaquettes qui sont une source riche en microRNA susceptibles de fausser l’interprétation des résultats. Cette extraction globale ne permet pas de différencier les différentes formes circulantes de microRNA. C’est pourquoi, une ultracentrifugation (−110 000 g) ou une autre procédure plus praticable (précipitation sélective, ultrafiltration…) est indispensable pour séparer les exosomes et les microvésicules. En ce qui concerne les délais de conservation, les échantillons peuvent être conservés au moins 72 heures à +4 °C et plusieurs mois à −20 °C ou à −80 °C. D’après une étude ayant montré que la concentration de certains microRNA diminue à 4 °C [44], il serait préférable de congeler immédiatement les échantillons à −80 °C. L’effet des cycles de congélation/décongélation est controversé, avec des résultats souvent contradictoires.
Malgré le fait que les microRNA présentent globalement une meilleure stabilité par rapport aux mRNA, on note une différence considérable dans le profil d’expression des microRNA en fonction des conditions de conservation des échantillons à cause d’une dégradation inhomogène des microRNA dans les liquides biologiques. On remarque ainsi que les microRNA associés à des formes vésiculaires sont plus stables dans le sérum et résistent mieux au traitement par la RNase que ceux non associés à des vésicules. Comme de nombreux microRNA sont inégalement répartis entre les formes non vésiculaires et les formes vésiculaires, cela pourrait expliquer des comportements différents dans des conditions favorisant les mécanismes de dégradation. Mais la séquence de chaque microRNA ainsi que la structure des formes non vésiculaires et leur association ou non avec leurs cibles doivent constituer des facteurs différenciant importants. Köberle et al. [45] ont constaté que l’utilisation d’inhibiteurs des RNases permet de conserver les microRNA sériques non vésiculaires, en protégeant notamment les microRNA les plus sensibles à la dégradation. Ces résultats sont encourageants, car ils apportent une issue à ce qui sinon constituerait un écueil dans l’essor des microRNA comme biomarqueurs, et permettent d’envisager de découvrir de nouveaux microRNA qui échappaient à la détection à cause d’une dégradation rapide. Au total, ces éléments sont à prendre en considération si l’on souhaite qualifier des microRNA en tant que biomarqueurs. Mais, en réalité, la problématique concernant la stabilité des biomarqueurs ne leur est pas réservée et se retrouve également dans le cas des peptides et des polypeptides, par exemple.
L’extraction des microRNA peut être réalisée par le mélange thiocyanate de guanidinium/phénol/chloroforme ou par des kits d’extraction commercialisés tels que miRNeasy® (Qiagen), mirVana® (Ambion) et PureLink® (Invitrogen). Au même titre que les méthodes de dosage, le profil d’expression des microRNA circulants est dépendant de la méthode d’extraction utilisée [46]. Par ailleurs, les microRNA dérivés des exosomes vésiculaires peuvent provenir d’un processus d’exocytose et donc avoir une composition en lipides différente de celle des microRNA cellulaires. C’est pourquoi il est important d’utiliser des méthodes d’extraction adaptées. L’utilisation du phénol combinée à l’ultrafiltration sur colonne permet d’obtenir un bon rendement [47]. L’état de jeûne peut influencer le profil d’expression des microRNA de deux manières. Premièrement, certaines molécules qui participent au transport des microRNA comme les HDL sont affectées par le jeûne. Deuxièmement, l’augmentation des lipoprotéines qui suit un repas interfère avec la phase de l’extraction [48]. D’autres variables pré-analytiques peuvent aussi influencer les résultats telles que l’âge, le sexe, l’origine ethnique, l’activité physique, le rythme circadien, la prise de médicaments ou encore l’altitude [49].
4.2.2 Stratégies de normalisation pour la quantification relative des microRNA
Il existe deux méthodes de quantification des microRNA : la quantification absolue et la quantification relative. Dans la première, une courbe d’étalonnage est établie à partir de microRNA synthétiques qui sont amplifiés parallèlement aux échantillons biologiques. Cependant, ces oligonucléotides synthétiques dilués dans de l’eau n’ont pas forcément la même composition que les microRNA endogènes. Dans la seconde, il est nécessaire de normaliser les résultats par rapport à des gènes de référence endogènes ou exogènes, afin de déterminer avec précision les niveaux d’expression des microRNA circulants. En l’absence de consensus, les stratégies de normalisation diffèrent considérablement d’une étude à l’autre, ce qui rend les résultats non comparables et difficilement interprétables. Dans la quantification relative, la valeur du cycle seuil des microRNA cibles est comparée à celles des gènes de références. Cette approche vise à atténuer les différences dues à la qualité et au traitement des échantillons. Parmi les gènes de références endogènes, on retrouve les RNU6A et RNU6B, qui ont été très largement utilisés. Pourtant, ils appartiennent à une classe de petits RNA nucléaires (snRNA) ayant des propriétés différentes de celles des microRNA. Le microRNA miR-16 est également très couramment utilisé pour la normalisation des résultats. Il a été décrit comme étant stable et hautement exprimé dans le sang. Cependant, l’hémolyse peut modifier son taux et son expression est dérégulée dans certaines pathologies comme le cancer de la prostate. Plusieurs combinaisons de microRNA ont été proposées comme références en fonction de la pathologie concernée. Par exemple, l’association de miR-16 avec miR-93 semble être une bonne solution pour la normalisation des résultats chez les patients ayant un cancer gastrique, et l’évaluation de la stabilité de l’expression de 11 gènes de références dans des exosomes circulants a montré que la combinaison de plusieurs microRNA (miR-221, miR-191, let-7a, miR-181a et miR-26a) est la plus appropriée pour la normalisation de microRNA sériques spécifiques du foie [50]. Parmi les références exogènes, on retrouve essentiellement des microRNA synthétiques ou appartenant à C. elegans comme cel-miR-39. L’utilisation des gènes de références endogènes permet de limiter les différences dues à l’échantillonnage alors que les références exogènes sont utiles pour minimiser la variabilité due à la technique d’analyse. La meilleure solution pour la normalisation des résultats semble donc être la combinaison de ces deux types de gènes de référence [51].
5 Les applications des microRNA comme biomarqueurs
Dans les pathologies humaines, les différents intérêts des biomarqueurs ne se limitent pas au diagnostic ou au suivi thérapeutique. On peut attendre d’eux une précocité leur permettant de prédire l’apparition de la maladie avant même la manifestation de ses symptômes ou celle de ses complications (approche de stratification des risques). Ce caractère de prédictivité est tout particulièrement considéré dans les cancers, pour lesquels sont recherchés des marqueurs de l’agressivité ou du risque de métastases ainsi que de la réponse au traitement.
Les microRNA étant impliqués dans un nombre important de pathologies, nous ne pourrons pas traiter ici toutes les maladies dans lesquelles les microRNA pourraient être utilisés comme biomarqueurs. Nous avons choisi d’aborder quelques applications actuelles des microRNA circulants ainsi que leurs perspectives dans des domaines où leur émergence en tant que biomarqueurs semble imminente, à savoir les cancers, les maladies cardiovasculaires, le diabète sucré, les maladies du système nerveux central, les pathologies hépatiques et rénales.
5.1 Cancers
Les cancers représentent la troisième cause de mortalité dans le monde après les maladies cardiovasculaires et infectieuses, et la deuxième cause de décès en Europe. En 2012, 14,1 millions de nouveaux cas et 8,2 millions de décès ont été recensés dans le monde [52]. Les causes connues des cancers aboutissent toutes, d’une manière ou d’une autre, à l’altération du ADN. Le cancer est donc une maladie fondamentalement génétique. Plusieurs points plaident en faveur de l’implication des microRNA dans le développement des maladies cancéreuses. D’abord, environ 50 % des gènes codant pour des microRNA se trouvent dans des sites fragiles qui présentent des amplifications, des délétions ou encore des translocations de séquences de ADN au cours du développement des tumeurs. Par ailleurs, les modifications épigénomiques tumorales générant des régions hyperméthylées ou au contraire hypométhylées ont un retentissement au niveau de la transcription des gènes de microRNA. Or, les microRNA régulent l’expression de nombreux gènes codant des protéines, soit oncogènes, soit suppresseurs de tumeurs, d’où leur nom « oncomiR ». Il a été démontré récemment que l’hypoxie accompagnant le processus cancéreux inhibe la synthèse des enzymes Drosha et Dicer, ce qui diminue le pool global de microRNA, favorisant ainsi la croissance tumorale et l’apparition des métastases [53]. En outre, il existe de nombreuses interactions entre les fonctions des microRNA et les grandes voies de l’oncogenèse (Fig. 4). Les microRNA peuvent donc servir de marqueurs tumoraux utiles au dépistage, au diagnostic, à la classification et au pronostic des maladies cancéreuses, ainsi qu’au suivi thérapeutique de ces maladies. La première utilisation des microRNA comme biomarqueurs dans le domaine de la cancérologie remonte à 2008, quand Lawrie et al. ont découvert le rôle potentiel de miR-21 dans le diagnostic et le pronostic du lymphome diffus à grandes cellules B [54]. Depuis, le nombre de microRNA pouvant servir comme biomarqueurs des cancers ne cesse d’augmenter dans la littérature. Le concept de « biopsie liquide » a connu un essor considérable au cours de ces dernières années. On inclut dans les biopsies liquides la recherche dans les liquides biologiques du ADN tumoral, des cellules tumorales et des microRNA [55]. Deux avantages majeurs en découlent. Premièrement, ces examens sont non invasifs et, deuxièmement, il est possible de suivre l’évolution de la cinétique de ces marqueurs lors du suivi thérapeutique.
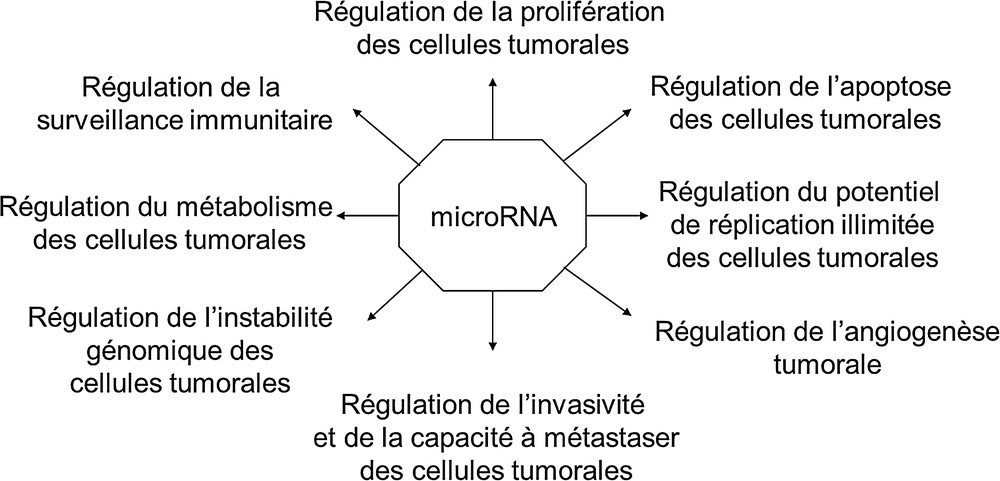
Interaction des microRNA avec les grandes voies de l’oncogenèse Parmi la dizaine de caractéristiques distinctives nécessaires aux cellules tumorales pour qu’elles acquièrent les capacités spécifiques dictant leur survie, leur prolifération et leur dissémination, on en retrouve une large majorité pour lesquelles l’implication des microRNA a été établie.
5.1.1 Cancer du poumon
Il s’agit du cancer dont le taux de mortalité est le plus élevé dans le monde, à la fois chez la femme et chez l’homme. Malheureusement, le diagnostic est le plus souvent établi tardivement lorsque les métastases sont installées et que la guérison devient difficile. La tomodensitométrie (TDM) à faible dose est une technique d’imagerie qui permet le dépistage de cette maladie à un stade précoce, ce qui réduit de manière significative la mortalité. C’est pourquoi cette technique a été recommandée pour le dépistage du cancer du poumon chez les sujets à haut risque [56,57]. Les microRNA pourraient constituer un outil de sélection de ces personnes à risque, diminuant ainsi le risque d’exposition inutile à des radiations chez les sujets non atteints. Dès 2004, il a été observé qu’environ la moitié des gènes de microRNA connus à l’époque se trouvaient dans des régions génomiques associées aux cancers ou dans des sites fragiles, et que plusieurs microRNA transcrits à partir de ces régions délétées étaient faiblement exprimés au cours du cancer du poumon [58]. Au cours de la même année, Takamizawa et al. ont également rapporté une diminution de l’expression de let-7 au niveau du tissu tumoral dans les cancers du poumon non à petites cellules [59]. Par la suite, plusieurs autres études ont retrouvé une dérégulation des microRNA au cours du cancer du poumon [60]. Les exosomes dérivés des tumeurs sont abondants dans les liquides biologiques et contiennent des concentrations élevées en microRNA, qui reflètent le processus tumoral et son influence sur les cellules voisines et éloignées, ce qui en fait des biomarqueurs et des cibles thérapeutiques potentielles. Deux tests sont actuellement en phase avancée dans le processus de validation en tant que biomarqueurs. Ces tests ont été développés indépendamment par deux instituts du cancer à Milan : le « European Institute of Oncology » (EIO) et l’« Istituto Nazionale dei Tumori » (INT). D’une part, l’INT a développé à partir des échantillons plasmatiques de 939 participants comprenant 69 patients atteints du cancer du poumon et 870 sujets témoins, une signature de 24 microRNA appelée microRNA signature classifier (MSC), qui permet le diagnostic précoce de ce cancer. Le test MSC a montré une sensibilité, une spécificité et une valeur prédictive négative (VPN) respectivement de 87, 81 et 99 %. Ces résultats sont comparables à ceux de la TDM à faible dose, qui présente une sensibilité, une spécificité et un taux de faux positifs respectivement de 79, 81 et 19,4 %. L’association du test MSC à la TDM à faible dose permet aussi de réduire le taux de faux positifs à 3,7 % [61]. D’autre part, l’EIO a développée sur les sérums de 1115 sujets à risque enrôlés dans l’étude Continuous Observation of Smoking Subjects (COSMOS) une signature de 13 microRNA, qui est appelée « miR-Test ». Parmi ces 13 microRNA, cinq sont communs avec le test MSC. Le miR-Test offre une sensibilité, une spécificité et une VPN respectivement de 77,8, 74,8 et 99 % [62]. Malgré une spécificité inférieure à celle de la TDM à faible dose, le miR-Test pourrait trouver sa place dans la sélection des patients à risque qui nécessitent la réalisation de la TDM à faible dose grâce à sa forte VPN. Le test MSC et le miR-Test sont en cours d’évaluation dans des études prospectives sur deux nouvelles cohortes, respectivement bioMILD et COSMOS2. Les microRNA sont associés à des mutations touchant des molécules impliquées dans la signalisation comme EGFR (Epidermal Growth Factor Receptor), Ras ou Alk, ainsi que des suppresseurs de tumeurs tels que PTEN (phosphatase and tensin homologue deleted on chromosome 10) ou p53 [63]. Les microRNA pourraient donc aussi avoir un intérêt pronostique en détectant les patients qui risquent de développer une tumeur agressive et qui nécessitent un traitement agressif dès le départ. Par exemple, miR-21 est l’un des microRNA les plus étudiés et a pour cibles les gènes des suppresseurs de tumeurs PTEN et PDCD4 (Programmed Cell Death 4). La surexpression de miR-21 dans le sang est associée aux métastases ganglionnaires, à un stade avancée de la maladie et à un faible taux de survie dans le cancer du poumon, mais aussi dans d’autres cancers [64]. Les microRNA peuvent aussi être utiles dans le suivi thérapeutique. D’autant plus que certains d’entre eux sont eux-mêmes impliqués dans le phénomène de résistance à la chimiothérapie. Ainsi, une augmentation de miR-22 dans le sang prédit une mauvaise réponse au Pémétrexed (un analogue de l’acide folique) chez les patients atteints du cancer du poumon non à petites cellules [65].
5.1.2 Cancer de la prostate
Le cancer de la prostate représente la tumeur maligne la plus fréquemment diagnostiquée chez l’homme et la deuxième cause de décès par cancer après celui du poumon. Sur le plan expérimental, des études réalisées chez la souris montrent qu’une diminution concomitante de miR-15 et miR-16 associée à l’élévation du taux de miR-21 au cours du cancer de la prostate active de manière aberrante la voie du TGF-β (Transforming Growth Factor β), favorisant les métastases et l’ostéolyse par invasion des cellules cancéreuses [66]. L’utilisation d’un panel de quatre microRNA sériques pour le diagnostic du cancer de la prostate a montré une aire sous la courbe de 0,783 et une valeur prédictive positive (VPP) de 80 %. Parmi ces microRNA, on retrouve let-7a, qui est un suppresseur de tumeur et miR-141, qui est au contraire un oncogène [67]. Les concentrations de miR-107 et miR-574-3p sont également beaucoup plus élevées dans les urines d’hommes atteints du cancer de la prostate que chez des hommes sains [68].
5.1.3 Cancer du sein
C’est la deuxième principale cause de décès par cancer chez la femme. Les microRNA miR-137 et miR-138 régulent l’activité de KDM5, une histone déméthylase, qui contrôle la prolifération et la migration des cellules du cancer du sein [69]. Le microRNA miR-21 intervient également dans les processus de prolifération et de migration des cellules cancéreuses en inhibant Smad7 au travers des voies de signalisation de l’EGF et du TGF-β [70]. L’analyse par puce ultrasensible d’une cohorte de 1280 échantillons sériques de patientes atteintes du cancer du sein provenant de la National Cancer Center Biobank (NCCB) par comparaison à des sujets témoins et à des sujets atteints d’autres types de cancer a montré l’intérêt de la combinaison de cinq microRNA (miR-1246, miR-1307-3p, miR-4634, miR-6861-5p et miR-6875-5p) dans la détection précoce de la maladie [71]. Les microRNA circulants pourraient également avoir une valeur pronostique. En effet, il a été suggéré dans une étude récente qu’un panel de quatre microRNA sériques pourrait constituer un facteur prédictif de rechute et de survie globale chez les patientes atteintes d’un cancer du sein triple négatif [72]. Ce dernier doit son nom à l’absence des récepteurs hormonaux des estrogènes, de la progestérone ainsi qu’à l’absence de la surexpression du récepteur HER-2. Le cancer du sein triple négatif représente près de 15 % des cancers du sein, et il est associé à un pronostic plus péjoratif.
5.1.4 Cancer du pancréas
En 2012, le cancer du pancréas a représenté la quatrième cause de décès dans l’Union européenne et la septième dans le monde [73]. Ce cancer est de très mauvais pronostic. Le seul traitement curatif est la chirurgie, mais celle-ci n’est envisagée que dans les stades précoces. Certains microRNA circulants permettent non seulement de différencier les patients atteints d’un cancer du pancréas des sujets sains, mais aussi des patients présentant d’autres types de cancers (œsophage, estomac, foie et côlon) avec une exactitude supérieure à celles du CA19-9 (carcinoma antigen 9-9) et de l’ACE (antigène carcinoembryonnaire) [74]. Par exemple, le dosage de miR-744 au niveau sérique pourrait servir au diagnostic, au pronostic et au suivi de la chimiothérapie [75]. Deux microRNA salivaires possèdent également un bon pouvoir discriminatoire pour détecter le cancer du pancréas résécable, avec une sensibilité et une spécificité acceptables [76]. Il est intéressant de noter que, contrairement à ce qu’on observe dans la majorité des cancers, l’augmentation de miR-21 dans le sérum serait de bon pronostic dans le cancer du pancréas [77].
5.1.5 Cancer hépatocellulaire
Le cancer hépatocellulaire est le sixième cancer le plus fréquent et la troisième cause de décès par cancer dans le monde [78]. La recherche de nouveaux marqueurs pour cette pathologie est particulièrement intéressante, vu le manque de spécificité des marqueurs actuels. De nombreux microRNA sont impliqués dans le développement de ce cancer, en modulant diverses voies de transduction du signal. Ainsi, il a été démontré que l’inhibition de miR-181 réduisait le nombre des cellules cancéreuses hépatiques et que l’ajout de miR-181 exogène leur permettait de reprendre leur croissance [79]. Une étude comparative des profils d’expression des microRNA provenant des cellules de carcinomes hépatocellulaires avec ceux des exosomes dérivés de ces cellules a montré que ces exosomes favorisent la croissance cellulaire, la migration et l’invasion des cellules cancéreuses, et sert de véhicule pour le transport des microRNA qui induisent ces signaux [80]. Le dosage de miR-126 en association avec l’AFP (Alpha Fœto-Protéine) constitue un marqueur non invasif pour le diagnostic et le suivi des patients ayant un cancer hépatocellulaire et une hépatite B [81]. En outre, un panel de sept microRNA (miR-29a, miR-29c, miR-133a, miR-143, miR-145, miR-192 et miR-505) a été proposé pour la détection et la classification des cancers hépatocellulaires à partir d’une étude multicentrique portant sur des sujets contrôles, des porteurs inactifs d’Ag HBs, des personnes atteintes d’hépatite B et de cirrhoses induites par l’hépatite B, ainsi que des patients présentant des cancers hépatocellulaires provenant de quatre hôpitaux de Chine. Cette étude s’est déroulée en trois étapes. La première cohorte comprenait 257 participants. Deux groupes de validations de 352 et 139 participants ont ensuite été recrutés. La troisième partie était une étude cas témoins de validation portant sur 27 patients atteints de carcinomes hépatocellulaires et 135 témoins [82].
5.1.6 Cancer colorectal (CCR)
Le CCR est particulièrement fréquent en France avec 42 152 nouveaux cas et 17 722 décès en 2012. Plusieurs études ont établies des signatures pouvant servir au dépistage précoce du CCR. Ainsi, Pu et al. ont analysé les niveaux d’expression plasmatiques des microRNA chez 103 patients atteints de CRC et 37 sujets témoins. Le microRNA miR-221 permet de différencier les deux groupes avec une sensibilité de 86 % et une spécificité de 41 % [83]. Les microRNA miR-29a et miR-92a ont également un intérêt diagnostique pour ce cancer, avec une sensibilité de 73 % et une spécificité de 79 % [84]. Le microRNA miR-92 a été validé chez 180 participants comprenant 90 patients atteints de CRC, 20 patients ayant un cancer gastrique, 20 patients présentant une maladie inflammatoire des intestins et 50 sujets contrôles pour différencier le CCR du cancer de l’estomac [85]. En outre, les microRNA circulants peuvent servir au pronostic ou à l’identification des patients ayant un risque important de rechutes. En effet, l’augmentation de miR-141 dans le plasma est associée à un risque important de métastases et à un mauvais pronostic [86], alors que le microRNA miR-29-c prédit une rechute précoce [87].
5.1.7 Cancers du système nerveux central (SNC)
Ces dernières années, un certain nombre d’études ont révélé également l’intérêt que pourraient avoir les microRNA circulants comme biomarqueurs des cancers du SNC. Il est ainsi possible, grâce à ces microRNA, de différencier les tumeurs cérébrales primitives des métastases cérébrales provenant d’autres tumeurs. Par exemple, miR-9/9*, qui est fortement exprimé dans le cerveau, est révélé dans les tumeurs du SNC et absent dans d’autres tumeurs [88]. Les glioblastomes sont les tumeurs les plus agressives et mortelles du SNC ; c’est pourquoi la recherche de biomarqueurs prédictifs de la survie des patients et du risque de récurrence de la tumeur est primordiale. Comme pour les autres pathologies cancéreuses, plusieurs signatures de microRNA circulants ont été décrites. Un modèle prédictif des glioblastomes combinant cinq microRNA plasmatiques a montré une aire sous la courbe de 0,99 pour discriminer des sujets porteurs d’une tumeur cérébrale des sujets contrôles [89]. Le LCR des patients porteurs de tumeurs cérébrales contient des microRNA pour certains caractéristiques du type de tumeur [90], dont une partie sous forme d’exosomes provenant de la tumeur et qui pourraient eux aussi servir de biomarqueurs [91]. Le médulloblastome est la tumeur du SNC la plus fréquente chez l’enfant. Il est dû à une altération du développement des cellules progénitrices cérébelleuses. Les concentrations de plusieurs microRNA comme miR-124, miR-125 et miR-9 sont abaissées, suggérant leurs rôles en tant que suppresseurs de tumeurs [92].
5.2 Maladies cardiovasculaires
Les microRNA sont impliqués dans la régulation de l’organogenèse du cœur à partir de cellules souches embryonnaires cardiaques, ainsi que dans le fonctionnement du système cardiovasculaire chez l’adulte. Plusieurs microRNA sont dérégulés dans les maladies cardiovasculaires telles que l’infarctus du myocarde, l’athérosclérose, l’insuffisance cardiaque, la fibrillation auriculaire, l’hypertrophie et la fibrose cardiaque [93]. L’athérosclérose est responsable de la majorité des évènements cardiovasculaires ; elle se caractérise par des dépôts de lipides et d’éléments fibreux dans la paroi des artères. La formation de cette plaque d’athérome est la résultante d’interactions entre les cellules immunitaires, les cellules musculaires lisses et l’endothélium vasculaire. Les microRNA régulent les fonctions de ces cellules ainsi que le métabolisme du cholestérol [94]. Certains microRNA comme miR-155 peuvent exacerber les stades précoces de l’athérosclérose en stimulant la réaction inflammatoire et le captage de lipides par les macrophages. À l’inverse, d’autres microRNA tels que miR-126 possèdent une action antiathérogène [95]. L’infarctus du myocarde (IDM) est une pathologie qui met en jeu le pronostic vital du patient, d’où l’intérêt d’établir le diagnostic rapidement. L’étude du profil d’expression des microRNA par qRT-PCR a objectivé une élévation de certains microRNA circulants, comme miR-499, miR-208b, chez 32 patients atteints d’IDM, comparativement à 36 sujets témoins [96,97], permettant le diagnostic de l’IDM avec une bonne sensibilité et une bonne spécificité dans les 4, 8, 12, 24 et 72 heures qui suivent l’infarctus [98]. Par ailleurs, dans une étude menée sur des sérums d’une cohorte de 444 patients avec IDM, la mesure quantitative de certains microRNA par qRT-PCR révèle une association indépendante entre ces molécules et la troponine ultrasensible. De plus, miR-133a et miR-208b sont associés au risque de mortalité, ce qui en fait des marqueurs pronostiques potentiels [99]. Une signature spécifique de microRNA a été découverte dans le plasma de patients hypertendus [100]. Curieusement, l’un de ces microRNA (hcmv-miR-UL112) est codé par le cytomégalovirus humain, ce qui suggère un nouveau lien entre l’infection par ce virus et l’hypertension artérielle essentielle. Néanmoins, l’abondance de ce microRNA est très variable chez les patients, et donc son intérêt comme biomarqueur de l’hypertension artérielle reste à confirmer.
5.3 Diabète sucré
Le diabète est une maladie dont l’incidence ne cesse d’augmenter. Ainsi, 382 millions de patients diabétiques ont été recensés en 2013, et on en prévoit 592 millions en 2035 [101]. Actuellement, le diagnostic du diabète est essentiellement basé sur la glycémie à jeun, l’HbA1c et l’HGPO (hyperglycémie provoquée par voie orale). Chacun de ces tests possède ses limites ; les cliniciens pourraient donc, en fonction du contexte, utiliser un, deux ou trois de ces tests pour arriver à un bon diagnostic [102]. La recherche de nouveaux biomarqueurs est donc très active. De nombreux microRNA circulants sont dérégulés au cours du diabète et peuvent servir de biomarqueurs. On retrouve par exemple, chez les diabétiques de type 1, une dérégulation du miR-375 [103], miR-25 [104], miR-21, miR-126 et miR-210 [105]. On observe aussi une dérégulation de plusieurs microRNA chez les patients diabétiques de type 2, ainsi que chez les personnes obèses [106]. Les microRNA sont impliqués dans le développement du diabète et de ses complications en ciblant des gènes responsables de l’insulinorésistance (miR-103, miR-107 et miR-802) et en agissant sur des gènes qui jouent un rôle important dans l’apparition de la néphropathie diabétique (miR-93 et miR-192) [107]. Une méta-analyse récente a identifié 40 microRNA dérégulés dans le diabète de type 2 [108]. Zampetaki et al. ont analysé, durant une étude prospective, les échantillons plasmatiques d’une cohorte de 822 personnes. Parmi eux, 80 patients diabétiques de type 2 et 19 sujets normoglycémiques qui ont développé un diabète de type 2 au cours des 10 ans de l’enquête. La comparaison de l’expression des microRNA dans le plasma des patients diabétiques de type 2 par rapport à des sujets contrôles appariés sur l’âge et le sexe a conduit à l’identification de 13 microRNA pouvant servir de biomarqueurs du diabète. Cinq de ces microRNA (miR-15a, miR-28-3p, miR-126, miR-223 et miR-320) ont permis d’identifier 70 % des patients diabétiques. Les 24 patients restants étaient bien équilibrés et avaient des valeurs de glycémie à jeun et d’HbA1c comprises dans les intervalles de références. Il est également important de noter que 52 % des sujets normoglycémiques qui ont développé un diabète pendant les 10 ans du suivi étaient classés d’emblée comme diabétiques en utilisant le profil d’expression des cinq microRNA [109]. Les microRNA pourraient donc servir au dépistage précoce de sujets diabétiques de type 2 qui ne pourraient l’être par les examens biologiques classiques. Toutefois, ces résultats doivent être confirmés par d’autres études. De la même manière, trois microRNA sériques (miR-132, miR-29a et miR-222) ont été retrouvés comme prédictifs du diabète gestationnel avec une sensibilité de 66,7 % et une spécificité de 63,3 % [110].
5.4 Maladies du SNC
Environ 70 % des microRNA connus se trouvent au niveau du cerveau. Les microRNA contribuent au développement, à la différenciation et à la plasticité synaptique des cellules neuronales. La dérégulation des microRNA a été associée à de nombreuses pathologies touchant le SNC, quelles que soient leurs origines (neurodégénérative, neuro-inflammatoire, neurotraumatique…), comme la maladie d’Alzheimer, la maladie de Huntington, la sclérose latérale amyotrophique, la sclérose en plaque, la schizophrénie et les troubles bipolaires. Par exemple, l’augmentation de la concentration de miR-29a dans le LCR permet de différencier les patients atteints de la maladie d’Alzheimer des sujets contrôles [111], et l’augmentation dans le sang de miR-132 au cours de cette maladie a été décrite dans trois études indépendantes [112]. Cependant, il serait intéressant de vérifier par d’autres études que cette différence d’expression des microRNA circulants est bien le reflet de la maladie et non pas de la différence d’âge entre les patients étudiés et les sujets témoins.
5.5 Pathologies hépatiques
Le foie est un organe central du métabolisme qui est la cible de nombreuses agressions par les virus, l’alcool, les médicaments et autres xénobiotiques. Le microRNA miR-122 est le plus abondant du foie, où il représente plus de la moitié des microRNA exprimés au niveau de cet organe ; en outre, le foie est quasiment le seul organe exprimant miR-122. Ce microRNA est impliqué dans de nombreuses fonctions physiologiques et pathologiques, comme la régulation du métabolisme lipidique et la suppression de tumeurs [113]. Les essais cliniques du Miravirsen, un « antagomir » inhibiteur de miR-122, chez des patients atteints d’hépatite C (génotype 1) ont montré une diminution dose dépendante du RNA viral, ce qui confirme le rôle de miR-122 dans le développement de cette maladie [114]. Dans le cadre de la recherche de nouveaux biomarqueurs non invasifs, l’analyse par qRT-PCR des plasmas de 83 patients atteints d’hépatite B, de 15 patients ayant des maladies du muscle squelettique et de 50 sujets témoins a montré que miR-122 constitue un bon marqueur non invasif des dommages hépatiques provoqués par les virus, l’alcool et les produits chimiques [115].
5.6 Pathologies rénales
L’estimation du débit de filtration glomérulaire est essentielle à la recherche et à la classification de l’insuffisance rénale, ainsi qu’à l’ajustement de la posologie des médicaments à élimination rénale. Actuellement, il n’existe pas de marqueur idéal de la fonction rénale et les formules de calcul de la clairance de la créatinine présentent de nombreuses limites [116]. Plusieurs microRNA sont exprimés au niveau des reins et contrôlent la prolifération, la migration et la structure des cellules rénales. Des modifications dans certains de ces microRNA sont associées à des maladies rénales. Par exemple, la diminution de miR-30 est accompagnée d’une diminution du nombre de cellules rénales, d’une perturbation de la pression sanguine, du développement de lésions vasculaires et d’une fibrose extensive [117]. Le dosage des microRNA dans les sédiments urinaires de 56 patients atteints d’insuffisance rénale chronique ayant subi une biopsie rénale montre que l’expression de miR-21 et miR-216a est corrélée au déclin de la fonction rénale et au risque de progression vers une insuffisance rénale dépendante de la dialyse [118]. Le profil d’expression des microRNA dans les urines pourrait donc constituer un marqueur non invasif de la fonction rénale. Toutefois, les études publiées portant uniquement sur de faibles séries [119], des études plus larges sont donc nécessaires pour confirmer et éventuellement valider cette application. Le dosage des microRNA dans les urines pourrait également s’avérer utile pour le diagnostic et/ou le pronostic d’autres pathologies touchant le rein, comme le syndrome néphrotique ou la fibrose à IgA [120,121]. En outre, dans une étude récente, le dosage au niveau sanguin d’un panel de cinq microRNA a été validé sur 161 patients pour prédire le rejet à médiation cellulaire T après une transplantation rénale [122]. Enfin, l’analyse spécifique des microRNA vésiculaires dans les urines, notamment dans les exosomes, constitue vraisemblablement une perspective d’un grand intérêt en matière de biomarqueurs dans le domaine de la néphrologie.
6 Conclusion
La stabilité et l’accessibilité des microRNA dans les liquides biologiques en font de nouveaux biomarqueurs non invasifs particulièrement intéressants. Le champ d’application des microRNA circulants couvrirait non seulement les maladies cancéreuses, mais pourrait aussi s’étendre à de nombreuses autres pathologies. Toutefois, il est indispensable, pour minimiser la variabilité des résultats, de standardiser les procédures de traitement des échantillons, les méthodes de détection et surtout les stratégies de normalisation. En outre, les microRNA n’étant pas communément spécifiques d’une cellule, il est donc difficile de déterminer la contribution de chaque cellule, tissu ou organe au pool des microRNA mesurés dans le sang ou dans d’autres liquides biologiques. Mais la combinaison de plusieurs microRNA au travers de « signatures » constitue une alternative au manque de spécificité d’un microRNA pris isolément. L’autre percée que l’on entrevoit désormais concerne l’exploration des microRNA dans les vésicules circulantes, telles que les exosomes, qui grâce aux diverses méthodes spécifiques d’isolement actuellement disponibles, permettent d’envisager l’étude des biomarqueurs en fonction de leur cellule d’origine [123].
Déclaration de liens d’intérêts
Les auteurs déclarent ne pas avoir de liens d’intérêts.


