1 Introduction
Une épidémie de dengue sévit actuellement sur l’île de la Réunion, dans l’océan Indien. Plus de 5 000 cas ont été confirmés biologiquement entre janvier et juin 2018, et plus de 16 000 cas évocateurs de la dengue ont été signalés (Figure 1). Fin juin, du fait de l’arrivée de l’hiver austral, mais aussi de la lutte antivectorielle, l’épidémie recule. Cependant, il est difficile de prévoir si le nombre d’infections diminuera suffisamment pour empêcher une seconde vague épidémique à la fin 2018, lorsque les conditions climatiques seront plus favorables aux moustiques vecteurs de la maladie. C’est cette deuxième vague qui, en 2006, avait infecté presqu’un tiers de la population avec le chikungunya.
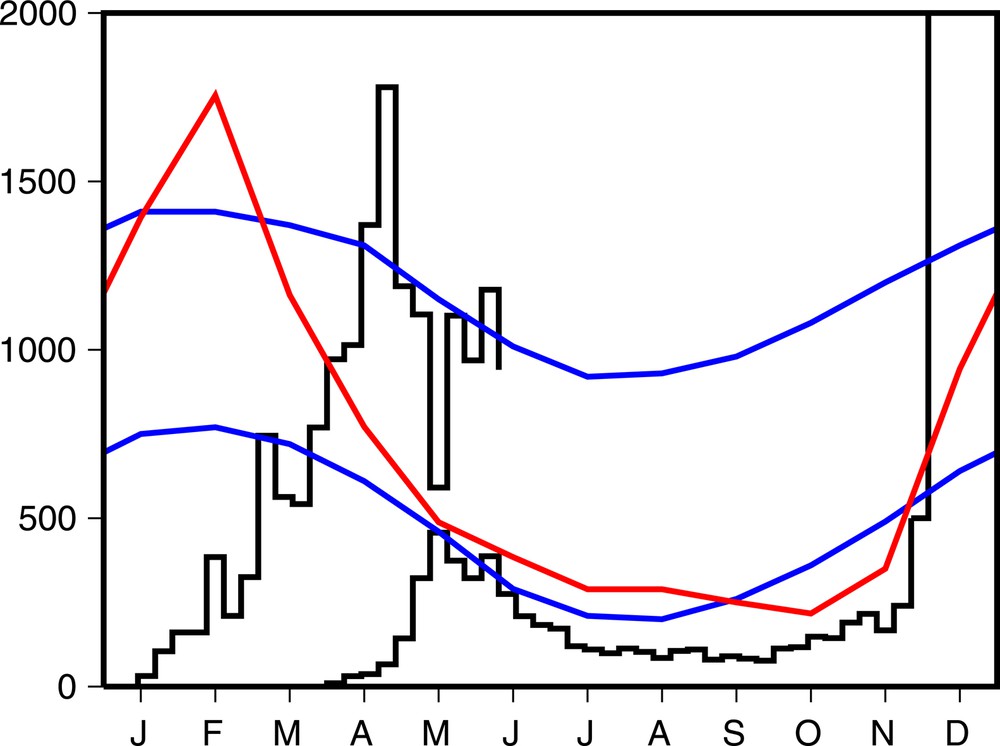
Courbe en noir du haut (échelle sur l’axe vertical de gauche) : estimation du nombre de cas hebdomadaires évocateurs de la dengue à la Réunion entre janvier et juin 2018, d’après [1]. Courbe en noir du bas (même échelle) : cas hebdomadaires de chikungunya en 2005, avant le grand pic de la fin 2005 et du début de 2006 [2]. Données climatiques à la station de l’aéroport de la Réunion, d’après [3] : en bleu, température minimale (de 18,0 à 23,7oC) et température maximale (de 25,2 à 30,1oC) ; en rouge, précipitations mensuelles (de 43 à 351 mm).
Il serait tentant de modéliser mathématiquement la propagation de la dengue de manière réaliste tout en limitant la complexité du modèle pour n’avoir que quelques paramètres inconnus, comme on a essayé de le faire pour le chikungunya dans [2]. Mais les incertitudes qui pèsent sur ces paramètres et sur leur dépendance par rapport aux variables climatiques sont si grandes que les résultats numériques risquent fort d’être assez douteux [4]. On se contentera ci-dessous d’un modèle épidémique extrêmement simplifié à transmission directe et non vectorielle, qui ne prétend donc pas pouvoir être appliqué au cas de la dengue à la Réunion. Cela n’aurait pas vraiment de sens de vouloir ajuster ses paramètres aux données épidémiques. L’objectif est plutôt d’attirer l’attention sur la question de la taille finale d’une épidémie dans un environnement saisonnier périodique, qui a été peu étudiée d’un point de vue théorique.
Même les modèles mathématiques les plus simples qui tiennent compte de la saisonnalité présentent de nombreuses difficultés. On sait depuis longtemps que pour les modèles de maladies endémiques, un coefficient périodique peut engendrer des oscillations avec une période différente, voire des oscillations chaotiques [5]. Dans l’étude [2], inspirée de l’épidémie de chikungunya à la Réunion, on a vu par ailleurs que même la notion classique de reproductivité (notée R0 à la suite de Lotka [6]) dans les modèles périodiques devait être définie avec quelques précautions pour que l’inégalité R0 > 1 traduise bien l’instabilité de la situation sans épidémie ; la difficulté apparaît notamment pour les modèles avec au moins deux compartiments infectés.
Dans [7], on a abordé le modèle S–I–R périodique, simple généralisation du modèle classique de Kermack et McKendrick [8, p. 75] :
| (1) |
En combinant méthodes numériques et méthodes analytiques, on étudie ici plus attentivement la taille finale de l’épidémie dans le cas particulier où
Si ϕ(t) = cos(ωt), si le nombre de personnes infectées au départ est petit devant la taille de la population (i ≪ N) ; si R0 = a0/b > 1 avec R0 proche de 1 et si i/N ≪ (R0 − 1)2, alors on détermine analytiquement le coefficient correcteur c en fonction des paramètres N, i, a0, b, t0 et ω du modèle. On trouve que l’environnement périodique augmente la taille finale de l’épidémie (c > 0) si le taux de contact infectieux vérifie a(t0 + τ) > a0, où t0 + τ est le moment où l’épidémie atteindrait son pic dans un environnement constant.
2 Formule exacte pour le coefficient correcteur
Rappelons tout d’abord qu’à partir du système différentiel (1), on montre que: Sɛ(t) + Iɛ(t) + Rɛ(t) = N pour tout t ≥ t0 ; Sɛ(t) > 0, Iɛ(t) > 0 et Rɛ(t) ≥ 0 pour tout t ≥ t0 ; Sɛ(t) est une fonction décroissante qui tend vers une limite Sɛ(∞) ; Rɛ(t) est une fonction croissante qui tend vers une limite Rɛ(∞) ; Iɛ(t) tend vers une limite qui ne peut être que 0 [7]. Intégrons la troisième équation différentielle de t0 à l’infini : cela donne . Par ailleurs, la première équation différentielle s’écrit . Intégrons de même pour obtenir
| (2) |
| (3) |
On peut évaluer numériquement cette intégrale. On utilise d’abord un logiciel de calcul numérique (tel que Scilab et sa fonction << ode >>, voir www.scilab.org/fr) pour résoudre le système différentiel (1) avec ɛ = 0). On obtient ainsi (S0(t), I0(t), R0(t)) pour un ensemble discret de valeurs de t, avec un petit pas de temps. On en déduit en particulier la valeur R0(∞). Puis on calcule l’intégrale avec cette même discrétisation du temps. On en déduit la valeur du coefficient c.
Prenons un exemple. Supposons que ϕ(t) = cos(ωt) avec ω = 2π/T, N = 10 000, i = 1, T = 12 mois, a0 = 10/mois et b = 5/mois. Alors la reproductivité est R0 = 2 ; la taille finale de l’épidémie dans un environnement constant est R0(∞) ≃7 968. La Figure 2 montre comment Rɛ(∞) varie en fonction de ɛ (0 ≤ ɛ ≤ 1) lorsque t0 prend trois valeurs différentes, qui correspondent à trois moments différents d’introduction du premier cas infecté dans la population : 0,5 mois, 2 mois ou 3 mois. La figure montre aussi l’approximation R0(∞) + N c ɛ pour ɛ petit, avec le coefficient correcteur c calculé selon la formule (3). On observe que le coefficient c peut être positif ou négatif, de sorte que la taille finale de l’épidémie peut être plus grande ou plus petite que dans un environnement constant. On observe aussi que pour t0 = 3 mois, la fonction Rɛ(∞) varie en fonction de ɛ de manière plus compliquée que pour les deux autres valeurs de t0 : en particulier, cette fonction n’est décroissante que tant que ɛ < 0,3.
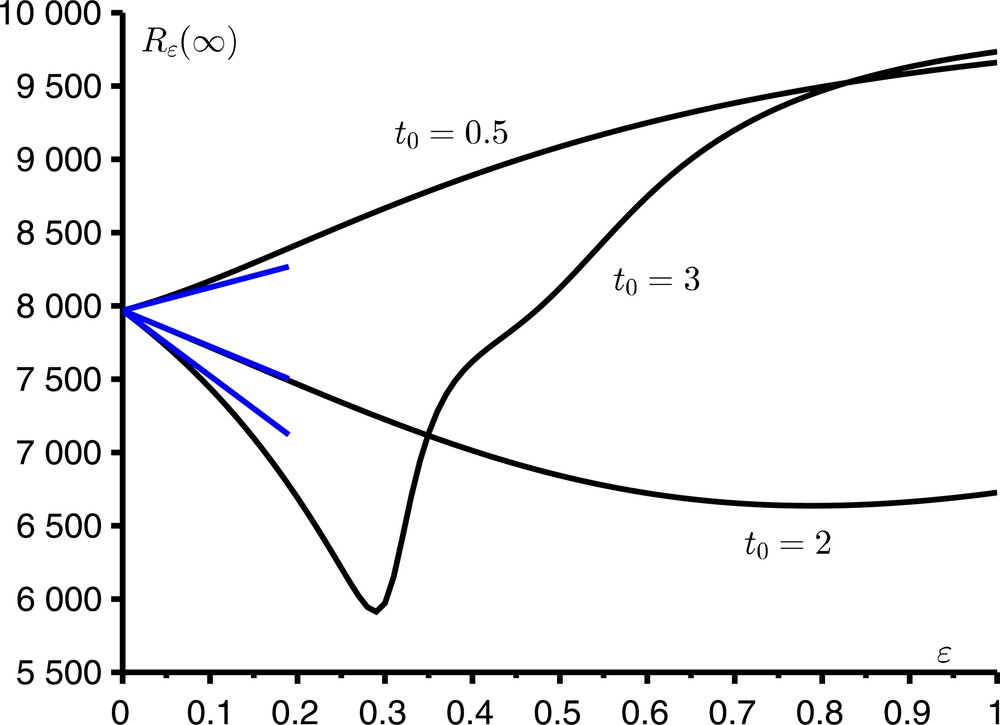
La taille finale de l’épidémie en fonction de ɛ pour trois valeurs différentes de t0. En bleu : l’approximation R0(∞) + N c ɛ.
3 Formules approchées
Kermack et McKendrick ont trouvé (voir par exemple [11, §2] ou [12, p. 235]) une approximation pour I0(t) à partir de l’équation exacte dR0/dt = b(N − s e−a0R0/(Nb) − R0) vérifiée par R0(t). Un développement limité de l’exponentielle e−x = 1 − x + x2/2 + o(x2) tronqué à l’ordre 2 conduit à une équation de Ricatti résoluble pour R0(t) et à une courbe en cloche symétrique pour I0(t),
| (4) |
Supposons plus précisément que R0 = a0/b → 1 avec R0 > 1 et que (i/N)/(R0 − 1)2 → 0 ; autrement dit, R0 est proche de 1 et i/N ≪ (R0 − 1)2. Alors i → 0 et on a bien R0(∞) → 0 d’après l’équation (2) avec ɛ = 0. Comme on a aussi i/N ≪ (R0 − 1), on trouve que
Cette dernière intégrale se calcule explicitement lorsque ϕ(t) = cos(ωt). En effet,
| (5) |
Comme exemple, prenons les mêmes valeurs des paramètres que dans la Figure 2, sauf que a0 = 6/mois pour avoir R0 = 1,2 plus proche de 1 ; [12, p. 240] indique d’ailleurs que l’approximation en cloche symétrique de Kermack et McKendrick n’est satisfaisante que pour R0 < 1,5. La Figure 3 compare l’expression exacte (3) du coefficient correcteur c avec les approximations (4) et (5). Avec nos valeurs numériques, ces deux dernières approximations sont indiscernables. Elles sont d’autant plus proches de la valeur exacte que R0 est proche de 1. Notons qu’ici i/N = 10−4 ≪ (R0 − 1)2 = 0,04. La figure s’interprète comme ceci : si l’épidémie démarrait à t0 = 0, les valeurs des paramètres conduiraient à un pic de l’épidémie environ τ ≃ 6,3 mois plus tard dans un environnement constant ; or, l’environnement périodique sera défavorable à ce moment (on sera dans le creux du facteur cos ωt) ; donc, la taille finale de l’épidémie sera moindre et c < 0.
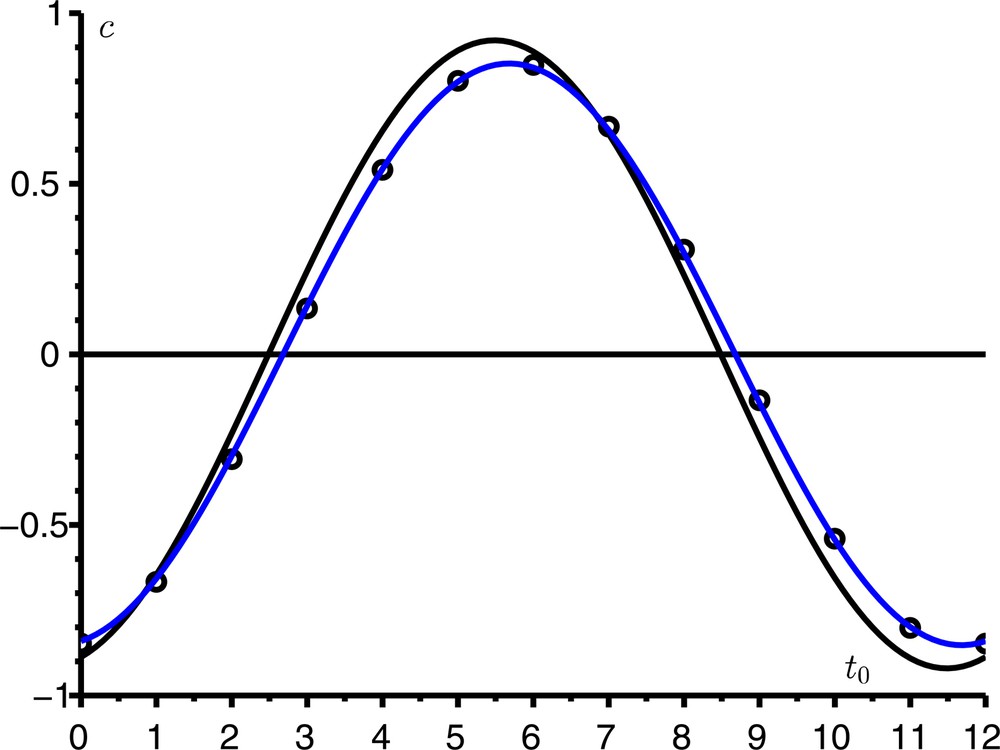
Le coefficient correcteur c pour la taille finale de l’épidémie en fonction de t0, l’instant du début de l’épidémie. Comparaison de l’expression exacte (3) [en noir] avec les approximations (4) [points] et (5) [en bleu].
4 Remarque
On peut adapter la formule exacte (3) pour le coefficient correcteur c à des modèles plus complexes. Supposons par exemple que l’épidémie se propage entre deux populations (comme des hommes et des vecteurs) suivant le schéma S–I–R, avec N1 = S1(t) + I1(t) + R1(t) et N2 = S2(t) + I2(t) + R2(t) :
5 Conclusion
On a déterminé de manière analytique comment la taille finale de l’épidémie était influencée par un taux de contact infectieux périodique de faible amplitude, lorsque la reproductivité R0 reste proche de 1. Il s’agit d’un petit progrès par rapport aux études précédentes, qui étaient soient qualitatives comme dans [7], soit purement numériques comme dans [10, figure 1]. Les hypothèses sont néanmoins assez restrictives. Elles évitent en particulier les cas où plusieurs pics épidémiques se produisent, puisque l’on reste proche de la situation à un seul pic des environnements constants. Or ce sont justement ces cas qui risquent de se présenter avec l’épidémie de dengue à la Réunion.


