1. Introduction
Transcription of protein-coding genes begins with the formation of a pre-initiation complex (PIC) composed of RNA polymerase II and several general transcription factors (TFs) [1]. PIC assembly is nucleated by the recruitment of TATA-binding protein (TBP) to promoter DNA, a focal point for regulated gene expression [2].
Transcription co-activator complexes are required for activated transcription in a chromatin environment. Two multi-protein co-activator complexes, TFIID and SAGA, are globally required for gene expression in yeast [3, 4] and can deliver TBP to the gene promoter [5]. The relative contribution of each of these two complexes to PIC formation at a particular gene, is dictated by gene-specific features including promoter sequence, histone modifications and transcription activator binding sites [6]. TFIID and SAGA have a homologous histone octamer like module, which was reported to be responsible for TBP delivery. They share five TBP Associated Factors, Taf5 and four Tafs containing a histone fold (HF) motifs (Taf6, 9, 10, and 12). SAGA also harbors three additional subunits in this module containing one (Ada1 and Spt7) or two (Spt3) HF domains. Important progress was reported in determining the architecture of TFIID, but the machinery that binds and delivers TBP could not be depicted in detail [7, 8].
The 1.6 MDa Spt-Ada-Gcn5 acetyltransferase (SAGA) complex is composed of 19 subunits organized in four modules with distinct functions: a histone acetyl transferase (HAT) module, a histone deubiquitinase (DUB) module, the 430 KDa Tra1 subunit which serves as a docking platform for sequence-specific activators, and a central module physically connected to all other modules and responsible for binding and delivery of the TATA-box binding protein (TBP) [9]. The functional diversity of SAGA allows it, in addition to PIC formation, to modulate transcription at different steps [10, 11] and to play an important role in DNA repair signaling [12].
Atomic structures of several individual isolated components of SAGA were obtained by X-ray crystallography. Cryo-EM studies positioned Tra1 in a map of holo-SAGA but all other subunit appeared together in one main lobe that remained poorly resolved [13].
2. High resolution structure of SAGA
The dynamic nature of SAGA hampered efforts to resolve its structure at high resolution, hindering attempts to place atomic models of subunits or domains within the main lobe. We posited that by improving our purification scheme, as well as by introducing SAGA natural ligands, the conformation of SAGA may be stabilized. These advances combined with local structure refinement resulted in a structure at a resolution which allowed us to position and model the majority of SAGA subunits [14].
At the periphery of SAGA, we identified the distinctive saddle shaped density of the conserved core of TBP. At the opposite pole of the main lobe, the DUB crystal structure fits into and occupies fully a large but not well resolved density that is nearly completely detached from and independent of SAGA. The second enzymatic module of SAGA which harbors the HAT activity adopts multiple conformations and orientations. Accordingly, the bulk of this module is visible only as an elongated featureless density at the tip of our map. Nevertheless, we could identify its docking site on SAGA (Figure 1).
Structure of the SAGA complex. (A) Composite structure of the Pichia pastoris SAGA complex. Blue: main lobe; tan: Tra1 lobe. SAGA core and Tra1 lobe are at 3.5 Å resolution, while the enzymatic modules are depicted as low resolution densities. (B) Fitted model where the subunits are labelled.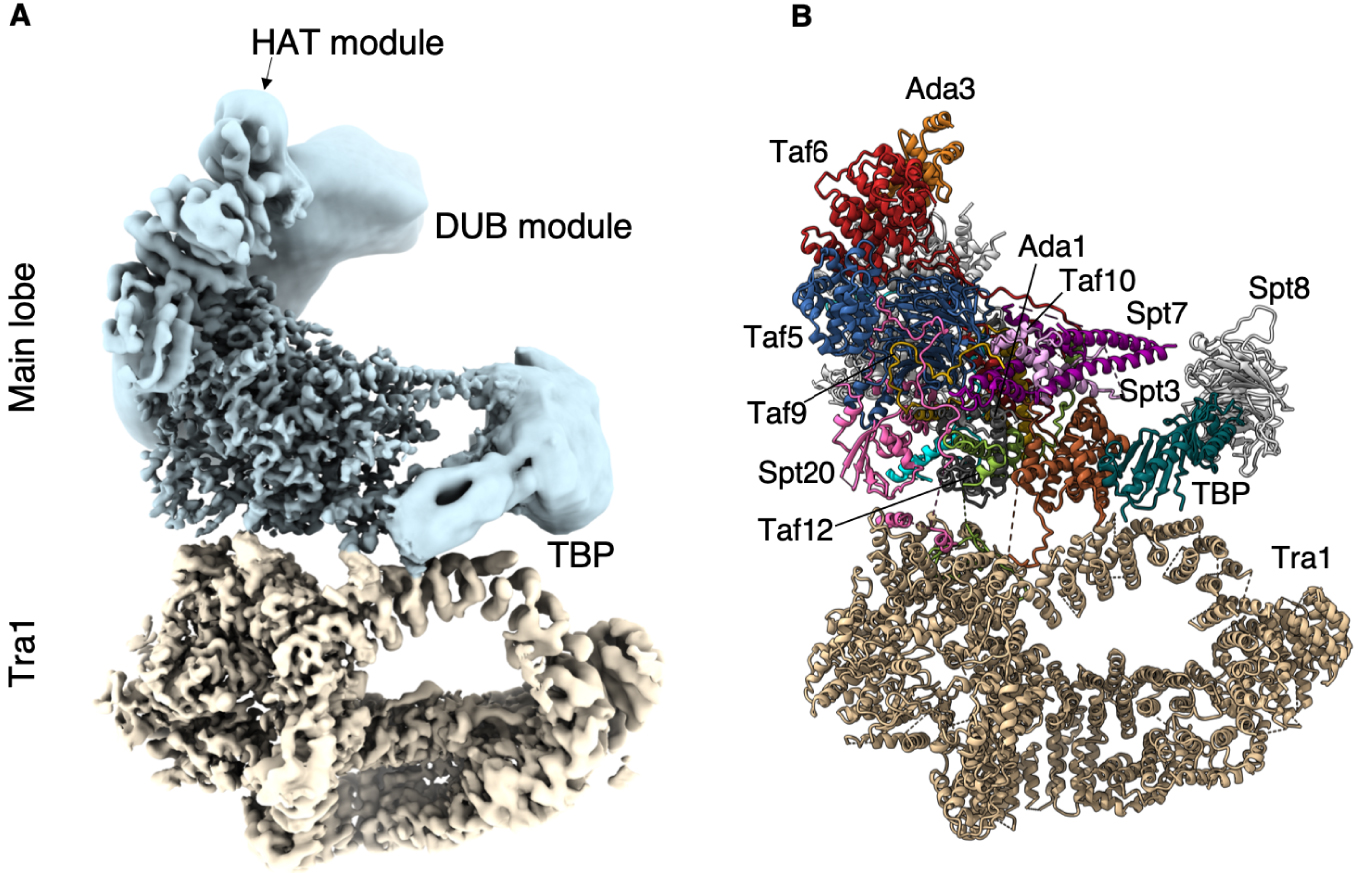
3. The central role of TAF5
The main lobe is dominated by the WD40 7-blade propeller of Taf5. This domain occupies a central position, serving as a hub connecting major SAGA functions. The flat top face of the WD40 propeller clamps together the Taf5 N-terminal domain (NTD) and the HEAT repeat domain of the TAF6 subunit, which holds the HAT enzymatic module. The opposite WD40 bottom face holds the histone-fold machinery that ultimately binds TBP, but also connects to the DUB module and to Tra1.
WD40 domains are recognized as potential hubs that mediate protein-protein interactions. The Taf5 WD40 in SAGA is a staggering manifestation of this capacity as it associates with at least 11 different protein domains representing 9 different subunits. Hence Taf5 is a centrally located master hub that orchestrates the architecture of the entire main lobe. It is tempting to speculate that such a hub serves to coordinate activities that reside at different extremities of the complex.
4. A histone octamer like structure in SAGA
Our structure offers the first atomic model of the full HF octamer that resides at the core of SAGA, and most probably, with minor variations, in the TBP-binding lobe of TFIID. It reveals a striking disparity between histone interactions as compared to the nucleosome, particularly for the incorporation of the TBP binding subunit Spt3.
In the nucleosome the interaction between neighboring histone pairs is mediated by a tightly packed four-helix bundle [15]. The orientation of SAGA HF pairs is gradually deviating from the nucleosomal arrangement resulting in a 30 degrees tilt of the last, Spt3 HF pair compared to its analogous nucleosomal histone pair. To secure incorporation of Spt3 into SAGA the C-terminal tail of Spt3 inserts into the center of the spiral disc.
We propose that this assembly of histone folds, deviating considerably from the symmetric arrangement in the nucleosome, is geared specifically towards a delicate balance between a rigid Spt3 that is firmly embedded into the octamer and an overly flexible Spt3 that associates with SAGA through loops alone (Figure 2).
Comparison of SAGA TBP delivery machinery with the histone octamer. (A) Histone octamer consisting of two heterodimers of H2A-H2B and H3-H4. (B) Taf5 WD40 domain coordinates the Taf histone-fold containing subunits to forma an octamer-like structure holding TBP at the periphery of the SAGA complex.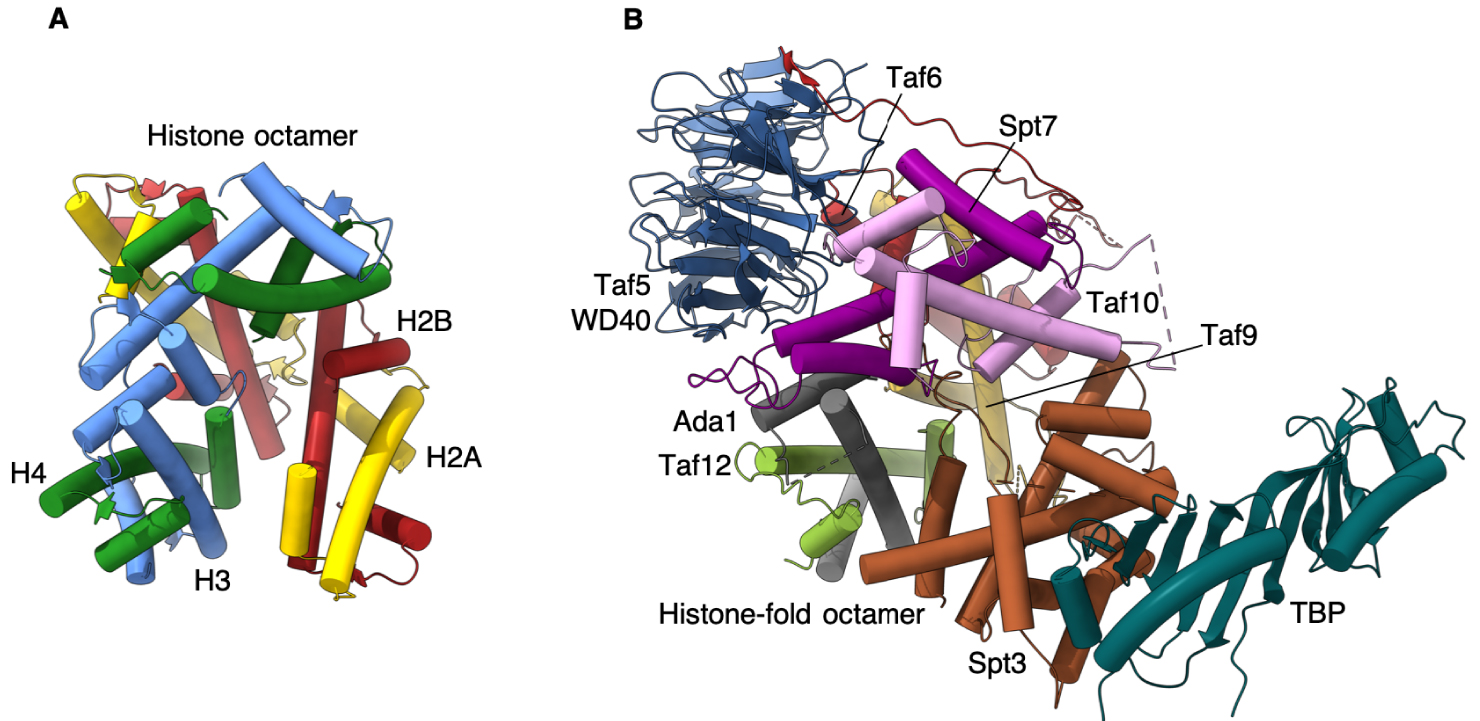
5. TBP binding machinery
TBP binds SAGA at the extremity of the main lobe orienting its DNA-binding cleft towards SAGA’s main body. The principle subunit in coupling TBP to SAGA and determining its position is Spt3. Another SAGA subunit, Spt8, was also reported to interact with TBP. In contrast to Spt3, we find that Spt8 is not part of the SAGA core. It is flexibly tethered via a fuzzy density to the outward tips of two long nearly parallel helices, extensions of each end of the Spt7 histone fold that reach 50 Å away from the main body of SAGA. The flexible tethering of Spt8 to SAGA core contributes to small movements of TBP, which might be important to transiently allow initial access to DNA or regulatory factors.
The space between TBP and the main lobe of SAGA is just wide enough to accommodate a double-stranded DNA bent by TBP. However, the path of DNA is obstructed by structural elements located distally to the TBP DNA binding cleft. Gel-Shift assays of TATA-DNA binding to SAGA-TBP under physiological conditions demonstrate that this mild steric hindrance is none-the-less sufficient to impede binding to TBP of even high-affinity DNA.
Our structure suggests that the TFIIA-stimulated mechanism for the concerted DNA binding and TBP release from SAGA includes the following steps: TFIIA first displaces Spt8 from TBP; it then promotes an initial binding of DNA to TBP. TBP could then tilt, letting DNA pass in the gap between Tra1 and SAGA main body, completing the binding to DNA.
6. Flexible enzymatic modules
The DUB and HAT modules are highly dynamic, permitting the exploration of a large conformational space in search of their chromatin substrates and the structure shows how these modules maintain robust binding to SAGA.
SAGA DUB module consists of the catalytic subunit Ubp8 as well as Sgf11, Sus1, and the Sgf73 N-terminal end. Two helices in the C-terminal part of Sgf73 are embedded within SAGA core. The DUB module is tethered to SAGA’s core through the 140aa linker connecting the anchoring domain to the N-terminal end of Sgf73 which is part of the DUB. We find little trace of this linker in the structure except for a short stretch contacting Spt20 and Taf5.
In contrast to the DUB, no parts of the HAT module are embedded within SAGA core. The HAT densities emerge, harboring two distinct helical domains, at the surface of the Taf6 HEAT repeats.
7. Intricate interaction network
Three distinct bridges serve to physically and possibly functionally couple Tra1 with the main lobe. The first bridge is established by a highly dispersed and extended domain of Spt20 touching Taf5, Taf9, Taf12, Ada1 and Spt7 as it traverses the surface of SAGA core on its way to Tra1. An exceptionally long loop that precedes the histone fold of Taf12 is responsible for establishing the second bridge. This loop forms a lasso shaped thread at the surface of the Tra1. The third bridge is formed by the loop connecting the second and third helices of cSpt3-HF as its infiltrates between two helices of Tra1.
8. Perspectives
Our work provides the architecture of the multi-subunit yeast SAGA complex and sheds light into its role in eukaryotic transcription initiation. The structure paves the way towards better understanding of how the TFIID and SAGA complexes, designed to deliver TBP, may share workload on different gene promoters. TBP is required for each transcription event in eukaryotes and interacts with a large number of partners with overlapping binding interfaces. Our results illustrate how TBP is hold with SAGA thus enlarging the regulatory repertoire controlling transcription initiation.
French version
1. Introduction
La transcription des gènes codant pour les protéines débute par la formation d’un complexe de pré-initiation (PIC) composé de l’ARN polymérase II et de plusieurs facteurs de transcription (TF) généraux [1]. L’assemblage du PIC est nucléé par le recrutement de la protéine de liaison à la boîte TATA (TBP) à l’ADN promoteur, un point focal pour la régulation de l’expression des gènes [2].
Des complexes co-activateurs de transcription sont nécessaires pour la transcription modulée par des activateurs de transcription dans un environnement chromatinien. Deux complexes multi protéiques de co-activation, TFIID et SAGA, sont requis pour l’expression génique dans la levure [3, 4] et peuvent déposer TBP sur le promoteur de la séquence codante du gène [5]. La contribution relative de chaque complexe à la formation de PIC est dictée par les caractéristiques spécifiques du gène, notamment la séquence du promoteur, les modifications post-traductionnelles des histones et les sites de liaison des activateurs de transcription [6]. TFIID et SAGA comportent tous deux un module protéique homologue semblable à l’octamère d’histone, qui serait responsable de l’interaction avec TBP. Ils partagent cinq facteurs associés à TBP (TAF), Taf5 et quatre Tafs supplémentaires contenant un motif de repliement protéique commun aux histones (HF) (Taf6, 9, 10 et 12). SAGA abrite dans ce module trois sous-unités supplémentaires contenant un (Ada1 et Spt7) ou deux (Spt3) domaines HF. Des progrès importants ont été réalisés dans la détermination de l’architecture de TFIID, mais les détails moléculaires de liaison à TBP ainsi que les mécanismes de libération de TBP n’ont pas été décrits à l’échelle atomique [7, 8].
Le complexe Spt-Ada-Gcn5 acétyltransférase (SAGA) est composé de 19 sous-unités organisées en quatre modules aux fonctions distinctes : un module d’histone acétyltransférase (HAT), un module d’histone désubiquitinase (DUB), la sous-unité Tra1 d’une masse moléculaire de 430 KDa qui sert de plate-forme d’interaction pour les activateurs transcriptionnels, et un module central physiquement connecté aux autres modules et responsable de la liaison et de la délivrance de la protéine TBP [9]. Au-delà de la formation du PIC, la diversité fonctionnelle de SAGA permet, de moduler la transcription à différentes étapes [10, 11] mais également de jouer un rôle important dans la signalisation de la réparation de l’ADN [12].
Les structures atomiques de plusieurs composants individuels de SAGA ont été obtenues par cristallographie aux rayons X. Les études par cryo-microscopie électronique (cryo-EM) de molécules uniques ont récemment positionné et déterminé un modèle atomique la sous-unité Tra1 dans une structure tri-dimensionnelle de SAGA mais toutes les autres sous-unités, regroupées dans le lobe principal, sont restées mal résolues [13].
2. Structure haute résolution de SAGA
La nature dynamique de SAGA a entravé les efforts visant à résoudre sa structure à haute résolution, restreignant les tentatives de placer des modèles atomiques de sous-unités ou de domaines dans le lobe principal. Nous avons stabilisé la conformation de SAGA en améliorant notre schéma de purification, ainsi qu’en introduisant des ligands naturels de SAGA tel que TBP. Ces progrès, combinés à un affinement local de la structure, ont permis d’obtenir une structure à une résolution qui nous a permis de positionner et de modéliser la majorité des sous-unités de SAGA [14].
À la périphérie de SAGA, nous avons identifié la densité caractéristique en forme de selle de la partie conservée de TBP. Au pôle opposé du lobe principal, la structure cristalline du DUB s’intègre et occupe pleinement une densité importante mais mal résolue qui est presque complètement détachée et indépendante de SAGA. Le deuxième module enzymatique de SAGA qui abrite l’activité HAT, adopte de multiples conformations et orientations. En conséquence, la majeure partie de ce module n’est visible que sous la forme d’une densité allongée et sans caractéristiques à l’extrémité de la structure. Néanmoins, nous avons pu identifier son site d’ancrage sur SAGA (Figure 1).
Structure du complexe SAGA. (A) Structure composite du complexe SAGA de la levure Pichia pastoris. Bleu : lobe principal ; brun : Lobe Tra1. Le cœur de SAGA et le lobe Tra1 ont une résolution de 3,5 Å, tandis que les modules enzymatiques flexibles sont représentés sous forme de densités à plus faible résolution. (B) Modèle atomique de SAGA où les sous-unités sont assignées.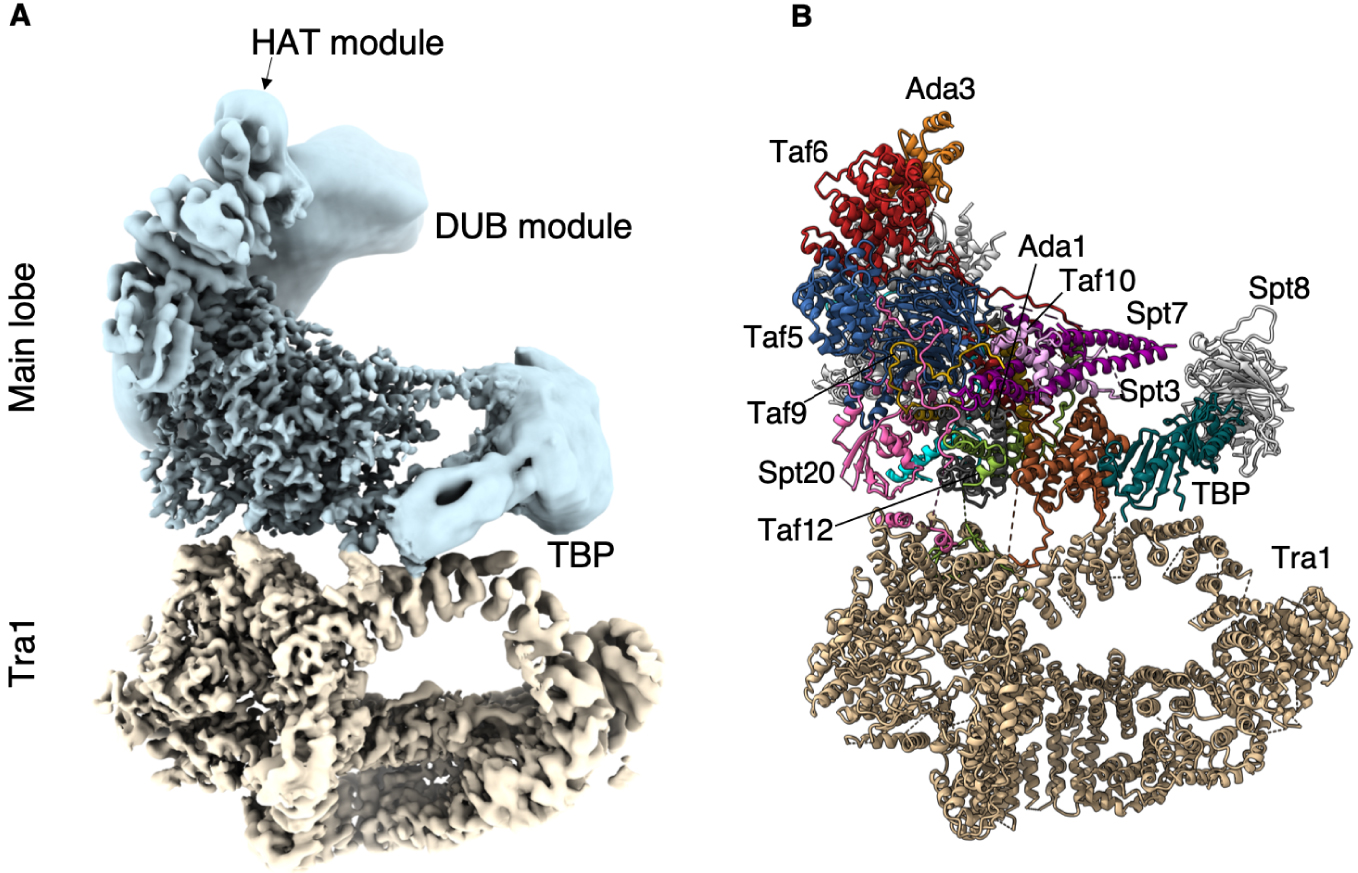
3. Le rôle central du TAF5
L’organisation du lobe principal est dominée par l’hélice WD40 à 7 pales de la sous-unité Taf5. Ce domaine occupe une position centrale et sert de charnière reliant les principales fonctions de SAGA. La face supérieure plate de l’hélice WD40 associe le domaine N-terminal (NTD) de Taf5 et le domaine répété HEAT de la sous-unité Taf6, qui interagit avec le module enzymatique HAT. La face opposée de l’hélice WD40 contient la machinerie de repliement d’histones qui non seulement s’associe à TBP, mais se connecte également au module DUB et à la sous-unité Tra1.
Les domaines WD40 sont reconnus comme des plateformes d’interactions protéine-protéine, et le domaine WD40 de Taf5 dans SAGA est une manifestation stupéfiante de cette capacité car il s’associe à au moins 11 domaines protéiques différents représentant 9 sous-unités distinctes. Ainsi, Taf5 est une plaque tournante centrale qui orchestre l’architecture de l’ensemble du lobe principal. Il est tentant de supposer qu’un tel centre permet de coordonner les activités qui se déploient aux différentes extrémités du complexe.
4. Une structure de type octamère d’histone dans SAGA
Notre structure offre le premier modèle atomique de l’octamère de domaines de type histone qui réside au cœur du SAGA, et très probablement, avec des variations mineures, dans le module de liaison à TBP présent dans TFIID. Elle révèle une disparité frappante avec les interactions des histones du nucléosome, en particulier pour l’incorporation de la sous-unité Spt3 qui interagit directement avec TBP.
Dans le nucléosome, l’interaction entre les paires d’histones voisines est médiée par un groupe de quatre hélices très serrées [15]. L’orientation des paires d’HF SAGA s’écarte progressivement de l’arrangement nucléosomal, ce qui entraîne une inclinaison de 30 degrés de la dernière paire d’HF Spt3 par rapport à la paire d’histones nucléosomales homologues. Pour consolider l’incorporation de Spt3 dans SAGA, l’extrémité C-terminale de Spt3 s’insère au centre du disque formé par la spirale des HF, un espace vide dans le nucléosome.
Nous proposons que cet assemblage de repliements d’histones, qui s’écarte considérablement de la disposition symétrique dans le nucléosome, soit spécifiquement orienté vers un équilibre délicat entre un Spt3 rigide qui est fermement encastré dans l’octamère et un Spt3 trop mobile qui ne s’associe à SAGA que par des boucles flexibles (Figure 2).
Comparaison du module TBP de SAGA avec l’octamère d’histone. (A) Octamère d’histone composé de deux hétérodimères de H2A-H2B et H3-H4. (B) Le domaine WD40 de Taf5 coordonne les sous-unités Taf, contenant un repliement histone, pour former une structure octamérique maintenant TBP à la périphérie du complexe SAGA.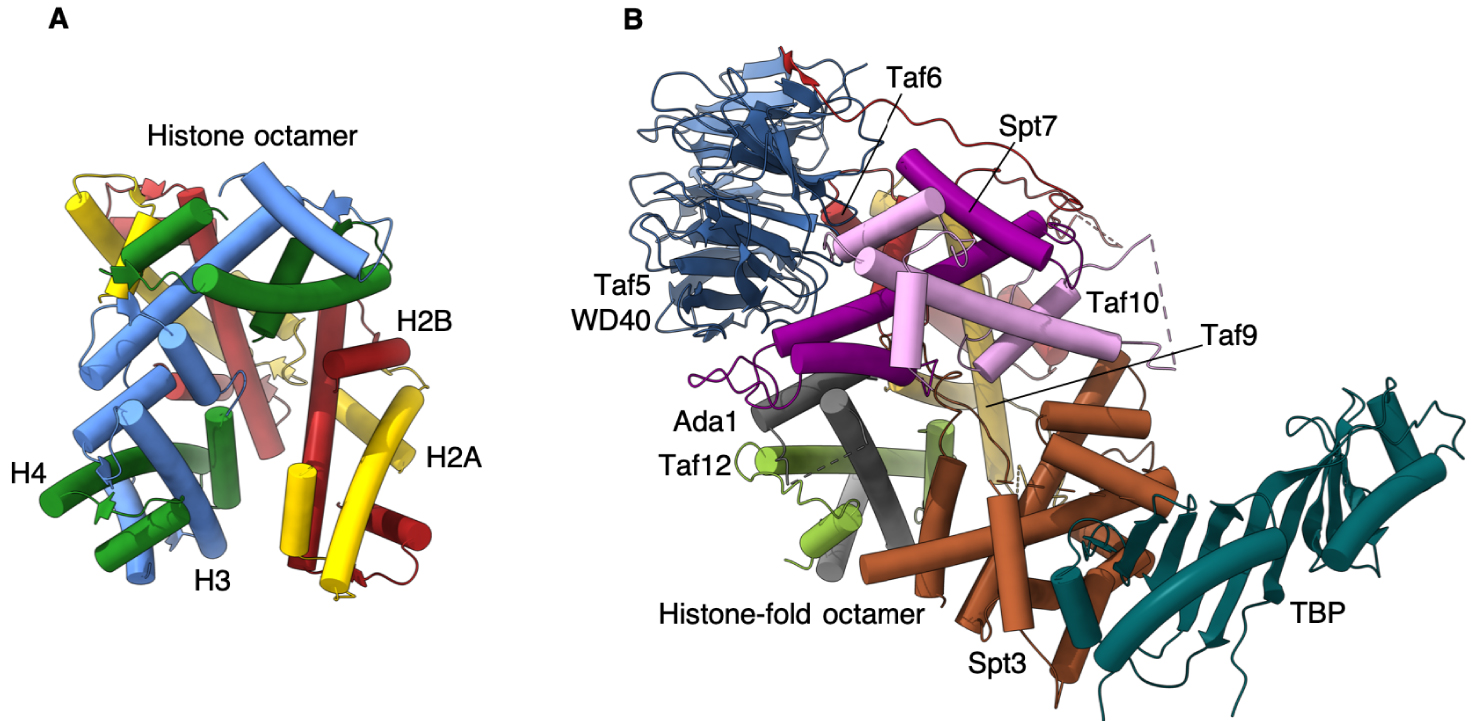
5. Mécanisme de liaison des TBP
TBP lie SAGA à l’extrémité du lobe principal, orientant son domaine de liaison à l’ADN vers le corps principal de SAGA. La principale sous-unité qui permet de coupler TBP à SAGA et de déterminer son orientation est Spt3. Une autre sous-unité de SAGA, Spt8, a également été reconnue comme interagissant avec TBP. Contrairement à Spt3, nous constatons que le Spt8 ne fait pas partie du cœur de SAGA, mais est attaché de manière flexible aux extrémités de deux longues hélices presque parallèles. Ces hélices s’étendent à chaque extrémité du domaine histone de Spt7 et tiennent Spt8 à une distance de 50 Å à l’écart du corps principal de SAGA. La fixation flexible de Spt8 au cœur de SAGA contribue aux mouvements de faible amplitude de TBP, qui peuvent jouer un rôle important pour donner une porte d’accès initiale à l’ADN ou à des facteurs de régulation.
L’espace entre TBP et le lobe principal de SAGA est suffisamment assez grand pour accueillir un ADN double brin courbé par TBP. Cependant, le chemin de l’ADN est obstrué par des éléments structuraux placés à distance du domaine de liaison à l’ADN de TBP. Les expériences de retard sur gel de l’ADN comportant une séquence TATA se liant à la SAGA-TBP dans des conditions physiologiques démontrent que ce léger obstacle stérique est suffisant pour empêcher la liaison à la TBP de l’ADN, même de haute affinité.
Notre structure suggère que le mécanisme stimulé par le facteur TFIIA pour permettre la liaison concertée de l’ADN et le détachement de TBP par SAGA comprend les étapes suivantes : TFIIA déplace d’abord Spt8 de TBP ; elle favorise ensuite une liaison initiale de l’ADN à TBP. TBP pourrait alors basculer, laissant l’ADN passer dans l’espace entre Tra1 et le corps principal de SAGA, permettant une interaction complète avec l’ADN.
6. Modules enzymatiques flexibles
Les modules DUB et HAT sont très mobiles ce qui permet l’exploration d’un grand espace conformationnel à la recherche de leurs substrats chromatiniens et la structure montre comment ces modules entretiennent une liaison robuste avec SAGA.
Le module DUB de SAGA se compose de la sous-unité catalytique Ubp8 ainsi que de Sgf11, Sus1 et de l’extrémité N-terminale Sgf73. Deux hélices de la partie C-terminale de Sgf73 sont incorporées dans le cœur de SAGA par une connexion protéique de 140 résidus reliant le domaine d’ancrage à l’extrémité N-terminale de Sgf73 qui fait partie du DUB. Nous trouvons peu de traces de ce lien dans la structure, à l’exception d’un court tronçon en contact avec Spt20 et Taf5.
Contrairement au domaine DUB, aucune partie du module HAT n’est intégrée dans le cœur de SAGA. Les densités de HAT émergent à la surface des répétitions HEAT de Taf6 sous la forme de deux domaines hélicoïdaux distincts.
7. Un réseau d’interaction complexe
Trois ponts protéiques servent à coupler physiquement, et éventuellement fonctionnellement, la sous-unité Tra1 avec le lobe principal. La première connexion est établie par un domaine très dispersé et étendu de Spt20 interagissant avec Taf5, Taf9, Taf12, Ada1 et Spt7 en traversant la surface du cœur de SAGA en direction de Tra1. Une boucle exceptionnellement longue qui précède le repliement histone de Taf12 est responsable de l’établissement du second pont. Cette boucle forme une connexion en forme de lasso à la surface de Tra1. Le troisième pont est formé par la boucle reliant les deuxième et troisième hélices du domaine histone C-terminal de Spt3 lorsque celle-ci s’infiltre entre deux hélices de Tra1.
8. Perspectives
Nos travaux ont permis de déterminer l’architecture du complexe multi-protéique SAGA de levure et mettent en lumière son rôle dans l’initiation de la transcription chez les organismes eucaryotes. La structure ouvre la voie à une meilleure compréhension de la manière dont les complexes TFIID et SAGA, conçus pour délivrer TBP, peuvent partager la charge de travail sur différents promoteurs de gènes. TBP est nécessaire pour chaque événement de transcription et interagit avec un grand nombre de partenaires avec des interfaces de liaison qui se chevauchent. Nos résultats illustrent la manière dont TBP est maintenu dans SAGA, élargissant ainsi le répertoire de mécanismes de régulation contrôlant l’initiation de la transcription.



 CC-BY 4.0
CC-BY 4.0