1. Introduction
Motility is an essential property for all living organisms. It critically depends on biological nanomachines called molecular motors. Their capacity to produce uni-directional force or movement within the crowded environment of a cell is essential for cell division, morphogenesis, intracellular trafficking, muscle contraction and numerous specialized cellular functions. More than 100 such nanomachines are encoded in the human genome. The cytoskeleton, made up of biological polymers, provides most of the mechanical and architectural properties of the cells. Molecular motors as well as numerous proteins finely tune the dynamics and organization of the cytoskeleton to control cellular processes and cell shape. Biological molecular motors are protein enzymes that produce force by using ATP as fuel and by engaging in cycles of interactions with their polarized cytoskeleton track, amplifying in the process large conformational changes essential for defining the direction of the force produced.
Three families of biological motors have expanded upon evolution: myosins work on actin filaments while kinesin and dynein motors produce force using microtubules (MTs) as tracks. Skeletal muscle myosin II, the motor that powers muscle contraction, was the first motor identified [1] and the later discovery of actin lead to the in vitro reconstitution of contraction, one of the first major advances in studies of cellular contractility [2, 3]. In 1969, Holtzer’s group described that all eukaryotic cells contain actin-based structures [4] and the role of non-muscle myosin II in cytokinesis, (last step of cell division), was uncovered in 1977 [5, 6]. Dynein was the first microtubule motor discovered in 1967 [7] and in 1985, the first kinesin was discovered as the driving force for axonal transport [8]. The astonishing wealth of biological motors really became obvious in the 90s upon the sequencing of the genomes that lead to the discovery of the myosin and kinesin superfamilies [9, 10]. All these proteins produce force via their motor domain and mechanical element, while their tail regions are diverse and play essential roles for assembly or recruitment of these motors.
Activation of these motors can lead to spatially controlled force production with distinct mechanical output, such as specific transport within cells, organization or contraction of the cytoskeletal tracks, among others. In complex cellular events such as cell division, distinct motors are at play, all working on either actin or microtubule tracks at specific times, but often in distinct regions of the cell. How post-translational modifications, partners, signals, or lipid membranes influence motor recruitment is currently under-studied. Direct binding of actin polymerization machineries and myosin motors can coordinate their specific recruitment [11]. Much remains to be learned about how motors are timely activated and the role each motor plays in a cellular process is often undefined.
In addition to their specific motor sequence, atypical features of the motor and the way partners interact with them can modulate their mechanical properties as they assemble and recruit the motors. Thus, defining their mechanical output in cells—what they actually power upon recruitment—is challenging. Structural insights and in vitro reconstitution studies defining the motility properties or possible mechanical output of these motors are precious to define how these motors operate. They can lead to testable hypotheses on the role the motors play in cells. The impact of mechanobiology for cell behavior and cell response to its external environment is clearly established. Future research direction must investigate how the mechanical output of motors contribute to cell fate in health and disease to provide mechanistic understanding of life processes as well as innovative treatments.
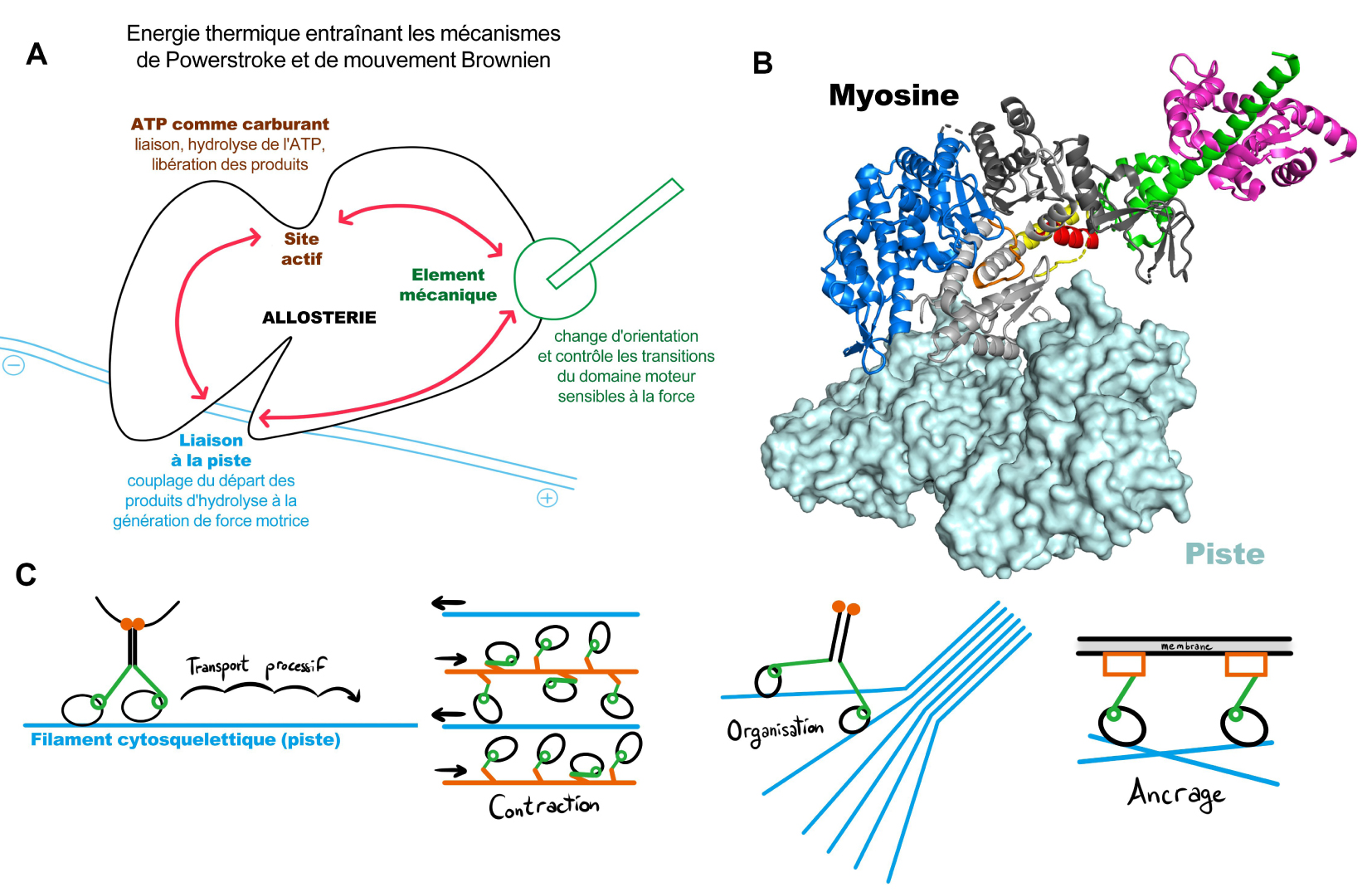
Like the chronophotographer Muybridge, when he analyzed the “horse in motion” [12, 13], the visualization of these nanomachines in different states of their motor cycle has enlighten how force is produced. Together with functional studies and molecular dynamics experiments, these structural studies have shed light on the role of allostery for force production [14, 15]. By the selection of conformations accessible to the motor along its cycle, three regions of the motor (allosteric sites) communicate: the ATP binding site, the cytoskeleton-binding site and the mechanical element responsible for directed movement along the cytoskeleton (Figure 1A,B). An event in any of these allosteric sites (i.e. ATP binding, track binding, external force applied on the mechanical element) is communicated to the other sites and this information is integrated to influence the conformations and the rate of the transitions the motor can explore along the motor cycle. Atomic resolution structures from X-ray crystallography of the unique reverse-direction myosin motor Myo6 have been particularly insightful to decipher how it undergoes its motor cycle. Since 2016, high resolution structures are also available for describing how motors bind to their track [14, 16, 17, 18], as cryo-electron microscopy (cryoEM) has undergone a “resolution revolution” [19]. In the first part of this review, the general conserved mechanisms essential for force production by myosin and kinesin motors will be summarized, as well as the current challenges that limit our current understanding.
Little is known at present on how molecular motors select their tracks in cells or can affect the cytoskeleton dynamics and organization. Interesting insights have emerged for motors that do not process on one track but instead function by reaching out to more than one track, such as the Myo10 myosin and the KIF20A kinesin. Such motors do not use the track to move or contract filaments. They actually influence the organization of the cytoskeleton with which they interact upon force production. In addition, what controls the directionality of movement produced by these motors has been enlightened from structural studies of a class 14 kinesin [20] and of the Myo6 motor [21]. Myo6 indeed produces force and moves processively on a single track taking 30–36 nm in the opposite direction when compared to all other members of the superfamily. Many puzzles about this myosin have been solved [21] but how dimerization of motors define the mechanical output they can play in cells remains highly debated. The second part of this review will describe how structural studies brought light in understanding of how motors can evolve towards new mechanical functions.
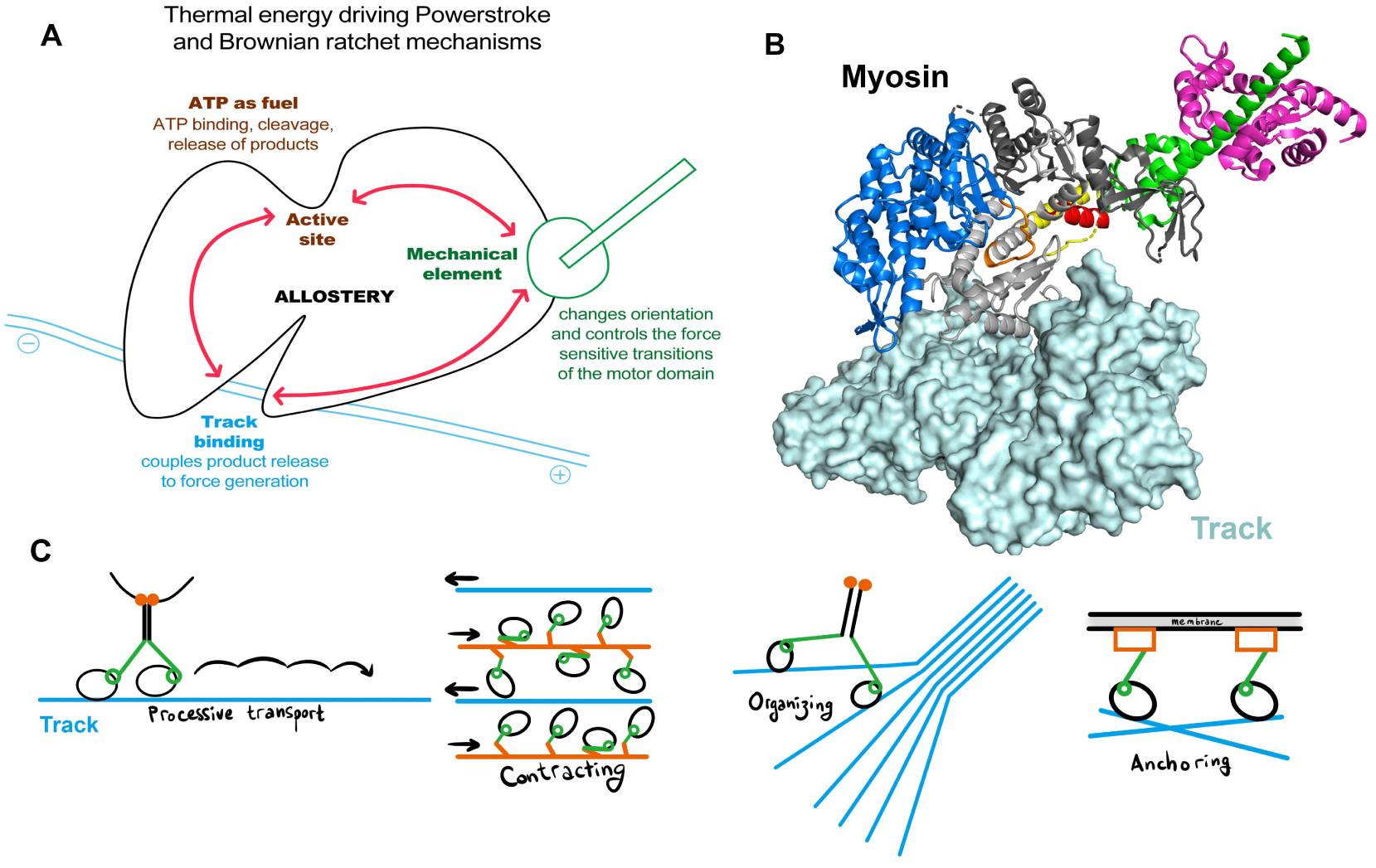
Allostery in molecular motors. (A) Allosteric sites in the motor domain that are essential for force generation. (B) Crystal structure of a myosin molecule at the end of its stroke bound to its F-actin track (light blue surface). The mechanical element is composed of the Converter (green) and the following helix to which light chains (purple) bind. (C) Interaction of a molecular motor with its track can lead to different types of mechanical outputs that require distinct motility properties of the motor. Both the way the motors are assembled and recruited as well as the kinetic and mechanical properties of their motor domain contribute to define the output nanomotors can produce in cell. Masquer
Allostery in molecular motors. (A) Allosteric sites in the motor domain that are essential for force generation. (B) Crystal structure of a myosin molecule at the end of its stroke bound to its F-actin track (light blue surface). The mechanical ... Lire la suite
The wealth of motor structures solved to date support the idea that all motors within a superfamily produce force by undergoing similar rearrangements. The motor domain sequence of different types of myosins, however, define their individual motor properties that underly their distinct roles in cells. How fast they move, how many steps they can perform on a single track prior to detachment (processive movement) and how they can operate under opposite force (load) is particularly important. However, no prediction from the motor sequence is possible for these crucial motor characteristics. In vitro experiments, in particular single molecule experiments, have been essential to define the work a motor can execute in cells [22, 23]. Depending on its own motility properties and the context of its recruitment, a motor can be suited for distinct mechanical output such as (a) transporting cargos along the cytoskeletal track (processive transporters), (b) contracting or organizing the cytoskeleton, or (c) acting as dynamic force sensors or anchors capable of withstanding an opposite load [24, 25, 26, 27] (Figure 1C). In addition, a wealth of data has described how motor activity can be impaired by single mutations and lead to diseases. This sequence information on the myosin superfamily provides an exceptional treasure trove to understand the essence of force production and how sequence variation can modify the kinetic and mechanical properties of motors for different purposes. In addition, new effectors that have emerged as promising drug candidates can modulate the force produced by a particular myosin, these will be discussed in the context of how these molecules can be used in targeted and personalized medicine.
2. General principles for force production by molecular motors
Unlike manufactured machines, molecular motors of nm-size work under a chaotic environment known as “brownian motion” [28]. Despite such thermally driven random molecular storm, these motors produce their own directed movement. In fact, they use chemical energy as fuel but also harness thermal agitation to undergo cyclic, unidirectional conformational rearrangements to produce mechanical work. By stereo-specific interactions with their track and using allostery, they can spatially rectify Brownian motion. The rearrangements they undergo while bound to the track are sensitive to the direction of the force applied to their mechanical element. Thus, motor proteins use cytoskeleton filaments as their tracks and as a means to convert chemical energy (provided by ATP hydrolysis) into mechanical energy (production of directed force) (Figure 1A). These nanomachines are highly efficient [29]. Such chemical-mechanical transduction has fascinated biophysicists, chemists and biologists for decades.
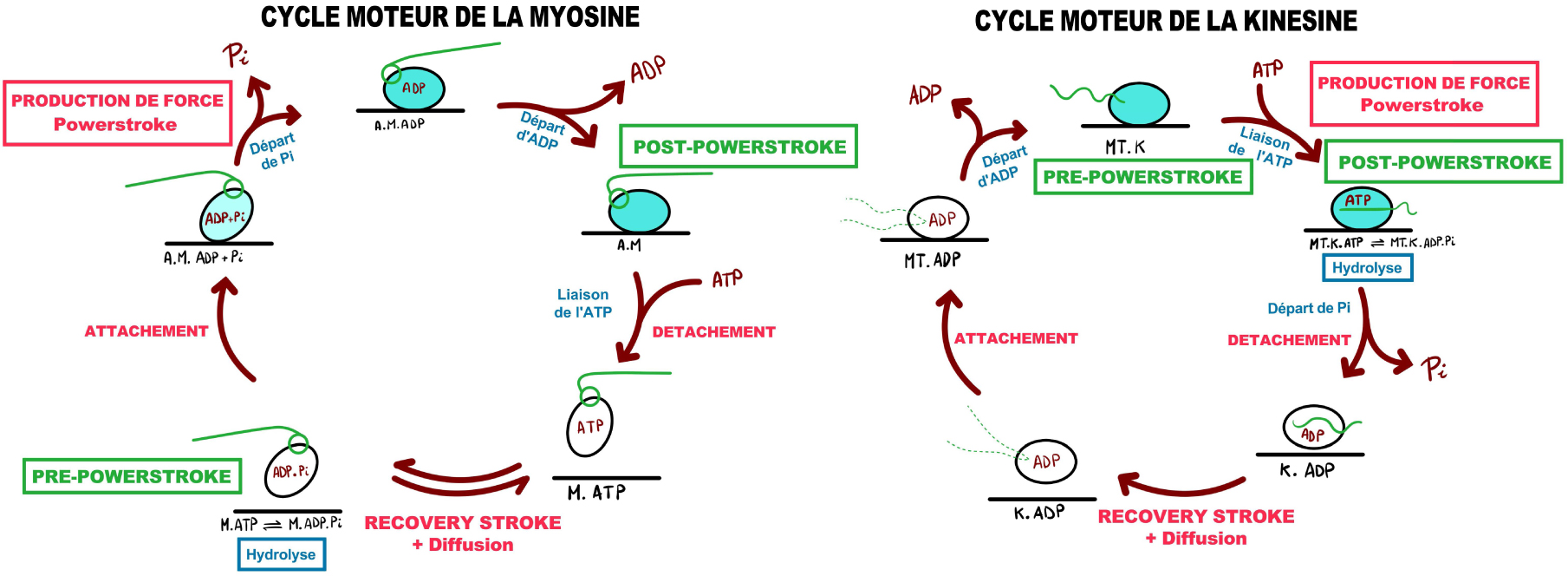
Motors are intrinsiquely poor ATPase enzymes. Binding to the track accelerates slow steps of their basal activity and ensures that ATP usage is coupled with force production on their track. Enzymologists provide essential clues about how these motors work [24, 25, 26, 27]. In 1971, transient kinetic studies had already set up how binding to the track depends on the nucleotide bound in muscle myosin II [30], allowing the description of the motor cycle used by all myosins to produce force (Figure 2). The major biochemical states and transitions kinesins undergo in their motor cycle have also been described [26]. Interestingly, major differences are found in these motor cycles: (1) the myosin-ATP states are not attached to the actin track while ATP-binding occurs when kinesin is bound to its MT track, (2) the ADP state is of high affinity for the track in myosin but of poor affinity in the case of kinesin. Overall, force production requires the ability to cycle and depends on the rates of the transitions in the cycle, not on how the motor evolved to allow this cycle to occur. Paramount in the motor design, however, is the capacity of the motor to guide its mechanical element in a directed motion when bound to the track. This large re-orientation of the mechanical element on the track is called the power stroke, while the states prior and after force generation are called pre-powerstroke and post-powerstroke (Figure 2). The cycle implies that while detached, the mechanical element will be repositioned to adopt a pre-powerstroke configuration, implying a recovery stroke for myosin motors while bound to ATP (Figure 2).
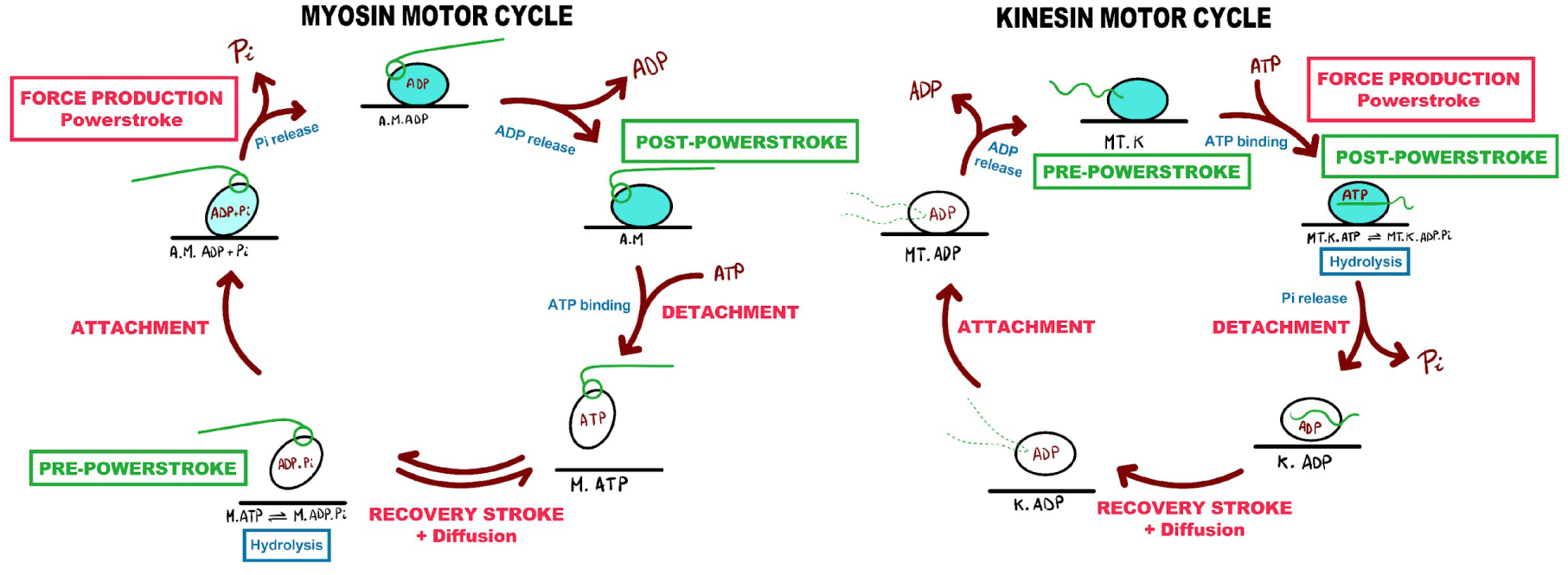
Motor cycles of myosin and kinesin motors. Molecular motors populate different conformational states along their cycle depending on the nucleotide bound in their motor domain. These conformations differ in their affinity for their track and in the orientation of their mechanical element. Here the conformational changes within the motor domain are not described although the colour in the motor indicates the affinity of the state of the track (dark green color for binding states of high actin affinity, white color for weak affinity states). The mechanical and chemical cycles of kinesin and myosin are presented. Note the difference in the orientation of the mechanical element along the cycle (lever arm for myosin, neck-linker for kinesin, represented in green). The cycles for kinesin and myosin are out of phase with respect to one another for the steps in which force is generated while they are strongly bound to the track. To generate force, actin filaments trigger Pi release from myosin, while microtubules act as nucleotide exchanger from ADP to ATP in kinesins. In the myosin cycle, ATP binding detaches the motor from its track and promotes repriming of the mechanical element (recovery stroke). Hydrolysis then occurs while myosin is detached from the track. Rebinding to the track accelerates the release of both Pi and then ADP, leaving the active site empty and ready to bind ATP. ATP binding detaches the motor that can restart a new cycle. In the kinesin cycle, detachment from the track occurs when kinesin has lost Pi from the active site. The ADP-state is indeed of poorest affinity for the track. Microtubule attachment allows to populate successively two states (nucleotide-free and ATP bound). ATP binding thus lead to force generation by directing the orientation of the mechanical element. Masquer
Motor cycles of myosin and kinesin motors. Molecular motors populate different conformational states along their cycle depending on the nucleotide bound in their motor domain. These conformations differ in their affinity for their track and in the orientation of their ... Lire la suite
What drives the direction of the cycle is the fact that the concentration in ATP is always higher than that of ADP in cells (ATP disequilibrium). It indeed causes ATP to bind rather than ADP in an empty active site. In addition, the track drives motor rearrangements uni-directionally and load is required to reverse the powerstroke. In the cycle, force is produced when the motor bound to the track undergoes a series of rearrangements that direct their mechanical element from the pre- to the post-powerstroke configuration. In myosins, this movement consists on a rotation of the lever arm, which lead to the “swinging lever arm hypothesis” [31]. The mechanical element is indeed called the lever arm and consists of the “converter” (the last subdomain of the motor) and the elongated region that follows. In kinesin, ATP binding drives the docking of the neck-linker and directs it in the sense of movement.
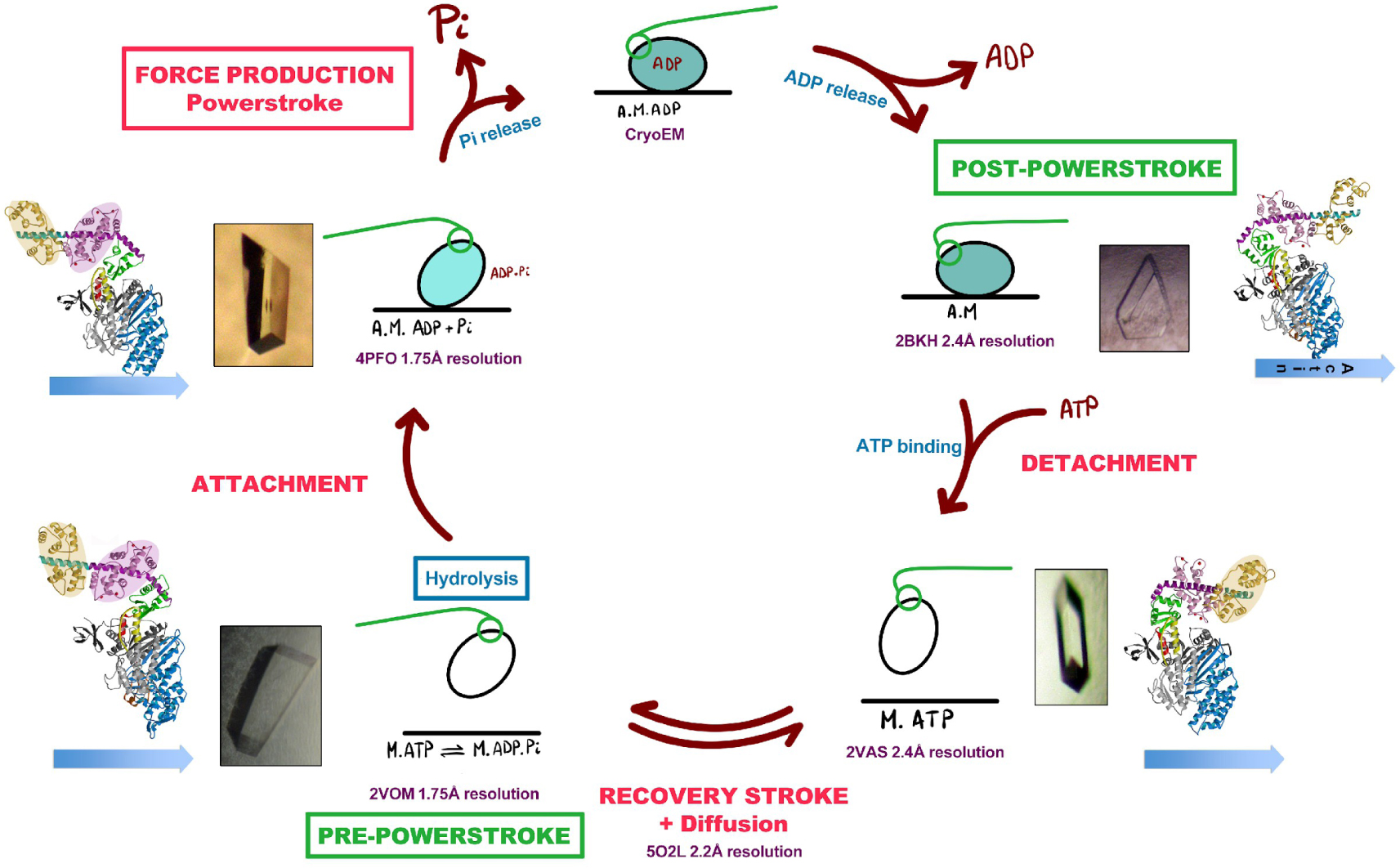
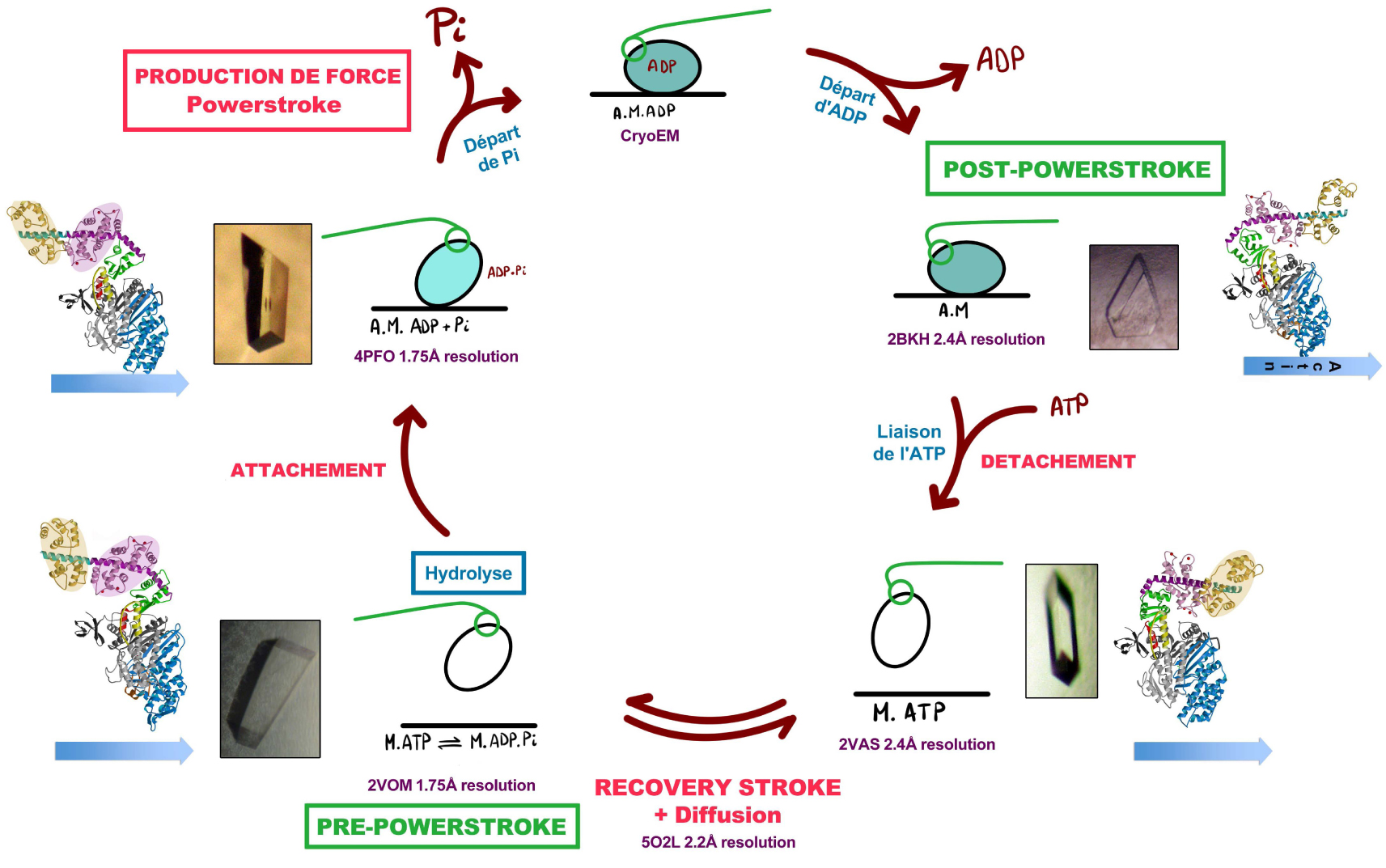
High resolution structures of Myo6 determined by X-ray crystallography. Atomic models of the Myo6 motor domain have allowed visualizing how major changes within the motor domain lead to force production. States populated by the motor are indicated with cartoons with a photo of the Myo6 crystals that allowed solving the structures presented colored by subdomains. Differences in the motor related to the nucleotide bound in the active site are amplified to direct its mechanical element (shown in the structures as the Converter (green circle), and the following α-helix that binds two calmodulin light chains (purple and yellow). The rearrangements explored by the motor to reorient its mechanical element by 180° during the recovery stroke (while detached from actin) greatly differ from those that drives force production during states the motor explores while attached to the track. Masquer
High resolution structures of Myo6 determined by X-ray crystallography. Atomic models of the Myo6 motor domain have allowed visualizing how major changes within the motor domain lead to force production. States populated by the motor are indicated with cartoons with ... Lire la suite
The current picture of how molecular motors work comes from several decades of studies in all disciplines, including molecular dynamic simulations that can provide insights on how transitions between snap-shots may occur in these nanoengines. High-resolution structures of a motor provides with atomic detail snap-shots of the states a motor can explore when bound to a particular nucleotide, or when bound to its track (as reviewed in [32]) (Figure 3). The conformation of three distant allosteric sites (track-binding site, ATP-binding site and mechanical element) as well as rearrangements within the motor that allow them to communicate by globally rearranging the motor domain as a whole can be described. These studies bring light on how the three essential allosteric sites depend on one another and how the motor accommodates binding events during its cycle. While critical information is missing on myosin states and rearrangements the motor explores during its powerstroke, a good description of the essential features of the pre-powerstroke and post-powerstroke states are available for both myosin [14] and kinesin motors [15] (Figure 2). Structural rearrangements lead to directing the mechanical element orientation thus generating and amplifying movement.
Dimeric processive motors can perform several steps along their track if the two heads coordinate their steps by gating. For this, the time spent in states where they are strongly attached to their track must be sufficiently long to ensure track binding of one head while the other head detaches, diffuses and reattaches. Note that diffusion is biased towards reattachment in the direction of movement thanks to the powerstroke of the attached head, as well as the repriming of the detached head. Thus, both the powerstroke and the biased Brownian diffusion required for reattachment to most suitable sites on the track contribute to stepping.
3. Force production by myosin motors—a structural perspective
The first description of the myosin motor at high resolution [33] and the wealth of structural insights gathered so far (reviewed in [14]) have been determinant for interpreting and guiding functional studies. Let us examine the atomic details of these nanoengines for a better understanding of their mechanism. First, X-ray crystallography structures of Myo2 provided three structural states of the motor of poor affinity for the track corresponding to the recovery stroke, (thus explored by the motor when it is bound with ATP) [34]. This provided the first view of the amplitude of the rearrangement of the lever arm in myosin motors [31] and how these changes replace the lever arm prior to force production [35, 36].
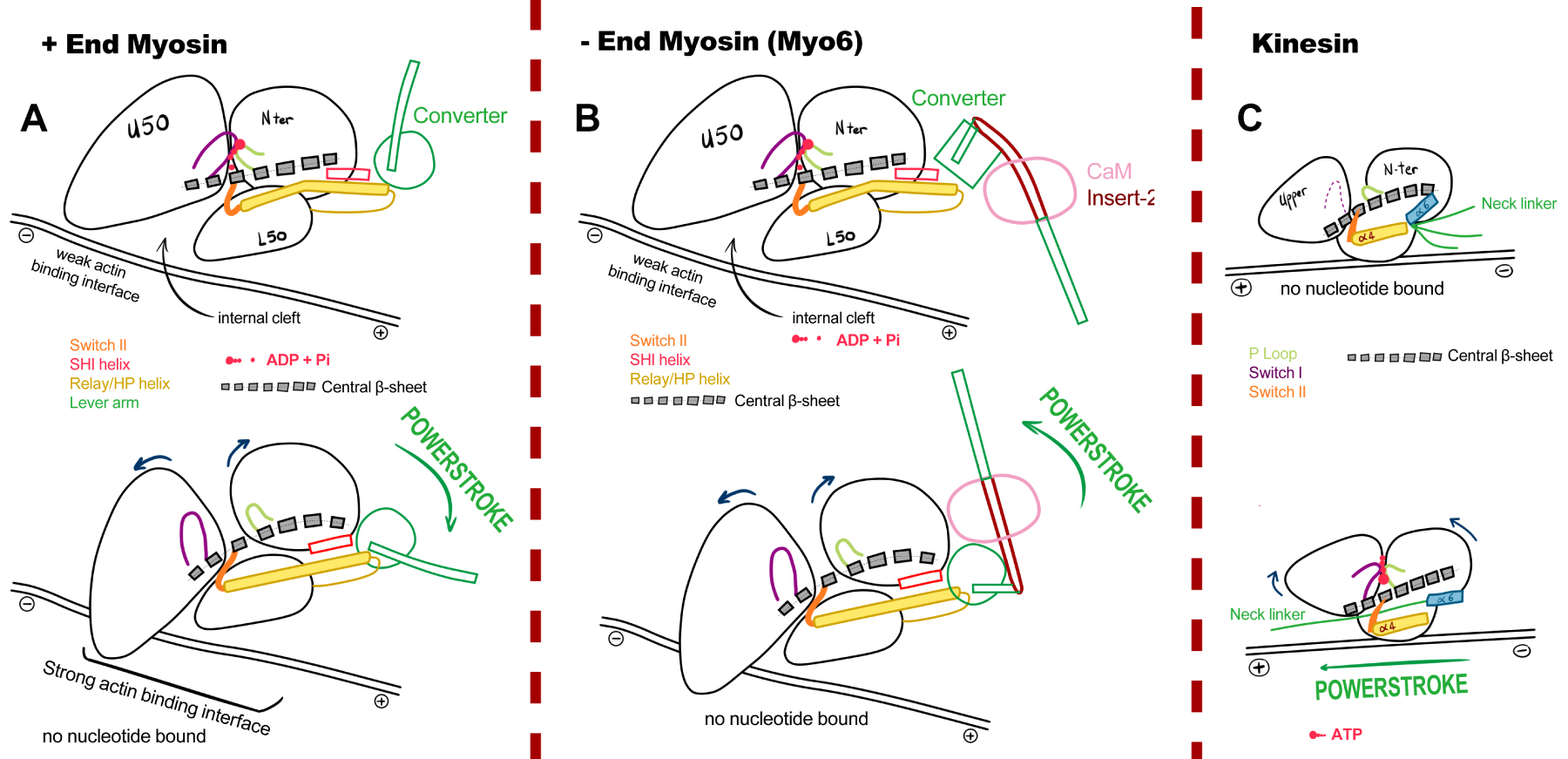
Motor domain conformations in the pre- and post-powerstroke states. The pre- and post-powerstroke states of the motors are shown at the top or bottom and allow appreciating the conformational changes within the motor linked to force production (powerstroke). Small movements within the motor domain are amplified and transmitted to the mechanical element. Its re-orientation lead to large directed movements. (A) and (B) Myosin rearrangements during the powerstroke allow closure of the internal cleft (between the U50 and L50 subdomains, coloured in blue and light grey in Figures 3 and 5) and thus the affinity for the track while elements of the active site control the release of the hydrolysis products. (B) In Myo6, the directionality is reversed by changes in the mechanical element that involves a change in the converter conformation as well as its interactions with a calmodulin (pink) bound to a specific 40 residue insert (brown). These variations in the mechanical element lead to an efficient 180° swing of the lever arm in the opposite direction compared to +-end myosins. (C) Kinesins undergo analogous rearrangements compared to myosin motors but these motors differ in how these conformational transitions control track affinity and the orientation of the mechanical element. Masquer
Motor domain conformations in the pre- and post-powerstroke states. The pre- and post-powerstroke states of the motors are shown at the top or bottom and allow appreciating the conformational changes within the motor linked to force production (powerstroke). Small movements ... Lire la suite
Determination of the Myo5 structures at 2 Å resolution in 2003 provided for the first time the visualization of how the motor binds strongly to the track [37], and how ATP binding promotes its detachment [38]. In 2016, cryo-electron microscopy (cryo-EM) studies have confirmed and extended this observation by describing the actin-myosin interface below 4 Å resolution [16]. All of the structures together highlighted a major change at the heart of the motor (in particular, its central beta-sheet) that drastically affect the nucleotide binding properties of the active site. A twisted conformation of the β-sheet separates nucleotide-binding elements, so-called P-loop and Switch-I, which are part of the N-term and U50 subdomains respectively (Figure 4A). ATP binding re-orientes the motor domain subdomains so that all nucleotide-binding elements can participate in ATP binding, which requires a flattened β-sheet conformation (Figure 4A). These rearrangements highlighted how formation of a strong actin interface requires the closure of an internal cleft within the motor, which is not compatible with strong ATP binding in the active site (details reviewed in [14]). From initial weak binding of ATP in an acto-myosin structure (with the internal cleft closed), rearrangements in the motor favouring strong binding to ATP populate states in which the internal cleft is opened. Distinct parts of the actin-binding interface are thus separated leading to weaker affinity for the track and detachment of the motor.
Interestingly, a motor without nucleotide can be crystallized in weak and strong actin-binding states [34, 37, 39]. The motor does not need actin or ATP to adopt these structural states. This suggests that the motor explores several conformations and works using conformational selection for this transition, although induced fit sensing locally the presence of ATP also plays a role to trigger efficient track detachment. Proper description of the free energy pathways for conformational transitions in the motor cycle is challenging. One of the fastest transition in the motor cycle is the repriming of the lever arm in an ATP-bound myosin detached from F-actin (i.e. the recovery stroke). Earlier studies had concluded that local sensing of the γ-phosphate of ATP would trigger a series of conformational rearrangements initiated at the active site that would guide the lever arm swing [40]. Recent myosin-ATP structures and molecular dynamic calculations do not support this scenario [41]. The current view indicates that the motor can first explore states in which the mechanical element is uncoupled and gets primed by thermal energy before stabilization can occur by formation of interactions at the active site [41]. The association of molecular dynamics simulations and ever-growing structural information is essential to reveal the mechanism of communication between allosteric sites.
The wealth of structures currently available indicate that all myosin motors undergo similar rearrangements along their motor cycle (Figures 3 and 4B). CryoEM structures have provided some answers regarding the last step of the powerstroke that controls ADP release [14, 17, 18, 42]. A major hole in our understanding is how the motor engages in interactions with the track at the beginning of force production, and how the track drives the rearrangements of the powerstroke. While data from a structural state of Myo6 provided clues on how actin promotes Pi release [43], these critical steps of the motor cycle remain poorly characterized.
4. Force production by kinesin motors
Suprisingly, considering the differences in their motor cycle (Figure 2), the first crystal structures of myosin [33] and kinesin revealed a striking structural similarity between the two motors, suggesting that they may share a common ancestor [44]. Notably, the motors possess similar nucleotide-binding elements and a central beta-sheet, which both undergo internal rearrangements that are analogous for the two motors and that are linked to the presence/absence of nucleotide in their binding site (Figure 4). As for myosin, three subdomains can be identified in the kinesin motor domain that change orientation in the pre- and post-powerstroke states. However, the track binding elements drastically differ in the two motors, as well as the mechanism that drives the mechanical element orientation. In kinesin, unlike myosin, no internal cleft is found to distinguish weak and strong binding interfaces for the track. In fact, when bound strongly to the track, kinesin interacts mainly via elements of its Lower subdomain, with much smaller participation of loops of the N-term and Upper subdomains (Figure 4).
Kinesin is half the size of myosin. The three allosteric sites (active site, track-binding site and the mechanical element) are much closer to one another than they are in myosin. Compared to myosin, the elements composing these allosteric sites undergo major order/disorder transitions that underlie force generation since less interaction stabilizes their conformation by interaction with other structural elements of the motor. Structural studies of kinesin bound to microtubules or tubulin have been particularly insightful for understanding the basis of the powerstroke during ATP binding [45, 46]. Interactions of the track with kinesin orders regions of the MT-binding interface that are close to the active site. These rearrangements overall lead to ADP release by opening the nucleotide-binding site. Upon ATP binding, the powerstroke occurs while the main and central region of the MT-binding interface remains unchanged. Thus, the kinesin stays anchored to the track while rearrangements in the motor accommodate ATP binding. Closure of the active site so that both P-loop and Switch-I participates in ATP binding requires flattening of the central β-sheet, as described for myosin (Figure 4). This leads to the rotation of the Nter and Upper subdomains around the central MT-binding elements, (which includes the α4 helix/L12 loop of the Lower subdomain). These rearrangements change drastically the environment of the last helix of the motor domain, the α6 helix, that preceeds the mechanical element. In pre-powerstroke structures (without ATP), steric hindrance with the α4 helix shortens α6 and directs the neck linker in a disordered, backwards conformation (Figure 4). Upon ATP binding, the steric hindrance with α4 is resolved due to the re-orientation of the subdomains. The α6 helix can elongate while the mechanical element (neck linker) docks on the motor domain surface and adopts a stable forward post-powerstroke orientation (Figure 4).
In summary, while the central rearrangements around the active site are similar in the two motors, the powerstroke in kinesin involves the control of the neck linker docking along the surface of the motor domain. Unlike kinesins, the equivalent of the α4 helix (the HP helix) is not involved in track binding in myosins. Instead, as part of the Relay, it participates in driving the lever arm rotation during the powerstroke together with the SH1 helix connector (Figure 4). Using analogous nucleotide-dependent rearrangements at the heart of the motor domain, kinesins and myosins have thus evolved in drastically different ways to produce force via the control of their mechanical element upon binding to the track.
5. How motors are tuned for distinct cellular functions
The motility properties of myosin motors are extremely diverse despite sharing a common mechanochemical cycle. They can thus perform vastly different biological functions. The tuning of this motility is the result of differences in the thermodynamics and kinetics of the motor cycle transitions, which are themselves encoded by sequence variations altering allostery within the motor. Sequence modifications can either impair or tune motor properties. Studies on divergent motors such as PfMyoA provide interesting illustrations of this point. A small number of critical differences compared to myosin sequences would lead to loss of motility if introduced in conventional myosins. However, PfMyoA is motile thanks to sequence compensation, in particular via a N-terminal extension that allow the motor to explore the same structural states as all other myosins [47, 48]. Interestingly, this PfMyoA motor can be tuned by a single phosphorylation site [47], found on its specific N-terminal extension that can modulate its performance under load and its velocity by controlling the last step of the powerstroke. Thus, at different stages of the life cycle of the Plasmodium falciparum parasite, the same motor is essential for quite different roles: it is used for fast cell motility (work under low load with a motor probably phosphorylated) or invasion of a host cell (work under high load with a motor best suited if unphosphorylated). This essential myosin for the parasite is thus considered as a prominent target for anti-malaria treatment.
The allosteric code that tunes motor properties is currently an enigma. Allostery is indeed a global property of the molecule and a single mutation may alter the rate of several steps of these precisely tuned nanomachines. Our current understanding of the transitions in the motor cycle are too limited to fully account for and predict what tunes force production. In addition, too little is known about how the track triggers these transitions, how divergence in the track binding regions can tune motor properties [14, 49] and how motors resist detachment from the track when opposite force is imposed on them. Ultimately, motility properties are governed by the rate of the transitions between states of the motor cycle, in particular those that occur while the motor is bound to the track. It is thus critical to gain an understanding of how the sequence of the motor encodes the kinetics and the force sensitivity of the transitions of the powerstroke.
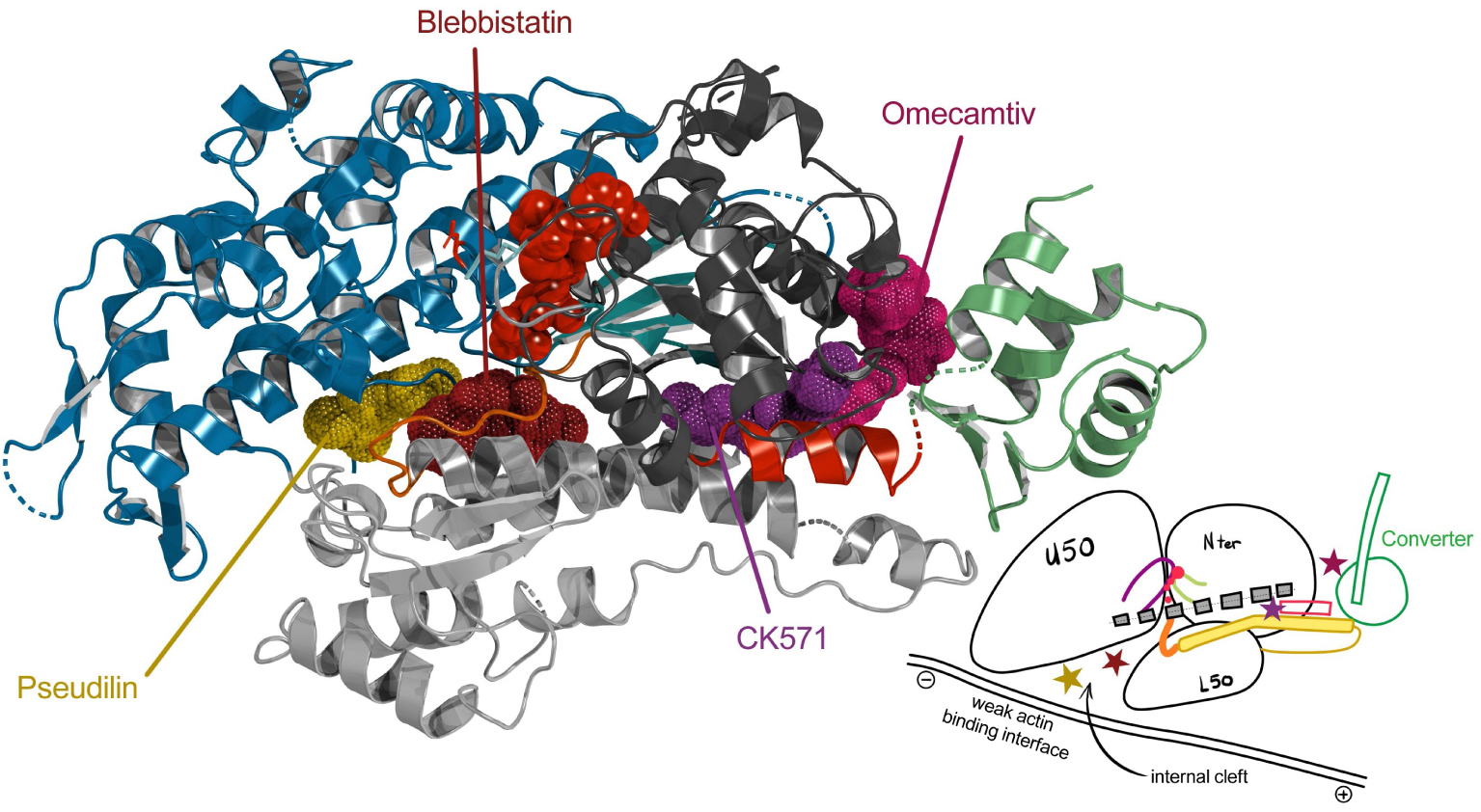
Binding sites of small molecule effectors in the myosin motor domain. The myosin motor domain structure is presented in cartoon on the left with the ATP molecule (red balls) and as a schematic drawing on the right. Binding sites for four allosteric small molecules that act as specific inhibitors (blebbistatin [50]; Pseudilin [51], CK-571 [52]) or activator (Omecamtiv mecarbil, [53]) are presented as colored surfaces (left) or colored stars (right). CK-571 binds in a pocket that opens only during the recovery stroke [52]. The other molecules bind in the pre-powerstroke state of myosin molecules. Masquer
Binding sites of small molecule effectors in the myosin motor domain. The myosin motor domain structure is presented in cartoon on the left with the ATP molecule (red balls) and as a schematic drawing on the right. Binding sites for ... Lire la suite
Sequence diversity in the mechanical element also tunes motor activity. Reverse directionality and the ability to organize cytoskeleton tracks are two striking examples of how the mechanical element can lead to novel motility properties. Comparison of +end and -end myosin motors in Figures 4A and B show that the main difference results from a small 39 residues insert that is unique to Myo6. This sequence binds calmodulin and is part of the converter. Unlike other myosins, the converter undergoes internal rearrangements upon force production that efficient reverse the direction of the lever arm by 180° between the pre- and the post-powerstroke state of this motor (Figure 4B). Unusual mechanical elements can also allow reaching out to more than one track. Structural studies of the Myo10 motor [54] and of the Kif20A motor have thus provided insights on how the length and the swing angle of the mechanical element can differ during the powerstroke in these motors compared to processive motors such as Myo5 and Kinesin 1. These adaptations allow attachment for the track while making a new step to occur preferably on nearby filaments onto which force can be produced allowing zippering and thus organization of the tracks.
There is no doubt that visualizing molecular motors at atomic resolution is a powerful means for understanding the origin of their distinct mechanical output in cells and more puzzles about how motors acquire distinct function will be uncovered in the future by studying tail regions of the motor and how partners influence motor clustering and their ability to interact with tracks.
6. Myosin and diseases
Allostery is key for force production. As the motor output can easily be altered by a single mutation in these precisely designed allosteric machines, several molecular motors have been linked to human diseases [55, 56]. For example, a large number of mutations in the genes coding for Myo6, Myo7a or Myo15a can lead to deafness since these motors are essential for the integrity of the hair cells of the inner ear [57]. Mutations in distinct regions of a motor can either lead to total or partial impairment of force production, or can also result in improper regulation of its activation in cells. Studies of the motility properties of these impaired motors and the cellular impairments they cause are interesting means to learn about force generation and how precise it must occur in cells.
Cardiomyopathies, which can lead to sudden cardiac death, provide a remarkable demonstration of how a small difference in the force a motor produces can influence the cell fate. Heart beat is powered throughout life by a myosin motor (β-cardiac myosin) that contracts cardiomyocytes. Distinct mutations in sarcomeric proteins have been identified to cause distinct forms of cardiomyopathies, slightly increasing or reducing the contraction produced. Over time, the heart cells sense this impairment and respond by changing their metabolism [58]. While the mechanisms leading to alterations in cell organization may vary depending on each mutation, it is becoming clear that mechanosensing is an important factor for the progression of the disease [59]. Milestone studies have uncovered the possibility of new treatments for such diseases by demonstrating that small molecules acting as allosteric effectors can modulate the force produced by myosin motors. Three drug candidates acting either as specific inhibitors or activators of cardiac myosin are under phase 3 clinical trials for treatment of distinct heart insufficiencies [60, 61, 62]. The pockets in which several of these specific allosteric effectors bind have been identified in the myosin molecule (Figure 5). Current research aims at exploiting these sites for discovering new molecules with high therapeutic potential for diseases such as spastic gait disorder [63] or malaria [48, 56].
7. Conclusion and perspectives
By acting locally to produce directed force at specific locations in cells, biological motors underpin essential functions, organizing or contracting cytoskeleton tracks or moving along polarized tracks. Several of these engines work together for exchange of membranous transport intermediates between compartments: some motors are required for scission of membrane tubes [64, 65], others participate in long-range and short-range transport and others assist fusion events. Even more motors coordinate their action for the movements and forces required in the distribution of the genetic material in a dividing cell. Forces built by these nanomotors are efficiently transmitted to control large cell rearrangements in processes such as cell division and cell migration. Integration of mechanical sensing and signalling is also critical to build highly organized structures (neuron protrusions, filopodia, microvilli, stereocilia) that define cell function and control their fate. It is thus indispensable to understand how forces produced in cells control cellular processes and much of it can be understood by figuring out how these nanomachines work to keep cells orderly.
Current insights from cell studies are often limited to the identification of the cell processes that are impaired when the whole cell is depleted from a particular molecular motor and to the characterization of a possible partner on a compartment. Going further to define the molecular output of a nanomotor in cells is not easy. As illustrated in this review, structural biology provides unsuspected insights at atomic detail that reveal how molecular motors perform distinct cellular functions. This can be the source of inspiration for design of chimeric motors or separation of function mutations that ultimately can test precisely how motors are recruited and which mechanical output they are responsible of in a particular cellular context. Modulating at will the force produced in cell by the discovery of specific allosteric effectors against distinct motors will also greatly advance this research. High-resolution structures of these motors bound to their track are missing to depict fully how they produce movement, in particular against load. Overcoming such fundamental questions regarding these fascinating allosteric proteins is essential as this will provide essential clues about how the motor domain sequence has evolved for performing different mechanical output. The discovery of new allosteric effectors is exciting as they can bring new insights on the transitions that control force generation. Such small molecules also provide emerging and promising therapeutical approaches for deadly diseases and thus can greatly improve human health in the future.
Acknowledgements
Research is a human adventure that provides joy when perseverance and intuition leads to discovery. I am thankful to my mentors Carolyn Cohen and Andrew Szent-Gyorgyi at Brandeis University and the structural biologists Professor Roger Fourme, Dr. Graham Bentley, Professor Roberto Poljak and Professor Joël Janin for their encouragements earlier in my career. I am immensely grateful to my collaborators Margaret Titus, Kathleen Trybus, among others, as well as to Lee Sweeney for our partnership over 20 years. I also express special thanks to beamline scientists at the Soleil synchrotron for their dedication and support. Special thanks also to Marie Juillé that draw the figures of this article. Finally, I dedicate this review to all members of the Structural Motility laboratory. I thank them deeply for their contribution in the understanding of these beautiful nanomachines.
French version
1. Introduction
La motilité est une propriété essentielle pour tous les organismes vivants. Elle dépend de façon critique des nanomachines biologiques appelées moteurs moléculaires. Leur capacité à produire une force ou un mouvement unidirectionnel dans l’environnement surpeuplé d’une cellule est essentielle pour la division cellulaire, la morphogenèse, le trafic intracellulaire, la contraction musculaire et de nombreuses fonctions cellulaires spécialisées. Plus de 100 nanomachines de ce type sont codées dans le génome humain. Le cytosquelette, constitué de polymères biologiques polarisés, fournit la plupart des propriétés mécaniques et architecturales des cellules. Des moteurs moléculaires ainsi que de nombreuses protéines règlent finement la dynamique et l’organisation du cytosquelette pour contrôler les processus cellulaires et la forme des cellules. Ces moteurs moléculaires biologiques sont des enzymes protéiques qui produisent une force en utilisant l’ATP comme combustible. Ils s’engagent dans des cycles d’interactions avec leur piste cytosquelettique polarisée, amplifiant ainsi les changements conformationnels essentiels pour définir la direction de la force produite. Ainsi nait un mouvement ou une force le long de la piste cytosquelettique.
Trois familles de moteurs biologiques se sont développées au cours de l’évolution : les myosines travaillent sur les filaments d’actine tandis que les moteurs de type kinésine et dynéine produisent une force en utilisant des microtubules (MTs) comme pistes. La myosine II du muscle squelettique, le moteur qui actionne la contraction musculaire, a été le premier moteur identifié [1] et la découverte ultérieure de l’actine a conduit à la reconstitution in vitro de la contraction, l’une des premières avancées majeures dans l’étude de la contractilité cellulaire [2, 3]. En 1969, le groupe de Holtzer a décrit que toutes les cellules eucaryotes contiennent des structures à base d’actine [4] et le rôle de la myosine II non musculaire dans la Cytocinèse, (dernière étape de la division cellulaire), a été découvert en 1977 [5, 6]. En ce qui concerne les microtubules, le premier moteur identifié a été la dynéine en 1967 [7]. En 1985, la première kinésine a été découverte comme force motrice du transport axonal [8]. L’étonnante richesse des moteurs biologiques est devenue réellement évidente dans les années 90, lors du séquençage des génomes qui a conduit à la découverte des superfamilles de la myosine et de la kinésine [9, 10]. Toutes ces protéines produisent une force via leur domaine moteur et leur élément mécanique, tandis que leurs régions de queue (en C-terminal) sont diverses et jouent des rôles essentiels pour l’assemblage ou le recrutement de ces moteurs.
L’activation contrôlée de ces nanomoteurs induit la production d’une force à un endroit précis de la cellule. Cependant, cette force peut conduire à diverses actions mécaniques en fonction des propriétés du moteur et de la manière avec laquelle dont il est recruté. D’une telle force peut donc résulter le transport spécifique de cargos sur de longues distances au sein des cellules, l’organisation ou la contraction de pistes cytosquelettiques, ou la résistance à des forces externes. Lors d’événements cellulaires complexes tels que la division cellulaire, des moteurs distincts sont en jeu, tous travaillant sur les pistes d’actine ou de microtubules à des moments précis, mais souvent dans des régions distinctes de la cellule. La manière avec laquelle les modifications post-traductionnelles, les partenaires, les signaux ou les membranes lipidiques influencent le recrutement des moteurs est actuellement sous-étudiée. La liaison directe des machineries de polymérisation de l’actine et des moteurs de myosine peut coordonner leur recrutement spécifique [11]. Il reste beaucoup à apprendre sur la façon avec laquelle les moteurs sont activés en temps voulu et le rôle que chaque moteur joue dans un processus cellulaire est souvent non élucidé.
En plus de leur séquence motrice spécifique, les caractéristiques structurales atypiques du moteur et la façon avec laquelle les partenaires interagissent avec lui peuvent moduler les propriétés mécaniques en modifiant l’assemblage de ces moteurs lorsqu’ils sont recrutés. Il est donc difficile de définir leur rôle dans les cellules, c’est-à-dire l’action réelle qu’ils exécutent en produisant une force après recrutement. Les études de reconstitution in vitro définissent les propriétés de motilité ou la production mécanique possible de ces moteurs et sont donc précieuses pour définir leur fonctionnement. Elles peuvent conduire à des hypothèses vérifiables sur le rôle que les moteurs jouent dans les cellules. L’impact de la mécanobiologie sur le comportement et la réponse des cellules à leur environnement est clairement établi. Les futures orientations de la recherche doivent étudier comment la production mécanique des moteurs contribue au destin des cellules en matière de santé et de maladie afin de fournir une compréhension mécanistique des processus vitaux ainsi que des traitements innovants.
Comme le chronophotographe Muybridge, lorsqu’il a analysé le « cheval en mouvement » [12, 13], la visualisation de ces nanomachines dans différents états de leur cycle moteur (cycle de production de force) a éclairé la manière avec laquelle la force est produite. Associées à des études fonctionnelles et à des expériences de dynamique moléculaire, les études de la structure de ces moteurs ont mis en lumière le rôle de l’allostérie dans la production de force [14, 15]. Par la sélection de conformations accessibles au moteur le long de son cycle, trois régions du moteur (sites allostériques) communiquent : le site de liaison à l’ATP (site actif), le site de liaison au cytosquelette et l’élément mécanique responsable du mouvement dirigé le long du cytosquelette (Figure 1A,B). Un événement dans l’un de ces sites allostériques (i.e. liaison de l’ATP, liaison à la piste, force mécanique externe appliquée à l’élément mécanique) est communiqué aux autres sites et l’ensemble de ces informations est intégré pour influencer les conformations accessibles et les transitions du cycle moteur. Les structures de résolution atomique issues de la cristallographie aux rayons X ont été particulièrement utiles pour déchiffrer de quelle fac˛on un moteur myosine produit une force. Depuis 2016, des structures à haute résolution sont également disponibles pour décrire comment les moteurs se lient à leur piste cytosquelettique [14, 16, 17, 18], car la cryo-microscopie électronique (cryoEM) a connu une « révolution de la résolution » [19]. Dans la première partie de cet exposé, les mécanismes généraux essentiels à la production de force par les moteurs myosine et kinésine seront décrits, ainsi que les défis actuels qui limitent notre compréhension.
Allostérie dans les moteurs moléculaires. (A) Sites allostériques du domaine moteur qui sont essentiels pour la génération de force. (B) Structure cristalline d’une myosine à la fin de la production de force. La surface de l’actine est représentée en bleu clair. (C) Actions mécaniques possibles d’un moteur lorsqu’il produit une force en interagissant avec le cytosquelette. Pour un moteur donné, ses propriétés mécaniques mais aussi la façon avec laquelle il est assemblé et recruté contribuent à définir l’action mécanique qu’il réalisera au sein d’une cellule à la suite de son recrutement. Masquer
Allostérie dans les moteurs moléculaires. (A) Sites allostériques du domaine moteur qui sont essentiels pour la génération de force. (B) Structure cristalline d’une myosine à la fin de la production de force. La surface de l’actine est représentée en bleu ... Lire la suite
Beaucoup reste à faire pour comprendre comment les moteurs moléculaires sélectionnent leurs pistes cytosquelettiques dans les cellules ou comment ils peuvent affecter la dynamique et l’organisation du cytosquelette. Des connaissances intéressantes ont été acquises sur les moteurs qui ne fonctionnent pas sur une seule piste mais sur plusieurs, comme la myosine Myo10 et la kinésine KIF20A. Ces moteurs n’utilisent pas seulement la piste pour se déplacer ou pour contracter des filaments. Ils peuvent aussi influencer l’organisation du cytosquelette avec lequel ils interagissent en produisant une force. En outre, ce qui contrôle la direction du mouvement produit par ces moteurs a été mis en lumière par des études d’une kinésine de classe 14 [20] et du moteur Myo6 [21]. Myo6 est la seule myosine caractérisée aujourd’hui comme produisant une force en sens opposé des autres myosines. Elle peut se déplacer de manière processive sur une seule piste en réalisant des pas de 30–36 nm (transport). Elle a également la capacité de résister à des forces extérieures et d’organiser des filaments. De nombreuses énigmes concernant cette myosine ont été résolues [21], mais la manière avec laquelle la dimérisation des moteurs définit l’action mécanique qu’ils peuvent accomplir dans les cellules reste très controversée. La deuxième partie de cette revue décrira comment ces moteurs ont pu évoluer pour effectuer de nouvelles fonctions mécaniques.
Les structures de moteurs résolues à ce jour confortent l’idée que tous les moteurs d’une superfamille produisent une force en subissant des réarrangements similaires. La séquence des domaines moteurs des différentes myosines, cependant, définit précisément leur propriété de motilité propre afin qu’elles puissent jouer des rôles distincts dans les cellules. La vitesse à laquelle elles se déplacent, le nombre de pas qu’elles peuvent effectuer sur une piste sans s’en détacher (mouvement processif) et la manière avec laquelle elles peuvent résister lorsqu’elles sont soumises à une force opposée (charge) sont des spécificités propres particulièrement importantes. Cependant, aucune prédiction n’est possible pour ces caractéristiques motrices cruciales à partir de la connaissance seule de la séquence du moteur. Les expériences in vitro, en particulier les expériences sur molécule unique, ont été essentielles pour définir le travail qu’un moteur peut exécuter dans les cellules [22, 23]. En fonction de ses propriétés de motilité propre et du contexte de son recrutement, un moteur peut effectuer diverses actions mécaniques distinctes telles que (a) le transport de cargos le long de la piste cytosquelettique (transporteurs processifs), (b) la contraction ou l’organisation du cytosquelette, ou (c) la fonction de senseur de force dynamique ou d’ancrage capable de résister à une charge opposée [24, 25, 26, 27] (Figure 1C). D’autre part, une multitude de données ont décrit comment l’activité motrice peut être altérée par des mutations et conduire à des maladies. Ces informations constituent un trésor exceptionnel pour décoder comment la séquence secondaire du domaine moteur peut ajuster les propriétés mécaniques d’un moteur pour des fins différentes, et comment une altération de séquence peut engendrer une pathologie. L’existence actuelle de candidats médicaments capables de moduler spécifiquement la force produite par une myosine sera discutée dans le contexte de l’intérêt de ces molécules pour une médecine ciblée et personnalisée.
2. Principes généraux de la production de force par les moteurs moléculaires
Contrairement aux machines manufacturées, les moteurs moléculaires de taille nanométriques fonctionnent dans un environnement chaotique appelé « mouvement brownien » [28]. Malgré cette tempête moléculaire aléatoire, ces moteurs produisent leur propre mouvement dirigé. En fait, ils utilisent l’énergie chimique comme combustible mais exploitent également l’agitation thermique pour subir des réarrangements conformationnels cycliques et unidirectionnels afin de produire un travail mécanique. Par des interactions stéréospécifiques avec les pistes cytosquelettiques et en utilisant l’allostérie, ils peuvent rectifier spatialement le mouvement brownien. Les réarrangements qu’ils entreprennent lorsqu’ils sont liés à la piste sont sensibles à la direction de la force appliquée à leur élément mécanique. Ainsi, les protéines motrices utilisent les filaments du cytosquelette comme leurs pistes et comme moyen de convertir l’énergie chimique (fournie par l’hydrolyse de l’ATP) en énergie mécanique (production d’une force dirigée) (Figure 1A). Ces nanomachines sont efficaces [29] et fonctionnent à partir de conditions compatibles avec la vie. Cette transduction chimico-mécanique fascine les biophysiciens, les chimistes et les biologistes depuis des décennies.
Les moteurs sont des enzymes ATPase intrinsèquement peu efficaces. Leur liaison à la piste cytosquelettique accélère les étapes lentes de leur activité basale et garantit que l’utilisation de l’ATP soit couplée à la production de force sur leur piste. Les enzymologistes ont fourni des faits essentiels pour comprendre le fonctionnement de ces moteurs [24, 25, 26, 27]. En 1971, des études de cinétique transitoire avaient déjà établi pour la myosine II du muscle comment la liaison à la piste (actine) dépend du nucléotide lié [30], permettant ainsi la caractérisation des états du cycle utilisé par toutes les myosines pour produire une force (Figure 2). Les principaux états biochimiques et les transitions que les kinésines subissent dans leur cycle moteur ont également été décrits [26]. Il est intéressant de noter que ces cycles moteurs présentent des différences majeures : (1) les états myosine-ATP sont détachés de la piste actine alors que la liaison ATP se produit lorsque la kinésine est fortement attachée à sa piste MT, (2) l’état ADP est de forte affinité pour la piste dans la myosine mais de faible affinité dans le cas de la kinésine. Dans l’ensemble, la production de force requiert la capacité à effectuer un cycle et les propriétés du moteur dépendent de la vitesse des transitions dans le cycle, mais ces deux moteurs ont évolué pour permettre à ce cycle de se produire de façon très différente. Cependant, ce qui est primordial dans la conception du moteur est la capacité du moteur à guider son élément mécanique dans un mouvement dirigé lorsqu’il interagit avec la piste. Cette grande réorientation de l’élément mécanique sur la piste est appelée « powerstroke » (force de frappe), tandis que les états précédant et suivant la génération de la force sont appelés pré-powerstroke et post-powerstroke (Figure 2). Le cycle implique que, lorsqu’il est détaché, l’élément mécanique revient vers une configuration pré-powerstroke, ce qui se produit pour les moteurs myosine par réarrangement alors qu’ils sont liés à l’ATP lors de la transition « recovery stroke » (transition de récupération) (Figure 2).
Cycles des moteurs myosine et kinésine. Les moteurs moléculaires adoptent des états conformationnels de façon cyclique en fonction du nucléotide lié dans leur site actif, ce qui contrôle l’affinité avec les pistes cytosquelettiques et l’orientation de leur élément mécanique. Ici, les changements conformationnels internes au domaine moteur ne sont pas décrits bien que la couleur du moteur reflète l’affinité pour la piste (vert foncé pour les états de forte affinité, blanc pour les états de faible affinité). Les cycles mécaniques et chimiques de la kinésine et de la myosine sont présentés. L’élément mécanique (bras de levier pour la myosine, « neck-linker » pour la kinésine, représenté en vert) change d’orientation au cours du cycle. Les cycles des kinésines et des myosines sont déphasés l’un par rapport à l’autre pour les étapes dans lesquelles la force est générée alors que les moteurs sont fortement liés à la piste. Pour générer une force, les filaments d’actine déclenchent la libération de Pi de la myosine suivie de celle de l’ADP, tandis que les microtubules agissent comme échangeur de nucléotide de l’ADP à l’ATP dans les kinésines. Dans le cycle de la myosine, la liaison de l’ATP détache le moteur de sa piste et les réarrangements permettent de réorienter librement son élément mécanique (« recovery stroke » ou transition de récupération). L’hydrolyse se produit alors que la myosine est détachée de la piste. Le ré-attachement à la piste accélère la libération de Pi puis de l’ADP, laissant le site actif vide. Il est alors prêt à lier l’ATP, détachant le moteur qui peut redémarrer un nouveau cycle. Dans le cycle kinésine, le détachement de la piste se produit lorsque la kinésine a perdu le Pi du site actif. L’état ADP est en effet celui de plus faible affinité pour la piste. L’attachement des microtubules permet de peupler successivement deux états (sans nucléotide puis avec ATP lié). La liaison de l’ATP conduit à la génération de force en dirigeant l’orientation de l’élément mécanique. Masquer
Cycles des moteurs myosine et kinésine. Les moteurs moléculaires adoptent des états conformationnels de façon cyclique en fonction du nucléotide lié dans leur site actif, ce qui contrôle l’affinité avec les pistes cytosquelettiques et l’orientation de leur élément mécanique. Ici, ... Lire la suite
Ce qui détermine la direction du cycle est le fait que l’ATP est toujours en plus forte concentration que l’ADP dans les cellules. Ainsi, l’ATP se lie plutôt que l’ADP dans un site actif vide. De plus, la piste entraîne des réarrangements unidirectionnels dans le moteur et une force externe inverse (charge) est nécessaire pour inverser le mouvement de la machine. Au cours du cycle moteur, la force de ces nanomachines est produite lorsqu’ils subissent une série de réarrangements alors qu’ils sont fortement attachés à la piste. Le mouvement de leur élément mécanique passe alors de l’orientation pré-powerstroke à l’orientation post-powerstroke. Dans les myosines, ce mouvement consiste en une rotation du bras de levier, et l’hypothèse du « bras de levier pivotant » [31] a été décrite sur la base de l’observation de structures de myosine avant d’être confirmée par des expériences fonctionnelles. L’élément mécanique est en fait appelé bras de levier pour une myosine et se compose du « convertisseur » (le dernier sous-domaine du domaine moteur) et de la région allongée qui suit. Dans la kinésine, l’élément mécanique est une région non structurée qui suit le domaine moteur, le « neck-linker ». Cet élément mécanique est dirigé dans le sens du mouvement lorsqu’il s’arrime au domaine moteur alors que ce dernier se lie à l’ATP.
Structures à haute résolution de la myosine Myo6 déterminées par cristallographie aux rayons X. Les modèles atomiques du domaine moteur de Myo6 ont permis de visualiser comment les changements majeurs au sein du domaine moteur peuvent conduire à la production de force. Les états peuplés par le moteur sont indiqués sous forme schématique, à côté de la photo des cristaux obtenus pour résoudre la structure de la Myo6 dans différents états du cycle. Ces structures sont représentées en adoptant des couleurs spécifiques pour les sous-domaines du domaine moteur pour mieux observer les changements de conformation (notamment entre la position relative des sous-domaines bleu et blanc contribuant au site d’interaction au filament d’actine). Les différences au sein du moteur sont amplifiées pour diriger son élément mécanique (représenté dans les structures par le Convertisseur (cercle vert), et l’hélice α qui le prolonge et qui interagit avec deux chaînes légères de type calmoduline (violette et jaune)). Les réarrangements explorés par le moteur pour réorienter son élément mécanique de 180° pendant le « recovery-stroke » (transition de récupération) lorsqu’il est détaché de l’actine diffèrent grandement de ceux qui permettent la production de force (lorsqu’il prend appui sur le filament d’actine. Masquer
Structures à haute résolution de la myosine Myo6 déterminées par cristallographie aux rayons X. Les modèles atomiques du domaine moteur de Myo6 ont permis de visualiser comment les changements majeurs au sein du domaine moteur peuvent conduire à la production ... Lire la suite
Le modèle actuel du fonctionnement des moteurs moléculaires provient de plusieurs décennies d’études dans toutes les disciplines. Les structures à haute résolution d’un moteur fournissent des instantanés en trois dimensions comprenant des détails atomiques sur les états qu’un moteur peut explorer lorsqu’il est lié à un nucléotide particulier, ou lorsqu’il est lié à sa piste [32] (Figure 3). Des simulations de dynamique moléculaire peuvent fournir des indications sur la façon avec laquelle les transitions s’effectuent entre ces états structuraux principaux. Trois sites allostériques distants (site de liaison à la piste, site de liaison à l’ATP et élément mécanique) sont réorganisés de façon coordonnée car ils communiquent via les réarrangements du domaine moteur dans son ensemble. Ces études mettent en lumière la dépendance entre les trois sites allostériques essentiels et la façon avec laquelle le moteur s’adapte aux événements de liaison au cours de son cycle. Bien qu’il manque des informations essentielles sur certains états structuraux et sur les réarrangements que le moteur explore pendant son cycle, une bonne description des caractéristiques essentielles des états pré- et post-powerstroke est connue pour les moteurs myosine [14] et kinésine [15] (Figure 2). Les réarrangements structuraux du moteur par la liaison au nucléotide et à la piste le conduisent à effectuer un cycle et à modifier l’orientation de l’élément mécanique, générant et amplifiant ainsi le mouvement.
Les moteurs processifs dimériques peuvent effectuer plusieurs pas le long de leur trajectoire si les deux têtes coordonnent leurs cycles moteurs. Pour cela, le temps passé dans les états fortement attachés à la piste doit être suffisamment long pour qu’une tête reste fixée à la piste tandis que l’autre tête se détache, diffuse et se rattache. Notez que la diffusion est biaisée pour favoriser le rattachement dans le sens du mouvement grâce à la force de frappe (powerstroke) de la tête attachée, ainsi qu’au mouvement inverse de récupération de la tête détachée. Ainsi, la force de frappe et la diffusion brownienne biaisée sont les deux principes requis pour produire un mouvement et une force par un moteur. Ces deux facteurs contribuent à définir l’action des moteurs en définissant les sites de rattachement les plus appropriés sur la piste.
3. La production de force par les moteurs myosine — une perspective structurale
La première description du domaine moteur de la myosine à haute résolution [33] et la richesse des connaissances structurales recueillies jusqu’à présent (examinées dans [14]) ont été déterminantes pour interpréter et diriger des études fonctionnelles. Examinons les détails atomiques de ces nanomoteurs pour mieux comprendre leur mécanisme. En premier lieu, la cristallographie aux rayons X a permis de décrire trois états structuraux du moteur Myo2 lié à l’ATP de faible affinité pour la piste (actine) correspondant aux états du moteur lui permettant de replacer son élément mécanique lors de la transition de récupération (« recovery stroke ») [34]. Ceci a fourni la première vue de l’amplitude du réarrangement du bras de levier dans les moteurs myosine [31] et comment ces changements placent le bras de levier avant production de force (état pré-powerstroke) [35, 36].
La détermination des structures du moteur Myo5 à une résolution de 2 Å en 2003 a permis pour la première fois de visualiser comment le moteur se lie fortement à la piste [37], et comment la liaison de l’ATP favorise son détachement [38]. En 2016, les études de cryo-microscopie électronique (cryoEM) ont confirmé et étendu cette observation en décrivant l’interface actine-myosine avec une résolution inférieure à 4 Å [16]. L’ensemble de ces structures a mis en évidence un changement majeur au cœur du moteur (en particulier, son feuillet β central) qui affecte de manière drastique les propriétés de liaison du nucléotide dans le site actif. Une conformation courbée du feuillet β central entraîne en effet la séparation d’éléments de liaison du nucléotide, appelés P-loop et Switch-I, qui font partie respectivement des sous-domaines N-term et U50 (Figure 4A). La liaison de l’ATP réoriente les sous-domaines du domaine moteur afin que tous les éléments du site actif puissent participer à la liaison de l’ATP, ce qui nécessite une conformation plus aplatie du feuillet β (Figure 4A). Ces structures ont mis en évidence le fait que la formation d’une interface de forte affinité pour l’actine nécessite la fermeture d’une fente interne du moteur, ce qui n’est pas compatible avec une liaison forte de l’ATP dans le site actif (détails passés en revue dans [14]). A partir d’une faible liaison initiale de l’ATP dans une structure acto-myosine (avec la fente interne fermée), des réarrangements dans le moteur favorisant une forte liaison à l’ATP peuplent les états dans lesquels la fente interne est ouverte, séparant les sous-domaines L50 et U50. Des parties de l’interface de liaison à l’actine sont ainsi séparées, ce qui entraîne une affinité plus faible pour la piste et le détachement du moteur.
Il est intéressant de noter qu’un moteur sans nucléotide peut être cristallisé dans des états structuraux de liaison d’affinité forte et faible pour l’actine [34, 37, 39]. Le moteur n’a pas besoin d’actine ou d’ATP pour adopter ces états. Cela suggère que le moteur explore plusieurs conformations et utilise la sélection conformationnelle pour cette transition, bien que l’ajustement induit par la détection locale de la présence d’ATP joue bien sûr un rôle pour déclencher le détachement efficace du moteur de la piste. La description correcte de l’énergie libre lors des transitions conformationnelles du moteur reste aujourd’hui un défi. L’une des transitions les plus rapides dans le cycle moteur est la réorientation du bras de levier alors que la myosine est liée à l’ATP et détachée du filament d’actine (c’est-à-dire lors du « recovery stroke »). Des études antérieures avaient conclu que la détection locale du γ-phosphate de l’ATP était l’élément déclencheur d’une série de réarrangements conformationnels initiés au niveau du site actif qui guiderait le mouvement du bras de levier [40]. Les structures récentes de la myosine liée à l’ATP et les calculs de dynamique moléculaire ne soutiennent pas ce scénario [41]. La vision actuelle propose que le moteur explore d’abord les états dans lesquels l’élément mécanique découplé du moteur amorce son basculement par l’exploration de conformations sans être réellement guidé. La stabilisation d’une conformation pré-powerstroke se produit finalement par la formation d’interactions au niveau du site actif à la fin de la transition [41]. L’association de simulations de dynamique moléculaire et d’informations structurales toujours plus nombreuses est essentielle pour révéler le mécanisme de communication entre les sites allostériques.
L’ensemble des structures actuellement disponibles indique que tous les moteurs myosine explorent des réarrangements similaires le long de leur cycle moteur (Figures 3 and 4B). Les structures CryoEM ont apporté quelques réponses concernant la dernière étape du cycle moteur qui contrôle la libération d’ADP [14, 17, 18, 42]. Une lacune majeure dans notre compréhension est la nature des interactions avec la piste au début de la production de force, et le rôle des interactions avec la piste pour diriger les réarrangements produisant la force. Bien qu’une structure de la Myo6 ait fourni des indices pour décrire comment l’actine favorise la libération de Pi [43], ces étapes critiques du cycle moteur restent mal caractérisées.
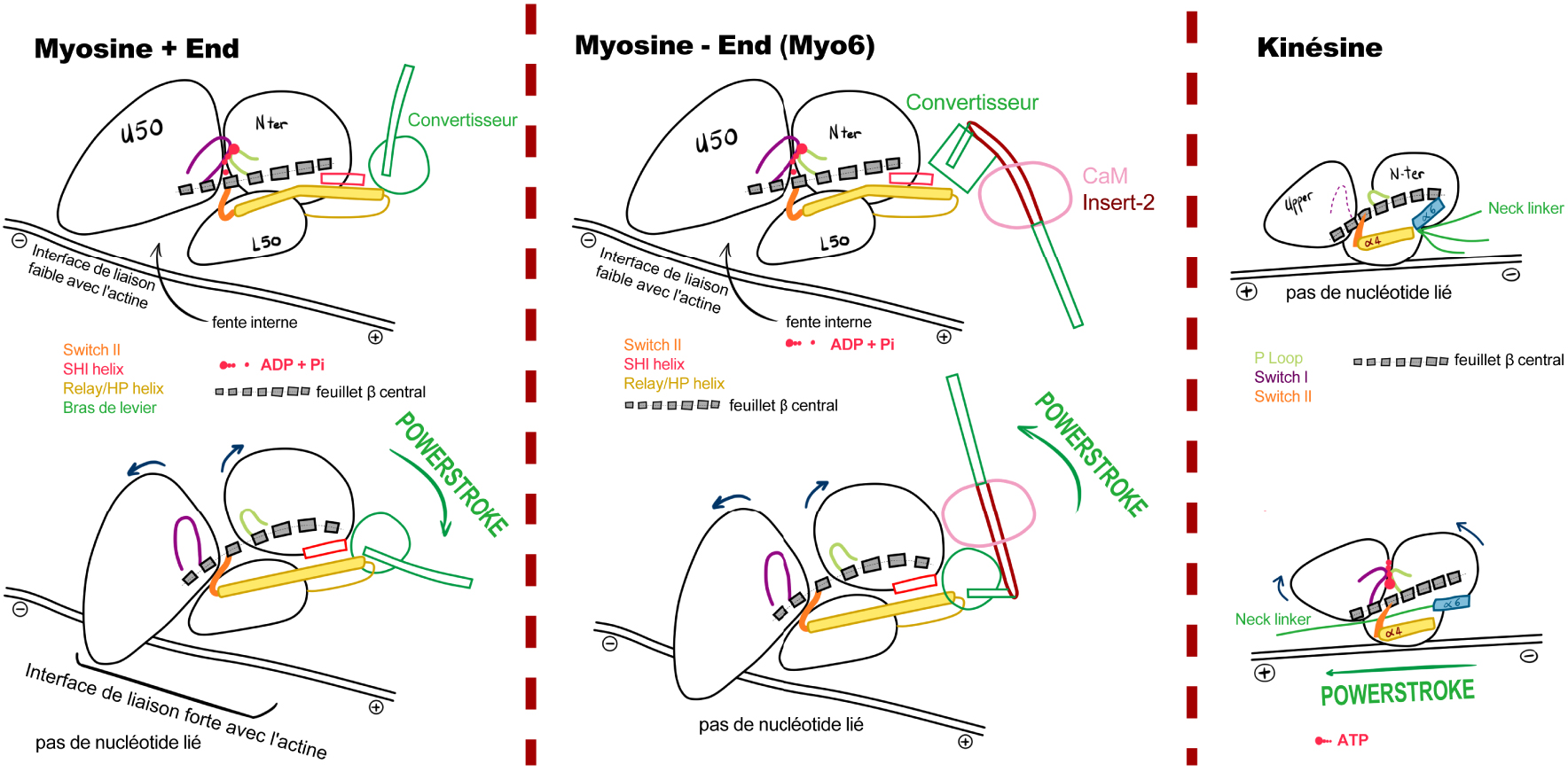
Conformations du domaine moteur dans les états pré- et post-powerstroke (avant et après production de force). Les états pré-powerstroke et post-powerstroke sont indiqués en haut et en bas et permettent d’apprécier les changements conformationnels au sein du moteur qui engendrent la production de force (powerstroke). Les petits mouvements dans le domaine moteur sont amplifiés et transmis à l’élément mécanique. Sa réorientation conduit à de grands mouvements dirigés. (A) et (B) Les réarrangements de la myosine nécessaires à la production de force permettent la fermeture de la fente interne (entre les domaines U50 et L50, colorés en bleu et gris clair dans les Figures 3 et 5) et modifient donc l’affinité pour la piste cytosquelettique, tandis que les éléments du site actif contrôlent la libération des produits de l’hydrolyse. (B) Dans la Myo6, la direction est inversée par des modifications de l’élément mécanique qui impliquent un changement de la conformation du convertisseur ainsi que la présence d’un insert spécifique de 40 résidus (marron) capable de recruter une calmoduline (rose). Ces variations de l’élément mécanique entraînent un pivotement efficace de 180° du bras de levier, dans la direction opposée de celle des myosines se dirigeant vers l’extrémité + des filaments d’actine. (C) Les kinésines subissent des réarrangements analogues à ceux des moteurs myosine, mais ces moteurs diffèrent dans la manière avec laquelle ces transitions conformationnelles contrôlent l’affinité de la piste cytosquelettique et l’orientation de l’élément mécanique. Masquer
Conformations du domaine moteur dans les états pré- et post-powerstroke (avant et après production de force). Les états pré-powerstroke et post-powerstroke sont indiqués en haut et en bas et permettent d’apprécier les changements conformationnels au sein du moteur qui engendrent ... Lire la suite
4. La production de force par les moteurs kinésine
De façon surprenante, compte tenu des différences de leur cycle moteur (Figure 2), les premières structures cristallines de la myosine [33] et de la kinésine ont révélé une similitude structurale frappante entre les deux moteurs, suggérant qu’ils pourraient partager un ancêtre commun [44]. En particulier, les moteurs possèdent des éléments similaires de liaison aux nucléotides et un feuillet β central, qui subissent des réarrangements internes analogues pour les deux moteurs en fonction de la présence ou l’absence de nucléotide dans leur site actif (Figure 4). Comme pour la myosine, on peut identifier dans le domaine moteur d’une kinésine trois sous-domaines qui changent d’orientation dans les états pré- et post-production de force. Cependant, les éléments de liaison à la piste diffèrent radicalement dans les deux moteurs, ainsi que le mécanisme qui commande l’orientation de l’élément mécanique. Dans la kinésine, on ne trouve pas de fente interne permettant de distinguer les interfaces de liaison faibles et fortes pour la piste. En fait, lorsqu’elle est fortement liée à la piste (MT), la kinésine interagit principalement par l’intermédiaire des éléments de son sous-domaine inférieur, avec une participation beaucoup plus faible des boucles des sous-domaines N-term et supérieur (Figure 4).
La kinésine est deux fois plus petite que la myosine. Les trois sites allostériques (site actif, site de liaison au cytosquelette et élément mécanique) sont beaucoup plus proches les uns des autres qu’ils ne le sont dans la myosine. Pour les kinésines, les éléments composant ces sites allostériques subissent des transitions d’ordre/désordre majeures qui sous-tendent la génération de force, car leur conformation est peu stabilisée par interaction avec d’autres éléments structuraux du moteur. Les études structurales de la kinésine Kin1 liée aux microtubules ou à la tubuline ont été particulièrement utiles pour comprendre comment la force motrice est produite pendant la liaison de l’ATP [45, 46]. Les interactions de la piste (MT) avec la kinésine commandent des régions de l’interface de liaison de l’ATP qui sont proches du site actif. Ces réarrangements conduisent globalement à la libération d’ADP en ouvrant le site de liaison au nucléotide. Lors de la liaison de l’ATP, le powerstroke se produit alors que la région centrale de l’interface de liaison au MT reste inchangée. Ainsi, la kinésine reste ancrée à la piste tandis que les réarrangements dans le moteur permettent la fixation de l’ATP. La fermeture du site actif de sorte que la P-loop et le Switch-I participent tous deux à la liaison de l’ATP nécessite l’aplatissement du feuillet β central, comme pour la myosine (Figure 4). Cela conduit à la rotation des sous-domaines Nter et Supérieur autour des éléments centraux de liaison au MT, (qui comprend l’hélice α4 et la boucle L12 du sous-domaine inférieur). Ces réarrangements modifient radicalement l’environnement de la dernière hélice du domaine moteur, l’hélice α6, qui précède l’élément mécanique. Dans les structures de pré-powerstroke (sans ATP), l’entrave stérique avec l’hélice α4 raccourcit l’hélice α6 et dirige l’élément mécanique dans une conformation désordonnée dirigée vers l’arrière (Figure 4). Lors de la liaison de l’ATP, l’encombrement stérique avec α4 est résolu en raison de la réorientation des sous-domaines. L’hélice α6 peut s’allonger tandis que l’élément mécanique (« neck linker ») s’arrime à la surface du domaine moteur et adopte une orientation stable vers l’avant permettant ainsi la production de force (Figure 4).
En résumé, alors que les réarrangements centraux autour du site actif sont similaires dans les deux moteurs, la force motrice dans les kinésines implique le contrôle de l’élément mécanique (« neck linker ») qui s’arrime à la surface du domaine moteur. Contrairement à la kinésine, l’équivalent de l’hélice α4 (l’hélice HP) n’est pas impliqué dans la liaison de la piste (F-actine) dans les myosines. Au lieu de cela, en tant que partie du relais, il participe avec un autre connecteur (l’hélice SH1) à l’entraînement de la rotation du bras de levier pendant le powerstroke (Figure 4). En utilisant des réarrangements analogues dépendants des nucléotides au cœur du domaine moteur, les kinésines et les myosines ont donc évolué de manière radicalement différente pour produire une force via le contrôle de leur élément mécanique lors de leur interaction avec la piste cytosquelettique.
5. Comment les moteurs sont réglés pour des fonctions cellulaires distinctes
Les propriétés de motilité des moteurs myosine sont extrêmement diverses bien qu’ils partagent un cycle mécano-chimique commun. Ils peuvent donc remplir des fonctions biologiques très différentes. Le réglage de cette motilité est le résultat de différences dans la thermodynamique et la cinétique des transitions du cycle moteur, qui sont elles-mêmes codées par des variations de séquence altérant l’allostérie au sein du moteur. Les modifications de séquence peuvent aider à la mise au point des propriétés du moteur, mais elles peuvent aussi altérer la fonction du moteur. Les études sur les moteurs divergents comme la myosine du parasite Plasmodium falciparum PfMyoA fournissent des illustrations intéressantes sur ce point. Un petit nombre de différences critiques par rapport à la séquence d’autres myosines entraînerait une perte de motilité si elles étaient introduites dans des myosines conventionnelles. Cependant, la myosine PfMyoA est capable de produire de la force grâce à la compensation de la séquence, en particulier via une extension N-terminale qui permet au moteur d’adopter les mêmes états structuraux que toutes les autres myosines [47, 48]. Il est intéressant de noter que la force produite par ce moteur PfMyoA peut être réglée par un seul site de phosphorylation [47], trouvé sur son extension N-terminale spécifique. Ce site de phosphorylation peut moduler sa vitesse et sa résistance à une force opposée en contrôlant la dernière étape du powerstroke. Ainsi, à différentes étapes du cycle de vie du parasite Plasmodium falciparum, la phosphorylation de ce même moteur lui permettrait de jouer des rôles très différents : ce moteur est crucial pour la motilité rapide des cellules du parasite (travail sous faible charge avec un moteur probablement phosphorylé) et pour l’invasion d’une cellule hôte par le parasite (travail sous forte charge avec un moteur probablement non phosphorylé). Cette myosine essentielle au parasite est donc considérée comme une cible privilégiée pour le traitement antipaludique.
Les règles encryptées dans la séquence à l’origine de la diversification des propriétés de motilité de ces moteurs sont actuellement non décodées. L’allostérie est en effet une propriété globale de la molécule et une seule mutation peut modifier la vitesse de plusieurs étapes du cycle précisément ajusté de ces nanomachines. Notre compréhension actuelle des transitions dans le cycle moteur est trop limitée pour expliquer et prédire comment la production de force est réglée. En outre, on en sait trop peu sur la façon avec laquelle la piste déclenche ces transitions, et comment le site d’interaction à la piste, variable d’un moteur à l’autre, pourrait contribuer à l’ajustement des propriétés du moteur [14, 49]. Enfin, les facteurs importants pour qu’un moteur résiste au détachement de la piste lorsqu’une force opposée est imposée sont aujourd’hui inconnus. En fin de compte, les propriétés de motilité sont régies par le rythme des transitions entre les états du cycle moteur, en particulier celles qui se produisent lorsque le moteur est lié à la piste. Rechercher comment la séquence du moteur encode la cinétique et la sensibilité à la force des transitions du powerstroke est donc un défi essentiel.
Les variations de l’élément mécanique d’un moteur à l’autre permettent également de modifier l’action de motilité effectuée par un moteur. L’inversion de la direction de la force produite et la capacité à organiser les pistes du cytosquelette sont deux exemples frappants de la manière avec laquelle l’élément mécanique peut conduire à de nouvelles propriétés de motilité. Les Figures 4A et 4B montrent que la principale différence entre la Myo6, la seule myosine se dirigeant vers l’extrémité négative (−) des filaments d’actine, et les autres myosines résulte d’un petit insert de 39 résidus unique à l’élément mécanique de Myo6. Cette séquence lie la calmoduline et fait partie du convertisseur. Contrairement aux autres myosines, le convertisseur subit des réarrangements internes lors de la production de force qui inversent efficacement la direction du bras de levier de 180° entre l’état avant et après production de force (Figure 4B). Des éléments mécaniques inhabituels peuvent également permettre à des moteurs dimériques d’atteindre plus d’une piste. Des études structurales du moteur Myo10 [54] et du moteur Kif20A ont ainsi permis de comprendre comment la longueur et l’angle d’oscillation de l’élément mécanique peuvent différer pendant le powerstroke de ces moteurs en comparaison à ceux des moteurs processifs tels que la myosine Myo5 et la kinésine Kin1. Ces adaptations permettent au moteur d’interagir avec des filaments différents, proches les uns des autres, sur lesquels une force peut être produite, permettant ainsi de les rassembler et de les organiser en réseau.
Il ne fait aucun doute que la visualisation des moteurs moléculaires à haute résolution est un moyen puissant pour comprendre comment la force peut les amener à effectuer des actions distinctes dans les cellules. D’autres énigmes sur la manière avec laquelle les moteurs acquièrent une fonction cellulaire particulière seront découvertes à l’avenir en étudiant les régions de la queue du moteur et la manière avec laquelle les partenaires influencent le regroupement des moteurs et leur capacité à interagir avec les pistes.
6. Myosines et maladies
L’allostérie est la clé de la production de force. Comme la force produite peut facilement être modifiée par une seule mutation dans ces machines allostériques conçues avec précision, plusieurs moteurs moléculaires ont été liés à des maladies humaines [55, 56]. Par exemple, un grand nombre de mutations dans les gènes codant pour Myo6, Myo7a ou Myo15a peuvent conduire à la surdité car ces moteurs sont essentiels pour l’intégrité des cellules ciliées de l’oreille interne [57]. Les mutations dans des régions distinctes d’un moteur peuvent soit entraîner une déficience totale ou partielle de la production de force, soit entraîner une mauvaise régulation de son activation dans les cellules. L’étude des propriétés de motilité de ces moteurs altérés et des altérations cellulaires qu’ils provoquent est un moyen intéressant d’en savoir plus sur la production de force et sur la précision avec laquelle elle doit être produite dans les cellules.
Les cardiomyopathies, qui peuvent entraîner une mort cardiaque subite, démontrent de manière remarquable comment une petite différence dans la force produite par un moteur peut influencer le destin des cellules. Les battements du cœur sont alimentés tout au long de la vie par un moteur myosine (la myosine cardiaque β) qui contracte les cardiomyocytes. Des mutations distinctes dans les protéines sarcomériques ont été identifiées pour provoquer des formes variées de cardiomyopathies, augmentant ou réduisant légèrement la contraction produite. Au fil du temps, les cellules cardiaques ressentent cette altération et réagissent en modifiant leur métabolisme [58]. Bien que les mécanismes conduisant à des modifications de l’organisation cellulaire puissent varier en fonction de chaque mutation, il devient évident que la mécanosensibilisation est un facteur important pour la progression de la maladie [59]. Des études marquantes ont permis de découvrir la possibilité de nouveaux traitements pour ces maladies en démontrant que de petites molécules agissant comme effecteurs allostériques peuvent moduler la force produite par les moteurs myosine. Trois candidats médicaments agissant soit comme inhibiteurs spécifiques soit comme activateurs de la myosine cardiaque sont en cours d’essais cliniques de phase 3 pour le traitement d’insuffisances cardiaques distinctes [60, 61, 62]. Les poches dans lesquelles plusieurs de ces effecteurs allostériques spécifiques se lient ont été identifiées dans la molécule de myosine (Figure 5). Les recherches actuelles visent à exploiter ces sites pour découvrir de nouvelles molécules à fort potentiel thérapeutique pour des maladies telles que les troubles spastiques de la marche [63] ou le paludisme [48, 56].
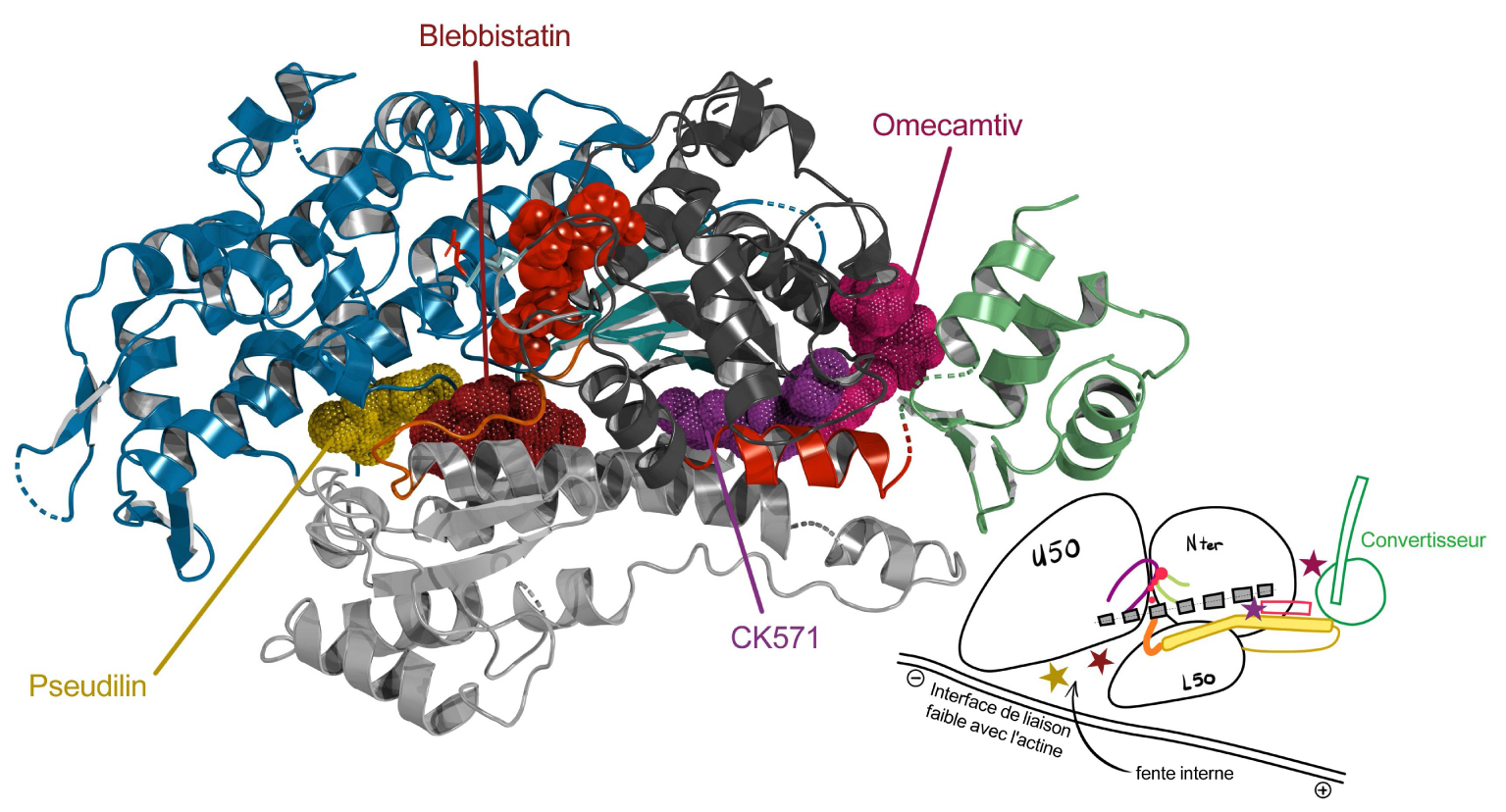
Sites de liaison de petites molécules effectrices dans le domaine moteur de la myosine. La structure du domaine moteur de la myosine est présentée sous forme de ruban à gauche avec la molécule ATP (sphères rouges) et en dessin simplifié à droite. Les sites de liaison de quatre petites molécules allostériques qui agissent comme inhibiteurs spécifiques (la blebbistatine [50]; la pseudiline [51], CK-571 [52]) ou comme activateur (Omecamtiv mecarbil, [53]) sont présentés sous forme de surfaces colorées (à gauche) ou d’étoiles colorées (schéma à droite). La molécule CK-571 (violet) se fixe dans une poche qui ne s’ouvre que pendant la transition de récupération [52]. Les autres molécules se lient dans des poches distinctes de l’état pré-powerstroke de la myosine. Masquer
Sites de liaison de petites molécules effectrices dans le domaine moteur de la myosine. La structure du domaine moteur de la myosine est présentée sous forme de ruban à gauche avec la molécule ATP (sphères rouges) et en dessin simplifié ... Lire la suite
7. Conclusion et perspectives
En agissant localement pour produire une force dirigée à des endroits précis dans les cellules, les moteurs biologiques sous-tendent des fonctions essentielles, organisant ou contractant les pistes du cytosquelette ou se déplaçant le long de pistes polarisées. Plusieurs de ces moteurs travaillent ensemble pour l’échange d’intermédiaires de transport membranaires entre les compartiments de la cellule : certains moteurs sont nécessaires pour la scission des tubes membranaires [64, 65], d’autres participent au transport à longue et courte distance et d’autres encore assistent les événements de fusion. D’autres encore coordonnent leur action pour les mouvements et les forces nécessaires à la distribution du matériel génétique dans une cellule en division. Les forces créées par ces nanomoteurs sont transmises efficacement pour contrôler les grands réarrangements cellulaires dans des processus tels que la division et la migration cellulaires. L’intégration de la détection et de la signalisation mécanistique est également essentielle pour construire des structures hautement organisées (protubérances des neurones, filopodes, microvillosités, stéréociles) qui définissent la fonction des cellules et contrôlent leur destin. Il est donc indispensable de comprendre comment les forces produites dans les cellules contrôlent les processus cellulaires et une grande partie de cette compréhension peut être obtenue en découvrant comment ces nanomachines fonctionnent pour maintenir les cellules en ordre et en bon état de fonctionnement.
Les connaissances actuelles des études cellulaires se limitent souvent à l’identification des processus cellulaires qui sont altérés lorsque la cellule entière est dépourvue d’un moteur moléculaire particulier et à la caractérisation d’un partenaire possible sur un compartiment. Il n’est pas facile d’aller plus loin pour définir l’action mécanique spécifique d’un nanomoteur dans les cellules. Les détails atomiques fournis par la biologie structurale apportent des informations capitales pour révéler comment les moteurs moléculaires remplissent des fonctions cellulaires distinctes. Cela peut être la source d’inspiration pour la conception de moteurs chimériques ou mutés précisément pour, en fin de compte, identifier comment les moteurs sont recrutés et de quelle action mécanique ils sont responsables dans un contexte cellulaire particulier. La modulation à volonté de la force produite dans la cellule par la découverte d’effecteurs allostériques spécifiques contre des moteurs distincts fera également progresser considérablement cette recherche. Il manque des structures à haute résolution de ces moteurs liés à leur piste pour dépeindre pleinement la production de force et comment un moteur résiste à une charge opposée. Répondre à ces questions fondamentales concernant de telles enzymes allostériques apportera des indices essentiels pour comprendre leur rôle précis dans la cellule et comment une mutation dans le gène d’un moteur peut engendrer une maladie. La découverte de nouveaux effecteurs allostériques est d’un intérêt majeur car ils peuvent apporter de nouvelles connaissances sur les transitions qui contrôlent la génération de force. Ces petites molécules permettent également d’envisager des approches thérapeutiques émergentes et prometteuses pour des maladies mortelles et peuvent donc améliorer considérablement la santé humaine à l’avenir.




 CC-BY 4.0
CC-BY 4.0