1. Régénération dans le SNC : un besoin non satisfait
Chez les mammifères adultes, les neurones du système nerveux central (SNC) ne régénèrent pas spontanément leurs axones, contrairement à ceux du système nerveux périphérique (SNP). Cela a des conséquences majeures pour les patients atteints de troubles neurologiques, qu’ils soient chroniques (maladies neurodégénératives) ou aigus (lésions traumatiques du cerveau ou de la moelle épinière), car cela entraîne une perte permanente et irréversible des fonctions motrices, sensorielles et/ou cognitives. À ce jour, aucun traitement n’est disponible, ce qui fait de la réparation du SNC l’un des plus grands défis de la recherche en neurosciences.
Au niveau cellulaire, lorsqu’un axone du SNC est lésé, sa partie distale dégénère tandis que la partie proximale forme un bulbe de rétraction, une structure incapable de soutenir la croissance de l’axone. Parmi les raisons de cette absence de régénération, l’environnement extracellulaire devient inhibiteur de la croissance après lésion, en raison de l’expression de facteurs répulsifs tels que les molécules associées à la myéline ou les composants de la bordure gliale [1, 2, 3]. Cependant, la modulation de ces facteurs inhibiteurs n’est pas suffisante pour favoriser la régénération [4], c’est pourquoi les chercheurs se sont intéressés aux propriétés intrinsèques de croissance des neurones eux-mêmes. En effet, au cours du développement, les neurones du SNC perdent progressivement la capacité à développer un axone [5]. De plus, la lésion elle-même altère davantage les propriétés de repousse des neurones adultes du SNC [6], ce qui coïncide avec la diminution intrinsèque des voies moléculaires de croissance. Au cours des 15 dernières années, la repousse axonale des neurones du SNC blessés a été réalisée par activation expérimentale ou co-activation de ces voies, par exemple mTOR (cible mécaniste de la rapamycine) [7], JAK/STAT [8], Klf (Krüppel-like factors) [9] ou c-myc [6].
Plusieurs de ces études pionnières sont basées sur le système visuel de la souris adulte, qui fait partie du SNC. Il est composé des yeux, des nerfs optiques et des différents noyaux cérébraux qui intègrent et traitent les informations visuelles. La lésion de l’axone est obtenue mécaniquement par pincement du nerf optique. Cette lésion affecte principalement les cellules ganglionnaires de la rétine (CGR), qui sont les seuls neurones de la rétine qui projettent leur axone pour former le nerf optique et connecter l’œil au cerveau (Figure 1).
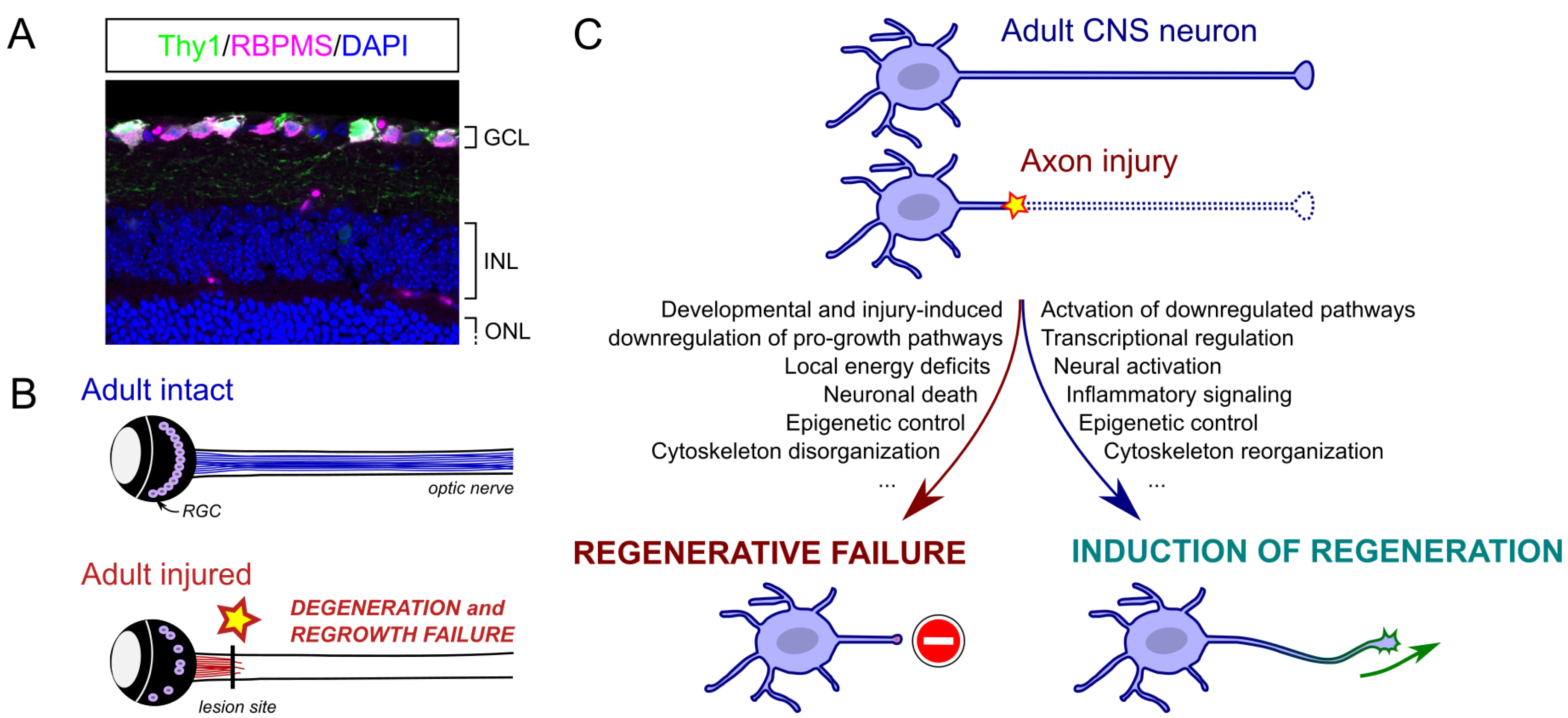
(A) Coupe d’une rétine adulte montrant le marquage des CGR avec expression spécifique de la GFP sous le promoteur Thy1 et avec l’anticorps anti-RBPMS. GCL : couche des cellules ganglionnaires ; INL : couche nucléaire interne ; ONL : couche nucléaire externe. Image tirée de [10]. (B) Schéma du modèle de lésion du nerf optique. Dans des conditions normales, les CGR projettent leur axone vers le cerveau via le nerf optique. Après une lésion du nerf optique, les axones dégénèrent de manière distale et ne parviennent pas à repousser. (C) Résumé des mécanismes moléculaires régulant la réponse à la lésion neuronale et les propriétés intrinsèques de régénération des neurones du SNC adulte. Masquer
(A) Coupe d’une rétine adulte montrant le marquage des CGR avec expression spécifique de la GFP sous le promoteur Thy1 et avec l’anticorps anti-RBPMS. GCL : couche des cellules ganglionnaires ; INL : couche nucléaire interne ; ONL : couche nucléaire externe. Image tirée de [Lire la suite
Cependant, malgré les progrès considérables réalisés dans ce domaine, de nombreuses limites subsistent. Premièrement, tous les neurones ne réagissent pas à ces manipulations expérimentales, en partie à cause de l’hétérogénéité des sous-populations neuronales. Dans la rétine adulte, plus de 30 sous-populations de CGR ont été caractérisées sur la base de la morphologie, des marqueurs moléculaires, de l’arborisation dendritique, des projections primaires [11] et, plus récemment, des programmes transcriptionnels qui conditionnent la régénération et la survie des CGR après une lésion [12, 13]. Cependant, la modulation de ces programmes transcriptionnels n’est pas suffisante pour promouvoir une régénération robuste, car la façon dont ils sont intégrés dans les différentes sous-populations n’est pas encore bien comprise. Même les modèles de régénération les plus robustes ne permettent pas à plus de 20 % des CGR de repousser, ce qui pourrait compliquer le passage à l’étape suivante : la reconnexion fonctionnelle.
Deuxièmement, les CGR en cours de régénération ne parviennent pas à atteindre leurs cibles initiales et aucun circuit fonctionnel n’a donc encore été obtenu [14, 6, 15]. Ceci est dû à la repousse désorganisée des axones en régénération, qui ne reprennent pas leur trajectoire initiale et ne peuvent pas réinnerver la bonne cible cérébrale [16, 17]. À cet égard, nos travaux récents ont permis de démontrer la possibilité et la pertinence de la modulation du guidage des axones pendant la régénération, afin de réorienter les axones qui repoussent sur le bon chemin [18] et dans la bonne cible cérébrale [19].
Troisièmement, les voies moléculaires qui ont été identifiées jusqu’à présent comme favorisant la repousse chez l’adulte ont potentiellement des effets secondaires : en effet, la plupart de ces voies, telles que mTOR, JAK/STAT et c-myc, activent plusieurs processus cellulaires et moléculaires. Cela explique comment la repousse des axones peut être obtenue dans le SNC adulte, mais cela explique également le potentiel oncogène de ces manipulations. Par conséquent, les stratégies expérimentales visant à promouvoir la régénération des axones sont des preuves de concept solides, mais leur application en tant que thérapie doit être envisagée avec prudence. C’est le cas du suppresseur de tumeur Pten, dont l’inhibition dans des neurones spécifiques chez la souris ne semble pas avoir d’effets néfastes à long terme [20], mais dont le large spectre de cibles peut en réalité entraîner de profonds changements cellulaires, et dont la manipulation doit être envisagée à de multiples niveaux (expression génique, dosage fonctionnel, activité de la phosphatase, modifications post-traductionnelles, etc.) [21, 22].
Néanmoins, les stratégies actuelles restent un paradigme expérimental précieux pour élucider les mécanismes moléculaires qui contrôlent la capacité de repousse du SNC. L’élucidation de ces mécanismes est fondamentale pour déclencher et soutenir la repousse des axones et, en fin de compte, pour aborder toutes les étapes ultérieures de la réparation des circuits qui commencent tout juste à être étudiées, par exemple le guidage des axones dans le cerveau mature, comme dans nos récentes études de preuve de concept [18, 23, 19]. L’étude de ces modèles permettra donc de comprendre comment ces voies fonctionnent pour favoriser la repousse des axones et la survie des neurones (ces deux processus pouvant être indépendants [24, 25, 26]), et d’affiner les cibles en aval qui sont nécessaires et suffisantes pour la régénération. Il est donc essentiel de découvrir d’autres mécanismes moléculaires contrôlant la régénération des axones afin de parvenir à une formation complète des circuits et à une récupération fonctionnelle.
2. La régulation traductionnelle en jeu pour contrôler l’expression des gènes
Jusqu’à présent, la plupart des études se sont concentrées sur la régulation transcriptionnelle pour débloquer les propriétés régénératives du SNC adulte. Dans le système visuel adulte, des programmes transcriptionnels de croissance ont été mis en évidence grâce au criblage à haut débit, via les puces à ARN [27] et le séquençage de l’ARN [26], et plus récemment via l’analyse unicellulaire des CGR (single-cell RNA-sequencing) [28, 12, 13]. Un autre niveau de régulation des gènes réside dans les mécanismes épigénétiques en jeu pour contrôler l’expression des gènes associés à la régénération (regeneration-associated genes, RAG). En effet, les programmes transcriptionnels régénératifs sont régis par une modification de l’accessibilité de la chromatine des régions régulatrices des RAG en réponse à une lésion, par exemple via la régulation de l’état d’acétylation des histones [29].
L’analyse transcriptomique permet d’identifier différents types de cellules en fonction de l’expression détaillée de l’ARNm. C’est le cas dans la rétine, où des analyses de single-cell RNA-sequencing ont permis de discriminer plusieurs dizaines de neurones, au cours du développement et tout au long de l’âge adulte [30, 31, 32, 33]. Pourtant, dans de nombreux cas, la caractérisation du transcriptome ne rend pas compte des propriétés fonctionnelles ou phénotypiques de sous-populations distinctes – et inversement, des sous-populations indiscernables peuvent en réalité arborer des transcriptomes distincts [34]. Ce phénomène est encore plus complexe dans le cas de situations pathologiques ou traumatiques. En ce qui concerne les lésions axonales et la régénération, la modulation des programmes transcriptionnels a été insuffisante pour récapituler la repousse axonale et la reconnexion du circuit neuronal. Les approches transcriptomiques sont donc insuffisantes pour prédire les principaux processus de régulation des gènes dans les neurones adultes lésés.
Ce phénomène est encore plus complexe dans le cas de situations pathologiques ou traumatiques. En ce qui concerne les lésions et la régénération des axones, la modulation des programmes transcriptionnels s’est avérée insuffisante pour récapituler la repousse des axones et la reconnexion des circuits neuronaux. En revanche, le processus de traduction de l’ARNm en protéines – l’élément fonctionnel de l’expression des gènes dans les cellules – a été négligé dans le contexte des lésions du SNC et de la régénération des axones. Alors que l’activation des voies mTOR [7, 35] et c-myc [36, 6] améliore la synthèse des protéines à un niveau global – directement ou indirectement – leur contribution exacte au contrôle de la traduction reste inconnue dans ce contexte.
En fait, il est maintenant bien décrit que les niveaux d’ARNm et de protéines ne sont que partiellement corrélés dans les cellules [37, 38, 39, 40] et, dans de nombreux cas, la caractérisation transcriptomique d’une cellule ne reflète pas l’expression réelle des protéines à un stade donné. Alors que la traduction a longtemps été décrite comme un processus invariable, il est apparu récemment que différents ARNm ne sont pas traduits de manière uniforme en protéines. Les mécanismes de régulation de la traduction comprennent l’activité différentielle des facteurs associés à la traduction [41], la rétention ou la dégradation sélective des ARNm [41], et, comme mis en évidence plus récemment, des variations dans la composition du complexe traductionnel lui-même [42].
3. Preuves de l’existence d’un ribosome spécialisé : le complexe traductionnel
Dans les cellules eucaryotes, le complexe traductionnel se compose principalement du ribosome, un complexe macromoléculaire formé d’environ 80 protéines ribosomiques et de 4 ARN ribosomiques (ARNr), qui s’assemblent au cours d’un processus strictement ordonné appelé ribogénèse. L’extension de la chaîne polypeptidique s’opère par la lecture d’un ARNm donné par le ribosome, qui traduit les séquences de codons en acides aminés à l’aide des ARN de transfert. Ce processus implique également des facteurs associés à la traduction qui contrôlent les étapes d’initiation, d’élongation et de terminaison [43] (Figure 2).
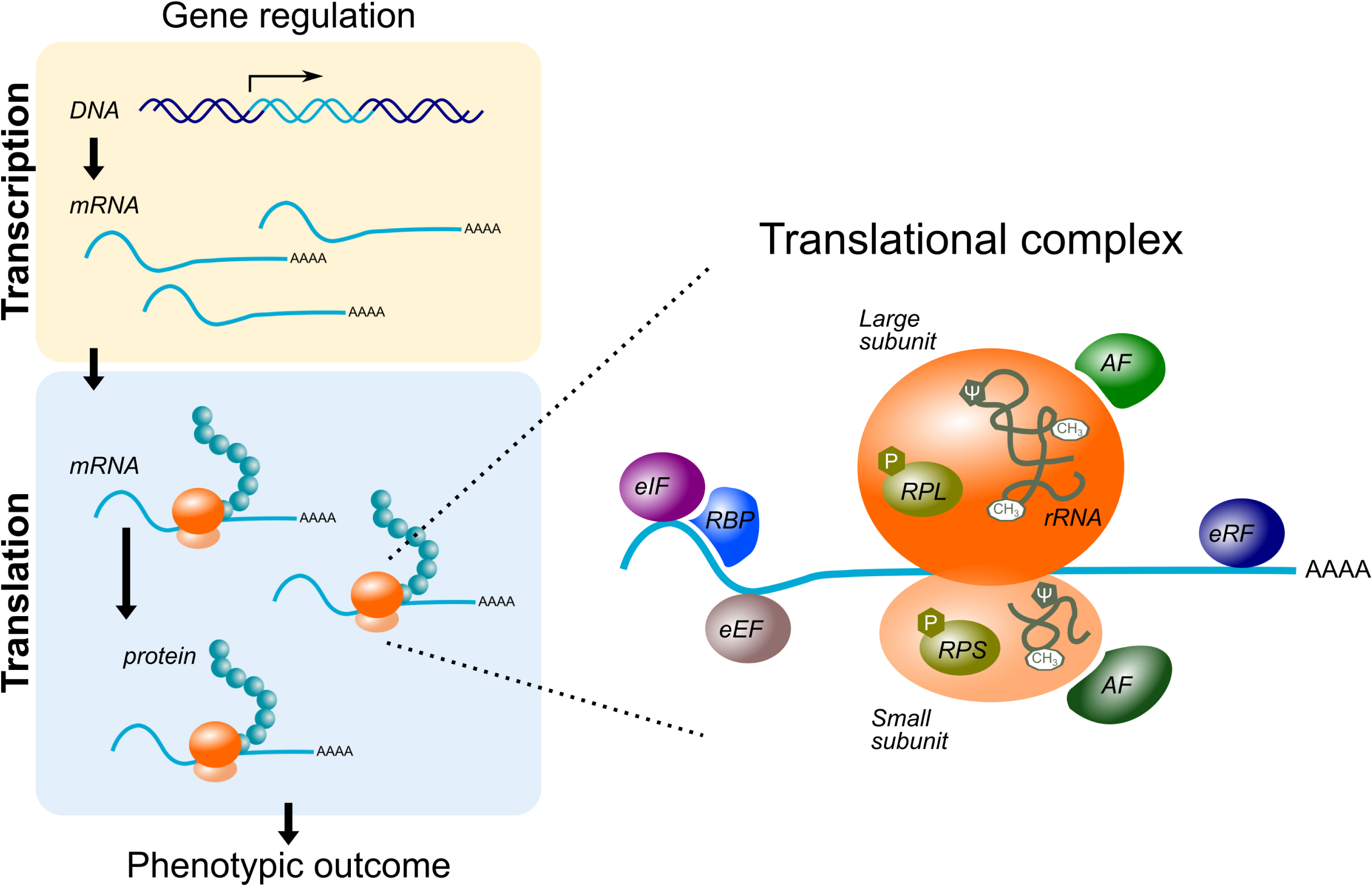
Représentation schématique du contrôle de l’expression des gènes dans les cellules : le flux d’information génétique va de l’ADN à l’ARNm (transcription), puis de l’ARNm à la protéine (traduction). Longtemps considérée comme un processus passif, la traduction apparaît désormais comme un régulateur actif dans le contrôle de l’expression des gènes. En particulier, dans des contextes pathologiques, mais aussi pour la spécification cellulaire, la fonction et la réponse induite par un stimulus externe (résultat phénotypique), le complexe traductionnel présente des variations dans sa composition. Ces variations sont associées à une spécialisation de sa fonction traductionnelle. eIF, eEF et eRF : facteurs eucaryotes d’initiation, d’élongation et de libération ; RBP : protéine liée à l’ARN ; RPL et RPS : protéines ribosomales de la grande et de la petite sous-unité ; rRNA : ARN ribosomal ; CH3 : méthylation ; ψ : pseudourydylation. Masquer
Représentation schématique du contrôle de l’expression des gènes dans les cellules : le flux d’information génétique va de l’ADN à l’ARNm (transcription), puis de l’ARNm à la protéine (traduction). Longtemps considérée comme un processus passif, la traduction apparaît désormais comme un ... Lire la suite
Longtemps considéré comme invariable dans sa composition, le complexe de traduction présente en fait des variations dans ses composants et sa composition et, par leur intermédiaire, régule activement le processus de traduction dans les cellules. Une illustration fondamentale réside dans les centaines de sites de modification de la 2’-O méthylation et de la pseudourydylation dans l’ARN ribosomal. Les travaux de Diaz et de ses collègues ont mis en lumière la façon dont le dérèglement de ces modifications contribue à la tumorigenèse en altérant la fidélité de la traduction et la traduction spécifique des ARNm [44, 45, 46]. Une autre illustration réside dans les modifications des protéines ribosomales (ribosomal protein, RP) : soit les modifications post-traductionnelles des RP, soit la stœchiométrie des RP dans les ribosomes. Ces observations sont issues de l’étude de ce que l’on appelle les « ribosomopathies », où des mutations somatiques ponctuelles affectent une RP ou un facteur de ribogenèse. Cela conduit à des altérations de l’homéostasie des ribosomes et/ou à une régulation altérée de la traduction, et entraîne le développement de pathologies, par exemple l’anémie de Blackfan-Diamond et les cancers [47].
En conditions physiologiques, des études transcriptomiques ont mis en évidence l’existence de RP paralogues qui confèrent une spécificité tissulaire et compensent leurs homologues canoniques. Les exemples incluent la protéine ribosomale L3-like (Rpl3l), qui est spécifique au muscle squelettique et contrôle la formation des myotubes [48] ; et la protéine ribosomale de type L10 (Rpl10l), qui est spécifique au testicule et contrôle la transition méiotique masculine [49]. On pense que ces paralogues remplacent leurs homologues canoniques dans le ribosome, confirmant en outre que la composition du complexe traductionnel lui-même contrôle le paysage traductionnel dans la spécification, le développement et la fonction des tissus.
Ces études ont ouvert la voie au concept de « spécialisation des ribosomes », un nouveau domaine de recherche très actif. Récemment, l’hétérogénéité des RP dans la composition des ribosomes purifiés a été mise en évidence à l’aide de la protéomique quantitative et a été reliée à une fonction spécialisée de régulateur de la traduction [50]. L’analyse protéomique de divers tissus et organes de souris adultes confirme également cette hétérogénéité inter-tissus au niveau des RP incorporés dans les ribosomes [51, 52, 53]. Il est intéressant de noter que, dans les neurones, plusieurs études indiquent un remodelage in situ des ribosomes par intégration de RP nouvellement synthétisés localement dans les compartiments distaux du neurone [54, 55]. Cela suggère un niveau supplémentaire de contrôle de la composition du complexe traductionnel dans les compartiments subcellulaires.
Enfin, un niveau supplémentaire d’hétérogénéité provient des facteurs associés à la traduction. Alors que les facteurs de traduction canoniques sont connus pour réguler le processus de traduction à un niveau global [41], des études récentes indiquent qu’ils jouent un rôle dans la traduction sélective (spécifique à l’ARNm). Par exemple, dans la structuration de l’embryon en fonction du gradient de Shh, le facteur d’initiation Eif3c contrôle la signalisation de Shh par la traduction sélective de l’ARNm Ptch1, un régulateur négatif de Shh [56].
En outre, dans différents contextes, il a été démontré que certains facteurs de traduction non canoniques (i) interagissent avec le ribosome ; et (ii) modifient son activité traductionnelle, au niveau global, mais aussi de manière sélective (c’est-à-dire pour des ARNm spécifiques). Parmi les exemples, citons FMRP, qui réprime la traduction d’ARNm cibles liés à la transmission synaptique [57], et Rack1, qui augmente la traduction d’ARNm spécifiques liés à l’autophagie [58]. Ces observations font de ces facteurs associés des composants à part entière du complexe traductionnel qui contribuent à son hétérogénéité et à sa spécialisation dans la fonction cellulaire. Récemment, Simsek et ses collègues ont dévoilé un « ribo-interactome » exhaustif des cellules souches embryonnaires de souris. Ils ont identifié plus de 300 protéines associées au ribosome susceptibles d’« accessoiriser » le ribosome, et donc en jeu pour contrôler l’expression des gènes dans des conditions physiologiques ou pathologiques spécifiques [59].
4. Régulation traductionnelle dans la régénération des axones du SNC
Nos travaux récents ont mis en lumière la régulation de la traduction en tant que processus essentiel de la régénération des axones du SNC : nous avons décrypté ce processus depuis les mécanismes moléculaires jusqu’aux implications in vivo pour la régénération des axones. En utilisant le modèle de référence de lésion du nerf optique, nous avons démontré que la repousse des axones des CGR adultes est contrôlée par la traduction sélective d’un groupe spécifique d’ARNm essentiels à la régénération de l’axone – mais pas par le niveau de traduction globale dans la cellule [60]. Ce mécanisme est régulé par l’association différentielle de facteurs spécifiques aux ribosomes en réponse à une situation pathologique, en l’occurrence la lésion des axones des CGR. À son tour, cela module le paysage traductionnel associé à la régénération des CGR, indépendamment de la régulation transcriptionnelle [60]. Cette étude s’appuie sur l’exemple spécifique de la protéine Huntingtin (HTT), identifiée comme une protéine clef de la réponse aux lésions neuronales [6, 61],
Ceci est assez inattendu puisque HTT est mieux connue pour son rôle neurodégénératif dans la maladie de Huntington lorsque le gène htt est affecté par une mutation d’expansion du CAG. En revanche, les rôles physiologiques de HTT restent mal connus.
Dans la rétine adulte, l’expression de HTT diminue dans les CGR après lésion du nerf optique. Cette diminution est corrélée avec la diminution du potentiel de régénération des CGR adultes lésés, ce qui renforce l’idée d’un rôle pro-régénérateur de HTT. Pour tester cette hypothèse, nous avons adopté une stratégie consistant à supprimer l’expression de HTT spécifiquement dans les CGR et à analyser son impact sur la régénération. Comme les CGR de type sauvage ne se régénèrent pas, nous avons supprimé HTT dans un contexte où la régénération des axones a été améliorée. À cette fin, nous avons utilisé l’activation de mTOR, par suppression de l’inhibiteur de mTOR Pten, dans les CGR. De manière très claire, la suppression de HTT dans les CGR adultes interrompt complètement la régénération des axones induite par l’activation de mTOR, ce qui signifie que HTT est essentielle à la régénération des axones adultes après une lésion (Figure 3A).
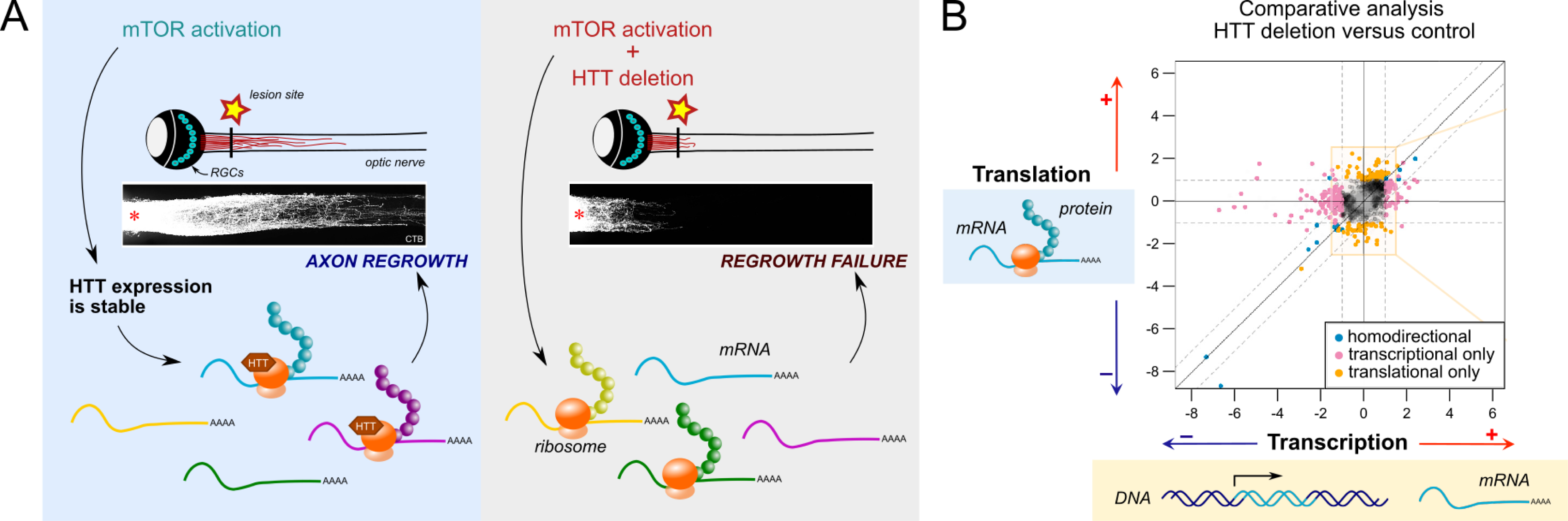
(A) La régulation translationnelle est en jeu pour contrôler la régénération des axones du SNC. Dans des conditions de régénération (activation de mTOR), l’expression de HTT reste stable après une lésion. HTT interagit avec les ribosomes et régule la traduction d’ARNm spécifiques associés à la régénération. Lorsque HTT est supprimée, le complexe traductionnel n’est plus accessoirisé avec HTT. Cela modifie l’identité des ARNm recrutés pour la traduction et fait basculer les neurones dans une incapacité à régénérer. (B) Analyse comparative du transcriptome et du translatome dans la condition de délétion de HTT par rapport à la condition contrôle. Chaque point représente un ARNm. La coordonnée x est la variation de l’abondance du transcrit (plus ou moins transcrit lorsque HTT est supprimée). La coordonnée y est sa variation dans l’association avec les ribosomes (plus ou moins traduit lorsque HTT est supprimé). Les points orange représentent les ARNm qui sont sélectivement régulés au niveau de la traduction, sans variation de leur régulation transcriptionnelle. Parmi ces ARNm se trouve Tox2, une cible traductionnelle pro-régénératrice de HTT. Données tirées de [60]. Masquer
(A) La régulation translationnelle est en jeu pour contrôler la régénération des axones du SNC. Dans des conditions de régénération (activation de mTOR), l’expression de HTT reste stable après une lésion. HTT interagit avec les ribosomes et régule la traduction ... Lire la suite
Sur la base d’une analyse non biaisée des interactants de HTT, nous avons montré que HTT interagit fortement avec les ribosomes, ce qui suggère un rôle pour HTT dans le processus de traduction. En évaluant la synthèse de protéines in vitro et in vivo, nous avons montré que HTT n’est pas nécessaire à la traduction globale. En revanche, des analyses combinées du transcriptome et du translatome ont montré qu’en l’absence de HTT, un sous-ensemble d’ARNm est traduit de manière différentielle, alors que leur niveau de transcription reste inchangé. Nous nous sommes concentrés sur l’une de ces cibles traductionnelles sélectives de HTT, Tox2, pour démontrer que (i) HTT contrôle la traduction de Tox2 dans les neurones ; et (ii) la traduction de l’ARNm de Tox2 en protéine est nécessaire à la régénération des axones. Cette étude élucide, dans le contexte d’une lésion du SNC chez l’adulte, la manière dont l’association de facteurs spécifiques tels que HTT aux ribosomes contrôle la repousse axonale via la régulation traductionnelle d’un sous-ensemble d’ARNm clefs pour la régénération [60] (Figure 3A–B).
Des preuves supplémentaires ont été récemment apportées concernant le rôle actif du complexe traductionnel dans la régulation du paysage traductionnel au cours de la régénération des axones. Premièrement, nos travaux et ceux d’autres chercheurs ont mis en évidence le rôle essentiel des kinases de la famille RSK dans la phosphorylation du facteur d’élongation eEF2 et de la protéine ribosomale RPS6 [62, 63]. Ces études démontrent que l’état de phosphorylation médié par les RSK est déterminant pour la régénération des axones en contrôlant le paysage traductionnel d’ARNm spécifiques. Deuxièmement, la manipulation d’une RP spécifique peut également déclencher la régénération des axones des CGR dans le nerf optique lésé, comme cela a été montré pour RPL7 et son isoforme RPL7a, dont les transcrits sont régulés à la baisse au cours du développement [64]. Si ce dernier résultat soutient le rôle de la spécialisation du complexe traductionnel dans la réponse régénérative du SNC, il reste à déterminer (i) si les RP expérimentalement surexprimées s’intègrent dans des ribosomes fonctionnels ; et (ii) si cela confère un rôle actif de régulation de la traduction au complexe traductionnel.
5. Perspectives
Bien que longtemps [IV1] étudiée sous l’angle transcriptomique et épigénétique, la capacité intrinsèque de régénération du SNC dépend d’un niveau supplémentaire de contrôle de l’expression des gènes : le contrôle de la traduction par la régulation du complexe traductionnel. Un tel mécanisme a été illustré par nos travaux récents montrant que HTT accessoirise le ribosome pour réguler la traduction sélective d’ARNm associés à la régénération dans les neurones lésés du SNC [60].On comprend de mieux en mieux que la transcriptomique ne peut à elle seule prédire l’expression des protéines, non seulement en raison du rôle régulateur actif du complexe de traduction, mais aussi des mécanismes d’accumulation et de dégradation des ARNm individuels [41]. Dans ce contexte, le profilage protéomique devrait apporter des informations essentielles pour donner une image complète de la régulation de l’expression des gènes, en particulier de l’intégration de la transcription, la traduction et la protéostase dans le contrôle d’un phénotype (ici : la réponse des neurones du SNC à une lésion et leur capacité à régénérer leur axone). Toutefois, la caractérisation des profils d’expression des protéines dans une population neuronale unique, et a fortiori dans des cellules uniques, reste un défi technique limité par la sensibilité actuelle de l’analyse protéomique basée sur la spectrométrie de masse, car les protéines ne peuvent pas être amplifiées comme les acides nucléiques. Malgré tout, certaines études récentes ont pu détecter des milliers de protéines dans une seule cellule et coupler les données transcriptomiques et protéomiques [65], grâce à des avancées majeures dans les technologies de protéomique « shotgun » telles que SCoPE-MS [66] et PiSPA [67]. Un tel profilage couplé fournit des informations très précieuses sur la régulation de l’expression des gènes à plusieurs niveaux. Cela peut également s’appliquer aux compartiments subcellulaires, comme cela a été fait récemment dans les cônes de croissance des neurones au cours du développement [68]. Aujourd’hui, l’analyse du translatome et le profilage des « footprints ribosomiques » (séquençage profond de fragments protégés par le ribosome) [69] offrent un moyen relativement accessible de faire le lien entre les niveaux de transcription et de synthèse des protéines, tandis que les efforts se poursuivront pour améliorer la sensibilité du profilage protéomique.
Nos résultats vont dans le sens d’un rôle de régulation de la traduction pour HTT. En effet, dans la maladie de Huntington, on a constaté que la mutation de HTT modifiait le processus de traduction, en altérant l’activité de facteurs de traduction tels que 4E-BP [70] ou en influençant l’arrêt des ribosomes au cours de la traduction [71]. Il est intéressant de noter que nous avons trouvé un rôle dans la traduction sélective, alors que les études sur HTT mutante montrent des altérations de la traduction globale. Des recherches supplémentaires seront donc nécessaires pour déterminer (i) quel sous-domaine de HTT interagit avec le ribosome (et en particulier s’il s’agit du domaine N-terminal où se trouve l’expansion polyglutaminique mutante) ; et (ii) si la mutation de HTT affecte son rôle traductionnel directement ou indirectement.
La question de savoir si ce mécanisme de contrôle de la traduction s’applique à d’autres sous-types de neurones reste ouverte. À l’appui de cette hypothèse, nous avons constaté que HTT est nécessaire à la régénération des axones des neurones des ganglions rachidiens dans le SNP après une lésion du nerf sciatique [60], et les travaux d’autres chercheurs ont mis en évidence que HTT est une protéine clef de la régénération des motoneurones du tractus corticospinal induite par la greffe de cellules progénitrices neurales dans la moelle épinière lésée [61]. Fait important, nos travaux ont montré une correspondance dans le contrôle du paysage traductionnel contrôlé par HTT entre les fibroblastes embryonnaires de souris et les CGR lésés [60], ce qui suggère un mécanisme partagé entre de nombreux types cellulaires et stades de développement. En fait, HTT est exprimée de manière ubiquitaire dans plusieurs régions du cerveau [72], et plus généralement dans de nombreux tissus et à différents stades de développement [73]. De plus, les études sur HTT dans plusieurs organismes modèles plaident pour une conservation des mécanismes de sa pathogénicité (induite par le domaine d’expansion polyglutamine muté) et de ses fonctions physiologiques, bien que celles-ci ne soient encore que très partiellement comprises [74, 73, 75]. Il est donc possible que le rôle de HTT en tant que facteur associé à la traduction soit un processus général en biologie cellulaire, mais nous ne pouvons pas exclure que les pools d’ARNm régulés traductionnellement soient en fait spécifiques d’un type cellulaire, d’un stade de développement ou d’un état physiopathologique donné, ce qui méritera d’être étudié à l’avenir.
Qu’est-ce qui contrôle la sélectivité du complexe de traduction pour traduire des ARNm spécifiques ? Certains éléments cis-régulateurs identifiés dans la séquence et/ou la structure des transcrits expliquent cette sélectivité. Il s’agit notamment des sites d’entrée des ribosomes internes (IRES) dans la région 5' non traduite (UTR), des cadres de lecture ouverts en amont (uORF), de l’oligopyrimidine terminale 5' (TOP 5') et des séquences de liaison aux micro-ARN
Qu’est-ce qui contrôle la sélectivité du complexe de traduction pour traduire des ARNm spécifiques ? Certains éléments cis-régulateurs identifiés dans la séquence et/ou la structure des transcrits expliquent cette sélectivité. Il s’agit notamment des sites d’entrée des ribosomes internes (IRES) dans la région 5' non traduite (UTR), des cadres de lecture ouverts en amont (uORF), de l’oligopyrimidine terminale 5' (TOP 5') et des séquences de liaison aux micro-ARN [76, 77, 78, 79]. Dans la régénération du SNC, il reste à déterminer si les cibles traductionnelles de HTT contiennent de tels éléments et, plus généralement, s’il existe des mécanismes consensus de recrutement et de traduction sélective des ARNm associés à la régénération.
Pour étudier plus avant la composition du complexe traductionnel, une approche non biaisée consisterait à déterminer par protéomique le ribo-interactome dans les neurones en réponse à une lésion de l’axone, comme cela a été fait récemment dans les cellules souches embryonnaires de souris [59]. Bien que travailler à partir de neurones soit assez difficile (faible quantité de protéines au départ), les progrès de la sensibilité protéomique devraient permettre cette caractérisation dans un avenir proche.
Enfin, l’angle de la régulation traductionnelle offre de nouvelles perspectives en termes de conception thérapeutique pour la réparation du SNC. Notre étude a permis d’identifier HTT comme un facteur pro-régénérateur, ainsi que ses différentes cibles traductionnelles dont la manipulation expérimentale peut favoriser la repousse des axones du SNC. Les travaux futurs se concentreront sur la manipulation des facteurs associés à la traduction afin de réguler la traduction des ARNm associés à la régénération.
En conclusion, la régulation de la traduction apparaît comme essentielle dans le contrôle des programmes de développement, de la spécification des types cellulaires, de la réponse au stress [80, 42, 81] et maintenant de la repousse des axones dans le SNC lésé [82]. Découvrir comment le complexe traductionnel est accessoirisé dans les neurones du SNC et comment il contribue à la régulation traductionnelle constitue une étape importante pour combler le fossé entre l’expression des gènes et le phénotype de repousse des axones.
Déclaration d'intérêts
Les auteurs ne travaillent pas, ne conseillent pas, ne possèdent pas d'actions ou ne reçoivent pas de fonds de toute organisation qui pourrait bénéficier de cet article, et n'ont déclaré aucune affiliation autre que leurs organismes de recherche.
Financement
Les travaux décrits dans cette revue et leurs auteurs ont été soutenus par des subventions de l'Agence Nationale pour la Recherche (ANR-18-CE16-0007 et ANR-21-CE16-0008-02), de la Fondation pour la Recherche Médicale et de l'UNADEV/AVISAN.
Remerciements
Nous remercions tous les auteurs qui ont participé à l’étude décrite dans cette revue, ainsi que le personnel des plateformes de l’Institut des Neurosciences de Grenoble.




 CC-BY 4.0
CC-BY 4.0