1 Introduction
La conception d’une enzyme pour une réaction donnée a été menée à bien par modélisation moléculaire et par mutagenèse dirigée [1]. Une autre approche consiste à isoler le catalyseur optimal à partir d’un grand répertoire de protéines distinctes. Ainsi, le criblage à haut débit permet d’analyser en série typiquement 105, voire 106 protéines distinctes [2]. La sélection est une stratégie différente, qui permet d’isoler un ou plusieurs catalyseurs à partir d’un ensemble plus grand, de 107 à 1014 protéines distinctes : la caractérisation des enzymes isolées est réalisée par amplification du gène correspondant [3]. Pour exploiter le potentiel des stratégies de sélection en ingénierie des enzymes, certaines limitations doivent être levées : (i) dans le cas des sélections in vivo, chaque enzyme est exprimée dans le cytoplasme d’une bactérie ; la généralisation de la méthode à la plupart des réactions chimiques est alors délicate, car les substrats passent souvent difficilement à travers les membranes cellulaires ; (ii) dans le cas de sélections in vitro de protéines (cf. Fig. 1) [4–10], c’est l’efficacité des sélections qui doit être optimisée pour diriger l’évolution d’enzymes. Cette efficacité est mesurée par un facteur d’enrichissement ; il est généralement défini pour un mélange de deux protéines active et inactive par la proportion en enzyme active après sélection, divisée par la proportion en enzyme active avant sélection. Des facteurs d’enrichissement de l’ordre de 103 ont été mesurés pour des sélections faisant appel à des réactions de transcription et de traduction couplées in vitro dans une émulsion d’eau dans l’huile [4]. Lorsque les techniques d’expression de protéines à la surface de phages filamenteux sont utilisées pour coupler une protéine au gène qui lui correspond, les facteurs d’enrichissement en enzymes actives obtenus jusqu’à présent sont de l’ordre de 102 pour des mélanges entre une enzyme et une autre protéine [5, 7–9], ou pour des mélanges entre une enzyme et une enzyme inactivée par mutagenèse dirigée [6].
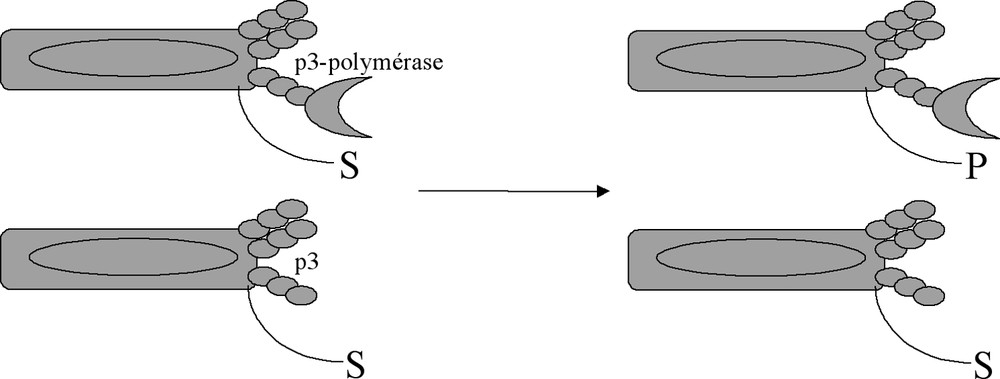
Principe de la sélection en fonction de l’activité catalytique. Le substrat S de l’enzyme est lié de manière covalente à une protéine de la surface du phage. Si une enzyme est exprimée à la surface du phage à proximité du substrat, le substrat est converti en produit P, qui est isolé par chromatographie d’affinité ; la phage-enzyme est ainsi extraite du mélange de phages-protéines. Si aucune enzyme n’est exprimée à la surface du phage, le substrat S n’est pas converti en produit P et le phage n’est pas isolé par chromatographie d’affinité pour le produit de la réaction.
Dans cet article, les réactions de pontage du substrat aux phages et de polymérisation de l’ADN à la surface des phages ont été étudiées pour optimiser une sélection de protéines en fonction de l’activité catalytique.
2 Résultats
Pour optimiser la sélection in vitro d’ADN polymérases en fonction de leur activité catalytique (cf. Fig. 2), notre étude a porté sur plusieurs paramètres : (A) pour diminuer des interactions non spécifiques phage–phage et phage–phase solide, des détergents ont été testés (B) pour minimiser les effets de réactions intermoléculaires (cf. Fig. 3), les conditions de la réaction de polymérisation ont été ajustées (C) pour adapter la sélection aux volumes importants des banques de phages ; l’influence de la concentration en substrat pontant 1 a été analysée.

Sélection in vitro en fonction de l’activité ADN-polymérase. La réaction de pontage conduit à la formation d’une liaison covalente entre l’amorce 1 modifiée par un groupement maléimidyle à son extrémité 5′ et un nucléophile à la surface du phage. La réaction de polymérisation est l’addition catalysée par la polymérase d’un nucléotide 3 à l’extrémité 3′ de l’amorce 1 hybridée au brin matrice 2. Le trait en gras entre O et T indique l’oxygène 5′ du nucléotide T de l’amorce.

La réaction intermoléculaire ou réaction en trans et la réaction intramoléculaire ou réaction en cis. Dans la réaction en trans, une phage-enzyme catalyse la réaction de conversion d’un substrat lié à un phage qui n’exprime pas d’enzyme : par chromatographie d’affinité pour le produit, un phage n’exprimant pas d’enzyme est isolé. Pour des temps de réaction longs ou pour des concentrations élevées en phages, la réaction en trans constitue un bruit de fond significatif pour la sélection en fonction de l’activité catalytique.
La réaction de polymérisation en trans (cf. Fig. 3) est favorisée dans des complexes constitués de plusieurs phages liés entre eux de manière non covalente. Un détergent, le monolaurate de polyoxyéthylène sorbitan, a été utilisé pour dissocier ces complexes. La comparaison des expériences (1) et (2) (Tableau 1) montre que le facteur d’enrichissement est multiplié par plus d’un facteur 5. L’augmentation du rendement de plus d’un ordre de grandeur suggère que l’activité catalytique des phages-polymérases est augmentée par la présence du détergent à des concentrations faibles (0,3%), ce qui pourrait induire le repliement de protéines. L’expérience (3) montre que l’effet du détergent n’est pas lié à l’étape de chromatographie.
Rendements et facteurs d’enrichissement de la sélection en fonction de l’activité catalytique mesurés pour des mélanges de phages-polymérases (sauvages S) et de phages-polymérases inactives (mutantes M) ou phages n’exprimant pas de polymérases (témoin T), dans des conditions expérimentales diverses. Les définitions du rendement et du facteur d’enrichissement sont données dans la partie expérimentale : (a) amorce 1 2,5 μM, dATP 100 μM, polymérisation et pontage pendant 90 min ; (b) amorce 1 2,5 μM, dATP 100 μM, incubation pendant 1 h avec détergent des phages-polymérases, pontage et polymérisation pendant la durée indiquée ; (c) dATP 1 μM, incubation des phages-polymérases pendant 1 h avec détergent, polymérisation pendant 5 min ; (d) dATP 100 μM, incubation des phages-polymérases pendant 1 h avec détergent, polymérisation pendant 5 min.
| Mélange de phages | rendement (%) | facteur d'enrichissement | conditions expérimentales | |
| (1) | S/T | 0,74 | 129 | (a) |
| (2) | S/T | 12 | 670 | (a) incubation avec détergent |
| (3) | S/T | 8,7 | 686 | (a) incubation et chromatographie avec détergent |
| (4) | S/T | 0,1 | 4 | (b) 20 sec |
| (5) | S/T | 7,2 | 255 | (b) 2 min |
| (6) | S/T | 13 | 1320 | (b) 90 min |
| (7) | S/T | 29 | 277 | (b) 1200 min |
| (8) | S/T | 1,8 | 1665 | (c) amorce 1 0,25 μM |
| (9) | S/T | 1,8 | 2747 | (c) amorce 1 0,75 μM |
| (10) | S/T | 1,3 | 3796 | (c) amorce 1 2,5 μM |
| (11) | S/T | 23 | 858 | (d) amorce 1 2,5 μM |
| (12) | S/T | 12 | 670 | (d) amorce 1 25 μM |
| (13) | S/M | 1,9 | 1104 | (c) amorce 1 2,5 μM |
Pour étudier la polymérisation en trans en fonction de la durée de réaction, la polymérisation est amorcée par l’ajout de désoxynucléotides triphosphates et bloquée par l’addition d’éthylène diamine tétra-acétate, qui chélate les ions magnésium, aux temps indiqués. Le facteur d’enrichissement montre un optimum en fonction du temps (4–7). Pour les temps longs, la réaction en trans n’est plus négligeable par rapport à la réaction en cis (7). Pour les temps courts (4), le rendement très faible indique que peu de dUTP dérivatisé par un groupement biotine est ajouté à l’extrémité de l’amorce ; les expériences (10) et (11) montrent que l’excès de dATP d’un facteur 100 par rapport au dUTP dérivatisé par un groupement biotine peut être supprimé. Les facteurs d’enrichissement les plus élevés sont ainsi obtenus. Cet excès avait été introduit initialement pour éviter l’incorporation non spécifique de dUTP dérivatisé par un groupement biotine. Par ailleurs, les expériences de (8) à (12) montrent qu’une concentration de 0,25 μM en amorce, modifiée par un groupement maléimidyle, le substrat pontant, est suffisante pour obtenir un facteur d’enrichissement supérieur à 103 ; cette concentration minimale sera utile pour l’application de la sélection in vitro à des banques de plus de 1013 phages-polymérases, qui correspondent à des volumes au moins égaux au millilitre. Enfin, la comparaison des expériences (10) et (13) suggère que la phage-polymérase mutante n’est pas totalement inactive et qu’elle possède une activité catalytique résiduelle, qui diminue le facteur d’enrichissement en enzymes actives par un facteur trois environ.
3 Partie expérimentale
Les désoxyoligonucléotides 1 et 2, dont les séquences respectives sont TAACACGACAAAGCGCAAGATGTGGCGT et TACAACAATAAAACGCCACATCTTGCG ont été synthétisés et caractérisés comme mentionné précédemment [6]. Le dUTP dérivatisé par un groupement biotine est le 5-[-N-[N-(N-biotinyl-ϵ-aminocaproyl)-γ-aminobutyryl]-3-aminoallyl]-uridine-5’-triphosphate (Roche).
Les phages-polymérase ont été préparées à partir du phage assistant KM13 [11] et des phagemides pHEN1–Stoffel pour la phage-polymérase sauvage, pHEN1–Stoffel mutant pour la phage-polymérase inactive et pHEN1 pour le phage n’exprimant pas de polymérase à sa surface [6]. La bactérie hôte utilisée pour produire les bactériophages filamenteux est la souche TG1 de E. coli. La séquence des inserts entre les sites de restriction NcoI et NotI, qui codent pour les polymérases, est donnée en annexe.
Le protocole pour la sélection in vitro de phages-polymérases en fonction de l’activité catalytique optimisée dérive de celui déjà publié [6]. Les modifications apportées à la sélection, qui ont conduit à un facteur d’enrichissement maximum, sont décrites ci-dessous. Un mélange d’environ 107 phages-polymérases actives et inactives ou un mélange de phages-polymérases actives et de phages n’exprimant pas de polymérase dans les proportions 1:1000 respectivement est incubé dans une solution aqueuse contenant 0,1 M NaCl et 25 mM Na2HPO4 à pH = 8 et à 25 °C pendant 1 h, en présence de 0,3% de monolaurate de polyoxyéthylènesorbitan (Sigma). Au mélange de phages, on ajoute ensuite le substrat pontant 1 2,5 μM (sauf si la concentration est précisée), le brin matrice 2 50 μM, du chlorure de magnésium (Merck) 9 mM dans un volume final de 11 μl, qui est incubé pendant 85 min à 35 °C sans agitation. La polymérisation est amorcée par l’ajout de dVTP 1 μM (V = A, C et G) et de dUTP dérivatisé par un groupement biotine 1 μM et arrêtée après une durée indiquée par l’addition d’éthylène diamine tétra-acétate (250 mM, 15 μl, pH = 8). Le mélange est alors incubé avec des billes superparamagnétiques recouvertes de streptavidine (Dynabeads M180, Dynal) dans un tampon TEN, 200 μl (0,3 M tris-hydroxyméthyle-aminométhane, 33 mM éthylène diamine tétra-acétate, 1,3 M NaCl, pH = 9) pendant 30 min sur une roue tournante à température ambiante. Les billes sont ensuite lavées à sept reprises avec le tampon TEN, contenant éventuellement 0,12% de monolaurate de polyoxyéthylènesorbitan, puis reprises dans le tampon PBS (0,1 ml). Un ajout de trypsine de pancréas de bœuf (10 μl, 10 g l–1, Sigma) rend non infectieux l’ensemble des phages qui n’expriment pas de protéines codées par le phagemide [11]. Des bactéries E. coli TG1 en excès (8 × 108 cellules, 1 ml de milieu de culture 2xTY) sont ensuite ajoutées aux mélanges de phages avant ou après sélection à 37 °C pendant 25 min, puis déposées sur des boîtes de Pétri contenant de l’ampicilline (100 mg l–1) et incubées à 37 °C pendant 12 h. Le nombre de colonies obtenu définit alors le nombre de phages avant ou après sélection. Le rendement est défini comme le rapport du nombre de phages-polymérases après sélection et du nombre de phages-polymérases avant sélection.
L’enrichissement est calculé après analyse de 48 colonies dérivant du mélange de phages avant sélection et de 48 colonies du mélange de phages après sélection. Chaque colonie de bactéries, qui dérive d’un phage après infection, comporte un phagemide, qui code ou non pour une polymérase et qui est utilisé comme matrice d’ADN pour l’amplification par réaction de polymérisation en chaîne.
Pour les mélanges de phages-polymérases (sauvage/mutant), le protocole d’amplification puis de digestion par l’enzyme de restriction AluI décrit précédemment [6] a été utilisé : la phage-polymérase sauvage se différencie après électrophorèse sur gel d’agarose à 2% par deux fragments de 57 et de 193 paires de bases, de la phage-polymérase mutante, qui comporte un fragment de 260 bases. Pour les mélanges de phages (sauvage/témoin) exprimant ou non une polymérase, l’amplification a été réalisée avec les désoxynucléotides 4 CTATGCGGCCCCATTCA, 5 ACAGCTATGACCATGATTACGCC et 6 GCTTGGGCAGAGGATCC ; la taille des fragments obtenus définit de façon univoque le phage exprimant l’enzyme sauvage (767 paires de base) ou le phage témoin (218 paires de bases).
4 Conclusion
L’expression de protéines à la surface des phages permet d’établir un lien entre une protéine et le gène correspondant. Le pontage du substrat sur les phages crée un second lien. C’est la superposition de ces deux liens qui permet de concevoir une stratégie pour isoler des enzymes à partir de banques de protéines par sélection in vitro. Cette sélection in vitro de protéines en fonction de l’activité catalytique est une sélection en fonction de l’affinité pour le produit de la réaction lié de façon covalente à la phage-enzyme qui a catalysé la réaction. Les sélections de protéines en fonction de l’affinité pour une molécule donnée, qui utilisent les techniques d’expression de protéines à la surface de phages filamenteux, et établies depuis plus d’une décade, conduisent typiquement à des enrichissements en protéines affines de l’ordre de 103 à 105 par tour de sélection [12–14]. Il paraît improbable que les enrichissements de notre sélection en fonction de l’activité catalytique puissent être supérieurs à ceux de sélections en fonction de l’affinité. Dans ce travail, nous avons montré qu’un enrichissement en enzymes actives d’un facteur 4 × 103 peut être obtenu dans le cas d’un mélange de phages-polymérases et de phages n’exprimant pas d’ADN-polymérases à leur surface. Cette sélection optimisée permet de diminuer le nombre de cycles d’amplification et de sélection nécessaires pour isoler après criblage de la population finale de protéines les meilleurs catalyseurs d’une banque.
Cette sélection in vitro de polymérases en fonction de l’activité catalytique et l’optimisation du facteur d’enrichissement devraient être aisément généralisables à d’autres enzymes. Cette sélection optimisée pourra être appliquée à des banques d’environ 1013 phages-polymérases contenant plus de 108 phages-polymérases distinctes pour extraire des enzymes dotées d’activités catalytiques nouvelles, comme des polymérases capables de copier des ARN (transcriptases inverses) ou des analogues d’acides nucléiques. Adaptée à d’autres stratégies d’expression de protéines à la surface de phages filamenteux [15], elle pourrait avoir des applications en génomique fonctionnelle.
Remerciements
Nous remercions O. Helynck, C. Gouyette et V. Huteau pour la synthèse d’oligonucléotides, Dr Vichier-Guerre, Dr Huynh-Dinh pour des conseils et des rapporteurs pour des critiques. E.O. a bénéficié d’une bourse d’ingénieur du CNRS. Ces travaux ont reçu le soutien du MENRT (ACI blanche/Chimie), du CNRS (programme PCV) et de l’Institut Pasteur.
Annexe
La séquence d’ADN entre les sites NcoI et NotI du phagemide qui code pour le fragment de Stoffel de l’ADN polymérase I de Thermus aquaticus est indiquée ci-dessous.
CCATGGCCTCTGGTGGCGGTGGCTGTGGTGGCGGTGGCAGCCCCAAGGCCCTGGAGGAGGCCCCCTGGCCCCCGCCGGAAGGGGCCTTCGTGGGCTTTGTGCTTTCCCGCAAGGAGCCCATGTGGGCCGATCTTCTGGCCCTGGCCGCCGCCAGGGGGGGCCGGGTCCACCGGGCCCCCGAGCCTTATAAAGCCCTCAGGGACCTGAAGGAGGCGCGGGGGCTTCTCGCCAAAGACCTGAGCGTTCTGGCCCTGAGGGAAGGCCTTGGCCTCCCGCCCGGCGACGACCCCATGCTCCTCGCCTACCTCCTGGACCCTTCCAACACCACCCCCGAGGGGGTGGCCCGGCGCTACGGCGGGGAGTGGACGGAGGAGGCGGGGGAGCGGGCCGCCCTTTCCGAGAGGCTCTTCGCCAACCTGTGGGGGAGGCTTGAGGGGGAGGAGAGGCTCCTTTGGCTTTACCGGGAGGTGGAGAGGCCCCTTTCCGCTGTCCTGGCCCACATGGAGGCCACGGGGGTGCGCCTGGACGTGGCCTATCTCAGGGCCTTGTCCCTGGAGGTGGCCGAGGAGATCGCCCGCCTCGAGGCCGAGGTCTTCCGCCTGGCCGGCCACCCCTTCAACCTCAACTCCCGGGACCAGCTGGAAAGGGTCCTCTTTGACGAGCTAGGGCTTCCCGCCATCGGCAAGACGGAGAAGACCGGCAAGCGCTCCACCAGCGCCGCCGTCCTGGAGGCCCTCCGCGAGGCCCACCCCATCGTGGAGAAGATCCTGCAGTACCGGGAGCTCACCAAGCTGAAGAGCACCTACATTGACCCCTTGCCGGACCTCATCCACCCCAGGACGGGCCGCCTCCACACCCGCTTCAACCAGACGGCCACGGCCACGGGCAGGCTAAGTAGCTCCGATCCCAACCTCCAGAACATCCCCGTCCGCACCCCGCTTGGGCAGAGGATCCGCCGGGCCTTCATCGCCGAGGAGGGGTGGCTATTGGTGGCCCTGGACTATAGCCAGATAGAGCTCAGGGTGCTGGCCCACCTCTCCGGCGACGAGAACCTGATCCGGGTCTTCCAGGAGGGGCGGGACATCCACACGGAGACCGCCAGCTGGATGTTCGGCGTCCCCCGGGAGGCCGTGGACCCCCTGATGCGCCGGGCGGCCAAGACCATCAACTTCGGGGTCCTCTACGGCATGTCGGCCCACCGCCTCTCCCAGGAGCTAGCCATCCCTTACGAGGAGGCCCAGGCCTTCATTGAGCGCTACTTTCAGAGCTTCCCCAAGGTGCGGGCCTGGATTGAGAAGACCCTGGAGGAGGGCAGGAGGCGGGGGTACGTGGAGACCCTCTTCGGCCGCCGCCGCTACGTGCCAGACCTAGAGGCCCGGGTGAAGAGCGTGCGGGAGGCGGCCGAGCGCATGGCCTTCAACATGCCCGTCCAGGGCACCGCCGCCGACCTCATGAAGCTGGCTATGGTGAAGCTCTTCCCCAGGCTGGAGGAAATGGGGGCCAGGATGCTCCTTCAGGTCCACGACGAGCTGGTCCTCGAGGCCCCAAAAGAGAGGGCGGAGGCCGTGGCCCGGCTGGCCAAGGAGGTCATGGAGGGGGTGTATCCCCTGGCCGTGCCCCTGGAGGTGGAGGTGGGGATAGGGGAGGACTGGCTCTCCGCCAAGGAGGCGGCCGC
La séquence codante pour le fragment de Stoffel mutant est obtenue en substituant GACGAGCTG par GCCGCACTG.


