Il manquait dans les exposés précédents un mot qui est central en biologie : le mot « information ». D’ailleurs, ce que nous sommes en train de faire est quelque chose de tout à fait particulier : nous échangeons de l’information. L’information que je vais essayer de vous transmettre aujourd’hui, c’est qu’aux quatre catégories de base de la nature (matière, énergie, espace et temps), il serait bon, au moins à titre heuristique, d’en ajouter une cinquième (information), et de chercher comment cette catégorie s’articule avec les quatre autres. Les êtres vivants sont des objets particuliers capables de piéger l’information, c’est ce que je vais essayer de vous montrer. Dans ce contexte, ce que nous appelons « sélection naturelle » devient un authentique principe de la physique. Ce principe, je vous l’énonce brièvement : « faire de la place en utilisant de l’énergie “pour éviter de détruire” ce qui marche ». Je dis bien : pour éviter de détruire. C’est ce que je vais essayer de vous montrer.
Les objectifs de la biologie synthétique sont nombreux : reconstruction pour comprendre, abstraction, ingénierie, évolution [1]. On a mentionné la reconstruction ce matin : le fait d’essayer de reconstruire met en évidence les points qui sont particulièrement difficiles à comprendre et on bute sur ce qu’on ne comprend pas. Essayer de voir les objets tels qu’ils sont avec leurs idiosyncrasies, pour découvrir d’autres propriétés plus abstraites, c’est l’attitude d’abstraction. Sur l’ingénierie, nous aurons un exposé, et c’est bien un aspect très important. Enfin, bien sûr, on va s’occuper d’évolution.
Ce que je vais retenir des organismes vivants est un peu différent de ce que nous avons entendu jusqu’à maintenant. Pour faire bref : j’essaie de considérer les êtres vivants comme des ordinateurs faisant des ordinateurs. L’idée est qu’ils contiennent un algorithme de construction. C’est l’algorithme de construction qui est transmis au cours des générations : cela nous évite le problème de la dualité entre préformation et épigenèse. Autre point important : la machine doit être séparée physiquement du support de l’algorithme. Ce processus ressemble à ce qui se passe dans les ordinateurs. Bien sûr, nos ordinateurs ne font pas des ordinateurs – pas encore. Mais, en ne considérant que les bactéries (je ne vais pas parler d’organismes plus compliqués à ce stade), on peut comparer le programme génétique à un programme d’ordinateur. Apparaît alors le point essentiel : il s’agit de construire (paradoxalement) de jeunes organismes à partir des vieux et pour cela, il y a des gènes qui sont des pièges à information.
Des ordinateurs qui font des ordinateurs, est-ce bien d’actualité ? Il existe une tentative pour faire des imprimantes laser 3D qui font des imprimantes laser 3D. Je vous invite à aller regarder le site1. Il est intéressant de voir ce qui s’y passe. La machine sait déjà reproduire la plupart de ses composants. Ce qui manque évidemment, c’est la ligne d’assemblage et le programme. On peut facilement imaginer des programmes qui se reproduisent et il y a déjà des robots qui savent répliquer des programmes en tant qu’éléments stables. L’idée d’un ordinateur qui se reproduirait n’est donc pas une utopie complètement délirante ; elle n’est pas plus délirante que la biologie synthétique telle qu’on la conçoit aujourd’hui. Et maintenant, où intervient l’information ?
J’ai l’habitude pour le faire comprendre de donner une image très simple qui remonte à 3000 ans. La pythie, à Delphes, posait la question suivante : j’ai une barque faite de planches, les planches s’usent une à une, je les change. Au bout d’un certain temps, toutes les planches ont été changées, est-ce la même barque ? Il va de soi que le propriétaire va répondre : « Oui, c’est ma barque ». Il a parfaitement raison. Quelque chose s’est conservée, qui n’est pas la matière de la barque, puisque l’on sait que tout a été changé. Ce quelque chose est ce qui permet à la barque de flotter, ce sont les relations entre les constituants de la barque ; ce qui fait la barque, c’est la manière dont les planches sont assemblées. C’est essentiellement une information, qui est exprimée dans la disposition des planches, et qui conserve à la barque sa propriété d’être une barque [2]. Nous touchons là une notion centrale pour la biologie.
Voici un exemple plus directement biologique. Massimo Vergassola et ses collègues se sont interrogés sur la façon dont certains papillons peuvent se rejoindre à très grande distance, 1 km ou plus, parfois [3]. La réponse classique consiste à dire que le papillon mâle qui va chercher sa femelle remonte un gradient chimique ; mais sur 1 km, avec la turbulence du vent et la présence d’obstacles (immeubles…), il ne peut pas y avoir de gradient chimique. Comment fait le papillon pour rejoindre sa femelle ? Vergassola et ses collègues ont montré qu’en mesurant quelque chose qu’on peut appeler « information » et traiter avec une théorie comme la théorie de l’information, et en considérant la femelle comme la source d’information, on peut aller vers la source en maximisant l’information ; ils appellent ce processus « infotaxie ». L’idée est que des organismes du type papillon, insectes somme toute assez simples sont capables de mesurer de l’information et d’utiliser cette mesure pour arriver à atteindre un objectif.
Avec la matière, l’énergie, l’espace et le temps, l’information est une catégorie du réel. Si l’on range les sciences en ordre croissant, eu égard au contenu informationnel de leur objet, on trouve d’abord la physique, puis la chimie, la biologie et les mathématiques. La biologie se trouve ainsi logée entre sciences très concrètes et sciences très abstraites. Cela explique une des difficultés de la biologie. On a tendance à penser que la biologie est facile à comprendre, parce qu’elle étudie une réalité concrète, proche de l’homme. Mais dès que l’on s’essaie à enseigner le code génétique et son cadre de lecture, on s’aperçoit qu’il met en jeu des propriétés très abstraites. En Chine, pour des gens qui n’ont pas l’écriture alphabétique, il est très difficile de faire comprendre, par exemple, qu’un message est une suite de codons, qu’un codon, qui correspond à un acide aminé, est un ensemble de trois nucléotides, et qu’un décalage de lecture d’un seul nucléotide change complètement le message. Sa proximité avec les sciences abstraites rend la biologie difficile. Notons aussi, incidemment, que sous cet angle la mathématique n’est pas platonicienne ; elle est plongée dans matière, espace, temps, énergie, comme le reste et sa composante informationnelle est très élevée.
Venons-en à un second point, lié à la notion d’information. Freeman Dyson, il y a 25 ans, a proposé une intéressante réflexion sur les origines de la vie, dans un livre qui porte ce titre, au pluriel, par conséquent [4]. Il montre de façon extrêmement convaincante qu’il ne peut pas y avoir qu’une origine de la vie ; il y en a au moins deux, et cela vient de ce qu’il faut séparer la reproduction (qui s’améliore avec le temps) de la réplication (qui, sauf si elle a accès à un réservoir extérieur, ne peut qu’accumuler les erreurs). Malheureusement, l’usage de ces deux mots est souvent mélangé par les gens qui les utilisent et c’est une malheureuse erreur conceptuelle. Reproduction et réplication sont deux choses différentes. La reproduction doit avoir précédé la réplication. Et il est très important de comprendre que dans les organismes vivants, il y a une information associée à la reproduction et une information associée à la réplication qui sont différentes.
Pour ces raisons, je propose une définition de la vie, qui va comme cela : la vie demande une machine et un programme. La machine, que dans le vocabulaire de la biologie synthétique on appelle le « châssis » (le mot français), est ce qui se reproduit. Elle contient des compartiments et un métabolisme. Les compartiments sont délimités par des membranes ou des peaux : en gros, on a ou bien une seule enveloppe qui enserre tout, ou bien de multiples casiers, l’important étant de marquer une frontière entre dehors et dedans. Le métabolisme est un processus dynamique, qui consiste en la conversion de molécules les unes dans les autres, avec gestion de l’utilisation de l’énergie. Tous les êtres vivants métabolisent. Il y a bien un état entre la mort et la vie qu’on appelle la dormance, où le métabolisme est mis en veilleuse, c’est l’état de la graine ou de la spore, mais on dira une graine ou une spore vivante quand on l’aura vue germer. Le programme, ou livre des recettes, c’est autre chose. Le programme se réplique, il produit une copie exacte de lui-même. Par ailleurs, il utilise l’information de façon récursive, ce qui est conceptuellement très profond. L’ancienne biologie des systèmes était fondée sur les concepts de proaction et de rétroaction (feedback). Un algorithme récursif est un algorithme qui fait appel à soi-même, cela renvoie à des propriétés qualitativement différentes de tout ce qui est proaction ou rétroaction.
Machine et programme sont distincts. La machine est capable de lire et écrire – j’insiste sur ce point, qui est fondamental. Par ailleurs, elle ne distingue pas les donnéess du programme. Cela évoque la définition de la machine de Turing, une machine qui peut lire et écrire. Le programme (qui est sur un support physique : le disque qu’on met dans la machine) comporte deux types de données, relatives à l’objectif à atteindre (programme au sens strict), et au contexte (données au sens strict). On notera que programme et données sont traités de la même manière par la machine. Aucune divinité extérieure ne dit à la machine : cela est un programme. Vous entrez un disque dans la machine, la machine ne sait pas qu’il y a quelqu’un qui a une intention vis-à-vis de ce programme.
Dans les expériences de transplantation de génome faite sous l’égide de Lartigue et al. [5], ce que je retiens, ce n’est pas la synthèse ou l’assemblage semi-synthétique d’un ADN complet d’une bactérie, c’est la séparation physique du programme et de la machine. À ma connaissance, cette expérience n’a pas encore été reproduite ailleurs dans le monde, mais je pense qu’elle est fondamentale du point de vue conceptuel. L’idée est de prendre un ADN d’une espèce particulière, de le mettre dans un organisme d’une autre espèce, dans des conditions où l’on s’arrange pour que seul l’ADN de l’espèce donneuse ait des chances de se multiplier et au bout d’un certain temps, on voit apparaître une machine correspondant au programme qui a été donné dans la première. Cette expérience, réalisée par Carole Lartigue et ses collègues chez Craig Venter, n’a pas été reproduite, à ma connaissance. Elle est conceptuellement si fondamentale que je pense qu’il est absolument essentiel que d’autres la réalisent. Je la crois faisable, mais je ne suis pas tout à fait sûr que les conditions qui ont été utilisées soient bonnes pour qu’elle réussisse facilement.
Quelles objections peut-on faire à ce que je viens d’expliquer ? Il y en a beaucoup, dont celle-ci : en dehors du programme génétique, la cellule contient déjà une quantité considérable d’information. Mais c’est exactement la même chose dans un ordinateur : quand vous mettez le disque avec le système d’exploitation, l’ordinateur contient déjà une information considérable. Or tout le monde admet qu’un ordinateur fonctionne selon les principes de la machine de Turing. Donc l’objection tombe. Ce n’est pas parce qu’il y a de l’information dans la machine qu’on n’a pas affaire à une machine de Turing.
Une seconde objection, plus cruciale pour la biologie synthétique, a trait à ce que le programme n’est pas une entité abstraite. Il faut un support au programme – par exemple, un disque compact. Mais imaginez que votre disque compact, vous l’avez laissé sur la plage arrière de la voiture un jour de grand soleil, et quand vous arrivez pour charger votre ordinateur, rien ne marche. Pourquoi ? Le programme n’a pas été effacé, il est là, mais le disque a été légèrement déformé, et étant déformé il ne peut plus être lu par le laser de la machine. Le programme doit avoir un support matériel. Cela s’illustre par une découverte absolument extraordinaire dans le système nerveux de certains vertébrés, des vertébrés qui voient la nuit. Vous savez que le bricolage qui est à la base de la construction des organismes vivants est tel que les rétines sont construites de façon aberrante. En fait, les cellules qui reçoivent la lumière sont derrière et non devant les neurones : la nature a mis un rideau devant, c’est complètement absurde, jamais un ingénieur n’aurait fait cela. On veut capter la lumière, la nuit on veut pouvoir capter un photon à la fois… Eh bien, ce qu’on observe est incroyable : au moment où la lumière baisse, les noyaux des neurones de la rétine de ces organismes se mettent à exprimer des gènes qui font que ces noyaux se réorganisent complètement, au point de devenir des lentilles qui concentrent la lumière sur les cellules qui se trouvent derrière. Il y a bien un aller-et-retour entre le support et le programme, y compris pour la modification du programme ! La conclusion qui s’impose est claire : on peut réussir à construire un génome artificiel, mais il n’est pas sûr qu’on saura le faire fonctionner en le mettant dans une cellule. Autrement dit, il ne suffit pas d’assembler un DNA dont la séquence est correcte, il faut aussi qu’il soit correctement replié.
Je reviens maintenant au cœur de ce que je voulais dire. Le problème est le suivant : la machine se reproduit, le programme se réplique. La reproduction, si l’on y réfléchit, est une chose extraordinaire : les bébés naissent très jeunes d’une mère adulte, ce qui suppose que le jeune va devoir recréer de l’information, et comment fait-il, à partir d’une machine qui n’a pas cette information ? Par ailleurs, le programme se réplique à chaque division cellulaire et la réplication mène à accumuler des erreurs. Comment, au cours des générations, peut-il y avoir accumulation d’information ? Je suis généticien : l’information est, soit recréée, soit inventée, et il doit s’agir d’une caractéristique universelle. Il doit donc exister des gènes codant ce processus d’acquisition d’information.
Pour les découvrir, nous nous trouvons face à un problème compliqué. Même si des fonctions sont universelles chez les organismes vivants qui sont soumis au triplet variation-sélection-amplification, c’est l’évolution qui domine. L’évolution crée les conditons d’apparition de fonctions, qui, pour exister doivent utiliser des structures, soit comme créations de novo, soit en détournant la fonction d’objets préexistants. Les choses vont rarement dans l’autre sens. Il est exceptionnel qu’une structure puisse vous dire réellement la fonction. J’ai un exemple simple : c’est l’été, je suis assis à mon bureau, qui est couvert de papiers, en train de lire un livre ; la fenêtre est ouverte, le vent se lève, que fais-je ? Je prends un parallélépipède lourd (le livre), je le pose sur les papiers pour qu’ils ne s’envolent pas. La fonction du livre ici est d’être un presse-papier. Cette image illustre que les fonctions ubiquistes ne peuvent pas être inférées à partir de la structure des gènes.
Pour détecter les fonctions ubiquistes, il y a en revanche un moyen : c’est d’étudier la persistance des gènes dans les génomes. Quand un organisme vivant a trouvé une solution à un problème, il a tendance (c’est une simple tendance, pas une nécessité) à la transmettre à sa descendance. Pour chercher les gènes persistants, on analyse la présence des gènes dans un grand nombre d’organismes. Certains d’abord sont présents dans un organisme, deux organismes, et le nombre de ces gènes décroît, puis tout d’un coup, il augmente, et l’on trouve une famille de gènes présents dans beaucoup d’organismes : ce sont les gènes persistants. On peut alors comparer ces gènes avec les gènes dits essentiels, ces gènes qu’on ne peut pas inactiver sans perdre la capacité totale des organismes vivants.
Le point fondamental ici est que, si l’on cherche les gènes persistants dans les organismes bactériens, on trouve à peu près 500 gènes, et ces gènes sont deux fois plus nombreux que les gènes dits essentiels. Ils ont tous les mêmes propriétés, en particulier ils sont transcrits avec la même orientation que le mouvement de la réplication, dans le sens du brin direct de l’ADN, une contrainte qui montre qu’ils ont des propriétés intrinsèques originales, liées à la façon dont le programme génétique est organisé dans le chromosome.
J’ai nommé « paléome » cet ensemble de gènes (du grec palaios, ancien). En effet, si l’on analyse les fonctions qu’ils codent, et leur organisation dans les génomes, on remarque qu’ils reconstituent un scénario plausible de l’origine de la vie [6]. Regardons un génome de bactérie (ici : l’exemple de Pseudomonas putida), comparé avec plusieurs centaines de génomes bactériens. Je groupe les gènes par 50 et analyse la contiguité de ces gènes dans le chromosome en fonction de leur rareté dans les génomes. On constate alors que les gènes très rares sont forment généralement des ensembles contigus, ce qui va bien avec leur origine : ils arrivent par transfert horizontal, mais ce qu’on sait d’eux le plus souvent, c’est qu’on ne connaît pas leurs fonctions. Quant aux gènes persistants, ils ont aussi tendance à rester ensemble, mais la plupart de leurs fonctions sont connues. Dans le groupe des gènes persistants (« paléome » : cœur du génome, nécessaire pour construire une usine cellulaire), il y a moins de 2000 gènes ; tandis que la partie variable, que j’appelle « cénome » (en souvenir de Karl Möbius, qui avait appelé « biocénose » le fait de former une communauté d’organismes), est en augmentation constante (ici : plus de 50 000 gènes), comme s’il y avait dans l’environnement un réservoir de gènes quasiment infini, non limité en l’état actuel de nos connaissances. Le « cénome » est l’ensemble des gènes qui permettent à l’organisme de vivre dans un environnement particulier (Fig. 1).
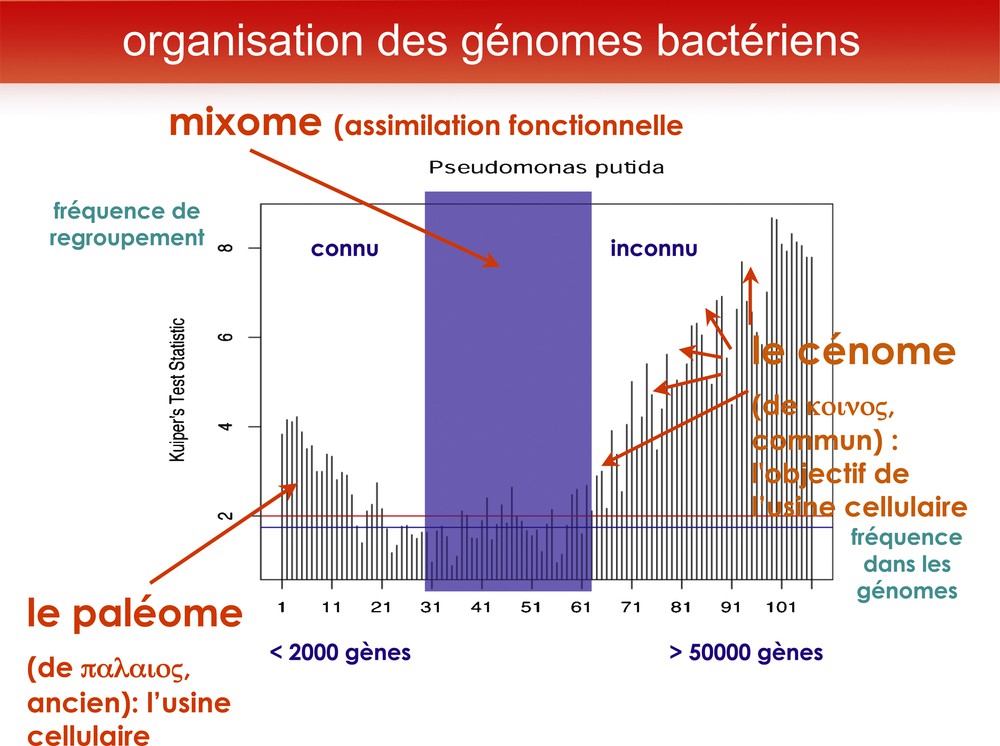
Pour découvrir les gènes du cœur de la cellule, nous avons comparé le génome d’une bactérie banale du sol, Pseudomonas putida, à celui de plusieurs centaines de bactéries. Nous avons regroupé les gènes par familles de 50, en portant en abscisse leur fréquence dans les génomes (très fréquents à l’origine, et de plus en plus rares quand on s’en éloigne). Et en ordonnée leur tendance à rester proches les uns des autres dans les génomes (la tendance à rester proche donne une position haute en ordonnée). Ce regroupement montre immédiatement une séparation en trois classes : les gènes très fréquents tendent à rester grouper, tout comme les gènes très rares. Le premier ensemble a été nommé paléome, car son organisation (non détaillée ici) permet de proposer un scénario plausible de l’origine de la vie. Le second ensemble, formé de gènes qui s’échangent horizontalement entre des organismes phylogénétiquement divers, forment le cénome (mot créé pour rappeler le concept de biocénose, communauté écologique d’organismes, proposé par Karl Möbius en 1877). Entre les deux, se trouve une région, le mixome, où les fonctions transmises horizontalement tendent peu à peu à être universelles et à s’assimiler au paléome.
L’ubiquité fonctionnelle n’implique pas l’ubiquité structurale. Mais la persistance à travers les générations permet d’identifier, dans les génomes bactériens, à peu près 500 gènes (le « paléome »), dont environ 250 (la moitié) sont « essentiels ». Les autres gènes, en nombre variable, sont ceux qui permettent d’occuper une « niche » écologique particulière.
Cela se résume de la façon suivante :
- • les gènes qui permettent de construire la vie sont les 250 gènes « essentiels » ;
- • les gènes qui permettent de perpétuer la vie (c’est-à-dire, de maintenir, de retrouver ou de créer l’information) sont présents dans les autres gènes du paléome ;
- • les gènes permettant de vivre dans un contexte donné sont ceux du cénome, découverts et étudiés par la métagénomique.
Le paléome lui-même est donc formé de deux parties : l’une qui inclut le constructeur et le réplicateur (celle des gènes « essentiels ») et une autre partie (celle des gènes persistants non essentiels). Cette dernière partie est inattendue. On y rencontre une première bizarrerie : il codent des fonctions qui sont celles de ‘patches’ ou ‘rustines’ métaboliques : je ne vais pas plus en parler, parce que cela supposerait entrer dans des considérations spécifiques de la chimie. Disons seulement qu’il n’y a aucune raison pour que les objets chimiques utilisés par la vie soient compatibles entre eux. On rencontre donc une série de contradictions et de très jolis problèmes de chimie – j’aimerais en discuter avec des chimistes. Dans l’espace réduit de la cellule, se côtoient des produits chimiques incompatibles entre eux. La seconde bizarrerie concerne ce qui est lié à la pérennisation de la vie. On trouve ici des gènes qui sont des gènes de dégradation (des ARN, des protéines) mais qui ont besoin d’énergie pour effectuer ce travail. C’est très paradoxal si l’on y réfléchit, parce que normalement la dégradation de quelque chose est productrice d’énergie, et non consommatrice. Alors, pourquoi diable cette dégradation utilise-t-elle de l’énergie ?
Revenons à l’information, dont je parlais au départ de cet exposé. Si je vous pose la question : pour créer de l’information, faut-il utiliser de l’énergie ? – intuitivement, vous me répondrez que oui. Cette notion intuitive était très répandue dans les années 1950, quand IBM cherchait à construire des calculateurs qui calculent de plus en plus vite dans des espaces de plus en plus petits. Si l’intuition était exacte, les transferts d’énergie qui allaient avoir lieu lors de la création d’information dans ces machines seraient tels qu’on se heurterait à une limite du calcul, tout simplement parce que le microprocesseur (il n’en existait pas encore, à l’époque, mais on imaginait réduire les unités de calcul) allait fondre. IBM a donc demandé à ses ingénieurs de prédire la limite du calcul. Un ingénieur du nom de Rolf Landauer2 démontra en 1961 que le calcul est réversible et que créer de l’information ne demande pas d’énergie [7]. J’insiste sur la date, parce qu’il est pour moi extraordinaire que ce résultat soit passé presque inaperçu, et qu’il reste largement ignoré, même des spécialistes de l’informatique. Charles Bennett3, l’un des pères du calcul quantique, poursuivit ces recherches et développa une autre idée de Landauer, que voici. Le problème n’est pas de générer de l’information, le problème est d’accumuler de l’information. Pour accumuler de l’information, il faut faire de la place et pour faire de la place, il faut dépenser de l’énergie. Cependant, ces chercheurs n’ont pas du tout discuté – c’est là qu’intervient ma propre contribution – ils n’ont pas cherché à voir où l’énergie était utilisée. Et c’est ce sur quoi je vais essayer de vous montrer qu’on peut faire des expériences. Ma conjecture est que l’utilisation de l’énergie n’est pas simplement pour faire de la place, elle est pour « éviter de détruire » ce qui est informatif. Si, par exemple, vous avez réalisé (de façon réversible) une division, le résultat informatif c’est le reste de la division : vous ne pouvez pas détruire au hasard, parce que vous risquez de perdre un morceau du reste, donc vous n’aurez pas votre résultat. Ce résultat, il faut absolument le protéger contre la dégradation, on fera de la place ailleurs, mais lui, il faut le garder. L’idée est qu’il faut faire de la place pour accumuler de l’information, mais il faut le faire en protégeant ce qui est riche en information. Cela requiert deux choses : découvrir les fonctions correspondantes et trouver la source d’énergie. Cela est un point crucial. Je n’aurai pas le temps de le détailler, mais je donnerai quelques éléments.
Schématiquement, on a donc un processus de dégradation énergivore, qui dépense l’énergie pour éviter de dégrader soit des gènes, soit des éléments fonctionnels. Comme ce processus est ubiquiste, on doit pouvoir trouver les fonctions correspondantes dans le paléome.
Vous cherchez donc une famille de systèmes qui s’attache à tous les objets de la cellule (une illustration concrète du « démon de Maxwell ») : quand il rencontre une entité non fonctionnelle, il la dégrade ; quand il identifie une entité fonctionnelle, il l’écarte du processus de dégradation, mais cela a un coût : il doit dépenser de l’énergie. La source d’énergie habituelle dans la cellule c’est l’ATP, j’ai donc initialement pensé à l’ATP ; puis je me suis dit que dans une vieille culture bactérienne il n’y a plus d’ATP, l’idée était donc mauvaise. Je suis retourné à mes gènes persistants et j’ai découvert quelque chose de vraiment incroyable. Dans les gènes du paléome, les gènes de synthèse et de dégradation, bref, du métabolisme en général, on trouve les gènes du métabolisme des polyphosphates. Les polyphosphates sont des minéraux présents dans toutes les cellules et qui sont en général considérés comme anecdotiques. Kornberg les a étudiés jusqu’à sa mort, et tout le monde a considéré que cela n’avait aucun intérêt, moi compris. Tout à coup, leur sens apparaît et l’on comprend tout l’intérêt de ce que signifie « minéral » ; un minéral qui, incidemment, peut avoir existé très tôt, au commencemant de la vie – stable à la dessication, stable à l’irradiation, une source d’énergie idéale, ultime en tout cas, pour effectuer le processus dont je parle.
Ce processus d’accumulation de l’information est aveugle, il accumule l’information quelles que soient les circonstances. L’information peut venir de la mémoire (en relisant le programme), elle peut être une création (comme on fait de la place, on peut mettre quelque chose à la place). Et je propose la conjecture que les mutations dites « adaptatives » sont de ce type et cette conjecture peut alors être testée. On sait détruire un gène dans une bactérie – disons, un gène qui lui sert à utiliser un grand nombre de sources de carbone ; on sait le détruire complètement, de sorte que, si on étale une centaine de ces bactéries sur une boîte de Petri avec un milieu qui contient assez d’aliments pour que chaque bactérie forme une colonie, et cette source d’énergie « inutilisable », elles se multiplient rapidement, puis, ayant épuisé ce qu’elles savent utiliser, elles subsistent sous la forme d’une centaine de colonies dont la taille ne change pas. On attend un, deux, trois jours, on observe ce qui se passe dans la boîte, rien ne se passe. Si l’on attend plus longtemps, soudain il apparaît à la surface de certaines colonies, des « papilles » qui se mettent à proliférer et envahir la boîte. Quelles sont leurs particularités ? Ces papilles sont des bactéries qui, contrairement à leurs parents, savent utiliser la source de carbone. Comment savoir s’il s’agit de « mutations » et lesquelles ? On ne pouvait pas le savoir avant d’être capable de séquencer rapidement les génomes. Dans mon laboratoire, nous avons séquencé sept génomes complets de ces bactéries et découvert que la plupart de ces mutations étaient dans un gène particulier. Nous avons pu en examiner d’autres en utilisant la PCR – je parle d’une expérience en cours. En scrutant ces mutations, on constate d’abord qu’elles sont différentes les unes des autres, ensuite qu’il arrive qu’il y en ait plusieurs dans le même gène, et même une duplication interne – d’une suite de 60 nucléotides (ce qui introduit 20 acides aminés dans la protéine correspondante), ce qui apparemment suffit à faire apparaître une fonction. Cela montre, et c’est le point crucial, que ces mutations ne préexistaient pas. On peut voir aussi qu’elles sont bien apparues au hasard, puisqu’elles sont réparties un peu partout et que les génomes des mutants qui sont apparus le plus tard contiennent beaucoup plus de mutations que ceux qui sont apparus très tôt. Les mutations apparaissent au hasard au cours du vieillissement, et si l’une d’entre elles permet la réponse adaptée, alors la bactérie qui a trouvé une solution se divise et produit des descendants. Mais il est évident que pour mettre au jour une fonction nouvelle, la cellule à l’origine de la papille a eu besoin de faire de la place, ce qui lui a permis de tester l’hypothèse de l’utilisation d’une nouvelle source de carbone. Ce modèle nous sert à tester la conjecture proposée plus haut : en inactivant un par un les gènes dont je parlais, nous pouvons identifier quels gènes peuvent être détruits sans empêcher la bactérie initiale de produire des mutations adaptatives, et lesquels sont indispensables à la fabrication de ces bactéries innovantes.
Je résume. Mon idée directrice est qu’il y a dans les organismes vivants des mécanismes de dégradation qui font de la place pour permettre l’accueil d’entités nouvellement synthétisées ; ces mécanismes ne sont pas passifs. Ils usent de l’énergie pour éviter la destruction d’entités fonctionnelles. Ce processus illustre ce qu’est la sélection naturelle, il est d’une grande généralité. Il est adaptable à toutes les échelles : par exemple, en gestion du personnel (même si malheureusement presque personne ne fait de cette façon), si l’on veut accroître la valeur de l’information que véhicule l’entreprise, il faut utiliser toute son énergie, non pas pour détruire, mais pour éviter de détruire ce qui marche. Notons aussi que dans le cerveau le processus peut très bien marcher avec des sources d’énergie différentes pour la mémoire et pour l’apprentissage. Comme il s’agit d’un processus ubiquiste, les fonctions auxquelles il correspond, avec les sources d’énergie utilisées doivent être codées dans le paléome. Ce processus est général : si une procédure marche, elle sera conservée (il n’est pas besoin qu’une divinité le décrète). Il a la particularité d’être myope : il est local, et n’a pas de vision globale du système auquel il participe, et cette myopie explique l’allure complètement bricolée des organismes vivants.
Quelles conclusions pour l’usine cellulaire ? En biologie synthétique, on essaie de construire sur la base d’un génome minimum fiable, on a des objectifs d’ingénierie, on veut une machine qui fonctionne, et surtout pas une machine qui improvise. Imaginez un avion qui tout d’un coup modifierait ses ailes et ses turbines, vous n’apprécieriez pas du tout d’en être passager. On souhaitera donc en général exclure les gènes qui permettent l’accumulation de l’information. On fera comme dans toutes les usines, des modèles standard, qui se périmeront, l’usine vieillira et mourra, il faudra reconstruire. C’est d’ailleurs ce qui se passe dans les productions de médicaments par génie génétique : on a des lots de semences, qui s’épuisent, et il faut à chaque fois repartir d’un stock d’autres semences. Le passage à grande échelle est ainsi sans doute impossible, ou très difficile (il faudrait asservir les démons de Maxwell aux objectifs humains). C’est un inconvénient, mais c’est aussi un avantage considérable, parce que cela minimise les risques associés à ces tentatives. On peut, bien sûr, imaginer qu’un jour notre espèce se lance dans la production d’organismes doués d’une capacité d’invention, ou d’improvisation, mais je ne crois pas qu’elle en soit aujourd’hui capable.
Le présent travail a été réalisé avec la contribution de Gang Fang, Eduardo Rocha, Andrew Martens, Agnieszka Sekowska, qui sont ici remerciés.
1 Replicating Rapid-prototyper: http://reprap.org.
2 Rolf Landauer (1927–1999), né à Stuttgart, émigra aux États-Unis en 1938, fit ses études secondaires à New York, servit dans la marine américaine, et obtint en 1950 un PhD de Harvard. Après deux ans à la NASA, il entra chez IBM à l’âge de 25 ans pour travailler sur les semi-conducteurs. Il fut l’un des grands noms de la recherche chez IBM.
3 Charles H. Bennett, né en 1943, obtint son PhD de Harvard (chimie) en 1970, et entra dans l’équipe de recherche d’IBM en 1972. Sur la base des travaux de Landauer, il montra que le calcul peut être effectué de façon logiquement et thermodynamiquement réversible, et qu’en ce qui concerne l’information, ce qui coûte est de la détruire, plutôt que de l’acquérir : un résultat contre-intuitif.


