J’aimerais d’abord dire quelques mots sur les liens possibles entre ces différentes approches ; ensuite, illustrer ce propos de manière concrète avec un exemple tiré de notre travail, où nous avons essayé de mêler de manière productive ces différents aspects en faveur de la découverte en biologie et si possible d’autres disciplines.
Au niveau le plus fondamental, la biologie systémique et la biologie de synthèse partagent une espèce de charnière faite des principes du « design » de la vie. Comment dans la nature s’implémentent un oscillateur, un interrupteur… Ces principes de « design » de la vie sont explorés par la biologie systémique dans son approche analytique. En bon français, le « design » de la vie serait désigné par le titre du livre célèbre de François Jacob publié en 1970 : La logique du vivant. La biologie de synthèse exploite cette logique du vivant que la biologie systémique explore.
Un autre aspect qui me semble digne d’intérêt regarde les relations qui existent entre l’expérimentation, l’investigation à la paillasse, l’investigation numérique suivie par les aspects théoriques. On a souvent parlé aujourd’hui de bio-inspiration, on a mis en avant le fait que la théorie peut en retour fournir des plans expérimentaux optimaux qui permettent de réduire fortement le coût de la partie expérimentale du travail, qui est en principe le coût le plus élevé de toute l’opération. Si l’on pense à la biologie de synthèse, qui a reçu ses lettres de noblesse par la publication d’un numéro spécial de Nature en novembre 2005, on pense souvent à l’ingénierie. Cela est dû à une définition, en deux mots, de la biologie de synthèse comme ingénierie de la biologie. De ce fait, cet aspect de la conception rationnelle de nouveaux objets se rattache de façon assez naturelle à l’application.
Le volet fondamental de la biologie de synthèse est donc bien réel. D’une manière générale c’est un bon outil lorsque l’on tente, par exemple, de discriminer entre contraintes et contingences. Si vous posez une question du type : il existe un élément métabolique comme le cycle de Krebs qui est relativement universel dans le monde vivant – est-ce dû à des contraintes physicochimiques ou est-ce dû à des contingences ? Notre Luca avait un cycle de Krebs. On peut se reporter également à la construction hiérarchique de systèmes qui sont au niveau de la cellule. On parlera de châssis pour les cellules minimales qui peuvent porter ces systèmes. Les champs d’application de la biologie de synthèse se situent typiquement en médecine : diagnostic, thérapeutique, médicaments et vaccins. Également dans la biotechnologie industrielle avec les biomatériaux. Du côté de l’agriculture, de l’agro-alimentaire, de l’environnement – avec la bio-énergie, les biosenseurs, la bioremédiation environnementale.
J’aimerais montrer de la façon la plus concrète possible, à travers un exemple que je connais bien et que nous avons développé, la manière dont on peut faire converger les approches « informatique » et « biologie systémique » vers une question unique avec des résultats qui se trouvent à la fois dans le domaine biologique et dans le cas précis, physique – la physique du polymère pour être précis. Qui amènent à se poser des questions nouvelles, des conceptions qui, elles, relèveraient plutôt de la biologie de synthèse. Tout part avec des questions qui ont l’air simple, et qui sembleraient résolues depuis longtemps, sur le contrôle de l’expression génique. Une expérience qui a déjà plus d’un demi-siècle : par Maaløe, un des pères fondateurs de la biologie moléculaire de l’école de Copenhague. Son expérience est très simple. Il fait pousser des bactéries dans un milieu minimal, et puis il ajoute du milieu riche et les bactéries changent de pente de croissance. Il mesure le logarithme du taux d’accumulation des ribosomes, donc un quart de la masse sèche à peu près de la bactérie ; il a deux droites. Il regarde l’intersection et constate que le changement de pente a eu lieu dans les dix à 30 secondes après le changement nutritionnel. C’était avant la découverte de l’opéron lactose. Si on parlait maintenant en termes modernes du résultat de cette expérience, on serait obligé de constater qu’en fait une centaine de gènes ont changé de niveau d’expression, brutalement et de manière concertée, en dix secondes. Les questions sont les suivantes : comment peut-on allumer ou éteindre des gènes avec une précision aussi adaptative, et comment une centaine d’entre eux peuvent-ils basculer d’une manière aussi concertée en dix secondes ?
La question n’a pas beaucoup de réponses, tout ce que je peux vous présenter c’est l’embryon d’une piste qui pourrait peut-être nous mener à un début de réponse à cette question très, très difficile. Voyons un peu : je prends le problème à l’envers, je commence par l’interprétation, et je ferai apparaître ce qui est de l’ordre des faits et ce qui est de l’ordre de l’interprétation. L’interprétation part de cela : si, dans un réseau d’interactions génétiques, l’on prend les gènes qui sont co-régulés, c’est-à-dire qui partagent un facteur de transcription (on prend ces gènes co-régulés ici dans la levure de bière ou dans le colibacille), on se rend compte qu’ils tendent à se trouver disposés à intervalles réguliers. Si maintenant on prend ce chromosome linéaire, où l’on a fait cette observation d’après la séquence, et l’on en fait une bobine, un solénoïde avec un tour par période, à ce moment là on récupère évidemment les gènes en alignement (Fig. 1). D’où la possibilité de créer des concentrations locales anormalement élevées, impliquant les deux partenaires, la protéine, le facteur de transcription, et l’ADN, c’est-à-dire les sites de liaison de ce facteur de transcription. Ajoutons à cela que si l’on regarde un autre facteur on trouvera souvent ces cibles, cette fois, partageant la même période. On a peut-être ici l’embryon d’un principe d’optimisation global du schéma transcriptionnel de toutes les cellules. D’où la possibilité éventuellement de mieux réaliser une bascule, un changement concerté, d’un grand nombre de gènes.
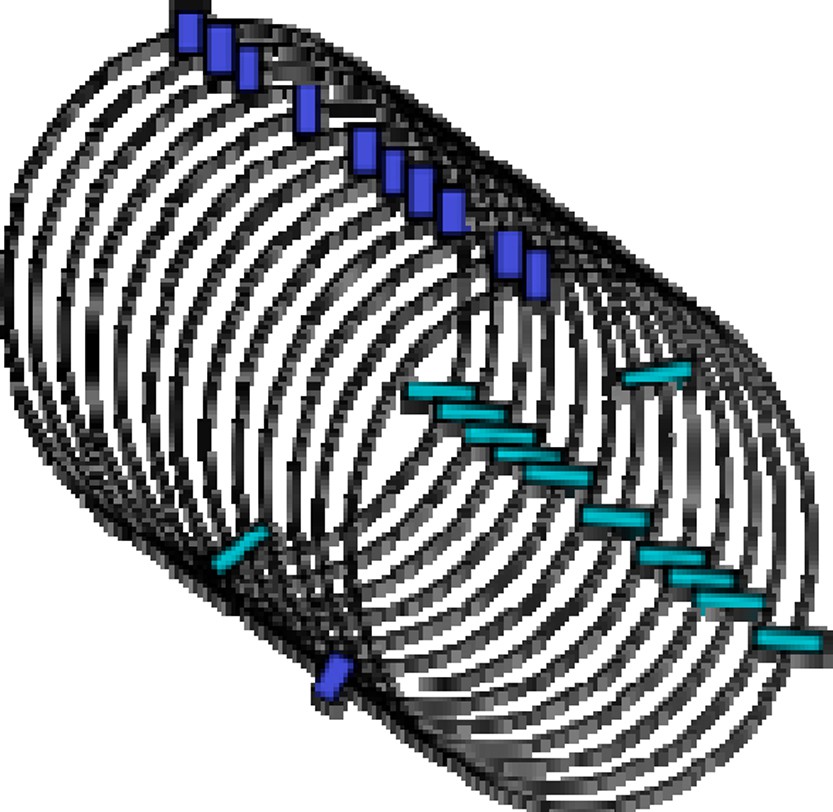
Arrangement solénoïde de la fibre chromosomique. Rectangles clairs, gènes-cibles d’un facteur de transcription ; rectangles foncés, gènes-cibles d’un second facteur. Dans cette configuration, les deux jeux de gènes-cibles sont regroupés chacun dans un petit volume, d’où des concentrations locales élevées qui entraînent l’optimisation du contrôle régulatoire de ces gènes. L’image du solénoïde ne doit pas être prise au pied de la lettre, mais plutôt utilisée pour aisément visualiser un ensemble considérable de regroupements positionnels.
Le plan de ce que je vais maintenant exposer très rapidement est le suivant. Par ailleurs, nous allons voir que la transcription se fait de façon localisée, qu’elle est sensible à la position et au regroupement. Les gènes co-régulés tendent à se positionner périodiquement. Cette périodicité favorise le regroupement tridimensionnel et concerne plusieurs facteurs de transcription. Le fait numéro un vient d’un certain nombre de laboratoires comme celui de Peter Cook. Quand on rend fluorescente l’activité de transcription, on a des points, et non pas un schéma diffus, donc il y a une focalisation des activités transcriptionnelles, chez les bactéries, comme dans un noyau de cellules humaines. Un autre fait qui est très important pour notre discussion a été récemment constaté : à un endroit comme celui-ci on retrouve en fait principalement le même facteur de transcription, il y a une certaine ségrégation, on n’aura pas de mélange de plusieurs facteurs de transcription dans un même point d’activité transcriptionnelle.
Le second point est celui-ci : la transcription est sensible à la position du gène le long de l’ADN. La première démonstration est venue de B. Müller-Hill, et puis cela a été modélisé dans le groupe de S. Leibler. Voici un facteur de transcription qui est justement le répresseur lactose de l’opéron, qui se fixe à deux sites ou davantage, avec une boucle d’ADN qui intervient entre ces deux sites. Ce qui a été montré de manière étonnante c’est que si vous ajoutez deux nucléotides dans cette boucle qui en fait 100, vous perturbez la régulation par deux ordres de grandeur environ. C’est ce que vous voyez ici entre le haut et le bas de cette courbe, au niveau de répression vous avez un facteur de 70 : c’est énorme. Maintenant il y a quelque chose d’amusant, il y a une périodicité de dix paires de bases ; c’est simplement dû au fait que la double hélice d’ADN a un tour de dix paires de base. En quelque sorte, la différence de deux ordres de grandeur sépare le cas où le facteur de transcription est lié aux deux sites et le cas où il est lié à un site.
Concernant les travaux de mon équipe, les données sont de deux types : les réseaux d’interactions transcriptionnelles qui nous disent si ce facteur a un certain nombre de cibles, et les données génomiques qui les positionnent à tel ou tel endroit de l’ADN. Nous avons mis au point un certain nombre de méthodes sophistiquées pour découvrir la régularité de ces positions. Pourquoi sophistiquées ? Parce que les données sont peu abondantes et contiennent des erreurs. Nous avons travaillé au départ sur des chromosomes artificiels (nous avons imposé les positions), puis sur les chromosomes naturels ; l’idée de base étant de passer en coordonnées solénoïdes, et de passer ensuite au solénoïde vu de face, pour établir un score qui est basé sur la formule de Shannon ; ce qui nous permet de récompenser non seulement le fort regroupement de gènes, mais aussi les arcs qui sont très longs et n’ont pas de gènes, donc aussi bien les vides que les forts regroupements. Ce nouvel algorithme a permis de découvrir d’intéressantes relations entre la régulation transcriptionnelle et la macrostructure du chromosome bactérien.
Nous avions une autre question pressante : est-ce qu’une périodicité de gènes placés le long de l’ADN favorise le regroupement en 3D de ces gènes et une mise en solénoïde du chromosome ? Pour cela il n’y a pas beaucoup de choix : l’expérience ne peut vraiment être faite sur la cellule, on peut la faire avec l’ordinateur. Nous avons mis sur pied un modèle physique du polymère dans lequel il y a une originalité : les interactions se font entre des points précis. Nous avons ainsi offert une petite contribution à la physique du polymère, et une à la biologie. Par ailleurs, au-delà de la transition de macro-phase bien connue pour un polymère, nous avons montré qu’il y a une transition de micro-phase dans laquelle on peut avoir un équilibre entre un cas où la plupart de nos sites sont regroupés et un cas où ils ne le sont pas. Du côté de la biologie, nous regardons un point où le chromosome artificiel comporte des gènes qui ont été placées périodiquement. Nous voyons un regroupement 100 % des cibles pour chacun des quatre facteurs de transcription. Nous prédisons une bonne optimisation collective de leurs régulations transcriptionnelles. En revanche, si les sites sont placés de façon aléatoire, nous avons toujours un grand nombre de gènes qui restent isolés, même s’il y a un petit regroupement. Le second point biologique réside dans le fait d’avoir plusieurs couleurs, donc plusieurs facteurs de transcription, ce qui favorise le regroupement et la mise en solénoïde ; nous avons un cas où le solénoïde est parfait. Donc voilà un certain nombre de prémisses du modèle qui sont renforcées par ce type d’observation.
Une proposition qui nous ramène assez près de la biologie de synthèse est celle des hyper-opérons. Qu’entendons-nous par là ? Dans la levure vous pouvez avoir deux gènes qui partagent un promoteur situé entre eux. Dans la bactérie vous pouvez aller plus loin car vous pouvez arranger les cistrons dans un même opéron. Cela on peut l’appeler la co-localisation en 1D. La co-localisation en 3D consiste à utiliser le principe que semble bien utiliser la levure et la bactérie, pour essayer de créer des hyper-opérons, c’est-à-dire des gènes qu’on va réguler de façon coordonnée en les disposant de manière périodique (la réalité est un peu plus compliquée). L’application biotechnologique semble assez évidente lorsque peu à peu nous essayons de sophistiquer notre approche de biologie de synthèse, quand nous essayons de mettre en place une suite d’une quinzaine de réactions catalysées par des enzymes étrangères. Il est important dans ce cas de co-réguler les différents gènes qui codent ces enzymes.
Je finis avec un petit mot en forme de point d’interrogation : on a vécu plus de 30 ans de génie génétique, est-ce qu’aujourd’hui on va commencer à s’intéresser au génie épigénétique ? Ce qui m’amuse, c’est que chaque fois que j’essaie de réfléchir à ce que sera le génie épigénétique, je retombe sur le bricolage de l’ADN, un bricolage orienté justement vers la construction de caractéristiques épigénétiques dans l’organisme que nous sommes en train de modifier, mais qui consiste à mettre des gènes à certains endroits, à bricoler avec l’ADN. Et donc le génie épigénétique, par manque d’imagination de ma part sans doute, est aujourd’hui encore basé sur les méthodes du génie génétique.


