1. Introduction
L'origine de la cellule eucaryote, l'eucaryogenèse, représente une transition évolutive majeure (« dans l'individualité »), au même titre que les origines de la vie et des organismes multicellulaires complexes [1, 2]. L'eucaryogenèse a entraîné une augmentation significative de la complexité cellulaire par rapport aux cellules procaryotes, généralement plus simples. Ce processus a été suivi d'une rapide et large diversification de plusieurs lignées. La plupart de la diversité eucaryote existante correspond à des organismes unicellulaires, généralement appelés protistes, mais plusieurs lignées comprennent des organismes multicellulaires avec des tissus plus ou moins spécialisés, tels que, entre autres, les animaux, les plantes, les champignons ou certaines algues [3, 4, 5] (Figure 1). Cette radiation phylogénétique a été accompagnée de l'adoption de différentes stratégies de vie. Bien que la plupart des eucaryotes soient des organismes phagotrophes qui peuvent engloutir des proies plus petites, certains d'entre eux ont acquis la photosynthèse et d'autres, tels que les champignons ou les oomycètes (Stramenopiles), sont devenus osmotrophes et souvent parasites, sécrétant des enzymes pour la digestion extracellulaire de molécules complexes [3, 4]. Grâce à ces différents modes de vie et en ayant généralement des cellules plus grandes, les eucaryotes ont colonisé des nouvelles niches écologiques et ont contribué à augmenter la complexité des réseaux trophiques dans les écosystèmes [6, 7].
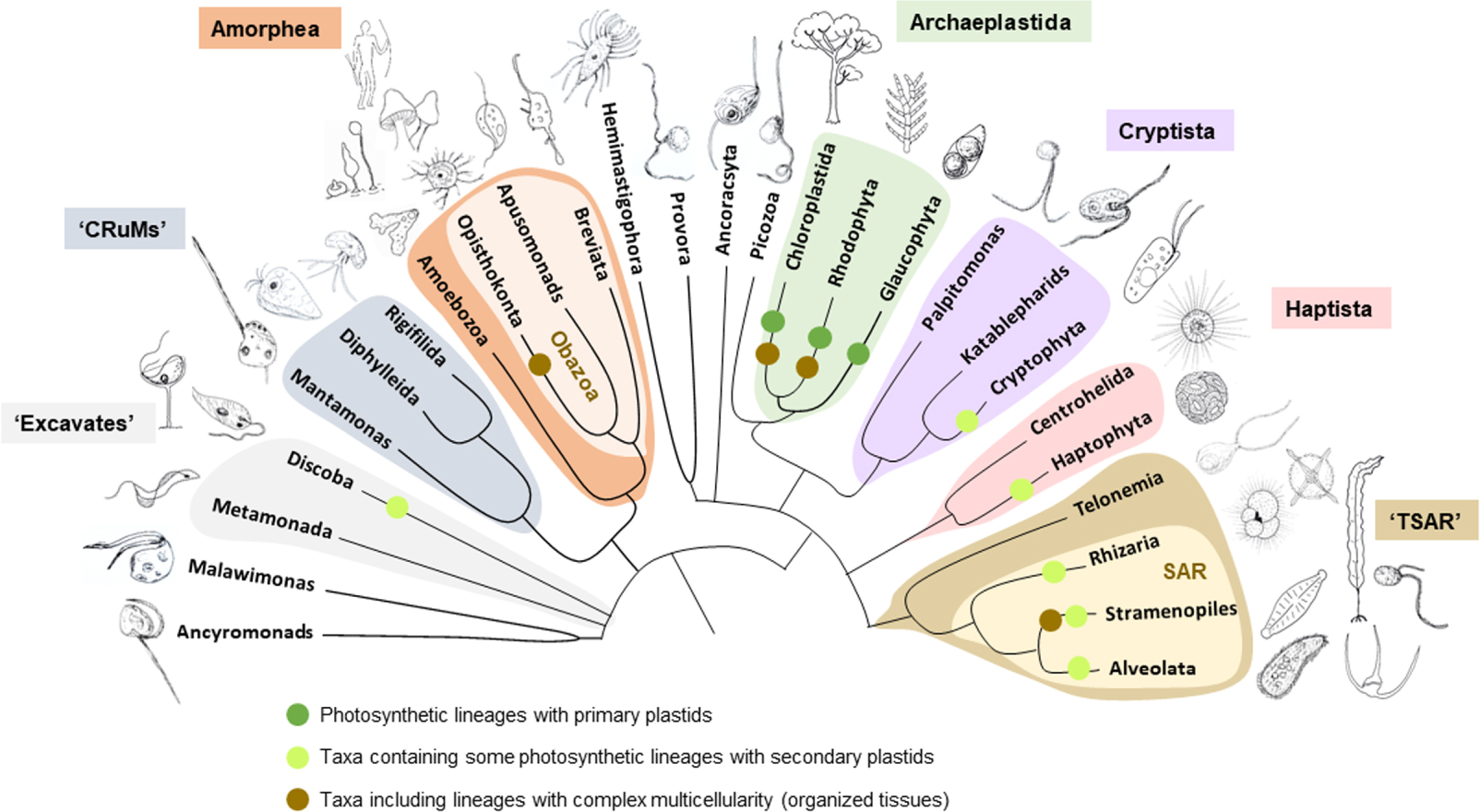
Schéma de l'arbre des eucaryotes montrant les relations phylogénétiques entre les principaux groupes et supergroupes eucaryotes. Les supergroupes eucaryotes (indiqués par des couleurs différentes) englobent généralement de nombreux phylums différents et représentent des divisions ancestrales majeures dans l'arbre eucaryote. Les dessins illustrent des exemples du type d'organismes inclus dans les taxons correspondants. La grande majorité de la diversité eucaryote correspond à des protistes unicellulaires, pour la plupart flagellés et hétérotrophes. La photosynthèse eucaryote a évolué par l'endosymbiose de l'ancêtre cyanobactérien des plastes à l'apparition d'Archeplastida, qui sont principalement photosynthétiques, et se sont ensuite propagés à d'autres lignées par endosymbiose secondaire. Les clades comprenant des membres photosynthétiques avec des plastes primaires (directement dérivés de leur ancêtre cyanobactérien commun) et des plastes secondaires (dérivés d'algues rouges ou vertes endosymbiotiques) sont indiqués par des cercles colorés. Les groupes comprenant des lignées multicellulaires complexes sont également indiqués : Opisthokonta comprend les animaux, les champignons et leurs parents unicellulaires ; Chloroplastida, les algues vertes et les plantes ; Rhodophyta, les algues rouges ; et Stramenopiles, les algues brunes et de nombreuses lignées photosynthétiques et hétérotrophes unicellulaires. Le reste des groupes comprend principalement des lignées unicellulaires (ou possédant une simple multicellularité filamenteuse ou coloniale). Le supergroupe classique « Excavates » peut ne pas être monophylétique ; il comprend le Discoba (y compris les euglènes photosynthétiques et divers protistes hétérotrophes) et le Metamonada (y compris des parasites bien connus, tels que Giardia et Trichomonas spp. et plusieurs protistes anaérobies libres). Le « CRuMs » est un clade relativement récent qui comprend plusieurs protistes flagellés hétérotrophes affichant souvent des filopodes. Les Amorphea comprennent, en plus des célèbres Opisthokonta, les Amoebozoa (amibes classiques), les apusomonades flagellées hétérotrophes et les breviates amoeboflagellés anaérobies/microaérophiles. En plus des algues vertes et des plantes (Chloroplastida) et des algues rouges (Rhodophyta), Archaeplastida comprend des algues glaucophytes, qui englobent une poignée d'espèces à faible abondance présentes dans les écosystèmes d'eau douce. Cryptista regroupe les algues cryptophytes photosynthétiques ainsi que les flagellés hétérotrophes. Haptista est un clade récemment reconnu qui englobe les héliozoaires centrohelidés et les algues haptophytes, dont certaines, comme Emiliania huxleyii, ont des exosquelettes calcifiants et sont abondantes dans les océans. Le supergroupe TSAR comprend les flagellés de telonemid et le clade SAR classiquement reconnu : Rhizaria (radiolaires, foraminifères, cercozoaires - y compris certaines espèces photosynthétiques - et lignées apparentées), Stramenopiles (lignées très diverses de nanoflagellés hétérotrophes mais aussi de diatomées unicellulaires, d'algues dorées, de bolidophytes et de phaeophytes multicellulaires ou d'algues brunes) et Alveolata (ciliés, dinoflagellés photosynthétiques et hétérotrophes et lignées apparentées). La position de la racine de l'arbre eucaryote n'est pas connue de manière fiable (indiquée par une multifurcation à la base de l'arbre).
En contraste avec la multicellularité complexe, qui est apparue indépendamment à plusieurs reprises parmi les eucaryotes [3, 5],l'origine de la cellule eucaryote a été unique, c'est-à-dire qu'elle n’a eu lieu qu'une seule fois pendant l'évolution de la vie sur Terre. Bien que la racine de l'arbre eucaryote reste à être solidement placée [5] (Figure 1), les analyses phylogénétiques moléculaires montrent que tous les eucaryotes existants forment un groupe monophylétique dans l'arbre de la vie [8],et partagent un même dernier ancêtre commun eucaryote (LECA, pour Last Eukaryotic Common Ancestor) [9, 10]. La génomique comparative permet d'inférer un LECA déjà assez complexe, qui ressemblait plus ou moins à un eucaryote moderne. Avec bien plus de 5 000 gènes [9], LECA avait déjà une grande flexibilité et complexité métabolique, possédant un cytosquelette développé et un système endomembranaire avec un noyau et des pores nucléaires, un réseau de transport vésiculaire complexe (appareil de Golgi, endosomes, etc.), un réticulum endoplasmique et des voies d'endocytose et d'exocytose. LECA était également doté de flagelles, d‘ubiquitination et de protéasomes, d’un système d’épissage d'introns, d'interférence d’ARN, de la capacité de mitose et de cytokinèse et, très probablement, de méiose et de sexe [9, 11]. LECA possédait également des mitochondries [12]. Bien que, dans les premiers temps, certaines lignées eucaryotes comprenant les Microsporidia et les Diplomonada (désignées à l’époque comme Archezoa), aient été considérées comme véritablement amitochondriales et semblaient se placer très profondément dans l'arbre eucaryote [13], il a été ensuite démontré qu'elles possédaient des organites dérivés des mitochondries et se plaçaient, avec des méthodes plus robustes d'inférence phylogénétique, au sein d'autres clades eucaryotes [12, 14]. Par conséquent, LECA possédait des mitochondries et était parfaitement comparable aux eucaryotes modernes.
Comment est apparue la cellule eucaryote ? Dans cette courte revue, loin d'être exhaustive, nous examinerons brièvement comment les idées initiales sur l'eucaryogenèse, largement basées sur des processus autogènes, ont évolué au fil du temps pour reconnaître de plus en plus le rôle crucial de la symbiose microbienne et sont devenues ouvertement symbiogénétiques de nos jours. Nous discuterons de l'impact de la découverte d'un groupe particulier de procaryotes, les archées Asgard, sur ce changement conceptuel. Nous commenterons enfin les défis actuels pour discriminer entre les modèles eucaryogénétiques existants et construire un modèle détaillé et consensuel d'eucaryogenèse.
2. Idées initiales sur l'origine des eucaryotes
2.1. Des modèles autogènes vers une vision à trois domaines primaires
La découverte et l'étude de la vie microbienne ont toujours été liées au développement d'outils technologiques, dont le premier était évidemment le microscope. Depuis l'époque d'Antonie van Leeuwenhoek, les progrès en microscopie optique ont permis de distinguer différentes formes microbiennes, ce qui a aidé à reconstruire le premier arbre phylogénétique universel basé sur les traits morphologiques par Ernst Haeckel [15]. La microscopie photonique a permis de distinguer deux types très différents de cellules, un type avec une morphologie assez simple et généralement de petite taille (génériquement appelé les bactéries) et un autre type de cellules avec une taille généralement plus grande et des formes très diverses, parfois portant une décoration à la surface (notamment certains protistes et microalgues). Au cours des premières décennies du XXe siècle, la microscopie électronique a permis de montrer que ces deux types de cellules étaient sous-jacents à des ultrastructures distinctes. En 1937, Edouard Chatton a forgé les termes « procaryote » et « eucaryote » pour eux [16], bien que la délimitation finale des procaryotes et des eucaryotes ne date que des années 1960 [17, 18]. La caractéristique définissante des eucaryotes était la présence d’un noyau (étymologiquement, du grec eu, vrai, et κάρυον, noyau, cœur). Chez les eucaryotes, la réplication de l'ADN et la transcription ont lieu dans le noyau tandis que la traduction a lieu dans le cytoplasme ; à l'inverse, dans de nombreuses cellules procaryotes, la transcription et la traduction sont souvent couplées, c'est-à-dire que la synthèse des protéines peut commencer alors que l'ARNm est en train d'être synthétisé.
Sous cette dichotomie de types structuraux cellulaires, la plupart des premiers modèles envisageaient que les cellules eucaryotes s’étaient développées à partir de cellules procaryotes (bactéries) par un processus de complexification. Ce processus serait autogène, c'est-à-dire impliquant le développement d'éléments déjà existants ou l'évolution de nouveaux éléments dans une cellule procaryote. Un exemple de tels modèles est celui proposé par Tom Cavalier-Smith en 1975, selon lequel non seulement le système d'endomembranes et le noyau, mais également les organites membranaires telles que les mitochondries et les plastes, dériveraient des thylakoïdes dans un ancêtre des eucaryotes de type cyanobactérien [19]. Beaucoup moins populaire, l'idée que les procaryotes se soient développés par réduction d'un ancêtre commun de type eucaryote a également été proposée [20, 21, 22]. Cependant, cette option laissait l'origine supposée de la complexité eucaryote à partir de systèmes précellulaires entièrement inexpliquée et ne s'est jamais imposé parmi les biologistes de l’évolution. Les modèles autogènes de transition bactérie-eucaryote suivaient un chemin linéaire (Figure 2A), mais celui-ci a rapidement été remis en question par la découverte d'un groupe d'organismes jusque-là négligé, les archées, et son impact sur l'arbre de la vie.
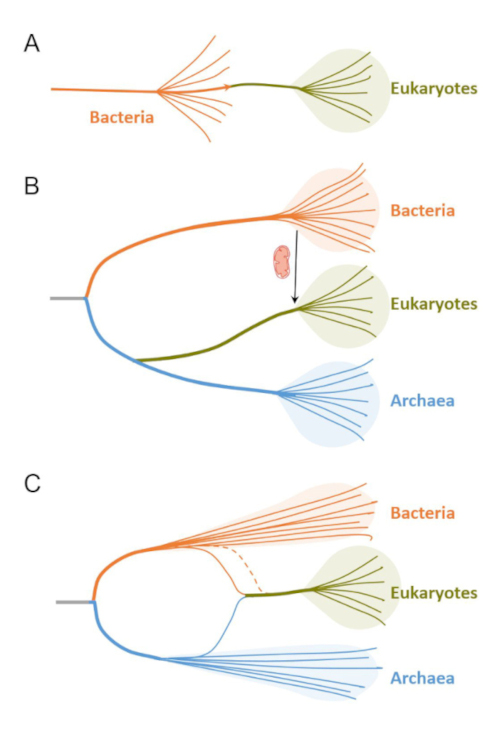
Evolution des concepts sur l'eucaryogenèse. (A) Premiers modèles selon lesquels les eucaryotes résulteraient de la complexification de bactéries plus simples. (B) Modèles basés sur l'existence de trois domaines primaires par lesquels les eucaryotes dériveraient d'une lignée proto-eucaryote sœur des archées qui ont développé des caractéristiques complexes, notamment le noyau, les endomembranes et la phagocytose, avant l'acquisition tardive de l'ancêtre alphaprotéobactérien des mitochondries. (C) Modèles privilégiés actuels par lesquels les eucaryotes dérivent de la fusion symbiotique d'un archéon lié à Asgard et d'un ou plusieurs partenaires bactériens (ligne pointillée). De ce point de vue, seuls les archées et les bactéries sont des domaines primaires ; les eucaryotes forment un troisième domaine, mais secondaire, de la vie.
La découverte des archées est une conséquence de ce que l'on appelle la « révolution moléculaire », qui a eu lieu au cours de la seconde moitié du XXe siècle et n'a cessé de se développer jusqu'à aujourd'hui. Tout système de classification biologique doit être basé sur la parenté phylogénétique. Sur la base de critères morphologiques, les espèces bactériennes étaient difficiles ou impossibles à distinguer les unes des autres et, pendant longtemps, les microbiologistes ont utilisé une taxonomie numérique reposant sur divers traits phénotypiques d'espèces clonales cultivées. Cependant, ces traits ne permettent pas d'établir des relations évolutives entre les taxons bactériens, si bien que Roger Stanier et ses collègues ont conclu avec pessimisme en 1963 que « le but scientifique ultime de la classification biologique ne peut être atteint dans le cas des bactéries » [23]. Heureusement, à peu près à cette époque, il est devenu clair que la séquence des polymères biologiques, des nucléotides ou des acides aminés, pouvait stocker de l'information évolutive [24], ce qui a ouvert la voie au développement de la phylogénie moléculaire. Sur cette base et dans le but d'établir une classification phylogénétique naturelle de toutes les formes de vie existantes, y compris les bactéries, Carl R. Woese et ses collègues ont été les premiers à utiliser des marqueurs moléculaires universellement conservés (ARNs de la petite sous-unité du ribosome) pour reconstruire un arbre phylogénétique universel [25, 26]. De manière surprenante, un clade d'organismes d'apparence bactérienne qui se développaient principalement dans des environnements extrêmes (sources chaudes et/ou acides, marais salants) ou qui étaient méthanogènes se séparaient des bactéries classiques et des eucaryotes dans des arbres phylogénétiques. Ce clade a d'abord été appelé Archaebacteria [25] avant qu’il ne soit officiellement défini comme un troisième domaine de la vie, les Archaea, avec ceux des Bacteria et des Eucarya [26]. La découverte des archées a posé un nouveau problème pour la compréhension de l'origine de la cellule eucaryote, car il y avait maintenant deux types structurels de cellules mais trois domaines phylogénétiques de la vie. D'où viennent donc les eucaryotes ?
Il est intéressant de noter que dans les premiers arbres phylogénétiques universels racinés, les archées apparaissaient comme le groupe frère des eucaryotes [26, 27, 28]. Quelques auteurs trouvaient même une plus forte similarité de certaines archées avec les eucaryotes sur la base du ribosome [29]. En outre, cette relation de parenté était cohérente avec la prise de conscience progressive du fait que les gènes et processus « informationnels » des archées, c'est-à-dire ceux impliqués dans la réplication et la réparation de l'ADN, la transcription et la traduction, ressemblaient davantage (ou uniquement) à leurs homologues eucaryotes [30, 31]. En conséquence, le scénario qui a émergé et qui a été dominant jusqu'à ces dernières années, postulait que les eucaryotes avaient évolué à partir d'une troisième lignée proto-eucaryote qui partageait un ancêtre commun plus récent avec les archées (Figure 2B). Cette troisième lignée primaire aurait développé toutes les caractéristiques eucaryotes typiques à l'exception de la mitochondrie, y compris un système endomembranaire, le noyau, un cytosquelette développé et, notamment, la phagocytose [32, 33]. La phagocytose aurait été cruciale pour l'acquisition de l'ancêtre mitochondrial dont il a été montré, à peu près au même moment, qu'il provenait d'anciennes bactéries endosymbiotiques.
2.2. L'origine symbiotique des mitochondries et des plastes
L'idée que certains organites membranaires des eucaryotes aient évolué à partir de bactéries endosymbiotiques remonte au début du vingtième siècle. En 1905, Konstantin Mereschkowsky a suggéré que les plastes provenaient d'algues bleues-vertes (cyanobactéries) [34, 35] et, en 1927, Yvan Wallin a proposé que les mitochondries provenaient de bactéries pourpres (alphaprotéobactéries) [36]. Cependant, ces idées sont restées controversées et ont disparu de la scène jusqu'à ce que, en 1967, Lynn Margulis (Sagan) les popularise à nouveau [37], plaidant pour l'importance de la symbiose dans l'évolution eucaryote [38]. En effet, les mitochondries et les plastes ne se formaient pas de novo, mais à partir de la division d'organites déjà existants et présentaient des caractéristiques qui pourraient bien être des vestiges d'une étape bactérienne libre antérieure, comme la possession d’un génome propre et des ribosomes de type bactérien [37]. La démonstration définitive de l'origine bactérienne de ces organites est venue des analyses phylogénétiques moléculaires des gènes organellaires (coxI, rbcL), qui ont permis de placer sans ambiguïté les mitochondries dans le groupe des Alphaproteobacteria et les chloroplastes dans celui des Cyanobacteria [39].
L'endosymbiose cyanobactérienne à l'origine des plastes a eu des conséquences évolutives et écologiques majeures, puisqu'elle a conduit à l'évolution des eucaryotes photosynthétiques. L'endosymbiose primaire avec l'ancêtre des plastes s'est avérée être unique et a donné naissance aux Archaeplastida, qui englobent trois clades majeurs : les algues glaucophytes ; les algues rouges ; et les algues vertes et plantes terrestres [40, 41].Par la suite, des endosymbioses secondaires (et même tertiaires) d'algues rouges ou vertes au sein d'un nombre pas encore bien établi d'hôtes eucaryotes hétérotrophes ont donné naissance à des lignées supplémentaires d'eucaryotes photosynthétiques [3, 42, 43] (Figure 1). Pendant longtemps, la lignée cyanobactérienne spécifique à l'origine des plastes est restée énigmatique. Cependant, il a été récemment démontré que l'ancêtre des plastes était lié au clade récemment découvert des Gloeomargaritales [44, 45]. Cet ordre de cyanobactéries comprend des espèces unicellulaires qui prospèrent exclusivement dans des écosystèmes d'eau douce ou terrestres, avec une préférence marquée pour les sources chaudes, ce qui suggère que la photosynthèse eucaryote a évolué dans des systèmes continentaux [44, 46]. On connaît également un deuxième événement indépendant d'endosymbiose primaire conduisant à une symbiose stable avec un niveau d'intégration élevé, bien qu'il ne soit pas aussi développé que celui des plastes. Il s'agit d'un membre du clade Synechococcus-Prochlorococcus au sein d'une amibe à thèques du genre Paulinella [47].
Comme dans le cas des plastes, avec le temps et l'augmentation du nombre de séquences mitochondriales provenant de divers eucaryotes, il est rapidement apparu que les mitochondries étaient monophylétiques et descendaient d'un ancêtre mitochondrial unique lié aux Alphaproteobacteria, qui était déjà présent chez LECA et qui était probablement aérobe facultatif [12]. Le passage de la bactérie libre à l'organite a impliqué, comme chez les plastes, le transfert de nombreux gènes vers le génome nucléaire eucaryote et la mise en place de voies de transport pour l'importation des produits fabriqués par l'hôte dans la mitochondrie [48]. Ce processus de réduction génomique a été plus drastique chez les protistes anaérobes, où les mitochondries ont perdu la respiration aérobe, et chez certaines lignées parasitaires, qui ont complètement perdu le génome mitochondrial et n'ont conservé que le codage de certains fonctions minimales, notamment l'assemblage des centres fer-soufre des protéines [49, 50]. La lignée alphaprotéobactérienne à l'origine des mitochondries reste encore à identifier. Bien que des affinités avec les Rickettsiales - qui englobent notamment des bactéries parasites intracellulaires - et les Pelagibacterales marines aient été proposées, elles résultent d'artefacts de reconstruction phylogénétique induits par un biais de composition similaire, à savoir des génomes riches en AT. Des analyses plus robustes comprenant un meilleur échantillonnage taxonomique et des méthodes de reconstruction phylogénétique plus précises placent actuellement les mitochondries en dehors des lignées alphaprotéobactériennes connues [51, 52].
2.3. Les premiers modèles symbiogénétiques sur l'origine des cellules eucaryotes
Bien que pendant la dernière partie du vingtième siècle et jusqu'à récemment, le scénario dominant de l'eucaryogenèse invoquait une troisième lignée proto-eucaryote sœur des archées qui, à un moment donné, aurait acquis des mitochondries par endosymbiose, plusieurs modèles ont commencé à remettre en question l'existence d'une telle lignée primaire pour laquelle il n'existait pas de preuves directes, surtout après avoir réalisé que LECA possédait déjà des mitochondries [12, 32]. Ces nouveaux modèles ont plutôt proposé que les eucaryotes avaient évolué à partir d'une symbiose directement établie entre les archées et les bactéries (Figure 2C).
Le premier scénario symbiogénétique de l'eucaryogenèse bien détaillé est la théorie de l'endosymbiose en série (Serial Endosymbiosis Theory, SET) proposée par L. Margulis [37, 53, 54]. Elle proposait non seulement que les mitochondries et les plastes dérivent d'anciennes bactéries endosymbiotiques, mais que l'hôte qui a acquis ces endosymbiotes avait également évolué à partir d'un événement symbiotique. Ce dernier aurait impliqué une archée sans paroi, de type Thermoplasma, et des spirochètes, qui auraient initialement établi une symbiose en oxydant le sulfure en soufre nécessaire à l'hôte [55] avant d'évoluer pour donner naissance aux flagelles eucaryotes [37, 53, 54]. En revanche, Margulis pensait que le noyau, que Mereschkowsky croyait également d'origine endosymbiotique [35], avait évolué de manière autogène [56]. Néanmoins, les preuves structurelles et phylogénétiques indiquant une origine symbiotique des flagelles eucaryotes manquaient, et à part l'idée d'une origine symbiotique pour la cellule eucaryote, l'hypothèse de Margulis n'a jamais été largement acceptée.
Dans les années 1990, à mesure que les connaissances sur la biochimie et la biologie moléculaire des archées progressaient, plusieurs modèles postulant une origine symbiotique des eucaryotes impliquant des archées et des bactéries ont été proposés indépendamment. Certains d'entre eux n'ont pas mis en avant de mécanisme particulier mais se sont appuyés sur des preuves croissantes de la nature chimérique des eucaryotes [57, 58]. En effet, si les processus informationnels eucaryotes sont de type archéen, la bioénergétique cellulaire (les gènes dits « opérationnels ») [30] et les phospholipides membranaires sont de type bactérien [58, 59]. Quelques modèles ont proposé que la symbiose métabolique, ou syntrophie, soit à la base de la symbiose eucaryogénétique [60]. L'un d'eux a été proposé par D. Searcy, qui a suggéré que les eucaryotes dérivaient d'une archée de type Thermoplasma dépendante du soufre, qui aurait incorporé une alphaprotéobactérie anaérobe facultative capable d'oxyder le sulfure en soufre [61]. Deux autres modèles étaient plus détaillés : l'hypothèse de Syntrophie et celle de l'Hydrogène (Syntrophy and Hydrogen hypotheses).
L'hypothèse de l'Hydrogène, de W. Martin et M. Müller, postule que les eucaryotes ont évolué à partir d'une archée méthanogène et d'une alphaprotéobactérie endosymbiotique, anaérobe facultative, qui fermenterait la matière organique dans des conditions anoxiques (nécessaires à la méthanogenèse), libérant de l'hydrogène qui servirait à réduire le CO2 en CH4 par l'hôte archéen [62]. L'hypothèse de Syntrophie, que nous avons proposée pour la première fois en 1998 [63] et affinée en 2006 [64], faisait appel à trois partenaires symbiotiques. Tout d'abord, une symbiose basée sur le transfert d'hydrogène inter-espèces s'établit entre une archée méthanogène endosymbiotique au sein d'une deltaprotéobactérie fermentatrice complexe, de type myxobactérien : l'hôte. Une deuxième endosymbiose impliquant une alphaprotéobactérie métaboliquement flexible, aérobe facultative mais aussi méthanotrophe, permettrait de recycler le méthane au sein du consortium symbiotique et conduirait à l'évolution de la mitochondrie. Ce modèle était ancré dans l'écologie microbienne, étant basé sur des interactions microbiennes répandues dans des milieux anoxiques [65]. Le modèle de Searcy et l'hypothèse de l'Hydrogène ont convergé pour proposer que l'endosymbiose mitochondriale et l'origine de la cellule eucaryote étaient un seul et même évènement. Les modèles de l'Hydrogène et de Syntrophie convergent vers la proposition d'une base métabolique similaire (transfert d'hydrogène entre espèces) pour la symbiose eucaryogénétique, mais diffèrent clairement quant au nombre de partenaires symbiotiques, au mécanisme spécifique de l'eucaryogenèse et aux forces sélectives mises en avant pour l'évolution du noyau eucaryote.
Malgré le soutien croissant des données phylogénomiques en faveur de l'existence de seulement deux domaines primaires, les bactéries et les archées, au lieu de trois [66, 67], ce qui a indirectement soutenu les modèles symbiogénétiques, ceux-ci sont restés moins populaires que le scénario dominant basé sur trois domaines, selon lequel les eucaryotes ont évolué à partir d'une lignée proto-eucaryote indépendante, sœur des archées. Cette idée dominante a radicalement changé avec la découverte des archées Asgard.
3. Des idées sur l'eucaryogenèse en pleine mutation
3.1. L'exploration du monde microbien : les archées d'Asgard
La « révolution moléculaire » qui a débuté dans la seconde moitié du vingtième siècle a non seulement permis de reconstruire des arbres phylogénétiques universels, mais a eu des implications supplémentaires de grande portée pour l'étude de la diversité, de la fonction et de l'évolution microbiennes dans les écosystèmes naturels. Bien que seule une infime partie des microorganismes présents dans la nature puisse être cultivée et étudiée en laboratoire, l'étude des gènes et des génomes directement à partir d'échantillons environnementaux est devenue fondamentale pour obtenir des informations sur cette « matière noire » microbienne. Depuis de nombreuses années, le séquençage d’amplicons des gènes d'ARNr 16S et 18S a permis de caractériser la diversité procaryote et eucaryote des communautés microbiennes naturelles, ce qui a conduit à l'établissement de grandes bases de données de référence contenant plusieurs milliers de séquences [68, 69]. Bien qu'extrêmement utiles pour explorer la composition des communautés dans l'espace et dans le temps, ces études de métabarcoding ne permettent pas de faire des inférences fonctionnelles basées sur les gènes codant les protéines. Pour cela, le développement de techniques de séquençage à haut débit et d'outils bioinformatiques, avec la possibilité de générer des métagénomes et des génomes assemblés à partir de métagénomes (MAG, Metagenome-Assembled Genomes), s'est généralisé et permet avec la génomique de cellules uniques d'accéder au contenu des génomes de nombreuses lignées microbiennes nouvellement identifiées [8, 70, 71, 72].
En 2015, A. Spang et ses collègues ont publié un article décrivant des MAG d'une lignée d'archées non cultivées, les Lokiarchaeota, assemblés à partir de sédiments d'eaux profondes à proximité du champ hydrothermal « Loki’s castle » dans la dorsale médio-Atlantique nord [73]. Ce groupe d'archées avait déjà été identifié par des séquences d'amplicons du gène de l'ARNr 16S comme le Deep Sea Archaeal Group (DSAG) ou le « groupe benthique marin B » [74, 75, 76], mais son potentiel métabolique et d'autres caractéristiques génomiques étaient inconnus. Les MAG de Lokiarchaeota ont révélé la présence d'un nombre relativement élevé de gènes qui avaient des homologues exclusivement chez les eucaryotes et qui n'étaient que très peu présents chez d'autres archées. De plus, les arbres phylogénomiques incluant les eucaryotes les plaçaient en tant que frères des Lokiarchaeota, nichés donc dans l'arbre des archées [73]. Cela a immédiatement suscité l'intérêt pour ce groupe d'archées et, peu de temps après, des membres de la même équipe de recherche ont publié d'autres MAG d'archées qui définirent plusieurs nouveaux groupes formant un clade avec les Lokiarchaeota : les Heimdallarchaeota, Thorarchaeota et Odinarchaeota. Ces groupes ont été collectivement appelés archées Asgard, en référence au panthéon des dieux nordiques [77]. Ces MAG ont révélé l'existence d'un nombre encore plus important de protéines dites « de signature eucaryote » (ESP, Eukaryote Signature Proteins) partagées avec les archées Asgard, notamment avec les Heimdallarchaeota, auxquelles les eucaryotes semblaient plus étroitement liés dans les arbres phylogénomiques [77] (Figure 3).

Arbre schématique des archées montrant le placement actuellement le plus probable des eucaryotes basé sur l'analyse phylogénomique de gènes hautement conservés. Les archées asgardes sont classées en classes selon la taxonomie GTDB [78]. Le cluster DPANN, nommé à l'origine d'après ses premières lignées membres reconnues (Diapherotrites, Parvarchaeota, Aenigmarchaeota, Nanoarchaeota et Nanohaloarchaeota) comprend principalement des membres parasitaires réduits [8, 70, 71, 72]. Les archées TACK (maintenant classées dans GTDB comme le phylum Thermoproteota) comprennent les taxons originaux Thaumarchaeota (GTDB Nitrosospheria), Aigarchaeota (GTDB Caldarchaeales), Crenarchaeota (GTDB Thermoprotei) et Korarchaeota (GTDB Korarchaeia) ainsi que des lignées relatives (Bathyarcheia, Methanomethylicia). Euryarchaeota, l'un des deux clades archéologiques définis par Woese avec Crenarchaeota [25, 26], est maintenant divisé en cinq phyles distincts dans GTDB (Halobacteriota, Thermoplasmatota, Methanobacteriota, Hadarchaeota, Hydrothermarchaeota).
De nombreux autres MAG d'archées Asgard ont été assemblés à ce jour. La plupart d'entre eux proviennent de métagénomes d’environnements dépourvus d'oxygène, notamment de sédiments marins et d'eau douce, de tapis microbiens ou de sources chaudes [77, 79], bien que quelques-uns aient été assemblés à partir de lacs hypersalins microoxiques [80]. Cet effort a conduit à la description du potentiel métabolique de nombreux autres clades d'archées Asgard, tels que les Helarchaeota, potentiellement impliquées dans l'oxydation anaérobe d’hydrocarbures [81] ; les Hermodarchaeota, dégradant probablement des alcanes et des composés aromatiques couplés à la réduction des nitrates [82] ; les Gerdarchaeota, qui utilisent le carbone organique et inorganique [83] ; les Sifararchaeota, qui semblent dégrader des polysaccharides et effectuer une méthylotrophie anaérobe [84] ; ou les Freyrarchaeota et les Wukongarchaeota, qui effectuent probablement de l’homoacétogenèse [85]. Bon nombre de ces groupes d'archées ont été reclassés en classes ou en ordres dans la Genome Taxonomy Database (GTDB) [78], les eucaryotes se situant toujours profondément à l'intérieur ou comme frères des Heimdallarchaeota dans les arbres phylogénomiques (Figure 3). Les capacités métaboliques affichées par les différents groupes d'archées Asgard, ainsi que celles déduites par la reconstruction métabolique pour leur dernier ancêtre commun, suggèrent que la plupart de ces archées (et leur ancêtre) sont des anaérobes stricts capables de produire et/ou de consommer de l'hydrogène (ou électrons). Ceci implique que ces archées sont principalement impliquées dans des symbioses métaboliques avec d'autres microorganismes qui se comportent comme des donneurs et/ou des accepteurs d'hydrogène (électrons) [85].
L'idée que les archées Asgard soient impliquées dans des interactions syntrophiques dans la nature semble confirmée par la culture des deux premières espèces de ce groupe, appartenant toutes deux au Lokiarchaeota. La première d'entre elles, Candidatus Prometheoarchaeum syntrophicum, vit en syntrophie avec une deltaprotéobactérie sulfato-réductrice, une archée méthanogène ou les deux [86]. Ces partenaires servent de puits pour l'hydrogène produit par l'archée Asgard. La deuxième archée Asgard cultivée, Candidatus Lokiarchaeum ossiferum, repose également sur la syntrophie avec une sulfato-réductrice du genre Halodesulfovibrio et une archée méthanogène du genre Methanogenium [87]. De manière intéressante, ces deux lokiarchées présentent une morphologie particulière avec des protrusions fines et plus ou moins longues qui peuvent parfois s'entrelacer [86, 87]. Elles révèlent également la présence d'un cytosquelette complexe à base d'actine, comme prédit à partir des séquences des MAG [88].
L'exploration de près de deux cents MAG d'archées Asgard a conduit à l'identification de plus de 500 ESP [85]. La plupart d'entre elles sont impliquées dans le transport intracellulaire, la sécrétion et le transport vésiculaire. Parmi celles-ci, on trouve de nombreuses GTPases, de l'ubiquitine et des protéines ESCRT-III, qui sont généralement impliquées dans le remodelage des membranes et le transport vésiculaire chez les eucaryotes. Cela suggère que le système ESCRT couplé à l'ubiquitine des eucaryotes a évolué par duplication et diversification de gènes à partir d'ancêtres archéens Asgard [89]. Les ESP comprennent également des actines et des protéines telles que les profilines [90] et la gelsoline/cofilines [91], impliquées dans la formation et la dynamique du cytosquelette d'actine. De plus, les homologues archéens de ces protéines peuvent complémenter les protéines eucaryotes [90, 91], indiquant que ces archées possèdent un cytosquelette d'actine complexe et suggérant que le cytosquelette eucaryote a évolué à partir de son homologue archéen. La situation est similaire pour la tubuline, dont les archées d'Asgard possèdent des homologues beaucoup plus proches des tubulines eucaryotes que FtsZ, un homologue procaryote de la même superfamille de gènes impliqué dans la formation de l'anneau de division cellulaire [92]. En outre, les tubulines des archées d'Asgard forment des protomères et des protofilaments très semblables aux microtubules eucaryotes, bien qu'elles forment des anneaux à la manière de FtsZ, constituant ainsi une sorte d'intermédiaire fonctionnel [93].
3.2. Eucaryogenèse par symbiogenèse
La découverte des archées Asgard, qui possèdent beaucoup plus de gènes en commun avec les eucaryotes que les autres archées, ainsi que la position phylogénétique des eucaryotes au sein de ces archées basée sur l’analyse de gènes hautement conservés, soutient fortement l’idée que les eucaryotes ont évolué par une sorte d’intégration symbiotique impliquant au moins deux partenaires procaryotes : une archée Asgard et l'ancêtre alphaprotéobactérien des mitochondries [73, 77, 94, 95, 96] (Figure 2C).Les données actuellement disponibles soutiennent en effet l'existence de seulement deux domaines primaires, Archaea et Bacteria [97]. Les eucaryotes constituent donc un troisième domaine de la vie, mais secondaire.
Bien que les symbioses microbiennes soient abondantes dans la nature, en particulier dans les environnements privés d'oxygène, l'eucaryogenèse ne s'est produit qu'une seule fois, ce qui suggère que le mécanisme qui a conduit à l'intégration stable des partenaires symbiotiques pour donner naissance à une cellule complexe n'était pas facile et/ou impliquait des aspects contingents difficiles à reproduire de novo. Un tel processus a probablement nécessité une période de coévolution plus ou moins longue au cours de laquelle de nombreux traits typiques des eucaryotes se sont développés [38]. Une première étape aurait été de rendre la symbiose obligatoire, ce qui aurait pu se produire par le transfert d'un ou plusieurs gènes essentiels du partenaire - ou des partenaires - bactérien(s) vers l'archée, suivi d'une perte de gènes chez le donneur bactérien. À partir de cette étape, le consortium aurait évolué comme une seule unité sélective et le génome de l'archée serait progressivement devenu le futur génome nucléaire, intégrant de plus en plus de gènes bactériens. Différents moyens de transfert horizontal de gènes, de la transformation naturelle à la médiation de virus, transposons et autres éléments génétiques mobiles, ont pu a priori intervenir pour véhiculer des gènes entre le génome des endosymbiontes et celui de l'hôte. Beaucoup de ces gènes codaient pour des fonctions redondantes chez l'archée. En conséquence, beaucoup d'entre eux ont été perdus, mais d'autres ont été conservés et sous-fonctionnalisés, donnant lieu à de nouvelles fonctions [38]. La duplication et la sous-fonctionnalisation de gènes semblent être à l'origine de nombreuses familles de gènes chez les eucaryotes [98, 99]. Certains gènes dupliqués, libérés des contraintes sélectives d'origine, ont évolué bien au-delà de la reconnaissance d'homologie, tandis que d'autres gènes ont probablement évolué de novo. Le mélange de gènes archéens et bactériens et l'accélération du taux d'évolution de nombre d'entre eux expliquent aisément l'origine chimérique des génomes eucaryotes ainsi que l'apparition de gènes eucaryotes spécifiques.
En plus de l'évolution du génome, l'évolution de certains traits typiques des eucaryotes, tels que le cytosquelette et la plupart des systèmes de remaniement membranaire, peut être attribuée en grande partie au partenaire archéen. D'autres, comme les fonctions opérationnelles liées à la mitochondrie ou aux phospholipides membranaires, peuvent être attribuées à des ancêtres bactériens [38, 94, 100]. En résumé, l'eucaryogenèse s'est produite par symbiogenèse, c'est-à-dire par l’intégration physique de partenaires procaryotes au cours d'un processus coévolutif qui a impliqué le chimérisme ainsi que la formation de caractères de novo, partiellement facilitée par le taux d'évolution accru de nombreux gènes dupliqués et/ou l'évolution de mécanismes spécifiques eucaryotes véritablement nouveaux.
3.3. Modèles actuels d'eucaryogenèse
Bien que les modèles actuels sur l'origine de la cellule eucaryote favorisent une symbiose entre une archée de type Asgard et au moins une alphaprotéobactérie, ils varient sur les mécanismes (le comment) et les forces sélectives (le pourquoi) impliqués dans le processus. Sur la base du type de symbiose établie par les archées Asgard avec d'autres microbes dans la nature, les interactions syntrophiques semblent être la base la plus probable pour la symbiose eucaryogénétique initiale [85, 96, 101, 102]. Actuellement, quatre modèles différents proposent un scénario évolutif plus ou moins détaillé basé sur des interactions syntrophiques. D'autres modèles suggèrent un scénario plus ou moins développé mais ne mettent pas en avant de base particulière pour la symbiose originelle entre l'archée Asgard et son ou ses partenaires bactériens (Figure 4).
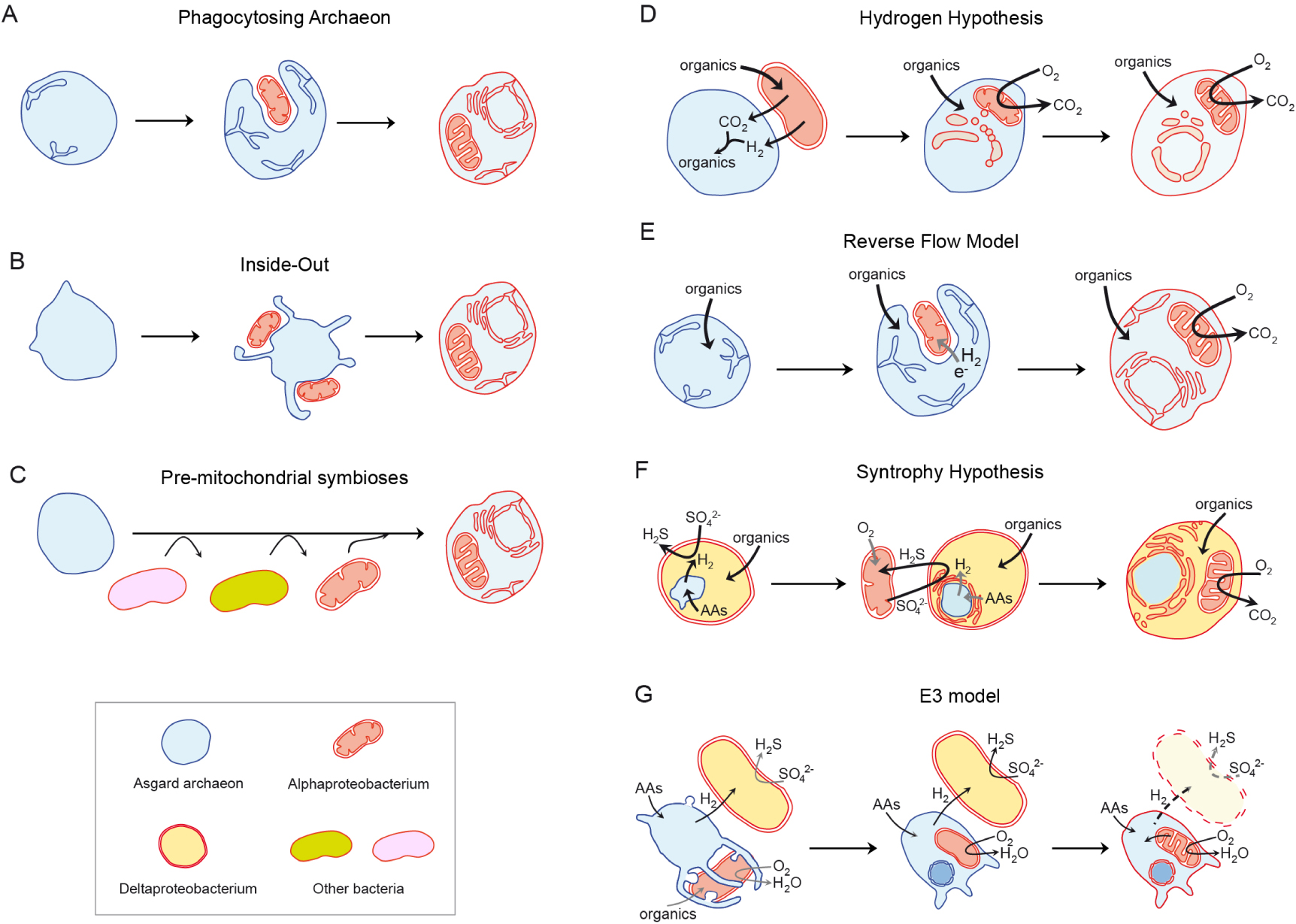
Modèles actuels d'eucaryogenèse basés sur la fusion symbiotique de partenaires archéaux et bactériens. (A–C) Représenter une sélection de scénarios sans base spécifiée pour la symbiose. (D–E) Correspondent à des modèles plus détaillés postulant des interactions syntrophiques spécifiques. (A) Modèle d'archéon phagocytaire : un archéon développe un cytosquelette, des endomembranes et une phagocytose avant l'engloutissement de l'ancêtre mitochondrial [103]. (B) Modèle Inside-Out : un archéon développe des extrusions qui engloutissent progressivement l'ancêtre mitochondrial [104]. (C) Modèle de symbioses pré-mitochondriales : plusieurs partenaires symbiotiques bactériens apportent des gènes et des traits à un archéon évoluant en proto-eucaryote avant la symbiose mitochondriale [100, 105]. (D) Hypothèse de l'hydrogène : une alphaprotéobactérie en fermentation libérant de l'hydrogène devient un endosymbiote dans un archéon de type Asgard et déclenche l'eucaryogenèse [106]. (E) Modèle de flux inverse : un archéon complexe producteur d'hydrogène devient phagotrophe et engloutit son partenaire alphaprotéobactérien en interaction [101]. (F) Hypothèse de syntrophie : un archéon de type Asgard produisant de l'hydrogène devient un endosymbiote dans une deltaprotéobactérie consommant de l'hydrogène et réduisant les sulfates ; les mitochondries dérivent d'une alphaprotéobactérie facultativement aérobie et oxydant les sulfures qui devient également endosymbiotique dans le consortium [107]. (G) Modèle E3 (Entangle-Engulf-Endogenize) : un archéon Asgard producteur d'hydrogène en symbiose avec une deltaprotéobactérie sulfato-réductrice change ce partenaire par l'ancêtre mitochondrial facultativement aérobie [86].
Parmi ces derniers, le scénario de l’Archée Phagotrophe postule que les eucaryotes ont évolué à partir d'une archée qui aurait développé un cytosquelette complexe, la capacité de produire et de remodeler des endomembranes et la phagocytose, ce qui aurait été nécessaire avant l'acquisition de l'ancêtre alphaprotéobactérien des mitochondries [103] (Figure 4A). Un modèle comparable serait le modèle « Inside-Out », qui inverse le scénario précédant en proposant qu'au lieu d'endomembranes, une archée Asgard aurait développé des extrusions cellulaires pour entourer progressivement les épisymbiotes alphaprotéobactériens [104]. Ces derniers finiraient par être complètement engloutis par un processus qui pourrait être assimilé à une « phagocytose lente » (Figure 4B).
Un autre modèle qui n'invoque explicitement aucune base spécifique pour les interactions symbiotiques à l'origine de la cellule eucaryote est l'hypothèse des Symbioses Pré-mitochondriales (ou des Symbioses en Série) [100, 105] (Figure 4C). Selon ce modèle, une symbiose entre un hôte archéen et l'ancêtre alphaprotéobactérien des mitochondries ne peut pas expliquer la nature chimérique complexe déduite pour l'ancêtre eucaryote. En particulier, la plupart des gènes d'ascendance procaryote supposés être présents chez LECA sont d'origine bactérienne, et non archéenne, et parmi ceux-ci, seule une fraction relativement mineure est d'origine alphaprotéobactérienne claire (ou, plus largement, protéobactérienne). Beaucoup de gènes bactériens chez LECA sont plutôt affiliés, entre autres, aux Deltaproteobacteria, Actinobacteria, Bacteroidetes ou Firmicutes (Figure 5). En outre, les gènes d'origines bactériennes spécifiques semblent être corrélés à des fonctions cohérentes, ce qui serait en accord avec des types particuliers de fonctions ayant été transférées à l'hôte archéen à partir de partenaires symbiotiques pré-mitochondriaux privilégiés au cours du temps [100]. En conséquence, ce modèle postule que des interactions symbiotiques successives avec d'autres bactéries ont précédé et facilité l'endosymbiose mitochondriale qui s'est finalement cristallisée dans l'origine de la cellule eucaryote [105].
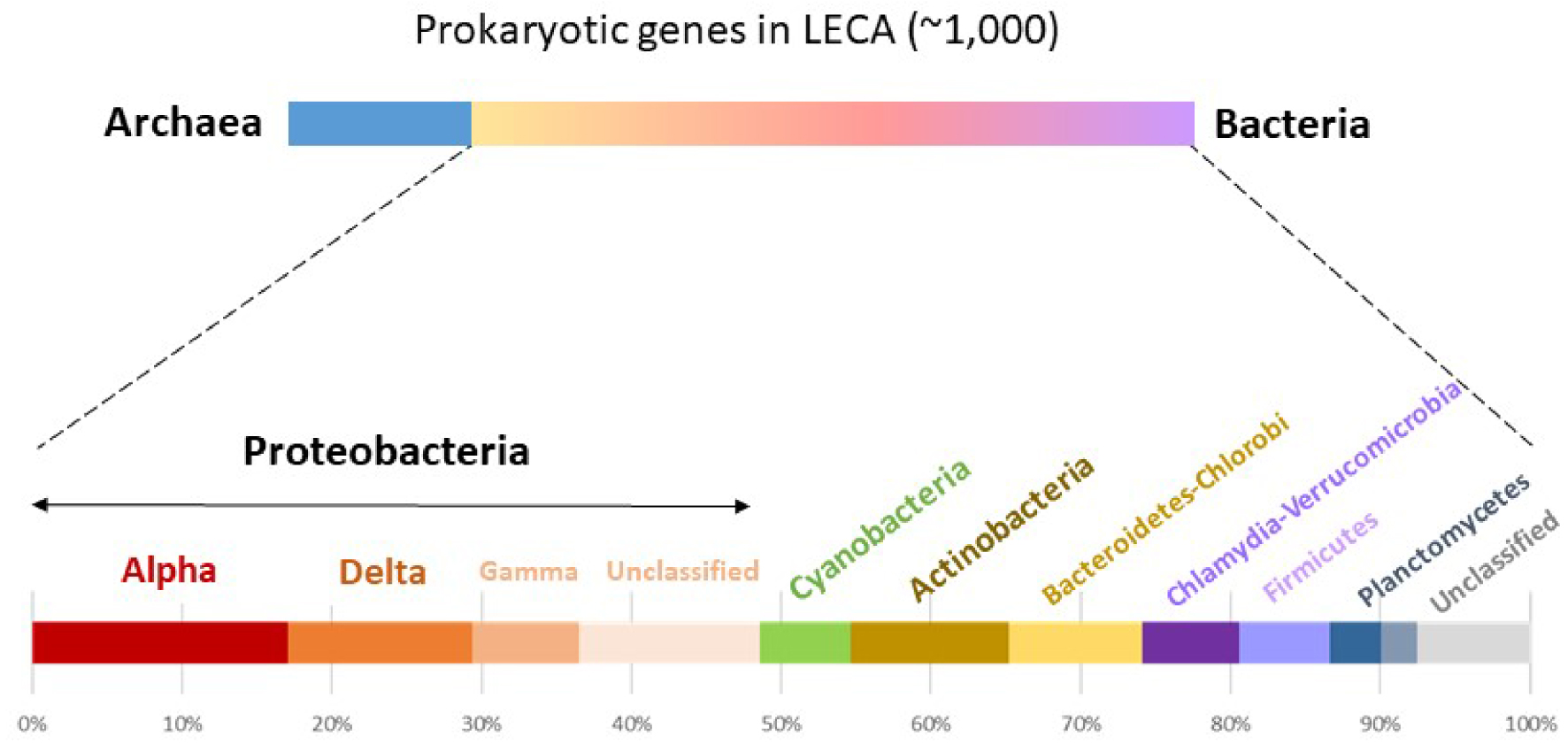
Origines phylogénétiques des gènes procaryotes présumés avoir été présents dans le LECA. Les chiffres et les proportions relatives sont basés sur la réf. [100, 105].
Parmi les modèles syntrophiques actuels pour l'eucaryogenèse, nous trouvons des versions révisées des hypothèses de l'Hydrogène et de Syntrophie, qui prennent désormais en compte un partenaire archéen de type Asgard. L'hypothèse révisée de l'Hydrogène postule que l'incorporation d'une alphaprotéobactérie productrice d'hydrogène dans un hôte archéen autotrophe dépendant de l'hydrogène aurait déclenché l'eucaryogenèse [106] (Figure 4D). À l'inverse, le modèle du « Flux Inverse », qui est un modèle d'Archée Phagotrophe adaptée à la syntrophie, invoque une archée Asgard productrice d'hydrogène qui développe progressivement un système endomembranaire et la phagocytose, ce qui lui permettrait ensuite d'engloutir une alphaprotéobactérie consommatrice d'hydrogène (ou d'électrons) (future mitochondrie) [101] (Figure 4E).
L'hypothèse révisée de Syntrophie (ou modèle de Syntrophie HS) postule que les eucaryotes ont évolué à partir de l'incorporation initiale d'une archée Asgard productrice d'hydrogène (futur noyau) au sein d'un hôte deltaprotéobactérien sulfato-réducteur avant l'acquisition de l'ancêtre alphaprotéobactérien des mitochondries, aérobe facultatif et oxydateur de sulfures (Figure 4G). Il s'agit de l'un des modèles les plus détaillés en ce qui concerne le contexte écologique de la symbiose et l'évolution du métabolisme, du génome et du système endomembranaire, y compris le noyau [107]. Le modèle E3 (Entangle-Engulf-Endogenize) proposé par l'équipe qui a obtenu la première archée Asgard en culture [86], postule que les eucaryotes ont évolué à partir de l'internalisation progressive d'une alphaproteobactérie (future mitochondrie), potentiellement un parasite, à l'intérieur d'une archée Asgard productrice d'hydrogène. Sur la base des interactions syntrophiques réelles observées chez Ca. Prometheoarchaeum, une symbiose tripartite initiale de ces deux partenaires avec une deltaprotéobactérie réduisant le sulfate est proposée, mais cette dernière disparaîtrait complètement du consortium dans les étapes suivantes (Figure 4F). En ce sens, bien que trois partenaires initiaux soient impliqués, seuls deux, un hôte archéen et l'ancêtre mitochondrial, évolueraient vers la cellule eucaryote, comme le suggèrent la plupart des autres modèles symbiogénétiques.
D'autres modèles, souvent des variantes des précédents, ont été proposés. Par exemple, un modèle plus récent postule l'intervention d'un symbiote supplémentaire à l'origine de la symbiose mitochondriale. Une seconde bactérie aurait coopéré avec l'ancêtre mitochondrial pour tuer les proies à l'aide de toxines et d'espèces réactives de l'oxygène avant d'évoluer vers une symbiose digestive basée sur l'acidification des vacuoles protophagocytaires par l'exportation de protons issus de la respiration [108]. Cependant, la plupart de ces scénarios d'eucaryogenèse est partielle et ne rend pas compte de tous les aspects impliqués dans l'évolution de la cellule eucaryote, laissant de nombreuses questions ouvertes.
4. Questions ouvertes
Certaines questions sur l'origine de la cellule eucaryote, telles que quand et où les eucaryotes ont évolué ou quelles lignées spécifiques d'archées Asgard et d'alphaprotéobactéries ont participé à l'eucaryogenèse, ont des réponses partielles ou des réponses qui seront affinées avec des données supplémentaires et l'amélioration des analyses phylogénomiques et de datation moléculaire. Les réponses à d'autres questions, telles que le nombre de partenaires ayant participé à la symbiose ou la façon dont les endomembranes eucaryotes et le noyau ont évolué, réfuteront certains modèles et aideront potentiellement à affiner les autres. Dans ce qui suit, nous évoquons brièvement certaines des interrogations actuelles sur l'eucaryogenèse.
Combien et quels partenaires bactériens ? Le fait de savoir quelles lignées spécifiques d'archées Asgard et d'Alphaproteobacteria sont les plus proches des partenaires procaryotes impliqués dans l'eucaryogenèse fournira des indices supplémentaires sur le potentiel métabolique du consortium symbiotique, et aidera probablement à mieux élucider l'évolution de certains traits eucaryotes. L'exploration de la diversité microbienne dans des écosystèmes naturels enrichis en archées Asgard pourrait aider à identifier de nouvelles lignées potentiellement plus proches de ces ancêtres procaryotes. Il est particulièrement crucial de déterminer si des partenaires bactériens supplémentaires ont participé à l'eucaryogenèse. En particulier, plusieurs modèles impliquent plus ou moins explicitement des deltaprotéobactéries [86, 100], bien que l'hypothèse de Syntrophie soit la plus détaillée quant à leur participation au processus [107]. Si l'on constate que certaines deltaprotéobactéries possèdent plus de gènes/traits en commun avec les eucaryotes, cela peut constituer une preuve de leur implication dans l'eucaryogenèse. Par exemple, l’origine phylogénétique de quelques voies eucaryotes est myxobactérienne (Deltaproteobacteria), y compris la bêta-oxydation mitochondriale d'acides gras [109] et, plus remarquablement, étant donné que les stérols membranaires sont souvent considérés des marqueurs typiques des membranes eucaryotes, la biosynthèse des stéroïdes [110]. Néanmoins, il sera difficile de déterminer si ces voies ont été acquises par le partenaire archéen Asgard ou par un stade symbiotique intermédiaire pré-LECA via transfert horizontal de gènes (HGT, pour Horizontal Gene Transfer) à partir de bactéries coexistantes dans l'environnement ou via ce que l'on appelle souvent le transfert endosymbiotique de gènes (EGT, pour Endosymbiotic Gene Transfer) à partir de symbiotes obligatoires prenant part au consortium eucaryogénétique. En outre, des situations intermédiaires sont possibles, avec deux ou quelques partenaires symbiotiques bactériens supplémentaires participant à l'eucaryogenèse, mais avec des transferts horizontaux de gènes provenant de donneurs bactériens externes aux stades LECA ou pré-LECA.
Quand et où les eucaryotes ont-ils évolué ? Les données actuelles basées sur des microfossiles qui sont interprétés sans ambiguïté comme des eucaryotes, avec des traits indiquant un cytosquelette développé et une décoration externe, soutiennent l'idée que les eucaryotes avaient déjà évolué au Protérozoïque moyen (1,5-1,6 Ga) [111, 112]. Ces dates sont cohérentes avec celles obtenues par certaines analyses de datation moléculaire, qui suggèrent que LECA a vécu dans la période 1,0 - 1,6 Ga [113]. Au fur et à mesure de l'amélioration du registre de microfossiles, mais aussi de l'échantillonnage des génomes au sein de l'arbre des eucaryotes et des méthodes de datation moléculaire, il est envisageable de voir se réduire l’incertitude de ces dates. Cependant, il pourrait être plus difficile d'établir la date à laquelle l'eucaryogenèse a commencé. Une limite maximale probable est celle de l'évolution de la photosynthèse oxygénique et le début de l'oxygénation de l'atmosphère, qui s'est produite vers 2,4-2,5 Ga, marquant le début du Grand Evénement d'Oxydation (GOE, Great Oxydation Event) [107, 114]. La disponibilité de l'oxygène aurait favorisé l'évolution de la respiration aérobe, dangereuse en raison des dommages oxydatifs qui en résultent mais aussi beaucoup plus efficace en termes de rendement énergétique que les respirations anaérobes. L'ancêtre des mitochondries était probablement un aérobe facultatif, respirant l'oxygène mais aussi capable de respirer le fumarate ou le nitrate et de fermenter. Il vivait probablement dans des environnements microoxiques et aurait pu se déplacer le long du gradient d'oxydo-réduction établi dans les sédiments ou les tapis microbiens. Cela aurait facilité son interaction avec les archées Asgard [107], qui sont pour la plupart et ancestralement des anaérobes stricts, situées dans les couches profondes des sédiments [77, 85].
La phagotrophie était-elle une condition nécessaire à l'eucaryogenèse ? Cette question est étroitement liée à celle de l'évolution précoce ou tardive des mitochondries, qui est souvent considérée comme discriminante pour les modèles eucaryogénétiques. Pour l'hypothèse de l'Hydrogène, une phagotrophie de type eucaryote n'était pas strictement nécessaire pour incorporer l'endosymbiote alphaprotéobactérien dans l'archée Asgard et cette endosymbiose initiale était la cause déclenchant l'eucaryogenèse [62, 106].En revanche, pour le modèle de l'Archée Phagotrophe et ceux apparentés [101, 103], l'évolution d'un système endomembranaire et de la phagocytose était obligatoire pour que l’engloutissement de bactéries devienne possible ; en un sens, un processus eucaryogénétique déjà engagé était nécessaire pour que l'ancêtre mitochondrial soit incorporé. Ces derniers modèles reposent sur l'idée que les procaryotes ne possèdent pas de cytosquelette complexe ni de système endomembranaire. Cependant, de nombreux procaryotes possèdent des systèmes endomembranaires, notamment les thylakoïdes des cyanobactéries et les compartiments membranaires des protéobactéries méthylotrophes, des bactéries magnétotactiques ou des planctomycètes anammox [115, 116, 117]. Certaines bactéries, comme le planctomycète Gemmata obscuriglobus 118] et les membres du groupe des Atribacteria [119], possèdent même des compartiments de type nucléaire. Les endomembranes chez les archées ont été rarement décrites mais semblent exister également [120]. De même, il existe des endosymbiotes bactériens dans d'autres bactéries, bien que le mécanisme d'internalisation soit inconnu [121]. Enfin, certaines bactéries planctomycètes peuvent réaliser une phagocytose via un nouveau mécanisme encore mal connu [122]. Ces observations suggèrent que les procaryotes, notamment les bactéries, peuvent développer des endomembranes et engloutir d'autres bactéries sans avoir besoin d'une phagocytose de type eucaryote. Ainsi, d'un point de vue mécanistique, un processus eucaryogénétique déjà en cours ne semble pas être une exigence pour l'incorporation de procaryotes endosymbiotiques.
Comment et pourquoi le noyau a-t-il évolué ? Bien que le noyau soit sans doute la caractéristique la plus singulière de la cellule eucaryote, peu de modèles d'eucaryogenèse abordent réellement son origine. La membrane nucléaire est continue avec le réticulum endoplasmique et, d'un point de vue mécanistique, il semble raisonnable de penser que les deux ont évolué ensemble par l'invagination de la membrane plasmique grâce à des systèmes de remodelage de la membrane, principalement d'origine archéenne (ESCRT, cytosquelette d'actine, GTPases) [89, 123, 124, 125, 126]. Cependant, l'évolution des caractéristiques plus spécifiques, comme les pores nucléaires, restent plus énigmatiques [94]. Certains modèles ont même spéculé que les virus étaient à l'origine du noyau eucaryote [127, 128], notamment sur la base de la ressemblance supposée des "usines virales" (lieux subcellulaires où se déroulent la réplication et la morphogenèse virales chez certains eucaryotes) [129] avec le noyau. Les virus géants étaient les candidats favoris pour une hypothétique eucaryogenèse virale, car ils contiennent plusieurs homologues de gènes cellulaires et, sur cette base, ont même été proposés comme un quatrième domaine de la vie frère des eucaryotes [130, 131]. Cependant, contrairement aux cellules et à leurs systèmes membranaires, qui se divisent à partir de systèmes préexistants (omnia cellula e cellula), les virus et les cellules (ou les noyaux cellulaires) n'ont aucune continuité structurelle. Les virus sont assemblés de novo à chaque génération ; les potentiels mécanismes qui expliqueraient une continuité évolutive et physique supposée entre les usines virales et les noyaux cellulaires font défaut. En outre, des analyses phylogénétiques utilisant des modèles complexes d'évolution des séquences et un échantillonnage taxonomique plus complet ont clairement montré que les gènes de type cellulaire des virus géants ont été transférés aux génomes viraux à partir de leurs hôtes et, par conséquent, ne reflètent pas une ascendance virale des eucaryotes [132, 133, 134, 135, 136].
Les forces sélectives qui ont conduit à l'évolution du noyau restent mystérieuses [94]. Certains auteurs ont suggéré que le compartiment nucléaire a évolué pour empêcher le cisaillement des chromosomes par les mouvements du cytosquelette pendant la mitose [137, 138]. Cependant, de nombreux eucaryotes désintègrent la membrane nucléaire au cours de la mitose (mitose ouverte) et s'accommodent bien des mouvements du cytosquelette ; de plus, les eucaryotes possèdent de nombreux chromosomes linéaires et de puissants mécanismes de réparation de l'ADN, ce qui devrait limiter toute rupture mécanique potentielle induite par les mouvements du cytosquelette [94]. Dans le cas de l'hypothèse de Syntrophie, le noyau aurait eu une origine partiellement autogène. Le génome avait une origine archéenne, dérivant de l'endosymbiote archéen, mais la membrane nucléaire aurait évolué à partir d'un système endomembranaire d'origine bactérienne qui se serait développé avec des fonctions de sécrétion pour transporter des enzymes hydrolytiques, dont la synthèse serait devenue progressivement centralisée chez l'archée, vers le périplasme bactérien. Le développement de la membrane nucléaire aurait favorisé le découplage de la transcription et de la traduction, facilitant ainsi la prolifération des introns, et aurait ensuite été retenue par l’évolution pour empêcher la synthèse aberrante de protéines après cette prolifération des introns [64, 107]. La prolifération des introns aurait été le principal déclencheur de l'évolution de la membrane nucléaire dans le cadre de l'hypothèse de l'Hydrogène ; la membrane nucléaire aurait évolué pour découpler la transcription et la traduction, évitant ainsi la synthèse délétère de protéines aberrantes [139]. Cependant, dès qu'un intron interrompt un gène essentiel en l'absence d'un mécanisme efficace d'épissage et de découplage transcription-traduction, la sélection naturelle éliminerait ces cellules. Ainsi, un mécanisme sélectif expliquant les étapes intermédiaires de l'évolution du système endomembranaire et de la membrane nucléaire doit dans ce cas être proposé.
Membranes eucaryotes de type bactérien, quelle origine ? Les phospholipides qui forment la membrane plasmique sont radicalement différents chez les bactéries et les archées en termes de constituants et de propriétés. Les phospholipides des membranes eucaryotes sont de type bactérien. Les phospholipides archéens sont composés de chaînes latérales isoprénoïdes liées par des liaisons éther à une molécule de glycérol-1-phosphate [140, 141]. Ce type de phospholipides membranaires confère aux archées une plus grande résistance à diverses conditions extrêmes ; ils empêchent notamment la fuite de protons à haute température [59]. Les phospholipides bactériens sont composés d'acides gras liés par des liaisons ester à une molécule de glycérol-3-phosphate. Il est connu depuis longtemps que plusieurs bactéries peuvent aussi utiliser des liaisons éther, notamment les thermophiles [142, 143], mais aussi les eucaryotes. Par conséquent, bien que la liaison éther/ester constitue une partie de ce que l'on appelle le « clivage lipidique » [144], le véritable fossé entre les phospholipides archéens et bactériens est déterminé par la stéréochimie opposée des molécules de glycérol-phosphate [141, 145], en combinaison avec la nature des chaînes latérales. Les archées et les bactéries synthétisent toutes deux des acides gras et des isoprénoïdes, mais ceux-ci sont exclusivement incorporés par l'un ou l'autre domaine dans leurs phospholipides membranaires [146]. À l'exception de l'hypothèse de Syntrophie, qui propose un hôte bactérien et, par conséquent, l'origine bactérienne de la membrane plasmique et des endomembranes (la membrane archéenne serait perdue au cours de l'évolution du noyau [64, 107]), la plupart des modèles d'eucaryogenèse postulent un hôte archéen [86, 101, 104]. Par conséquent, ces modèles doivent invoquer une transition phospholipidique d’une membrane de type archéen vers une membrane de type bactérien, ce qui est difficile à expliquer si l'on tient compte du fait que la membrane archéenne, y compris son protéome - qui était adapté à la physicochimie locale des lipides archéens - était adaptée à l’interaction avec l'environnement. Il est difficile de voir quelle force sélective pourrait promouvoir un changement aussi radical des phospholipides membranaires et du protéome associé [94]. Néanmoins, bien que les transitions entre membranes archéennes et bactériennes n'aient jamais été observées dans la nature, les liposomes de phospholipides hétérochiraux (mélange archéen + bactérien) semblent être stables [147]. Des tentatives pour réaliser une transition phospholipidique membranaire de type bactérien à archéen ont été effectuées chez Escherichia coli en surexprimant de manière hétérologue des gènes de biosynthèse de phospholipides archéens [148]. Cependant, les niveaux d'expression qui conduisent à l'incorporation de plus de 30% de phospholipides archéens dans E. coli entravent la croissance, entraînent une division cellulaire anormale et la sécrétion de vésicules et sont, in fine, délétères [148]. Des expériences futures devraient montrer si une telle transition est vraiment possible en conditions expérimentales de laboratoire. Néanmoins, il n'est pas certain que cette transition soit possible dans les communautés microbiennes naturelles où les eucaryotes ont évolué, car la capacité de survie des étapes de transition peut avoir été affectée négativement, empêchant que cette transition hypothétique se produise.
5. Perspectives
L'exploration de la diversité microbienne dans des écosystèmes peu étudiés et privés d'oxygène a facilité la découverte des archées Asgard, qui possèdent de nombreux gènes en commun avec les eucaryotes et sont leurs plus proches parents dans des arbres phylogénomiques basés sur des marqueurs conservés. Cette découverte a favorisé un changement conceptuel dans les scénarios sur l’origine de la cellule eucaryote, qui privilégient désormais une symbiose métabolique entre les archées et les bactéries comme point de départ de l'eucaryogenèse. Si les modèles existants tentent de rendre compte de certains aspects du déroulement de ce processus évolutif, très peu sont suffisamment détaillés pour expliquer l'évolution de toutes les caractéristiques typiques des eucaryotes. Des modèles plus explicites, expliquant notamment l'origine du noyau, les phospholipides membranaires de type bactérien des eucaryotes, ou l'héritage bactérien dans les génomes eucaryotes, sont nécessaires. Parallèlement à l'amélioration des analyses phylogénomiques et à une meilleure exploration du monde microbien, une meilleure compréhension de la biologie et de l'écologie des lignées procaryotes phylogénétiquement plus proches des eucaryotes devrait permettre de tester les modèles existants et/ou d'en affiner certains. Les nouveaux modèles d'eucaryogenèse devraient proposer des processus plausibles qui rendent compte de l’ensemble des traits et des patterns phylogénétiques observés dans un contexte écologique réaliste.
Conflits d’intérêts
Les auteurs n'ont aucun conflit d'intérêts à déclarer.
Remerciements
Cette recherche a été financée par des subventions du Conseil Européen de la Recherche : ProtistWorld (322669, P.L.G.), PlastEvol (787904, D.M.), et le Moore-Simons Project on the Origin of the Eukaryotic Cell, subvention de la Moore Foundation GBMF9739 (PL-G ; https://doi.org/10.37807/GBMF9739).




 CC-BY 4.0
CC-BY 4.0