1. Introduction
La divergence adaptative (différenciation des populations associée à l’adaptation à une nouvelle niche écologique) est un processus clé de la biologie évolutive. L’étude de ce processus est essentielle pour comprendre l’adaptation des organismes à leur environnement et la façon dont la biodiversité se développe. La domestication est un cas de divergence adaptative sous une forte pression humaine et à l’échelle temporelle humaine. Charles Darwin a utilisé des pigeons pour illustrer le processus et le pouvoir de sélection dans des formes de rendement radicalement différentes de la forme sauvage ancestrale en comparant les pigeons sauvages et domestiqués [15]. Depuis lors, la domestication est utilisée comme un bon modèle pour comprendre l’adaptation, car elle implique une sélection forte et récente pour des caractères qui peuvent être facilement identifiés. Cette sélection forte et récente par l’être humain laisse souvent des traces d’adaptation dans les génomes qui sont plus faciles à détecter que celles laissées par la sélection naturelle. La comparaison des populations domestiquées et non domestiquées aux niveaux phénotypique et génomique peut permettre d’identifier l’architecture génétique des caractères adaptatifs et les mécanismes génomiques permettant une adaptation et une différenciation rapides, comme cela a été démontré pour le maïs et les chiens par exemple [82, 23].
Dans les cultures domestiquées, de multiples événements convergents d’évolution vers des caractères phénotypiques similaires ont été documentés, tels que la perte de la dispersion des graines, la minimisation de la dormance des graines et l’augmentation de la taille et du nombre des graines, principalement due à des changements génomiques différents chez différentes espèces [35, 59] ; la perte de la dispersion des graines est une exception, avec la même base génétique dans le sorgho, le riz, le maïs et le millet sétaire [50, 42]. Les organismes domestiqués sont donc des modèles utiles pour les études de convergence au niveau phénotypique et génomique.
Les espèces domestiquées présentent également souvent une dégénérescence pour des caractères qui sont utiles dans l’environnement sauvage mais qui ne présentent pas d’avantages dans les environnements humains, ce qui conduit à une sélection relâchée. Les espèces domestiquées peuvent également dégénérer en raison d’une accumulation délétère d’allèles due à de forts goulets d’étranglement et/ou à un auto-stop avec des allèles sélectionnés positivement. L’accumulation d’allèles délétères a été démontrée par exemple dans le riz, les porcs et les chevaux [53, 46, 76, 57]. Les cas de dégénérescence de la fertilité comprennent une baisse de la fertilité des épillets latéraux chez l’orge domestiquée [66] et des très petites graines non fonctionnelles dans les cultivars de bananiers triploïdes [36, 67].
La domestication microbienne a été beaucoup moins étudiée que la domestication végétale ou animale, bien que les micro-organismes aient été utilisés pour la fabrication d’aliments et aient développé de nouveaux caractères dans le cadre de la sélection dirigée par l’homme. Les études sur les champignons domestiqués sont rares au-delà de l’espèce modèle de levure Saccharomyces cerevisiae utilisée pour la production de vin et de bière. Pourtant, les champignons domestiqués constituent des modèles eucaryotes très précieux pour l’étude de la divergence adaptative, avec leurs petits génomes, leur accès facile à la phase haploïde et leur traçabilité expérimentale pour les expériences in vitro [32, 33, 1, 10, 47]. De nombreux champignons ont été domestiqués, pour la fermentation et la maturation des aliments (par exemple pour la bière, le pain, le vin, la saucisse sèche ou le fromage), pour la production de métabolites secondaires utilisés en pharmacie (par exemple la pénicilline), ou pour leurs valeurs nutritionnelles et gustatives (par exemple les champignons de Paris et shiitake) [20]. La fermentation est utilisée par les populations humaines depuis des millénaires [60, 34] comme moyen de conserver les aliments périssables, et de conférer au produit de nouvelles caractéristiques organoleptiques et nutritionnelles. Les produits fermentés qui en résultent ont souvent une forte valeur économique et culturelle. La fermentation repose sur les capacités métaboliques des microorganismes à transformer un substrat en un produit présentant un mélange de métabolites responsables d’aspects, de textures, de saveurs et de caractéristiques nutritionnelles particuliers.
Les microorganismes de fermentation, qui résultent à l’origine d’une contamination environnementale, ont été améliorés par l’homme, grâce à un processus de sélection d’abord involontaire, puis plus conscient, sur de nombreuses générations microbiennes, pour les souches donnant les meilleurs produits en termes de caractéristiques sensorielles, de durée de conservation et de sécurité. Cette sélection par l’homme a eu lieu au cours des siècles et a conduit à la domestication microbienne, c’est-à-dire à la différenciation génétique des populations sauvages et alimentaires [2, 33, 1] et à la sélection de phénotypes spécifiques présentant un intérêt pour l’être humain [58]. L’étude des champignons utilisés et sélectionnés au cours des siècles pour la production alimentaire peut ainsi fournir des indications sur les processus d’adaptation. Pourtant, malgré leur importance économique et industrielle, les champignons utilisés par l’être humain ont été peu étudiés, à l’exception de la levure de boulanger Saccharomyces cerevisiae utilisée dans la production de bière, de vin et de pain [21, 48, 51, 62, 49, 19, 2, 77, 58, 27, 68], et le champignon filamenteux Aspergillus oryzae, qui est utilisé pour la fermentation des produits à base de soja et de riz en Asie [26, 28].
Les champignons sont encore d’excellents modèles pour étudier l’évolution et l’adaptation des eucaryotes, compte tenu de leurs nombreux atouts expérimentaux [32]. De nombreux champignons ont de petits génomes, ils sont faciles à cultiver dans des conditions de laboratoire, ont des temps de génération courts, peuvent survivre de longues périodes au congélateur, sont faciles à transformer génétiquement, et les champignons présentent une riche diversité de lignées et de niches écologiques. Les champignons peuvent être utilisés pour répondre à des questions de biologie de l’évolution en combinant des approches complémentaires, telles que des méthodes génomiques et expérimentales. Les études sur les champignons domestiqués ont en fait déjà permis de faire la lumière sur les processus génomiques impliqués dans l’adaptation aux environnements anthropiques, notamment l’expansion des familles de gènes pour des voies métaboliques spécifiques, la perte de gènes qui ne sont plus nécessaires dans le nouvel environnement, l’acquisition de gènes par transfert vertical, l’hybridation et l’introgression [62, 49, 2, 58, 6, 27, 47]. L’existence de lignées multiples et distantes génétiquement de champignons utilisés pour la production de produits alimentaires similaires crée une opportunité idéale pour étudier la répétabilité de l’évolution, en fournissant plusieurs réplications indépendantes récentes d’adaptation à la même niche écologique sous sélection forte.
Les champignons du fromage ne sont pas tous étroitement liés, mais plutôt dispersés dans l’arbre généalogique des champignons. On trouve les champignons Scopulariopsis sur Ossau-Iraty et Tommes, Bisfusarium domesticum sur Saint-Nectaire et Reblochon, Sporendonema casei sur Cantal et Salers, Penicillium camemberti sur Camembert et Brie, P. roqueforti dans tous les fromages bleus du monde, Debaryomyces hansenii, Geotrichum candidum, Kluyveromyces marxianus et Saccharomyces cerevisiae dans de nombreuses sortes de fromages. Phylogénie dessinée sur la base de LifeMap [16]. Images de fromages : Scopulariopsis : Ossau Iraty, B. domesticum : Saint-Nectaire, S. casei : Cantal, P. camemberti : Camembert, P. roqueforti : Roquefort, D. hansenii : Reblochon, G. candidum : fromage de chèvre. Les photos de Saint-Nectaire, Reblochon et Cantal sont tirées de Wikipédia.
La domestication des champignons pour la fabrication du fromage constitue un système modèle particulièrement attrayant. En effet, le fromage est l’un des aliments fermentés les plus populaires dans le monde entier. On le trouve en Europe, en Amérique, en Asie et au Moyen-Orient avec différentes variétés de fromage, comme l’Anthotyros et la feta en Grèce, le Comté, le Reblochon et le Roquefort en France et le Chhurpi au Népal. Les champignons sont utilisés pour la maturation du fromage et jouent un rôle essentiel dans la fermentation, la texture, la couleur et la saveur du fromage. En outre, les multiples espèces fongiques éloignées sur le plan phylogénétique qui prospèrent dans la niche écologique du fromage offrent une “expérience” fascinante de sélection similaire récente et des possibilités uniques d’évaluer la répétabilité de l’évolution, d’autant plus que plusieurs lignées éloignées ont été utilisées pour des produits similaires (Figure 1). Nous passons ici en revue les études récentes sur l’histoire et le processus de domestication de deux espèces fromagères emblématiques, Penicillium roqueforti et P. camemberti, utilisées respectivement pour la fabrication de fromages bleus et de fromages à pâte molle (Figure 1). Après un bref aperçu des champignons Penicillium, nous résumons les connaissances récentes acquises sur leur histoire démographique, leur diversité génétique et leur structure de population, ainsi que sur leurs changements phénotypiques et génétiques dans le cadre de la domestication.
2. Les champignons Penicillium
Penicillium est un genre de champignons des Ascomycota, décrit pour la première fois en 1809 par Link. Le nom Penicillium vient du mot latin qui signifie pinceau, reflétant la forme conidiophore des champignons Penicillium [81]. Le genre Penicillium contient 354 espèces acceptées taxonomiquement, isolées dans le monde entier dans divers environnements, par exemple le sol, l’air et la nourriture [22, 81, 17]. La plupart des espèces de Penicillium peuvent se reproduire par voie sexuée et asexuée. Pour la reproduction asexuée, les champignons Penicillium génèrent des conidies en chaîne (spores asexuées) via des structures spécifiques appelées conidiophores. Les conidiophores de Penicillium sont plus ou moins ramifiés, certaines espèces de Penicillium ayant des conidiophores uniquement composés de phialides d’où sortent les conidies (Penicillia monoverticillate), tandis que d’autres espèces ont des conidiophores avec une (biverticillate) ou plusieurs (Penicillia terverticillate) branches (appelées metula) ; ces schémas de ramification des conidiophores ont été utilisés par Pitt pour classer les espèces de Penicillium avant l’avènement de l’identification moléculaire. Les conidies de Penicillium sont généralement vertes ou bleues/vertes, la couleur changeant toutefois en fonction des conditions de croissance, telles que le milieu de culture, la lumière, l’humidité et la température [74]. La dispersion asexuée des spores peut se produire par dispersion aérienne, aquatique ou animale lorsque les spores germent en hyphes qui vont croître et se développer en un nouveau mycélium. Les champignons Penicillium peuvent également se reproduire par voie sexuée, lorsque les hyphes de deux individus haploïdes portant des types d’accouplement opposés, appelés MAT1-1 et MAT1-2, se rencontrent dans des conditions adéquates. Ensuite, les noyaux fusionnent (caryogamie) et une méiose se produit, donnant naissance à huit ascospores. Ces ascospores sont enveloppées dans des asques, eux-mêmes étant à l’intérieur du cléistothecium, le corps fructifère des Ascomycètes.
Les champignons Penicillium sont bien connus en tant que champignons saprophytes décomposant les fruits et les cultures, en tant que contaminants alimentaires, et certaines espèces sont des agents pathogènes des cultures humaines ou alimentaires. Par exemple, P. expansum et P. digitatum sont des agents de dégradation post-récolte des agrumes et des poires [8, 55], P. marneffei est un agent pathogène pour l’homme [52] ; P. oxalicum, P. viridicatum et P. cyclopium sont couramment utilisés pour endommager les cultures [61] ; P. glabrum, P. allii, P. sclerotigenum, P. roqueforti et P. paneum sont des agents d’altération des aliments [22, 17]. Les champignons Penicillium ont également d’énormes impacts positifs sur la société humaine : Alexander Fleming a accidentellement découvert en 1928 la capacité de production d’antibiotiques de P. rubens, nommé à l’époque P. notatum, puis P. chrysogenum et récemment rebaptisé P. rubens [38], ce qui lui a valu le prix Nobel de médecine en 1945. Comme beaucoup de champignons, les champignons Penicillium produisent des toxines dans certaines conditions pour inhiber la croissance et la reproduction de leurs concurrents [41, 14], qui peuvent avoir un impact toxicologique sur l’homme selon les conditions d’exposition. En outre, les champignons Penicillium sont utilisés pour la fermentation des aliments, étant par exemple inoculés pour la production de fromage et de viande séchée [78, 54, 20, 12]. En effet, les champignons Penicillium utilisés dans les aliments fermentés permettent de conserver le lait cru ou la viande périssable, en transformant les aliments crus grâce à leur activité métabolique telle que la lipolyse et la protéolyse, en générant des barrières physiques à la surface, ainsi que des goûts et arômes spécifiques.
3. Domestication du Penicillium roqueforti
Penicillium roqueforti est l’espèce de moisissure utilisée comme ferment secondaire pour la production de Roquefort et de tous les fromages bleus [9, 74, 14], avec plus de 16 synonymes tels que P. glaucum, P. gorgonzolae ou P. stilton. Penicillium roqueforti est un champignon saprophyte parfois trouvé dans le sol ou la matière organique en décomposition, bien que son réservoir naturel soit encore inconnu. Il est fréquemment isolé à partir de matières végétales mal stockées dans des conditions micro-aérophiles telles que l’ensilage, où il peut produire des toxines ayant des effets néfastes sur le bétail ou les humains [65, 64, 63, 5, 3, 14]. Sa tolérance au froid et aux acides organiques, en particulier lorsque l’activité de l’eau est supérieure à 95%, fait de cette espèce un contaminant alimentaire fréquent du pain, de la bière, de l’orge, du riz, des olives, des fruits, des fromages à pâte dure, de la farine ou de la viande séchée [69]. L’espèce a été décrite en 1906 par Charles Thom [79]. Il s’agissait en fait d’un complexe de plusieurs espèces, aujourd’hui appelé section Roquefortorum [40], comprenant également P. carneum, P. paneum et P. psychrosexualis, que l’on pouvait distinguer sur la base d’analyses moléculaires, morphologiques et biochimiques [24, 7, 25, 75, 43, 37, 39].
Au sein de l’espèce P. roqueforti, plusieurs populations différenciées ont été identifiées sur la base de marqueurs microsatellites [73, 30]. Sur la base de génomes entiers, quatre populations génétiquement et phénotypiquement différenciées ont été identifiées [18] (Figure 2). Deux populations sont utilisées pour la fabrication du fromage et les deux autres populations vivent dans d’autres environnements [18]. Parmi les deux populations de fromage, l’une comprend des souches liées à la production française de Roquefort en appellation d’origine protégée (AOP), tandis que la population non-Roquefort comprend des souches isolées dans le monde entier de tous les autres fromages bleus, comme le Stilton en Angleterre, le Cabrales en Espagne, le Gorgonzola en Italie, la Fourme d’Ambert en France. Les deux autres populations se trouvent sur le bois, l’ensilage et comme contaminants dans divers produits alimentaires (Figure 2).
Les deux populations de fromage présentent une diversité génétique beaucoup moins importante que les deux autres populations [18]. Toutefois, la population de Roquefort a conservé une diversité génétique légèrement plus riche que la population non-Roquefort, probablement en raison de l’obligation d’utiliser des souches locales dans le cadre du cahier des charges de l’AOP [18]. La perte drastique de diversité génétique dans la population non-Roquefort reflète une forte sélection pour la production à l’échelle industrielle et le commerce mondial qui ont conduit à l’utilisation d’une seule lignée clonale dans tous les fromages bleus analysés dans le monde [18].
Histoire de la domestication du Penicillium roqueforti pour la fabrication des fromages bleus. D’une population ancestrale avec une niche écologique jusqu’alors inconnue, les quatre populations identifiées de P. roqueforti se sont diversifiées en s’adaptant aux niches écologiques créées par l’homme, avec deux populations domestiquées indépendamment pour la fabrication de fromage, et deux populations contaminant la nourriture et l’ensilage. L’une des deux populations ne se trouve que dans les fromages appartenant à l’appellation d’origine protégée (AOP) Roquefort et abrite une certaine diversité génétique, tandis que l’autre population est une lignée clonale unique utilisée pour tous les autres fromages bleus du monde. Les goulets d’étranglement se sont produits dans toutes les populations, étant les plus forts dans les populations de fromages. Les populations fromagères ont développé des caractères spécifiques bénéfiques pour la fabrication du fromage, tels que les arômes, la coloration bleue des cavités du fromage, la lipolyse, la protéolyse et la tolérance au sel, avec des spécificités différentes dans les deux populations. La population de Roquefort produit une plus grande quantité de spores lorsqu’elle est cultivée sur du pain, le milieu de culture ancestral de Roquefort-sur-Soulzon. a : spores de P. roqueforti prélevées sur du pain inoculé, cuit auparavant en produisant des croûtes brûlées, comme on l’a longtemps fait pour l’inoculation du fromage bleu. b : tranche de fromage expérimental ressemblant au Roquefort. c : Morceau de fromage bleu avec des spores de P. roqueforti grossies en incrustation, portées dans les “pinceaux” typiques des champignons Penicillium. d : Boîtes de Pétri telles qu’elles sont utilisées aujourd’hui pour la culture stérile de P. roqueforti. e : Colonie de P. roqueforti sur une boîte de Pétri avec un milieu malté. f : Molécule de 2-heptatone responsable de l’un des arômes typiques du fromage bleu de Roquefort. g : Illustration de l’histoire de la divergence déduite des quatre populations de P. roqueforti identifiées, deux étant utilisées pour la fabrication du fromage et ayant souffert de forts goulots d’étranglement, une contaminant les aliments et le bois d’œuvre et une l’ensilage et également les aliments. h : Colonie de P. roqueforti sur une boîte de Pétri avec un milieu de fromage, avec des caractéristiques de mesures de taux de croissance. i : Tubes avec tributyrine utilisés pour mesurer le taux de lipolyse. j : Boîte de Pétri avec deux souches de P. roqueforti et structures sexuées (appelées cléistothèces, en marron) induites dans la zone de confrontation. k : Expérience de compétition sur une boîte de Pétri avec deux souches de P. roqueforti, montrant une plus grande capacité de compétition et une couleur plus foncée de la souche non-Roquefort. l : Acquisition horizontale de gènes dans la lignée non-Roquefort. m : Morceau de fromage roquefort. n : Balle d’ensilage dans un champ. o : Yaourt contaminé par P. roqueforti. p : Pain contaminé par P. roqueforti. q et r : Fromages bleus non Roquefort (Fourme d’Ambert et Bleu d’Auvergne). s : Fromages de Roquefort sur leurs étagères en bois dans les caves, l’une étant enveloppée de la typique feuille de plomb.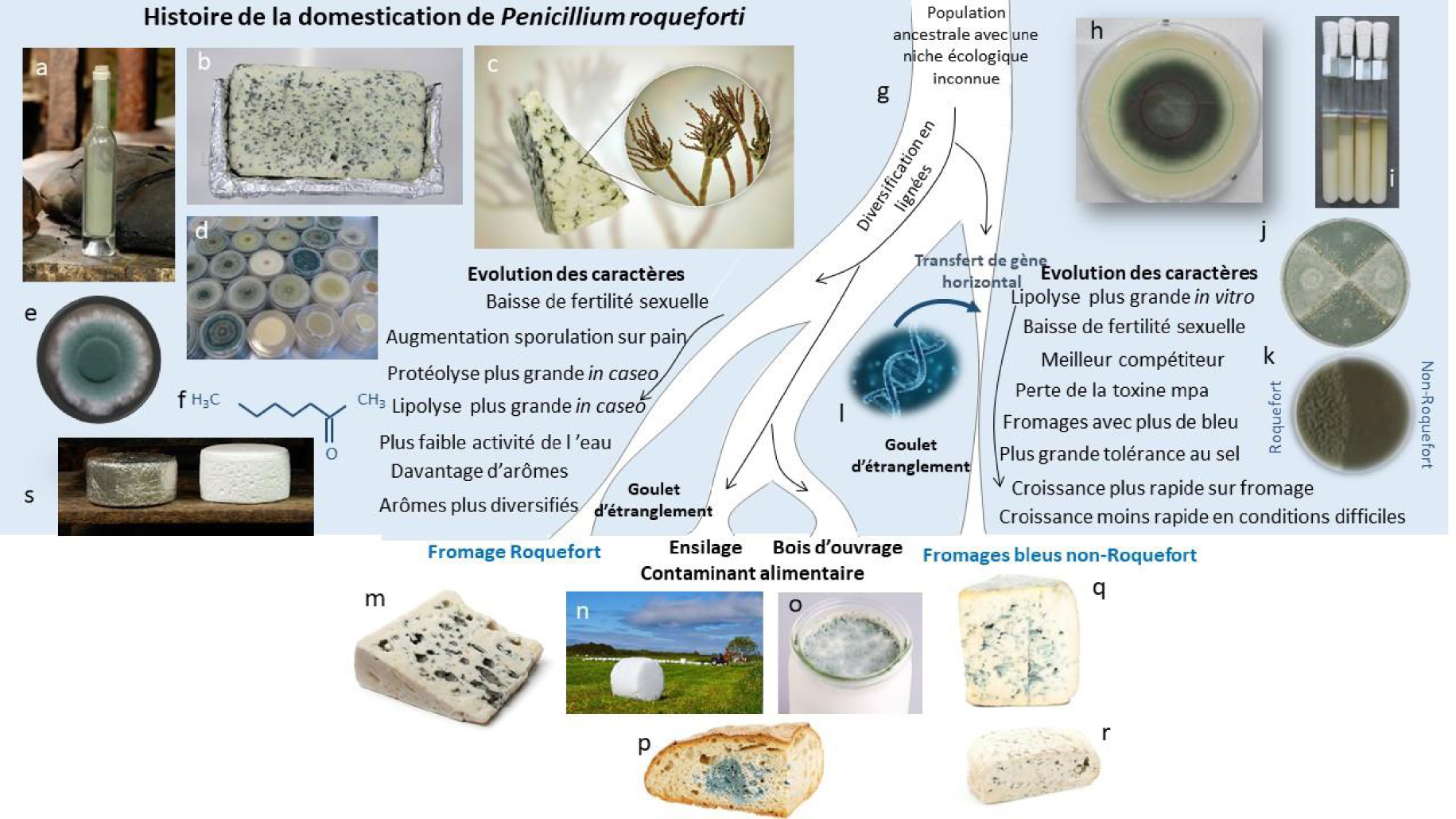
La lignée non-Roquefort a acquis de grands fragments génomiques par des transferts horizontaux de gènes [13, 73, 70]. Deux des régions transférées horizontalement semblent présenter un intérêt particulier en raison de la fonction de certains gènes qu’elles portent. Ces deux régions à transfert horizontal, appelées Wallaby et CheesyTer, ont une longueur respective de 600 et 80 kb et portent des gènes ayant des fonctions liées au métabolisme du lactose/lactate et à la compétition avec d’autres microorganismes [13, 70].
La comparaison des scénarios de divergence des populations dans le cadre du calcul approximatif bayésien a révélé que les populations d’ensilage, de bois et de contaminants alimentaires étaient les plus proches génétiquement les unes des autres, ayant divergé le plus récemment, tandis que les deux populations de fromage ont divergé plus tôt et étaient le résultat de deux événements de domestication indépendants [18] (Figure 2g). En effet, malgré un échantillonnage plus important des populations de fromage, en particulier pour la population non-Roquefort, de forts goulets d’étranglement ont été détectés dans les deux populations de fromage, étant un peu plus faibles dans la population roquefortaine [18] (Figure 2g). Deux goulets d’étranglement successifs ont même pu se produire dans la population de Roquefort [18]. Ces goulets d’étranglement sont le résultat de la domestication et plus probablement aussi de la sous-traitance récente de la production de ferments à un petit nombre de sociétés spécialisées, et peut-être de la sélection récente d’une lignée clonale performante après l’acquisition de gènes conférant des caractères avantageux par transferts horizontaux pour la population non-Roquefort [13, 70]. Les tests comparant les modèles démographiques ont également détecté un flux de gènes entre les deux populations non fromagères, mais pas avec les populations fromagères [18]. Les dates de domestication estimées à partir des derniers événements de flux de gènes entre les populations ont suggéré que la population de Roquefort aurait divergé il y a environ sept fois plus longtemps que la population non fromagère, il y a environ 760 et 140 générations, respectivement [18]. Cependant, l’estimation du temps absolu à partir du nombre de générations est difficile dans le cas des champignons, en particulier dans les lignées clonales.
Les souches de fromage ont des caractéristiques spécifiques, notamment en termes de couleur, de fertilité, de taux de croissance et de capacité à exclure d’autres micro-organismes en concurrence sur un milieu de culture [73, 30, 72, 18] (Figure 2i). Les souches non-Roquefort se développent plus rapidement sur milieu fromager que toutes les autres souches et excluent mieux les concurrents que toutes les autres souches [70] (Figure 2k). Ces capacités sont probablement liées, au moins en partie, aux régions transférées horizontalement Wallaby et CheesyTer (Figure 2), car elles portent des gènes ayant des fonctions liées au métabolisme du lactose/lactate et à la concurrence avec d’autres micro-organismes [13, 70]. La population non-Roquefort a également montré une lipolyse plus rapide, une plus grande tolérance au sel et une colonisation de la cavité du fromage [18], et la perte de la capacité à produire de l’acide mycophénolique en raison d’une délétion de 174 pb dans le gène mpaC [29] (Figure 2). La population de Roquefort a plus grande capacité de sporulation sur milieu à base de pain [18].
Principaux résultats d’une étude récente [11], dans laquelle des analyses de fromages expérimentaux fabriqués selon le mode de production du Roquefort (e) et par inoculation de souches provenant des quatre populations de Penicillium roqueforti (a) ont montré des caractères bénéfiques dans les deux populations de fromage, produisant une surface bleue plus élevée sur les tranches de fromage (b), une lipolyse et une protéolyse plus fortes in caseo, et en particulier dans la population de Roquefort (alors que la population non-Roquefort s’est avérée avoir le taux de lipolyse le plus élevé in vitro [18]), (c) et des quantités plus importantes d’arômes positifs (comme les méthylcétones, avec par exemple, dans d, la 2-pentanone et la 2-heptanone, et les alcools secondaires, avec par exemple, dans d, le 2-heptanol et le 2-nonanol, les quantités d’alcool multipliées par quatre par rapport aux méthylcétones).
La lente croissance de la population de Roquefort sur milieu fromager et son activité lipolytique plus faible observée in vitro par rapport à la population non Roquefort devraient ralentir la maturation du fromage. Ce ralentissement de la maturation peut être lié à la nécessité de conserver les fromages de Roquefort, fabriqués à partir de lait de brebis disponible uniquement entre février et juillet, sur de longues périodes et historiquement sans réfrigération [56], et à l’exigence d’un affinage d’au moins 90 jours dans le cahier des charges de l’AOP Roquefort. La meilleure sporulation sur pain de la population de Roquefort fait également probablement partie du syndrome de domestication, en raison de la méthode de conservation et de production des spores de moisissure sur le pain de seigle [56, 4, 44, 45, 80] (Figure 2a). Les performances de la population non-Roquefort en termes de croissance sur milieu fromager [70], de colonisation des cavités fromagères, de tolérance au sel, de lipolyse in vitro [18], et de perte de capacité de production de toxines [29], résulteraient donc d’une sélection très récente de caractéristiques bénéfiques pour la production moderne et rapide de tous types de fromages bleus.
Une analyse de 52 composés volatils produits par les deux populations de fromages dans des modèles fromagers très simplifiés a permis d’identifier des profils de composés volatils significativement différents entre les deux populations de fromages, notamment en distinguant les composés typiques des fromages bleus au goût prononcé et une plus grande diversité de composés volatils produits par la population de Roquefort [18]. Des empreintes de sélection positive ont également été identifiées dans plusieurs gènes potentiellement impliqués dans la production de composés volatils [18].
Une étude récente a examiné les différences entre les populations de P. roqueforti dans le contexte de la domestication pour la fabrication de fromage en comparant les fromages bleus fabriqués avec les quatre populations fongiques suivant des conditions aussi proches que possible des spécifications de production commerciale de type Roquefort [11] (Figure 3). Les populations de P. roqueforti ont eu un impact mineur sur la diversité bactérienne des fromages et aucun sur l’abondance des principaux microorganismes. Les populations de P. roqueforti ont produit des fromages avec des pourcentages plus élevés de surface bleue en raison d’une croissance plus rapide et/ou d’une meilleure sporulation, peut-être liée à une meilleure tolérance aux faibles niveaux d’oxygène. Les populations de fromages ont également produit des fromages avec différentes séries et des quantités plus élevées de composés volatils souhaités. La population Roquefort de P. roqueforti en particulier a produit des composés aromatiques plus variés et en plus grande quantité, ce qui a donné des notes plus fruitées et plus fromagères aux fromages ; la production de ces composés étaient principalement à sa lipolyse et sa protéolyse les plus efficaces in caseo. Il est frappant de constater que la lipolyse et la protéolyse étaient effectivement les plus efficaces dans les fromages pour la population de P. roqueforti de Roquefort [11], tandis que la lipolyse était en revanche la plus efficace in vitro pour la population de P. roqueforti non Roquefort [18], ce qui montre l’importance des études in caseo. La population de Roquefort produit également des fromages à faible activité de l’eau, ce qui peut prévenir l’apparition de micro-organismes contaminants. Il y avait donc une forte influence des populations de P. roqueforti sur plusieurs aspects importants de la sécurité, de l’apparence et de la saveur du fromage [11].
Des empreintes de dégénérescence ont été trouvées chez les populations de P. roqueforti domestiquées. La culture clonale de souches de P. roqueforti utilisées pour la production de fromage, et leur maintien isolé des populations dans d’autres environnements depuis le 19ème siècle, a entraîné une dégénérescence des fonctions sexuelles, avec une perte de fertilité à différents stades dans les deux populations de fromage [72]. La population de fromage de Roquefort a pu effectuer les premières étapes de la reproduction sexuée mais a montré une baisse de la fertilité post-zygotique, alors que la population non-Roquefort n’initie même souvent pas la reproduction sexuée [72]. Les souches non-Roquefort se développent plus lentement sur un milieu pauvre en nutriments, ce qui peut être dû à une dégénérescence des fonctions non utilisées [70]. Les importants goulets d’étranglement dans les populations de fromage [18] peuvent également avoir contribué à la dégénérescence.
4. Domestication du Penicillium camemberti
Le Penicillium camemberti est un autre champignon fromager emblématique, utilisé pour l’affinage des fromages à pâte molle comme le Camembert et le Brie, tout en étant phylogénétiquement éloigné de P. roqueforti [13]. Le complexe d’espèces Penicillium camemberti comprend trois espèces, P. biforme, P. camemberti et P. fuscoglaucum, toutes présentes dans l’environnement du fromage, P. biforme et P. camemberti étant spécifiques à l’environnement du fromage tandis que P. fuscoglaucum se trouve principalement dans les environnements naturels [71]. Au début du 20ème siècle, un mutant blanc a été généré dans l’industrie fromagère, par mutagenèse artificielle à partir d’un champignon gris-vert, à l’époque nommé P. commune, aujourd’hui divisé en deux espèces étroitement apparentées, P. biforme et P. fuscoglaucum [31, 71]. Le brie était en effet d’aspect gris-vert jusqu’au 20ème siècle. On peut le voir sur le tableau “Symphonie des fromages en brie majeur ; Nature morte au fromage” de Marie Julesx Justin (1847; https://www.pop.culture.gouv.fr/notice/joconde/M0794015842).
Une récente étude de génomique des populations a déduit un événement de domestication ancien séparant le champignon fromager P. biforme de P. fuscoglaucum, que l’on trouve principalement dans d’autres environnements que le fromage, par exemple le caoutchouc, les racines, le bois, les noix, l’eau ou les aliments contaminés. Un événement de domestication beaucoup plus récent a été détecté, séparant la lignée clonale de P. camemberti de celle de P. biforme [71] (Figure 4). La lignée clonale de P. camemberti a été trouvée dans des fromages à pâte molle de type Camembert, tandis que P. biforme a été trouvé dans des fromages à pâte dure comme la Tomme ou l’Ossau Iraty (Figure 4). Une adaptation phénotypique à la fabrication du fromage a été trouvée chez P. biforme par rapport à P. fuscoglaucum, avec une couleur plus blanche, une croissance plus rapide sur le milieu fromager dans des conditions de caves d’affinages (basse température et forte humidité), une tolérance au sel et des niveaux plus faibles de production de toxines de l’acide cyclopiazonique (Figure 4).
Historique de la domestication du Penicillium camemberti et du P. biforme pour la fabrication de fromages à pâte molle ou dure (a). Issu d’une population ancestrale dont la niche écologique était jusqu’alors inconnue, P. fuscoglaucum a divergé d’abord, se trouvant surtout dans des environnements non fromagers, puis P. biforme a été domestiqué pour la production de fromage, et plus tard les deux variétés de P. camemberti, var. camemberti et var. caseifulvum, avec de forts goulots d’étranglement. Le processus de domestication a conduit à une adaptation concernant la couleur (qui devient plus blanche), la capacité d’exclusion compétitive, la production de toxines (perte de la production d’acide cyclopiazonique CPA), la croissance radiale et/ou verticale (duveteuse). L’environnement d’origine des lignées est montré (de gauche à droite : racines pourries, fromage de chèvre, camembert et Saint-Marcellin), ainsi que des photos de la colonie de mycélium sur des boîtes de Pétri avec du fromage salé en milieu (coloré en vert avec un colorant alimentaire) en cercles. (b). Mycélium du P. camemberti var. camemberti blanc et du P. biforme gris-vert cultivés en secteurs dans la même boîte de Pétri.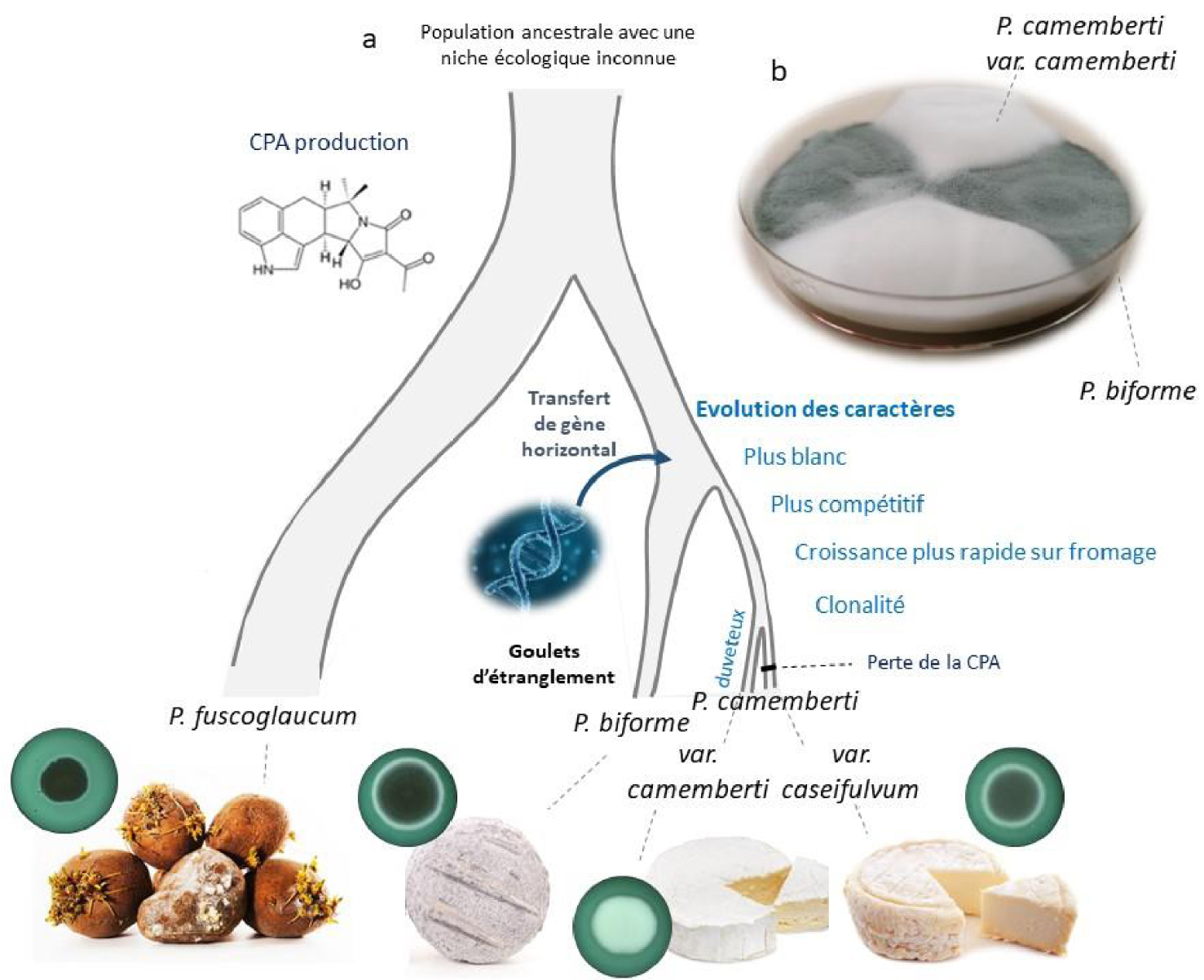
Deux lignées différenciées de P. camemberti ont été identifiées, avec des caractéristiques phénotypiques contrastées, qui peuvent donc être considérées comme deux variétés domestiquées différentes. L’une des variétés de P. camemberti contient les mêmes régions transférées que la population non Roquefort de P. roqueforti, CheesyTer et Wallaby [13], à l’exception de trois souches isolées du fromage camembert avant 1905 qui ne contiennent pas CheesyTer [71]. Cette variété de P. camemberti, appelée var. camemberti, présente une croissance radiale plus faible, mais était plus duveteuse que l’autre variété de P. camemberti et que P. biforme et P. fuscoglaucum (Figure 4), ce qui permet de former la croûte blanche et duveteuse typique des fromages Camembert et Brie. La variété gris-vert P. camemberti, appelée variété caseifulvum, est présente dans les fromages autres que le Camembert, comme le St Marcellin ou la Rigotte de Condrieu, porte CheesyTer mais pas toute la région de Wallaby, et est incapable de produire l’acide cyclopiazonique en raison d’une mutation de décalage du cadre de lecture dans un des gènes impliqués dans sa biosynthèse [71]. Les deux variétés de P. camemberti excluent les concurrents fongiques plus efficacement que P. biforme, la lignée blanche duveteuse de P. camemberti étant plus efficace à l’exclusion que la lignée gris-vert de P. camemberti.
Ainsi, le processus de domestication a conduit à la diversification de P. camemberti en deux variétés, P. camemberti var. camemberti et P. camemberti var. caseifulvum, présentant des différenciations génétique et phénotypique (en termes de taux de croissance, de couleur, de capacités concurrentielles et de production de toxines). En outre, les deux variétés de P. camemberti présentaient des empreintes de goulots d’étranglement importants, représentant des lignées clonales (Figure 4a). Les deux variétés de P. camemberti poussent plus lentement dans des conditions difficiles que P. biforme ou P. fuscoglaucum, ce qui suggère que l’adaptation à la fabrication de fromage a entraîné une dégénérescence des fonctions utiles dans les environnements sauvages [71]. Une autre empreinte de la dégénérescence est le niveau général très faible de production de spores chez P. camemberti, encore plus chez la variété camemberti, ce qui pose problème pour la production de fromage.
5. Conclusion
Les études récentes sur P. roqueforti et P. camemberti ont montré que l’aspect et le goût typiques des fromages bleus et des fromages à pâte molle sont le résultat d’une sélection humaine sur P. roqueforti et P. camemberti, constituant ainsi une domestication, et que différentes variétés au sein de chaque espèce ont acquis des spécificités pour la fabrication du fromage. Ceci a des implications importantes pour notre compréhension des processus d’adaptation et de domestication. Plusieurs caractéristiques d’adaptation convergente ont par exemple été trouvées dans les champignons fromagers domestiqués, les mêmes régions génomiques conférant des avantages pour la croissance du fromage ayant été acquises horizontalement à la fois chez P. roqueforti et P. camemberti, une même tolérance au sel, une plus grande capacité concurrentielle et la perte de production de toxines. Des goulots d’étranglement importants et une clonalité ont également été documentés dans les populations de fromage des deux espèces, ainsi qu’une capacité réduite à croître dans des conditions difficiles.
Les études récentes sur les champignons du fromage sont également importantes pour améliorer la production de fromage. La découverte de deux lignées clonales différentes utilisées pour la fabrication du fromage chez P. roqueforti, avec des phénotypes contrastés et des types sexuels opposés [18], ainsi que l’élaboration d’un protocole pour induire la reproduction sexuée [73], ouvrent la voie à l’amélioration des souches par la génération de descendants. Ces descendants peuvent en outre permettre d’associer des génotypes et des phénotypes pour élucider la base génétique de caractères importants pour la fabrication du fromage et l’adaptation. Penicillium camemberti présente des traces de dégénérescence sous la forme d’un niveau de production de spores dramatiquement bas, ce qui rend difficile la réplication et l’inoculation de cette moisissure pour les producteurs de fromage. Alors que P. camemberti représente une seule lignée clonale avec un seul type sexuel [71], les deux types sexuels ont été trouvés chez P. biforme [73], ouvrant la voie à la génération de recombinants pour l’amélioration des souches.
Les produits de viande séchée, comme le salami, le jambon et d’autres charcuteries comme les produits de viande séchée de canard ou de dinde, utilisent également des champignons Penicillium pour la fermentation, P. nalgiovense et P. salamii servant de ferment, ainsi que P. camemberti [78, 54, 12]. Il serait donc intéressant d’étudier les traces de domestication de ces champignons Penicillium. De plus, P. nalgiovense et P. salamii sont distants génétiquement, ce qui suggère que ces champignons Penicillium peuvent être des modèles idéaux pour étudier l’adaptation parallèle.
Remerciements
Ce travail a été financé par l’ERC starting grant GenomeFun 309403, un financement de la fondation Louis D. grant (Institut de France), l’ANRT, le LIP SAS, et l’ANR FUNGADAPT (ANR-19-CE20-0002).
English version
1. Introduction
Adaptive divergence (population differentiation associated with adaptation to a new ecological niche) is a key process in evolutionary biology. Studying this process is essential for understanding the adaptation of organisms to their environment and how biodiversity arises. Domestication is a case of adaptive divergence under strong human pressure and at human timescale. Charles Darwin used pigeons to illustrate the process and power of selection in yielding forms drastically different from the ancestral wild form by comparing wild and domesticated pigeons [15]. Ever since, domestication has been a good model for understanding adaptation, as it involves strong and recent selection for traits that can easily be identified. This strong and recent selection by humans often leaves footprints of adaptation in genomes that are easier to detect than those left by natural selection. Comparing domesticated and non-domesticated populations at phenotypic and genomic levels can allow elucidating the genetic architecture of adaptive traits and the genomic mechanisms allowing rapid adaptation and differentiation, as shown in maize and dogs for instance [82, 23].
In domesticated crops, multiple convergent events of evolution towards similar phenotypic traits have been documented, such as the loss of seed shattering, the minimization of seed dormancy and the increase in seed size and number, mostly arising through different genomic changes in different species [35, 59]; the loss of seed shattering is an exception, with the same genetic basis in sorghum, rice, maize and foxtail millet [50, 42]. Domesticated organisms are thus useful models for studies of convergence at both the phenotypic and genomic levels.
Domesticated species also often show degeneration for traits that are useful in the wild environment but do not provide benefit in human-made environments, which leads to relaxed selection. Domesticated species can also degenerate because of deleterious allele accumulation due to strong bottlenecks and/or to hitchhiking with positively selected alleles. Accumulation of deleterious alleles has been shown for example in rice, pigs and horses [53, 46, 76, 57]. Cases of degeneration in fertility include decreased fertility of lateral spikelets in domesticated barley [66] and non-functional very small seeds in triploid banana cultivars [36, 67].
Microbial domestication has been much less studied than plant or animal domestication, although microorganisms have been used for making food, and have evolved new traits under human-directed selection. Studies on domesticated fungi are rare beyond the model yeast Saccharomyces cerevisiae used for wine and beer production. Yet, domesticated fungi constitute highly valuable eukaryotic models for the study of adaptive divergence, with their small genomes, easy access to the haploid phase and experimental tractability for in vitro experiments [32, 33, 1, 10, 47]. Many fungi have been domesticated, for food fermentation and maturation (e.g. for beer, bread, wine, dried sausage or cheese), for producing secondary metabolites used in pharmaceutics (e.g. penicillin), or for their nutritional and gustatory values (e.g. button and shiitake mushrooms) [20]. Fermentation has been used by human populations for millennia [60, 34] as a way to preserve perishable food, and to confer new organoleptic and nutritional traits to the product. The resulting fermented products often have strong economic and cultural values. Fermentation relies on the metabolic abilities of microorganisms to transform a substrate into a product exhibiting a mixture of metabolites responsible for particular aspects, textures, flavors and nutritional traits.
Fermenting microorganisms, originally resulting from environmental contamination, have been improved by humans, via an initially unintentional, and then more conscious, selection process along microbial generations for strains yielding the best products in terms of sensorial traits, shelf life and safety. This selection by humans has occurred over centuries and led to microbial domestication, i.e., genetic differentiation from wild populations [2, 33, 1] and the selection of specific phenotypes of interest for humans [58]. The study of fungi used and selected over centuries for food production can thus provide insights into adaptation processes. Yet, despite their economic and industrial importance, the fungi used by humans have been little studied, with the exception of the budding yeast Saccharomyces cerevisiae used in the production of beer, wine and bread [21, 48, 51, 62, 49, 19, 2, 77, 58, 27, 68], and the filamentous fungus Aspergillus oryzae, which is used to ferment soy and rice products in Asia [26, 28].
Fungi are yet excellent models for studying evolution and adaptation in eukaryotes, given their many experimental assets [32]. Some fungi have small genomes, are easy to culture in laboratory conditions, have short generation times, can survive long periods in the freezer, are easy to transform genetically, and fungi present a rich diversity of lineages and ecological niches. Fungi can be used to address evolutionary biology questions through combinations of complementary approaches, such as genomic and experimental methods. Studies on domesticated fungi have as a matter of fact already shed light on the genomic processes involved in adaptation to anthropic environments, including gene family expansion for specific metabolism pathways, the loss of genes no longer required in the new environment, gene acquisition by horizontal gene transfers, hybridisation and introgression [62, 49, 2, 58, 6, 27, 47]. The existence of multiple, distantly related lineages of fungi used for the production of similar food products creates an ideal opportunity for studying the repeatability of evolution, by providing several recent independent replicates of adaptation to the same ecological niche under strong selection.
The domestication of fungi for cheese-making constitute a particularly attractive model system. Indeed, cheese is one of the most popular fermented food around the world, being found in Europe, America, Asia and Middle East with different cheese varieties, such as the Anthotyros and feta in Greece, Comté, Reblochon and Roquefort in France and Chhurpi in Nepal. Fungi are used for cheese maturation, playing a critical role in cheese fermentation, texture, color and flavor. Furthermore, the multiple phylogenetically distant fungal species thriving in the cheese ecological niche provide an exciting “natural experiment” of recent similar selection and unique opportunities to assess the repeatability of evolution, especially as several distant lineages have been used for similar products (Figure 1). Here, we review recent studies on the history and process of domestication in two emblematic cheese-making species, Penicillium roqueforti and P. camemberti, used for making blue and soft cheeses, respectively (Figure 1). After a brief overview on Penicillium fungi, we review the recent insights gained on their demographic history, their genetic diversity and population structure, and their phenotypic and genetic changes under domestication.
Cheese fungi are not all closely related, being instead scattered across the fungal tree of life. Scopulariopsis fungi can be found on Ossau-Iraty and Tommes, Bisfusarium domesticum on Saint-Nectaire and Reblochon, Sporendonema casei on Cantal and Salers, Penicillium camemberti on Camembert and Brie, P. roqueforti in all blue cheeses worldwide, Debaryomyces hansenii, Geotrichum candidum, Kluyveromyces marxianus and Saccharomyces cerevisiae in many different kinds of cheeses. Phylogeny drawn based on LifeMap [16]. Cheese pictures: Scopulariopsis: Ossau Iraty, B. domesticum: Saint-Nectaire, S. casei: Cantal, P. camemberti: Camembert, P. roqueforti: Roquefort, D. hansenii: Reblochon, G. candidum: goat cheese. Pictures of Saint-Nectaire, Reblochon and Cantal are from Wikipedia.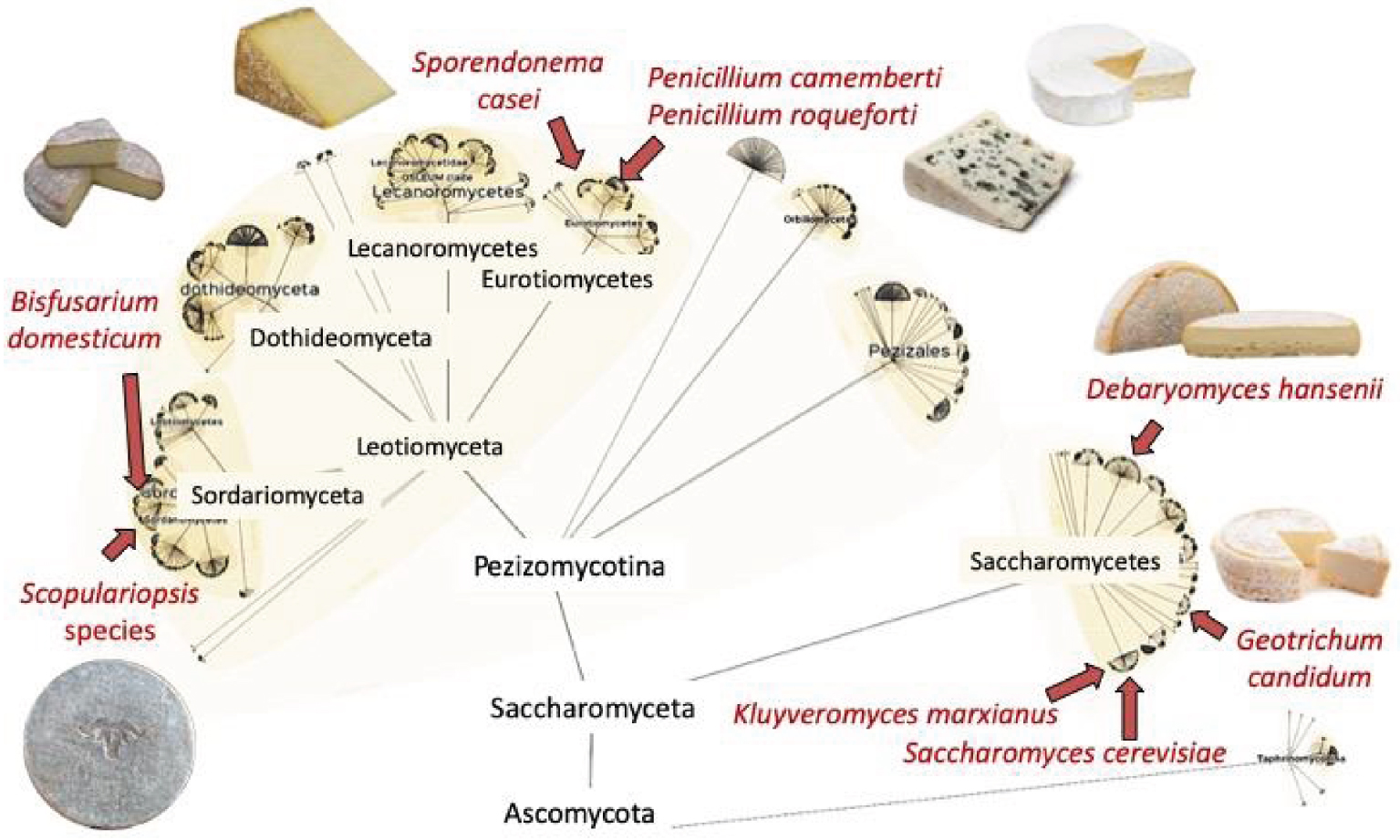
2. Penicillium fungi
Penicillium is a genus of Ascomycota fungi, first described in 1809 by Link. The name Penicillium comes from the Latin word meaning brush, reflecting the conidiophore shape of Penicillium fungi [81]. The Penicillium genus contains 354 accepted species, isolated worldwide from various environments, e.g. soil, air and food [22, 81, 17]. Most Penicillium species can perform sexual and asexual reproduction. For asexual reproduction, Penicillium fungi generate conidia in chains (asexual spores) via specific structures called conidiophores. Conidiophores of Penicillium are more or less branched, with some Penicillium species having conidiophores only composed of phialides from which conidia go out (monoverticillate Penicillia), while some other species have conidiophores with one (biverticillate) or more (terverticillate Penicillia) branches (called metula); these conidiophore branching patterns were used by Pitt to classify Penicillium species before the advent of molecular identification. Penicillium conidia are typically mostly green or blue/green, the color however changing depending on growth conditions, such as culture medium, light, humidity and temperature [74]. Asexual spore dispersion can occur by aerial, aquatic or animal dispersion when spores germinate into hyphae that will grow and develop into a new mycelium. Penicillium fungi can also undergo sexual reproduction, when hyphae from two haploid individuals carrying opposite mating types, named MAT1-1 and MAT1-2, meet under appropriate conditions. Then nuclei fuse (karyogamy) and meiosis occurs, giving rise to eight ascospores. These ascospores are enveloped into asci, themselves being inside the cleistothecium, the fruiting body of Ascomycetes.
Penicillium fungi are well known as saprophytic fungi decaying fruits and crops, as food spoilers, and some species are pathogens of human or food crops. For instance, P. expansum and P. digitatum are post-harvest spoiling agents of citrus and pears [8, 55], P. marneffei is a human pathogen [52] P. oxalicum, P. viridicatum and P. cyclopium are commonly found damaging crops [61] P. glabrum, P. allii, P. sclerotigenum, P. roqueforti and P. paneum are common food spoilers [22, 17]. Penicillium fungi also have huge positive impacts on human society: Alexander Fleming accidentally discovered in 1928 the antibiotic production ability of P. rubens (named at that time P. notatum, then P. chrysogenum and recently renamed P. rubens [38]), which made him win the 1945 medicine Nobel Prize. As many fungi, Penicillium fungi produce toxins under certain conditions for inhibiting competitor growth and reproduction [41, 14], which can have a toxicological impact on humans depending on exposure conditions. Furthermore, Penicillium fungi are used for fermenting food, being for example inoculated for cheese and dry-cured meat production [78, 54, 20, 12]. Indeed, Penicillium fungi used in fermented food enable preserving perishable raw milk or meat, by transforming the raw food thanks to their metabolic activity such as lipolysis and proteolysis and by generating physical barriers on the surface, and also for the generation of special taste and aromas.
3. Domestication of Penicillium roqueforti
Penicillium roqueforti is the species of mould used as a secondary ferment for the production of Roquefort and all blue cheeses [9, 74, 14], with more than 16 synonyms such as P. glaucum, P. gorgonzolae or P. stilton. Penicillium roqueforti is a saprophytic fungus commonly found in soil or decaying organic matter, although its natural reservoir is still unknown. It is frequently isolated from poorly stored plant material in micro-aerophilic conditions such as silage, where it can produce toxins with adverse effects on livestock or humans [65, 64, 63, 5, 3, 14]. Its tolerance to cold and organic acids, particularly when water activity is greater than 95%, makes this species a frequent food contaminant of bread, beer, barley, rice, olives, fruit, hard cheeses, flour or dried meat [69]. The species was described in 1906 by Charles Thom [79], and was in fact a complex of several species, now referred to as the Roquefortorum section [40], also including P. carneum, P. paneum and P. psychrosexualis, which could be distinguished on the basis of molecular, morphological and biochemical analyses [24, 7, 25, 75, 43, 37, 39].
Within the species P. roqueforti, several differentiated populations have been identified based on microsatellite markers [73, 30]. Based on whole genomes, four genetically and phenotypically differentiated populations have been identified [18] (Figure 2). Two populations are used for cheese making and the two other populations are found in other environments [18]. Of the two cheese populations, one encompasses strains related to the French Roquefort protected designation of origin (PDO) production, while the non-Roquefort population encompasses strains isolated worldwide from all other blue cheeses, such as Stilton in England, Cabrales in Spain, Gorgonzola in Italy, Fourme d’Ambert in France. The other two populations are found on lumber, silage and as contaminants in various food products (Figure 2).
The two cheese populations display much less genetic diversity than the two other populations [18]. However, the Roquefort population has retained a slightly richer genetic diversity than the non-Roquefort population, probably due to the requirement of using local strains as part of the PDO specifications [18]. The drastic loss of genetic diversity in the non-Roquefort population reflects a strong selection for industrial scale production and worldwide trade that have led to the use of a single clonal line in all analysed blue cheeses in the world [18].
Domestication history of Penicillium roqueforti for making blue cheeses. From an ancestral population with a so far unknown ecological niche, the four identified P. roqueforti populations have diversified by adapting to human-made ecological niches, with two populations independently domesticated for cheese-making, and two populations spoiling food and silage. One of the two cheese populations is only found in cheeses belongining to the Roquefort protected designation of origin and harbors some genetic diversity while the other population is a single clonal lineage used for all other blue cheeses worldwide. Bottlenecks occurred in all populations, being strongest in the cheese populations. Cheese populations evolved specific traits beneficial for cheese-making, such as aromas, blue coloration of cheese cavities, lipolysis, proteolysis and salt tolerance, with different specificities in the two populations. The Roquefort population produces greater spore quantity when grown on bread, the ancestral medium of cultivation in Roquefort-sur-Soulzon. a: spores from P. roqueforti collected from inoculated bread with burned crusts, as was done for long for blue cheese inoculation. b: Slice of experimental Roquefort-like cheese. c: Piece of blue cheese with P. roqueforti spores magnified in inset, borne in the typical “brushes” of Penicillium fungi. d: Petri dishes as nowadays used for sterile P. roqueforti cultivation. e: P. roqueforti colony on a Petri dish with malt medium. f: 2-heptatone molecule responsible for one of the typical Roquefort blue cheese aromas. g: Illustration of the inferred divergence history of the four identified P. roqueforti populations, two being used for cheese-making and having suffered from strong bottlenecks, one thriving in spoiled food and lumber and one in silage and also contaminated food. h: P. roqueforti colony on a Petri dish with cheese medium, with traits of growth rate measures. i: Tubes with tributyrin used to measure lipolysis rate. j: Petri dish with two P. roqueforti strains and sexual structures (named cleistothecia, in brown) induced at the confrontation zone. k: Competition experiment on a Petri dish with two P. roqueforti strains, showing greater competitive ability and darker color of the non-Roquefort strain. l: Horizontal gene acquisition in the non-Roquefort lineage. m: Roquefort cheese piece. n: Silage ball in a field. o: Yoghurt spoiled by P. roqueforti. p: Bread spoiled by P. roqueforti. q and r: Non-Roquefort blue cheeses (Fourme d’Ambert and Bleu d’Auvergne). s: Roquefort cheeses on their wood shelves in caves, one being enveloped in the typical lead sheet.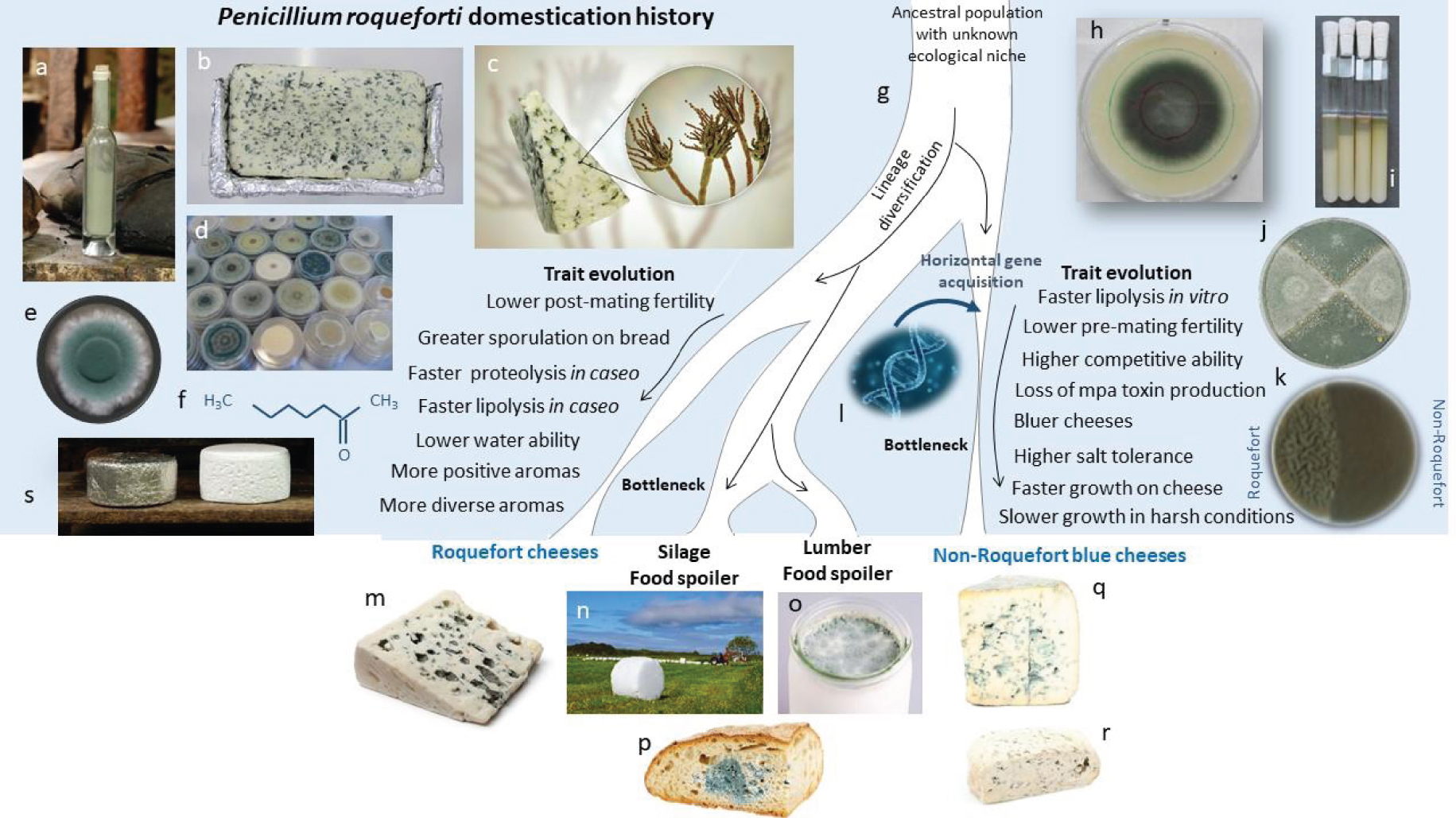
The non-Roquefort lineage has acquired large genomic fragments by horizontal gene transfers [13, 73, 70]. Two of the horizontally-transferred regions appeared of particular interest because of the putative function of some genes that they carried. These two horizontally-transferred regions, called Wallaby and CheesyTer, are 600 and 80 kb long, respectively, and carry genes with functions related to lactose/lactate metabolism and competition against other microorganisms [13, 70].
Comparisons of population divergence scenarios under the approximate Bayesian computation framework revealed that the silage, lumber and food contaminant populations were the genetically closest to each other, having diverged most recently, while the two cheese populations diverged earlier and were the result of two independent domestication events [18] (Figure 2g). Indeed, despite a larger sampling of the cheese populations, especially for the non-Roquefort population, strong bottlenecks were detected in the two cheese populations, being a bit weaker in the Roquefort population [18] (Figure 2g). Two successive bottlenecks may even have occurred in the Roquefort population [18]. These bottlenecks are the result of domestication and also probably of the recent subcontracting of ferment production to a small number of specialist companies, and possibly to the recent selection of a high-performance clonal lineage after the acquisition of genes conferring advantageous traits by horizontal transfers for the non-Roquefort population [13, 70]. Tests comparing demographic models also detected gene flow between the two non-cheese populations, but not with the cheese populations [18]. Domestication dates estimated from the last gene flow events between the populations suggested that the Roquefort population would have diverged about seven times longer than the non-Roquefort population, approximately 760 and 140 generations ago, respectively [18]. However, estimating absolute time from generation numbers is challenging in fungi, especially in clonal lineages.
Main findings from a recent study [11], in which analyses of experimental cheeses made following the Roquefort mode of production (e) and by inoculating strains from the four Penicillium roqueforti populations (a) showed beneficial traits in the two cheese populations, producing a higher blue surface on cheese slices (b), stronger lipolysis and proteolysis in caseo, and especially the Roquefort population (while the non-Roquefort population was found to have the highest lipolysis rate in vitro [18]), (c) and higher quantities of positive aromas (such as methyl ketones, with for example shown in d the 2-pentanone and the 2-heptanone, and secondary alcohols, with for example shown in d the 2-heptanol and the 2-nonanol, with alcohol quantities multiplied by four compared to methyl ketones to be visible on the same axis).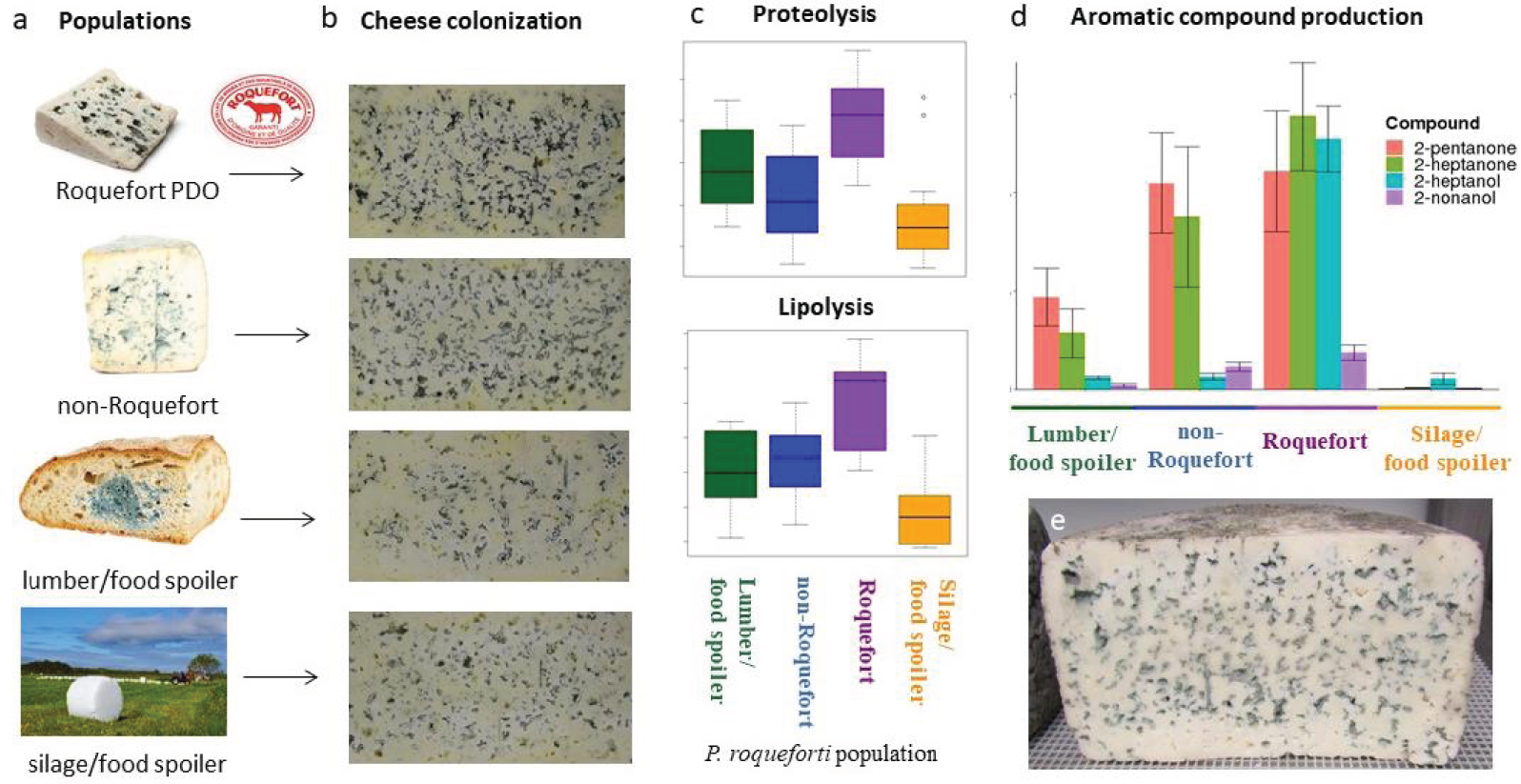
Cheese strains have specific characteristics, in particular in terms of colour, fertility, growth rate and ability to exclude other microorganisms competing on a culture medium [73, 30, 72, 18] (Figure 2). Non-Roquefort strains grow faster on cheese medium than all other strains and exclude competitors better than all other strains [70] (Figure 2k). These capabilities are likely at least partly related to the horizontally-transferred regions Wallaby and CheesyTer (Figure 2l) as they carry genes with functions related to lactose/lactate metabolism and competition against other microorganisms [13, 70]. The non-Roquefort population also showed faster lipolysis, greater salt tolerance and cheese cavity colonization [18], and the loss of ability to produce mycophenolic acid due to a 174 bp deletion in the mpaC gene [29] (Figure 2). The Roquefort population showed greater sporulation on bread medium [18]. The slow growth of the Roquefort population on cheese medium and its lower lipolytic activity observed in vitro compared to the non-Roquefort population are expected to slow down cheese maturation. Such slow ripening may be linked to the need to preserve Roquefort cheeses, made from sheep’s milk available only between February and July, over long periods and historically without refrigeration [56], and to the requirement of at least 90 days of maturation in the Roquefort PDO specifications. The better sporulation on bread of the Roquefort population is also likely part of the domestication syndrom, due to the method of conservation and production of mould spores on rye bread [56, 4, 44, 45, 80] (Figure 2a). The performance of the non-Roquefort population in terms of growth on cheese medium [70], colonisation of cheese cavities, salt tolerance, lipolysis in vitro [18], and loss of toxin production ability [29], would thus result from a very recent selection of beneficial characteristics for the modern and rapid production of all types of blue cheeses.
An analysis of 52 volatile compounds produced by the two cheese populations in highly simplified cheese models identified significantly different volatile compound profiles between the two cheese populations, notably distinguished by compounds typical of blue cheeses with a pronounced taste and a greater diversity of volatile compounds produced by the Roquefort population [18]. Footprints for positive selection have also been identified in several genes potentially involved in volatile compound production [18].
A recent study investigated the differences among P. roqueforti populations in the context of domestication for cheesemaking by comparing blue cheeses made with the four fungal populations following conditions as close as possible to commercial production Roquefort-type production specifications [11] (Figure 3). The P. roqueforti populations had a minor impact on the cheese bacterial diversity and none on the main microorganism abundance. The cheese P. roqueforti populations produced cheeses with higher percentages of blue area due to faster growth and/or better sporulation, possibly related to a better tolerance to low oxygen levels. The cheese populations also produced cheeses with different sets and higher quantities of desired volatile compounds. The Roquefort P. roqueforti population in particular produced higher diversities and quantities of aromatic compounds resulting in more fruity and cheesy notes in cheeses, which was mostly due to its most efficient lipolysis and proteolysis in caseo. Strikingly, lipolysis and proteolysis were indeed found highest in cheeses for the Roquefort P. roqueforti population [11], while lipolysis was in contrast found highest in vitro for the non-Roquefort P. roqueforti population [18], showing the importance of studies in caseo. The Roquefort population also produced cheeses with lower water activity, which can prevent the occurrence of spoiler microorganisms. There was thus a strong influence of P. roqueforti populations on several important aspects of cheese safety, appearance and flavour [11]. Footprints of degeneration have been found in P. roqueforti domesticated. The clonal culture of P. roqueforti strains used for cheese production, and their maintenance isolated from populations in other environments since the 19th century, has led to degeneration of sexual functions, with loss of fertility at different stages in the two cheese populations [72]. The Roquefort cheese population could perform the first steps of sexual reproduction but showed decreased post-zygotic fertility, while the non-Roquefort population even often not even initiate sexual reproduction [72]. Non-Roquefort strains grow more slowly on nutrient-poor medium, which may be due to degeneration on unused functions [70]. The strong bottlenecks in cheese populations [18] may have also contributed to degeneration.
4. Domestication of Penicillium camemberti
Penicillium camemberti is another emblematic cheese-making fungus, used for maturing soft cheeses such as Camembert and Brie, while being phylogenetically distantly related to P. roqueforti [13]. The Penicillium camemberti species complex includes three species, P. biforme, P. camemberti and P. fuscoglaucum, all present in the cheese environment, P. biforme and P. camemberti being specific to the cheese environment while P. fuscoglaucum is mostly found in natural environments [71]. At the beginning of the 20th century, a white mutant was generated in the cheese industry, by artificial mutagenesis from a grey-green fungus at that time named P. commune, now split into two closely related species, P. biforme and P. fuscoglaucum [31, 71]. The Brie cheeses indeed used to be of grey-green aspect until the 20th century. This can be seen on the painting “Symphonie des fromages en brie majeur; Nature morte au fromage” by Marie Julesx Justin (1847; https://www.pop.culture.gouv.fr/notice/joconde/M0794015842).
A recent population genomic study inferred an ancient domestication event separating the cheese-making P. biforme fungus from P. fuscoglaucum, mostly found in other environments than cheese, e.g., rubber, root, wood, walnut, water or contaminated food. A much more recent domestication event was detected, separating the P. camemberti clonal lineage from P. biforme [71] (Figure 4). The P. camemberti clonal lineage was found in Camembert-like soft cheeses, while P. biforme was found in hard cheeses like Tomme or Ossau Iraty (Figure 4). Phenotypic adaptation to cheese-making was found in P. biforme as compared to P. fuscoglaucum, with a whiter color, faster growth on cheese medium under cave conditions (low temperature, and high humidity), salt tolerance and lower levels of the cyclopiazonic acid toxin production (Figure 4).
Domestication history of Penicillium camemberti and P. biforme for making soft or hard cheeses (a). From an ancestral population with a so far unknown ecological niche, P. fuscoglaucum first diverged, thriving mostly in non-cheese environments, then P. biforme was domesticated for cheese production, and later the two P. camemberti varieties, var. camemberti and var. caseifulvum, with strong bottlenecks. The domestication process led to adaptation regarding color (becoming whiter), competitive ability, toxin production (loss of CPA—cyclopiazonic acid—production), radial and/or vertical (fluffiness) growth. Environment of origin of the lineages are shown (from left to right: rotten roots, goat cheese, Camembert cheese and Saint-Marcellin cheese), as well as pictures of mycelium colony on Petri dishes with salted cheese medium (colored in green with a food colorant) in circles. (b). Mycelia of the white fluffy P. camemberti var. camemberti and the grey-green P. biforme grown as sectors in the same Petri dish.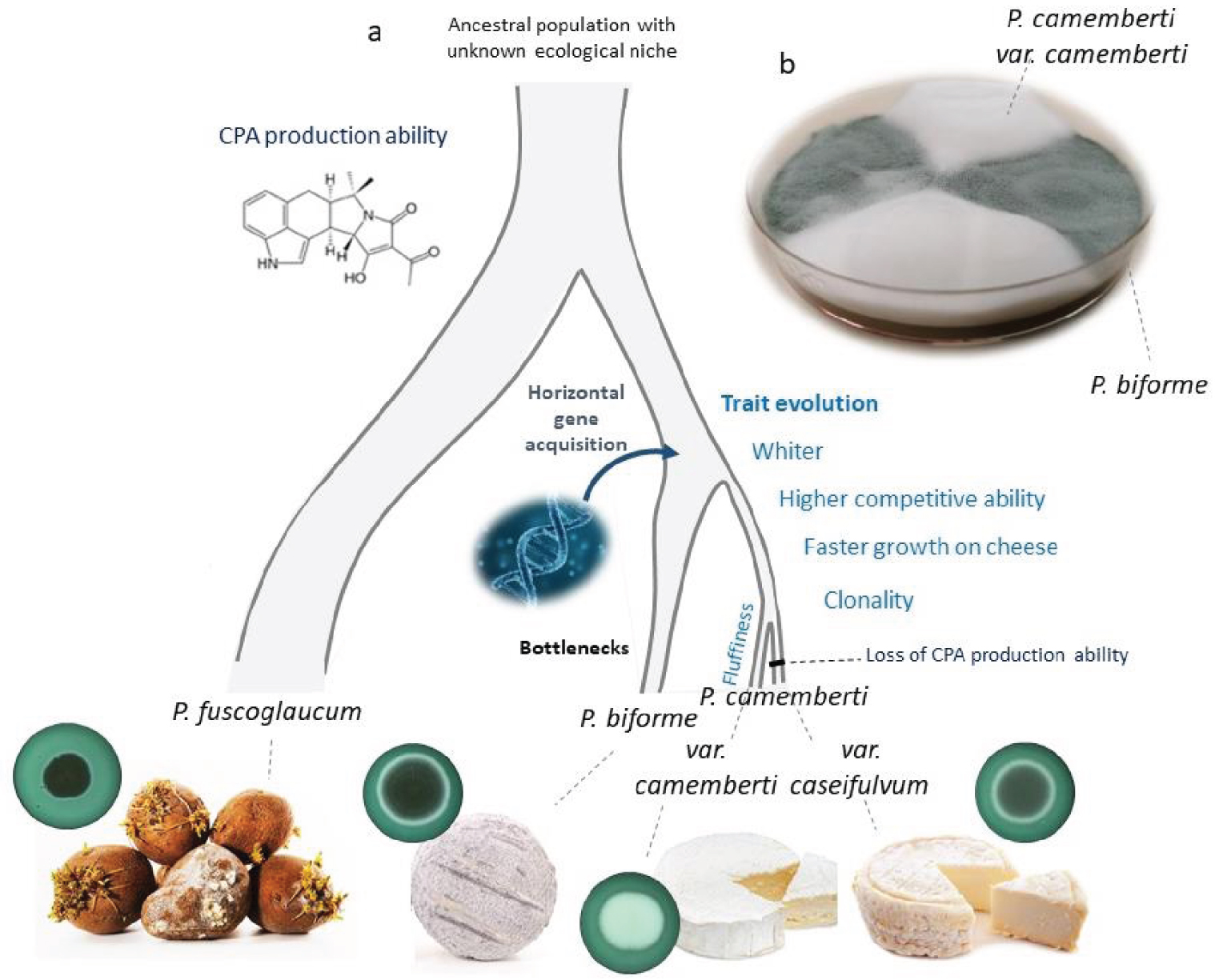
Two differentiated P. camemberti lineages were identified, with contrasting phenotypic features, that can therefore be considered as two different domesticated varieties. One of the P. camemberti varieties contained the same transferred regions as the non-Roquefort population P. roqueforti, CheesyTer and Wallaby [13], except three strains isolated from Camembert cheese before 1905 which do not carry CheesyTer [71]. This P. camemberti variety, called var. camemberti, displays weaker radial growth, but was fluffier than the P. camemberti other variety and than P. biforme and P. fuscoglaucum (Figure 4), which allows forming the white fluffy crust typical of Camembert and Brie cheeses. The grey-green P. camemberti variety, called the caseifulvum variety, is found in cheeses other than Camembert, such as St Marcellin or Rigotte de Condrieu, carries CheesyTer but not the entire Wallaby region, and is unable to produce the cyclopiazonic acid toxin due to a frameshift mutation in one of the genes involved in its biosynthesis [71]. Both P. camemberti varieties exclude fungal competitors more effectively than P. biforme, the fluffy white Camembert lineage being more effective at exclusion than the grey-green P. camemberti lineage.
Footprints of domestication are thus found in P. camemberti and P. biforme, with genetic differentiation between cheese and strains from other environments, and the identification of specific phenotypic traits beneficial for cheese-making. Furthermore, the domestication process led to diversification of P. camemberti into two varieties, P. camemberti var. camemberti and P. camemberti var. caseifulvum, also displaying genetic and phenotypic differentiation (in terms of growth rate, color, competitive abilities and toxin production).
Furthermore, both P. camemberti varieties showed footprints of strong bottlenecks, representing clonal lineages (Figure 4a). Both P. camemberti varieties grew more slowly in harsh conditions than P. biforme or P. fuscoglaucum, suggesting that adaptation to cheese making has led to a degeneration of functions useful in wild environments [71]. Another degeneration footprint is the general very low spore production level in P. camemberti, even more in the camemberti variety, which raises issues for cheese production.
5. Conclusion
The recent studies on P. roqueforti and P. camemberti have shown that the typical appearance and flavour of blue cheeses and soft cheeses are the result of human selection both on P. roqueforti and P. camemberti, thus constituting domestication, and different varieties within each species have acquired specificities for cheese-making. This has important implications for our understanding of adaptation and domestication processes. Several footprints of convergent adaptation have for example been found in the domesticated cheese fungi, with the same genomic regions conferring benefits for growth on cheese having been horizontally acquired both in P. roqueforti and P. camemberti, salt tolerance, higher competitive ability and the loss of toxin production. Strong bottlenecks and clonality were also documented in the cheese populations of the two species, as well as the reduced ability to grow in harsh conditions.
The recent studies on cheese fungi are also important for improving cheese production. The finding of two different clonal lineages used for cheese-making in P. roqueforti, with contrasted phenotypes and opposite mating types [18], together with the development of a protocol for inducing sexual reproduction [73], pave the way for strain improvement by generating progenies. Such progenies can further allow associating genotypes and phenotypes for elucidating the genetic basis for important traits for cheese-making and adaptation. Penicillium camemberti shows footprints of degeneration in the form of a dramatically low level of spore production, which renders difficult the replication and inoculation of this mold for cheese producers. While P. camemberti represents a single clonal lineage harboring a single mating type [71], both mating types were found in P. biforme [73], paving the way to generate new progenies for strain improvement.
Dry-cured meat products, such as salami, ham and other charcuteries like duck or turkey dry-cured meat products, also use Penicillium fungi for fermentation, P. nalgiovense and P. salamii serving as a starter, and also P. camemberti [78, 54, 12]. It would thus be interesting to study the footprints of domestication in these Penicillium fungi. Furthermore, P. nalgiovense and P. salamii are distantly related, which suggests that these Penicillium fungi can be ideal models to study parallel adaptation.



 CC-BY 4.0
CC-BY 4.0