1 Introduction
La détermination par résonance magnétique nucléaire (RMN) de la structure tridimensionnelle de la protéine humaine p8MTCP1 a montré un repliement original, constitué de trois hélices associées par trois ponts disulfure [1]. Les deux premières hélices sont maintenues antiparallèles par deux ponts disulfure, et forment un motif « double hélice » d'environ 40 acides aminés. La troisième hélice, plus hydrophile, n'interagit que faiblement avec le motif « double hélice » [2]. Un peptide de 38 acides aminés, α2p8, correspondant au motif « double hélice » de la protéine humaine p8MTCP1 a déjà été synthétisé et caractérisé [3]. La structure tridimensionnelle de ce peptide (Fig. 1a), déterminée par RMN, montre la conservation du repliement avec deux hélices α antiparallèles. La stabilisation de l'édifice par deux ponts disulfure à l'interface des deux hélices suggère qu'un grand nombre de mutations sont possibles, sans que soit altéré le repliement du peptide. Ce nouveau motif structural minimal, facile à produire par synthèse chimique ou par sur-expression dans E. coli, représente une plate-forme structurale stable pouvant être utilisée comme base pour la conception de nouvelles mini-protéines mimant, notamment, des faisceaux d'hélices impliqués dans la fonction d'un grand nombre de protéines [4].
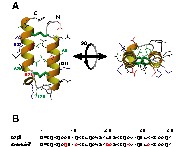
(a) Représentation en ruban de la structure RMN moyenne du peptide α2p8 (code PDB : 1ei0). Les chaînes latérales des acides aminés sont représentées en vert pour les résidus hydrophobes, en bleu pour les résidus chargés positivement, en noir pour les autres résidus. Les acides aminés remplacés pour la conception d'amphip8 sont notés. Les deux vues sont séparées par une rotation de 90° selon l'axe horizontal. (b) Alignement des séquences primaires des peptides α2p8 et amphip8. Les acides aminés mutés sont colorés en rouge.
Dans cet article, nous décrivons la conception d'un peptide amphiphile de 38 acides aminés (nommé amphip8) basé sur le motif minimal structural α2p8 (Fig. 1b). L'intégrité structurale du peptide de synthèse a été contrôlée par RMN.
2 Matériels et méthodes
2.1 Synthèse peptidique
Le peptide amphip8 de 38 acides aminés a été synthétisé sur une résine Fmoc-PAL-PEG-PS (Applied Biosystems, France) avec un synthétiseur Pioneer™ en flux continu. Le produit final, analysé en HPLC sur phase-inverse, présente un pic majeur représentant environ 65% du matériel total élué (données non montrées). Les quatre cystéines du peptide amphip8 ont été protégées avec le même groupement protecteur, le trityl. Le peptide est déprotégé et coupé de la résine avec le réactif K (82,5% TFA, 5% phénol, 5% thioanosole, 5% H2O, 2,5% éthanedithiol) pendant environ 4 h. L'oxydation du peptide est réalisée à température ambiante dans un tampon Hepes (5 mM) à pH 8,5 pendant 2 j sous agitation à l'air libre.
La pureté et l'identité du peptide purifié ont été vérifiées par HPLC analytique et par spectrométrie de masse en electrospray (masse déterminée : 4160,02 Da, masse théorique : 4158,85 Da). Le peptide oxydé a été obtenu avec un rendement de 30,4%.
2.2 Mesures RMN
Toutes les expériences RMN, i.e., DQF-COSY, z-TOCSY et NOESY ont été enregistrées à différentes températures (15 °C, 25 °C, 35 °C et 45 °C) sur un spectromètre Bruker AMX 600 équipé d'une sonde triple résonance (1H–13C–15N) avec gradient-z. La concentration de tous les échantillons était de 2 mM dans H2O (5% de D2O pour le lock) ou dans D2O à pH 5,6.
3 Résultats
3.1 Conception et synthèse
Les faisceaux d'hélices sont largement impliqués dans la fonction d'un grand nombre de protéines. Le motif minimal structural α2p8 permet d'entrevoir la construction d'une petite protéine amphiphile (amphip8), qui possèderait, sur un côté, une surface hydrophile et chargée et, sur l'autre, une surface hydrophobe apte à interagir avec des faisceaux d'hélices de protéines de taille plus importante. L'analyse de la structure du peptide α2p8 (code de la Protein Data Bank : 1ei0) suggère la possibilité d'introduire quelques mutations susceptibles de modifier la lipophilie du peptide, sans en dégrader le motif structural.
Il est important de rappeler que la séquence primaire d'α2p8 provient en grande partie de la séquence correspondant aux deux premières hélices de la protéine humaine p8MTCP1 [1]. Pour p8MTCP1, une troisième hélice vient, par son extrémité NH2 terminale, compléter le cœur hydrophobe de la protéine. De ce fait, α2p8 possède déjà des acides aminés hydrophobes exposés au solvant, tels que : Ala7, Leu14, Ile28 et Ala35. Afin d'étendre cette surface hydrophobe pour notre nouveau peptide amphip8, les résidus Gln11, Glu21 et Lys32 ont été remplacés par des alanines.
Sur la face opposée, déjà riche en acides aminés chargés (Lys5, Glu9 et Lys12 pour l'hélice I ; Glu30 et Lys33 pour l'hélice II) l'Ala26 a été remplacée par une lysine. Ce choix a été motivé tout d'abord par l'objectif de remplacer un résidu hydrophobe par un résidu chargé, mais aussi par celui de tenter de créer un pont salin avec la chaîne latérale du Glu30 et stabiliser ainsi l'hélice II.
L'Ala8, correspondant à une cystéine orpheline dans la séquence de p8MTCP1, est très exposée au solvant. Elle se trouve au milieu de l'hélice I, sur la face opposée à l'espace inter-hélice. Bien qu'elle n'altère pas la géométrie structurale de la protéine native [2], cette alanine est remplacée pour amphip8 par une glutamine plus hydrophile. En revanche, l'Ala36, qui est sur la face exposée au solvant de l'hélice II, a été conservée.
Finalement, la Leu20, située dans la boucle entre les deux hélices, a été remplacée par une glycine. Cette mutation devrait améliorer la flexibilité de la charnière entre les deux hélices et favoriser la structuration du peptide lors de la formation des deux ponts disulfure par oxydation ménagée des quatre cystéines (voir § Matériels et méthodes).
3.2 Caractérisation conformationnelle
Nous avons débuté une étude structurale du peptide amphip8 par RMN du proton à 600 MHz. Le peptide réduit, sans pont disulfure, présente un spectre RMN caractéristique d'un peptide qui n'est pas replié, avec un « entassement » des résonances de protons amides (HN) sur une largeur spectrale inférieure à 1 ppm, ainsi qu'une très faible dispersion des protons alpha (Hα) et des protons des groupements méthyle (Fig. 2a). En revanche, le spectre RMN du peptide oxydé est plus éclaté, traduisant une structuration du peptide (Fig. 2b). La présence d'un nombre important de pics de corrélations NOESY dans la zone des protons amide (spectre non montré) traduit la présence d'une structuration secondaire en hélice. Nous avons donc poursuivi l'étude RMN sur le peptide oxydé.

Spectres RMN (600 MHz, 25 °C) du peptide amphip8 (2 mM, dans H2O, 5% D2O, pH 5,6). (a) Le peptide est réduit. (b) Le peptide est oxydé avec formation de deux ponts disulfure.
L'attribution séquentielle des résonances des protons a été réalisée selon la méthode classique de Wüthrich [5]. La procédure implique : (1) l'identification des systèmes de spins des acides aminés sur la base des spectres 2D-TOCSYet (2) la connectivité de ces systèmes de spins selon la séquence en utilisant les pics de corrélations séquentiels dαN et dNN des spectres 2D-NOESY. Cette étape d'attribution des résonances des protons du squelette peptidique a été facilitée par la faible différence générale observée entre les déplacements chimiques des protons des acides aminés correspondants de α2p8 et d'amphip8.
Cependant, des différences significatives apparaissent pour plusieurs acides aminés quand on compare leurs déplacements chimiques avec ceux du peptide α2p8 (Fig. 3). Au niveau des HN, certains acides aminés mutés (20, 21, 26 et 32) subissent des variations importantes, alors que les positions 8 et 11 ne semblent pas être touchées. Mais on note surtout des changements importants pour des résidus hydrophobes (Ile10, Leu14, Leu31 et Ala35) qui sont disposés sur la même surface dans la structure modèle du peptide α2p8. Le HN de la Ser18 est aussi déplacé, ainsi que ceux des Gln25 et Gln29 qui se trouvent sur la face externe de l'hélice II. En ce qui concerne les Hα, des acides aminés mutés, surtout les positions 8, 20 et 26, subissent des déplacements importants. Mais également d'autres acides aminés présentent des variations plus importantes, comme l'Ile10, la Ser22, la Cys24, l'Ile28 et l'Ala35.
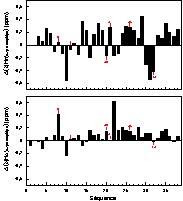
Différences des déplacements chimiques calculées pour les protons HN et Hα (respectivement en haut et en bas) entre les peptides α2p8 et amphip8 en fonction de la séquence. La position des acides aminés mutés pour amphip8 est indiquée.
4 Discussion
Les six mutations choisies pour concevoir amphip8 sur la base de la plate-forme structurale α2p8 ne concernent pas l'interface hydrophobe des hélices, qui peut apporter une contribution majeure à la stabilisation de l'édifice en faisceaux d'hélices [6]. Ces mutations ne sont donc pas censées altérer la structuration du peptide. Ceci est conforté par la présence de nombreux pics de corrélations NOESY dans la région des protons amide. Cependant, les variations de déplacements chimiques des protons observées entre α2p8 et amphip8 ne s'expliquent pas par la simple présence directe de quelques mutations ponctuelles. La formation d'un dimère, après mutation, pourrait expliquer en partie ces différences de déplacements chimiques. En effet, la construction d'une petite protéine amphiphile possédant une surface hydrophobe pourrait induire une dimérisation du peptide visant à enfouir les acides aminés hydrophobes. Il devient donc important de déterminer la structure RMN de ce peptide amphip8 afin d'élucider clairement les variations de déplacements chimiques des protons observées entre α2p8 et amphip8.


