1 Introduction
En Europe, chaque année, les tumeurs malignes du cerveau touchent environ sept personnes sur 100 000, ce qui représente 30 000 nouveaux cas par an, avec une très forte mortalité (plus de 75%) [1]. Le diagnostic de tumeur cérébrale repose, dans un premier temps, sur l'imagerie (IRM et scanner) associée aux données cliniques (âge, symptômes, antécédents...). Ce bilan permet de confirmer le diagnostic, de préciser la localisation de la lésion et de définir de manière incertaine (60 à 90% de diagnostics exacts) la nature de cette lésion (tumeur ou non, type et éventuellement grade de la tumeur). Le diagnostic final est donné par l'analyse histopathologique de biopsies ou de prélèvements d'exérèse, procédures invasives. Or, les tumeurs cérébrales sont très hétérogènes et l'échantillon analysé n'est pas toujours prélevé dans la zone la plus agressive ou représentative de la tumeur. Il est clair qu'une méthode non invasive, qui permettrait une caractérisation de la tumeur dans son ensemble, présenterait un grand intérêt pour le diagnostic, mais aussi pour le suivi des patients.
De nombreux travaux indiquent que la spectroscopie RMN du proton (SRM 1H) présente des potentialités intéressantes pour caractériser les tumeurs cérébrales ; de plus, elle est non invasive [2–6]. La majeure partie des études in vivo par SRM 1H des tumeurs cérébrales chez l'homme utilise des techniques dites à un voxel qui consistent à acquérir le spectre d'un volume sélectionné en zone tumorale, puis à le comparer à celui acquis en zone saine. Il y a quelques années, les critères de classification des tumeurs se basaient sur les rapports d'intensités ou de surfaces de chacun des pics par rapport au pic correspondant en zone saine ou sur les rapports des intensités de deux résonances caractéristiques de deux entités chimiques différentes dans la même zone tumorale. L'ensemble de ces études a montré que des différences sont observées dans les rapports de pics suivant le grade et/ou le type de la tumeur, mais elles ne sont pas statistiquement significatives. Aucun de ces critères n'est assez discriminant pour caractériser les tumeurs cérébrales avec une spécificité suffisante [3]. Les origines de la grande variabilité observée sont diverses : (i) le volume étudié, considéré comme représentatif de la masse tumorale, est contaminé par un volume variable de tissu sain ; (ii) l'hétérogénéité interne des tumeurs cérébrales peut entraîner des profils spectraux différents selon la zone tumorale étudiée (proportion variable de nécrose dans cette zone, zone de bas grade, zone de haut grade...). Des études plus récentes [7–10] s'orientent vers des analyses utilisant des outils statistiques de classification qui analysent simultanément les principales résonances observées dans un spectre proton de tumeurs cérébrales in vivo. Cependant, les spectres traités proviennent toujours de volumes pouvant présenter des effets de volume partiel et ne rendent pas compte de l'hétérogénéité tumorale. L'imagerie spectroscopique (acquisition des spectres de chacun des voxels d'un plan de coupe) met en évidence l'hétérogénéité des distributions des métabolites détectés par SRM 1H et est potentiellement plus riche en information que la spectroscopie à un voxel. Cependant, il est difficile d'extraire facilement des informations intéressantes pour le diagnostic des tumeurs. En effet, une image spectroscopique est constituée de 64 à 100 spectres contenant chacun de rois à six pics. Des images de la distribution de chacun de ces métabolites peuvent être obtenues (images métaboliques), mais il est difficile de combiner l'information contenue dans chacune de ces images en une seule image donnant des informations voxel par voxel sur la nature de la tumeur sous-jacente.
Nous avons récemment proposé une nouvelle approche d'analyse des images spectroscopiques 1H des tumeurs cérébrales, basée sur l'utilisation de techniques statistiques de classification des spectres de chaque voxel (analyse linéaire discriminante) [11]. Des « images nosologiques » sont ainsi obtenues, où chaque pixel est coloré différemment en fonction de la classe histopathologique attribuée. Le but de l'étude présentée ici est de déterminer la valeur ajoutée des images nosologiques pour le diagnostic des tumeurs cérébrales par rapport à l'IRM conventionnelle.
2 Méthodes
Nous avons étudié rétrospectivement 56 patients âgés de 20 à 86 ans (moyenne 52,6), porteurs de tumeurs cérébrales confirmées par analyse histopathologique à partir de biopsie (14 gliomes de bas grade, 24 gliomes de haut grade, 10 métastases, 8 méningiomes). L'acquisition a été réalisée sur un imageur Philips 1.5 T.
Les images IRM conventionnelles dans les trois plans de coupe perpendiculaires de l'espace ont été réalisées par imagerie d'écho de spins avec pondération T1 avant puis après injection de gadolinium, et avec pondération T2.
L'imagerie spectroscopique a été réalisée à l'intérieur d'un volume PRESS (temps d'écho (TE) 272 ms, temps de répétition (TR) 1500 ms, field of view (FOV) ou champ de vue 160 mm), avec 16 pas de codage dans chaque direction spatiale. Après filtrage (Hanning) et transformée de Fourier 3D (2D spatiales, 1D spectrale), les images métaboliques correspondant aux 6 pics principaux (créatine, composés à choline, N-acétylaspartate, alanine, lactate, et lipides) ont été reconstruites par ajustement d'une fonction modèle (gaussienne) sur chacun des pics. Ces images métaboliques ont ensuite été interpolées aux dimensions de l'image anatomique de référence (image pondérée T2). Un profil spectral a été reconstruit en chacun des pixels, prenant en compte l'information des six métabolites principaux plus celle de l'image T2. Chacun de ces profils a ensuite été classé par analyse linéaire discriminante (LDA) selon une base d'apprentissage composée de 77 profils répartis en sept classes :
- • 24 tumeurs gliales de haut grade (HG) ;
- • 14 tumeurs gliales de bas grade (BG) ;
- • 10 métastases (Méta) ;
- • 8 méningiomes (ME) ;
- • 9 nécroses (NE) ;
- • 5 liquides cérébro-rachidien (LCR) ;
- • 7 tissus sains (NT : non tumoral).
À chaque classe a été assignée une couleur, puis chaque pixel a été coloré en fonction de la classe histopathologique attribuée : l'image ainsi obtenue a été appelée « image nosologique » (Fig. 1). Chacune des couleurs de cette image correspond donc à un type et à un grade de tumeur.
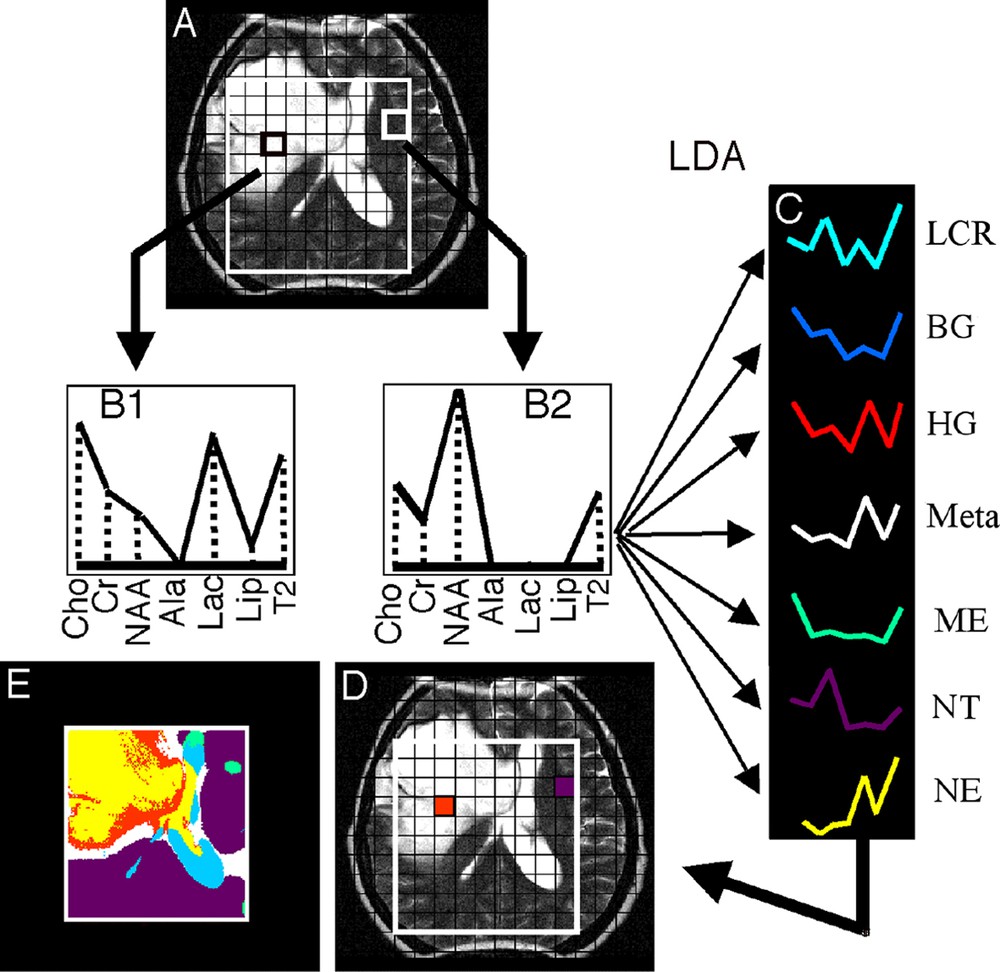
Construction des images nosologiques : pour chaque voxel de l'image de référence contenu dans le volume d'intérêt (carré blanc) (A), un profil spectral est obtenu (B1 et B2), correspondant aux six principaux pics de métabolites contenus dans les spectres RMN (choline, créatine, N-acétylaspartate, alanine, lactate et lipides) et au signal de l'image de référence anatomique T2 (signal de l'eau). Ces profils sont classés suivant sept groupes d'appartenance (C) histopathologique (liquide céphalo-rachidien, gliome de bas grade, gliome de haut grade, métastase, méningiome, tissu non tumoral, nécrose) par analyse linéaire discriminante (LDA). Une couleur est attribuée au voxel en fonction de la classe d'appartenance la plus probable (D). L'image interpolée ainsi colorée est appelée image nosologique (E). Un seuil peut-être défini pour laisser non colorés les pixels dont l'appartenance n'est pas clairement déterminée.
Nous avons ensuite évalué l'apport de ces images pour le diagnostic radiologique de tumeur cérébrale. Les images IRM conventionnelles et nosologiques ont été analysées indépendamment par deux neuroradiologues, en deux sessions. Pendant la première session, chaque neuroradiologue a interprété les images IRM conventionnelles seules pour la moitié des cas, et les images conventionnelles plus l'image nosologique pour l'autre moitié. Trois mois plus tard, les images conventionnelles et l'image nosologique ont été présentées pour la première moitié des cas et les images conventionnelles seules pour la seconde moitié.
3 Résultats
L'analyse statistique (Fig. 2) par courbes ROC (Receiver Operator Characteristic) montre une amélioration significative du diagnostic en utilisant les images nosologiques par rapport aux images conventionnelles seules (p < 0,01). En particulier, la présentation des images nosologiques conduit à une augmentation de l'aire sous la courbe pour les gliomes de bas grade, les gliomes de haut grade et les métastases, par rapport à l'imagerie conventionnelle seule. Les méningiomes étant tous correctement classés avec les images conventionnelles seules, aucune valeur ajoutée des images nosologiques n'a pu être mise en évidence.
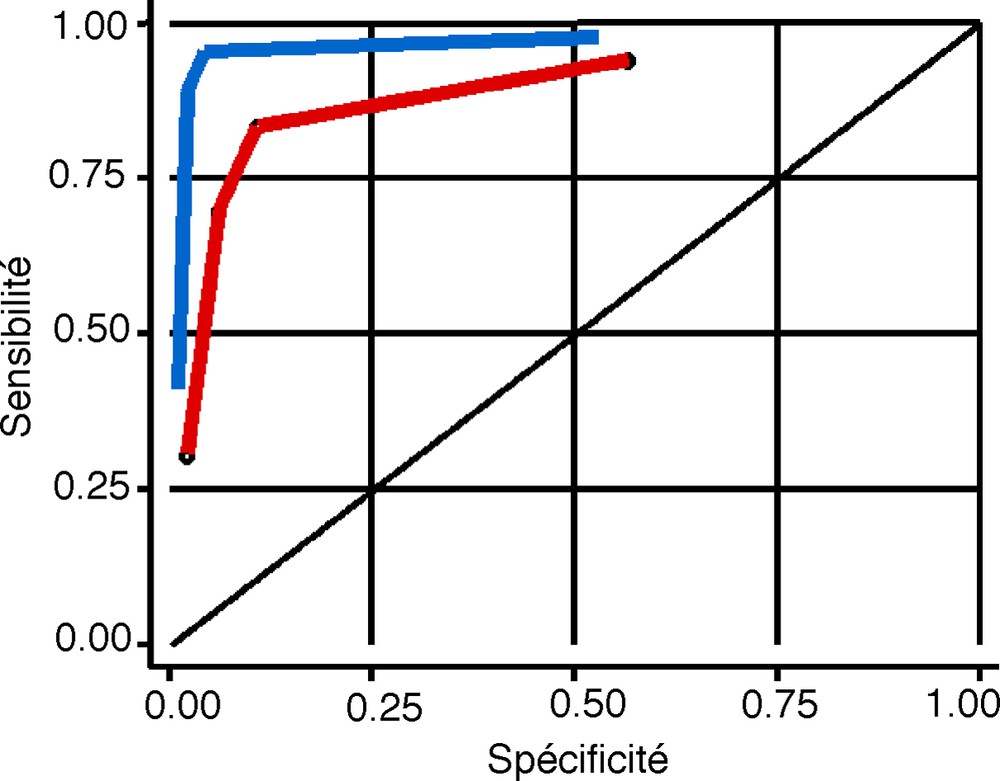
Une analyse de courbes ROC a montré que l'aire sous la courbe obtenue pour toutes les tumeurs confondues était de 0,9 sans tenir compte des images nosologiques (courbe rouge), et augmentait à 0,97 lorsque les images nosologiques étaient présentées (courbe bleue). Cette augmentation est statistiquement significative (p < 0,01).
4 Discussion
Seule une analyse histologique permet de préciser formellement le type et le grade d'une tumeur cérébrale. Cette analyse est effectuée sur un échantillon prélevé au niveau de la lésion par biopsie ou exérèse. Cet acte chirurgical comporte des risques pour le patient (1–2% de mortalité). De plus, l'échantillon prélevé n'est pas nécessairement représentatif de toute la tumeur, car celle-ci peut présenter une certaine hétérogénéité. Enfin, dans le suivi des tumeurs, en particulier sous traitement, des biopsies répétées ne sont pas souhaitables, ni parfois praticables. Pour remédier à ces limitations, la spectroscopie par résonance magnétique nucléaire (RMN du proton) peut présenter une alternative satisfaisante. C'est une technique non invasive qui permet de déterminer la composition chimique des tissus. Les données sont représentées sous forme de spectres, dont les pics signalent la présence de métabolites. À chaque volume élémentaire de tissu (voxel) correspond un spectre. Chaque spectre est comparé à une population de spectres provenant de tissu dont le diagnostic est sûr, puis chaque voxel est affecté à un groupe histopathologique. Cependant, ces examens conduisent à un important volume de données, qui est d'interprétation peu facile. C'est pourquoi il a paru nécessaire de mettre au point un système automatique de classification des tissus. Ce système utilisant des données spectroscopiques en combinaison avec des données d'imagerie permettrait alors d'établir le diagnostic de manière objective. Cette superposition d'une « image nosologique » à une image IRM classique rend plus facile la lecture des données spectroscopiques et fournit une aide significative au diagnostic des tumeurs cérébrales, en particulier en cas d'hésitation diagnostique (Fig. 3).
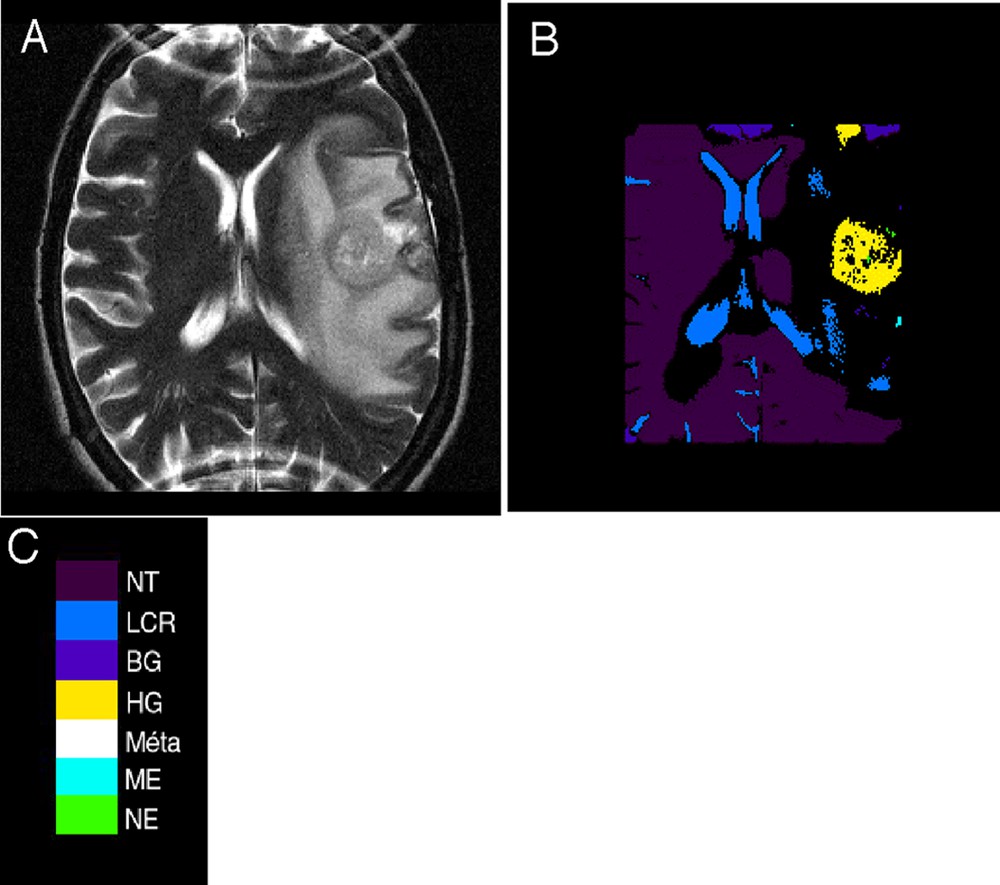
Exemple de tumeur temporale gauche primitive gliale de haut grade (diagnostic de glioblastome confirmé en histologie), dont le diagnostic radiologique hésitant entre glioblastome et métastase a été optimisé par l'image nosologique.
Cette nouvelle forme de représentation des données acquises par 1H MRS apparaît donc particulièrement intéressante pour le diagnostic, mais aussi pour le ciblage des zones biopsiques se focalisant sur les zones de haut grade de plus mauvais pronostic, ainsi qu'enfin pour le suivi des tumeurs cérébrales sous traitement.
5 Conclusion
Les informations fournies par imagerie spectroscopique 1H peuvent être condensées en une image nosologique unique, facilement interprétable par les radiologues et qui fournit un apport statistiquement significatif lors du diagnostic et de la prise en charge des tumeurs cérébrales.
Remerciements
Ce travail a été financé par EU IST-1999-10310 (INTERPRET).


